鱼类生活在富含病原体的水环境中,其鳃、皮肤、肠道是机体与外界环境接触的门户,这些黏膜组织及其表面的黏液构成了抵御病原微生物入侵的第一道防线[1]。鱼类黏膜相关淋巴组织(Mucosa-associated lymphoid tissues, MALTs)可以摄取并呈递抗原,其含有的抗体分泌细胞能分泌特异性抗体,这些特异性抗体与黏液中的溶菌酶、转移因子、补体等物质共同组成有效的免疫防线[2]。鳃是鱼类的呼吸器官,也是主要MALTs之一。鱼类鳃中含有大量的免疫相关因子和细胞,如抗菌肽、细胞因子、补体、主要组织相容性复合物(MHC)、淋巴细胞、巨噬细胞、中性粒细胞和抗体分泌细胞等[1, 3-5],它们对鱼类的免疫防御起着重要作用。近年来,越来越多的研究聚焦于病原微生物感染后鳃组织的免疫反应和调节机制[6-9]。本实验室前期对牙鲆鳃间淋巴组织的结构与应答特性进行了研究,发现鳃中存在大量T细胞聚集,也含有B细胞,在疫苗免疫后会显著增殖[10];鳃中含有的黏蛋白Muc2和Muc5ac,对病原具有缠裹和清除作用[11]。然而,鱼类鳃黏膜相关淋巴组织抗细菌感染的免疫应答机制仍不清楚。
转录组测序(RNA sequencing,RNA-seq)是解释功能基因组学元素和揭示细胞与组织分子机制的有力工具之一[12],已经广泛应用于鱼类功能基因组学及免疫防御机制的研究。通过转录组测序分析,在感染鲑鱼鳃痘病毒的大西洋鲑(Salmo salar)鳃组织中鉴定到与先天性抗病毒反应和免疫防御相关的免疫基因[6]。通过转录组测序分析,在银鲫(Carassius auratus gibelio)感染斜管虫(Chilodonella hexasticha)后的鳃组织中,分析到大量与免疫反应和细胞凋亡相关的途径,揭示了鱼类应对寄生虫感染的免疫调节机制[9]。通过转录组测序分析,在肿大细胞病毒属虹彩病毒感染的牙鲆(Paralichthys olivaceus)脾脏中,确定了鱼类抗病毒免疫基因及通路[13]。转录组测序技术为研究鱼类免疫应答机制提供了强有力的帮助。
鳗弧菌(Vibrio anguillarum)是水产养殖动物的一种主要致病菌,可引起多种养殖鱼类发生弧菌病,严重危害牙鲆(Paralichthys olivaceus)、大菱鲆(Scophthalmus maximus)等中国的重要经济鱼类[14-15]。患病鱼主要症状为体表溃疡,鳃、鳍等部位充血[16]。本文利用RNA-seq技术探究了感染鳗弧菌后24 h牙鲆鳃组织的基因表达情况,对感染早期免疫防御与调控相关的通路和基因进行了综合分析,解析了宿主和病原菌之间的相互作用以及牙鲆鳃组织的免疫反应机制,以期为深入理解硬骨鱼类抗感染黏膜免疫防御机制提供理论依据。
1 材料与方法 1.1 实验鱼与菌株实验所用健康牙鲆采购自山东省日照某养殖场,体型正常,大小均匀,体长为17~19 cm,体质量为60~63 g。实验前于水箱中暂养一周,水温19~21 ℃,连续充气。
感染用鳗弧菌由本实验室保种。将保种的鳗弧菌经连续划线法接种于灭菌的LB(Lysogeny broth)固体培养基,28 ℃培养24 h。挑取单菌落至灭菌的LB液体培养基中,28 ℃培养24 h后,离心并收集菌体,用无菌磷酸缓冲液(PBS)重悬菌体,以比浊仪测定菌液浓度,用PBS将菌液调整至1×107 CFU/mL[17-18],用于感染鱼体。
1.2 感染和样品采集将牙鲆随机分为两组,空白对照组养殖于无处理的海水中,实验组腹腔注射100 μL鳗弧菌液。注射菌液24 h后,从对照组和实验组中各随机抽取3尾鱼,解剖取鳃组织,并标记为CG组(对照组鳃组织)和VG组(实验组鳃组织),将这两组组织放入冻存管并立即投入液氮中冷冻,用于总RNA提取。
1.3 RNA提取、建库和测序用Trizol法提取牙鲆鳃组织中的总RNA,用琼脂糖凝胶电泳检测RNA质量。提取总RNA后,用Oligo(dT)富集真核mRNA,以超声波打断的mRNA为模板,以随机寡核苷酸为引物逆转录为cDNA。通过AMPure XP beads筛选cDNA,随后进行PCR扩增。在Omicshare公司(广州,中国)使用Illumina nova 6000平台进行测序。
1.4 数据质控和序列比对分析测序后,利用fastp[19]过滤低质量序列(Q<20)以及不明确的核苷酸和短读取序列(<30 bp),获得干净原始数据(Clean data)。随后分析碱基组成及质量分布,计算Q20、Q30、GC含量,以便直观展示数据质量。使用HISAT2[20]将质控到的序列比对到牙鲆参考基因组(https://www.ncbi.nlm.nih.gov/datasets/genome/GCF_001970005.1/)。同时,使用bowtie2[21]将干净原始数据与该物种的核糖体数据库进行比对,在不允许错配条件下,去除比对上核糖体的reads,并将保留下来的unmapped reads用于后续转录组分析。
1.5 表达量差异分析根据比对结果,利用Stringtie[22-23]重构转录本,RSEM[24]计算基因表达量,以FPKM(Fragments Per Kilobase of transcript per Million mapped reads)值对转录本的表达量进行标准化。利用DESeq2[25]软件筛选差异表达基因(Differential expressed genes,DEGs),将P<0.05且|log2(Fold Change)|>0.6作为筛选标准[26-27]。差异表达分析结果用火山图和差异散点图表示。
1.6 差异表达基因筛选及基因互作网络分析从差异表达基因中筛选与牙鲆免疫调控相关的差异表达基因,并以雷达图表示(OmicShare平台,https://www.omicshare.com/tools/)。使用STRING数据库(http://string-db.org)中的互作关系进行差异基因互作网络的分析。由于数据库中不包含牙鲆物种,将差异表达基因的序列应用blastx比对到STRING数据库中包含的参考物种的蛋白质序列上,利用比对上的蛋白质互作关系构建互作网络。
1.7 GO和KEGG通路富集分析将差异表达基因向GO(Gene Ontology)数据库(http://www.geneontology.org/)的各条目(Term)映射并计算其差异基因数,得到具有某个GO功能的基因列表及基因数目统计,应用超几何检验,找出与整个背景基因相比在差异表达基因中显著富集的GO条目。利用KEGG(Kyoto Encyclopedia of Gene and Genomes)针对Pathway显著性富集分析,以Pathway为单位,应用超几何检验,找出与背景基因相比在差异表达基因中显著性富集的Pathway。GO和KEGG均以P<0.05作为富集标准。
1.8 GSEA分析使用软件GSEA(Gene Set Enrichment Analysis)和MSigDB进行基因集富集分析,输入表达量表,通过Signal2Noise归一化方法对基因进行排序,用默认参数计算富集分数(ES)、P值和FDR值,以确定GO条目和KEGG通路中的一组基因在对照和实验组中是否显示出显著差异。
1.9 实时荧光定量PCR利用实时荧光定量PCR(qRT-PCR)验证转录组测序数据的可靠性。选取8个有代表性的免疫相关差异表达基因,使用Primer-Blast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/)设计特异性引物(见表 1),由青岛擎科生物科技有限公司合成。使用HiScript III RT SuperMix试剂盒(诺唯赞公司)将总RNA逆转录合成cDNA,以β-actin作为内参基因,使用2×SYBR Green PCR Master Mix试剂(ABclonal)和Lightcycler 480 Real-Time PCR仪(Roche)进行qRT-PCR扩增。配置体系为10 μL 2×Universal SYBR Green Fast qPCR Mix,0.8 μL PCR Primer(10 μmol/L,正向引物和反向引物各0.4 μL),2 μL cDNA模板和7.2 μL超纯水。反应程序如下:95 ℃,3 min;95 ℃ 5 s,60 ℃ 30 s,40个循环;60~95 ℃制作熔解曲线。每组测试设置3个平行样本,每个平行样本设置3次重复实验,采用2-△△Ct方法分析结果,数据以平均值±标准误(mean±SEM)表示,用GraphPad Prism 8.0绘制qRT-PCR和RNA-seq数据柱状图,利用Excel分析相关性。
|
|
表 1 引物序列 Table 1 Primer sequences |
采用Illumina测序平台构建健康(CG1、CG2、CG3)和感染鳗弧菌牙鲆(VG1、VG2、VG3)鳃组织的转录组文库,共获得294 633 606份原始数据,对6个转录组测序结果进行分析,利用软件过滤掉不合格的序列后,获得的clean reads数量分别为48 346 732、51 090 240、45 601 518、46 769 308、49 563 804、51 519 382,高质量reads均占原始数据的99.20%以上,高质量数据的碱基数分别为7 157 809 029、7 574 543 335、6 769 211 698、6 954 031 419、7 353 400 683、7 633 806 392,Q20碱基百分比在98.05%以上,Q30碱基百分比在94.47%以上,GC比例48.23%以上(见表 2),说明测序质量较好。6个样本能成功比对到参考基因组的reads比例在89.32%~90.11%之间(见表 3),说明参考基因组选择合理,测序数据满足后续分析的要求。
|
|
表 2 实验样品测序数据 Table 2 Sequencing data of experimental samples |
|
|
表 3 转录组数据序列比对情况 Table 3 Sequence alignment result statistics |
牙鲆鳃组织实验组与对照组相比(见图 1A),共有620个差异表达基因,包含334个上调表达和286个下调表达基因,多个差异表达基因参与了鳗弧菌感染鱼体后的病原入侵和机体免疫应答,列举的30个差异表达基因分别在图 1B和表 4中展示,图 1C展示了其雷达图。与CG组相比,il12ba、igdcc3、cd5、fcgbp、tcrcb2、pla2g4c、muc19、muc5b、igll1、cd22、tcrd、ptprc和bcl2l15下调表达,其中igdcc3下调表达最显著,其次是fcgbp和muc5b。而boka、lyg、cd209e、esama、fcgr3、hla-dpb2、cldn3d、serpinel、tlr13、ly6d、cd209c、nlrc3、h2-ab1、muc5ac、masp1、il17c上调表达,其中masp1上调倍数最大,其次是cldn3d和il17c(见表 4和图 1C)。VG组中,多种凝集素相关基因呈现不同的表达趋势。其中masp1、mrc1、sell、clec11A、clec10A、lgals3基因上调表达,且masp1上调倍数最高;而colec10、ladd和lgals1基因下调表达,且ladd下调最显著(见表 5)。构建了部分差异表达基因的互作网络图(见图 1D),该图由127个节点和97条边构成。其中fcgr3、nlrc3、tlr22、cd209e、esama是该网络的关键节点,作为核心基因在互作网络中起连接交互作用。此外,il12ba、boka、igdcc3、masp1、mmp9(NCBI_109630977)、hla-dqb2、cd209c、ldlra(NCBI_109643259)、ldlr(NCBI_109645966)、muc5b也处于中心节点位置。
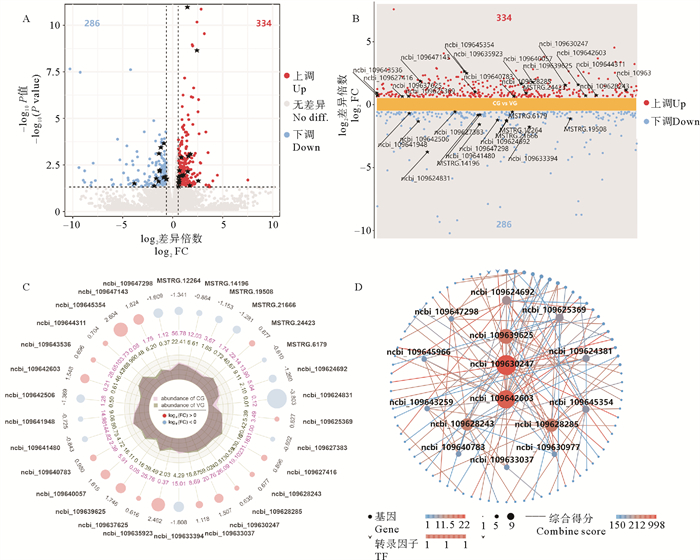
|
( A:火山图。红色(表达量上调)和蓝色点(表达量下调)为差异表达基因(DEGs)(判断标准为P<0.05,FC>1.5);灰色的点为没有差异;黑色星点表示免疫相关差异表达基因。B:差异散点图。C:免疫相关差异表达基因雷达图。灰色数值表示差异基因log2FC值;红色和蓝色圆圈分别表示上调基因和下调基因,圆圈大小根据log2FC值的大小而变化;粉色和绿色数值分别代表差异基因在CG组和VG组的平均表达量,圈中不规则形状表示各个轴上CG组和VG组的表达量丰度。D:免疫相关差异表达基因互作网络图。圆形节点表示基因名称;角型节点表示转录因子(TF)名称;节点大小及颜色表示连通性,节点越大,代表基因连通性越强;线段表示基因和基因之间相互作用关系,线段颜色表示综合得分,该值越大代表两种蛋白间的互作关系越强。A: Volcanic plot. The red (up-regulation) and blue (down-regulation) dots indicated differentially expressed genes (DEGs) (P < 0.05, FC>1.5). The gray dots indicated no difference, and the black star dots indicated immune related DEGs. B: Differential scatter plot. C: Immune related DEGs radar chart. The gray values represented the log2FC values of DEGs, while the red and blue circles represented upregulated and downregulated genes, respectively. The size of the circles varied according to the log2FC value; The pink and green values represented the average expression levels of DEGs in the CG and VG groups, respectively. The irregular shapes in the circles indicated the abundance of expression levels in the CG and VG groups on each axis. D: Immune related DEGs interaction network diagram. Circular nodes represented gene names, angular nodes represented transcription factor (TF) names, and node size and color indicate connectivity. The larger the node, the stronger the gene connectivity; The line segment represented the interaction between genes, and the color of the line segment represented the combined score. The larger the value, the stronger the interaction between the two proteins. ) 图 1 牙鲆鳃组织感染前后差异表达基因分析 Fig. 1 Analysis of differentially expressed genes before (CG group) and after (VG group) infection |
|
|
表 4 免疫相关的差异表达基因 Table 4 Immune related differentially expressed genes |
|
|
表 5 凝集素相关的差异表达基因 Table 5 Differentially expressed genes related to lectin |
对620个差异表达基因进行GO功能富集分析显示,差异基因被分别富集到生物过程(Biological process,BP)、分子功能(Molecular function,MF)和细胞组分(Cellular component,CC)三个类别体系中。对其进一步分析,得到45个功能亚类,包括22个生物过程,8个分子功能和15个细胞组分(见图 2)。图 3A和图 3B列出了显著富集的前20个子类功能条目,包括8个生物过程条目、6个分子功能条目和6个细胞组分条目。与机体炎症和免疫发生相关的条目发生显著富集,如自噬调控(GO:0010506)、免疫效应器过程(GO:0002252)、自噬(GO:0006914)和免疫效应器过程的调控(GO:0002697)。

|
( GO数目统计图。生物过程(BP)、分子功能(MF)和细胞组分(CC)为GO数据库中的三个一级分类。纵坐标表示三个大分类包含的二级分类term,横坐标表示差异基因中富集到各term中的差异基因数目,蓝色代表下调基因,红色代表上调基因,数字分别代表每个term中的下调基因和上调基因数量。条目从上到下依次为:生物过程:代谢过程、单一生物体过程、细胞过程、生物调节、生物过程调节、定位、多细胞生物过程、对刺激的反应、发育过程、信号、细胞组分组成或生物合成、生物过程负调控、免疫系统过程、生物黏附、多生物过程、生物过程正向调节、运动、生长、生殖过程、细胞杀伤、生物阶段、繁殖。分子功能:结合、催化活性、运输活性、分子传递活性、细胞核结合转录因子活性、信号传递活性、分子功能调节因子、转录因子活性,蛋白结合。细胞组分:细胞成分、细胞、细胞器、细胞膜、细胞膜成分、大分子复合体、细胞器成分、细胞外区域、细胞外区域成分、细胞连接、细胞膜内腔、细胞外基质、突触、突触成分、超分子复合体。GO number statistics chart. Biological process (BP), molecular function (MF), and cellular component (CC) are classified as three primary categories in the GO database. The vertical axis represented the secondary classification terms included in the three major categories, and the horizontal axis represented the number of differentially expressed genes enriched in each term. Blue represented down-regulated genes, red represented up-regulated genes, and the numbers represented the number of down-regulated and up-regulated genes in each term. Terms from top to bottom: Biological process: metabolic process, single-organism process, cellular process, biological regulation, regulation of biological process, localization, multicellular organismal process, response to stimulus, developmental process, signaling, cellular component organization or biogenesis, negative regulation of biological process, immune system process, biological adhesion, multi-organism process, positive regulation of biological process, locomotion, growth, reproductive process, cell killing, rhythmic process, reproduction. Molecular function: binding, catalytic activity, transporter activity, molecular transducer activity, nucleic acid binding transcription factor activity, signal transducer activity, molecular function regulator, transcription factor activity, protein binding. Cellular component: cell part, cell, organelle, membrane, membrane part, macromolecular complex, organelle part, extracellular region, extracellular region part, cell junction, membrane-enclosed lumen, extracellular matrix, synapse, synapse part, supramolecular fiber. ) 图 2 GO数目统计图 Fig. 2 GO number statistics chart |
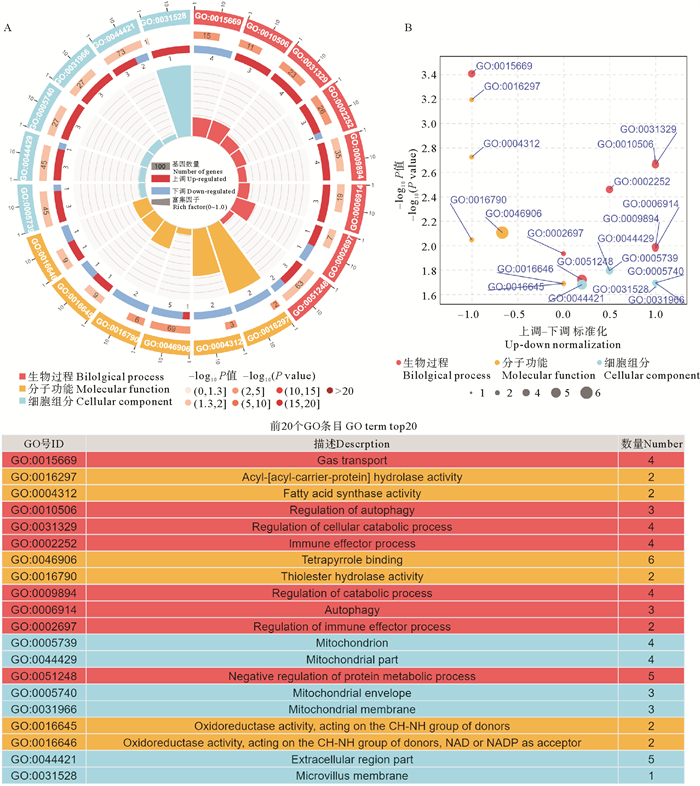
|
( A:富集圈图。从外到内,第一圈代表富集前20的GO子term,圈外为差异基因数目的坐标尺,不同的颜色代表不同的一级分类;第二圈代表该GO term中差异基因的数目以及P值,差异基因背景数量越多条形越长,P值越小颜色越红;第三圈是上下调差异基因数量,红色代表上调差异基因比例,蓝色代表下调差异基因比例,下方显示具体的数值;第四圈:各GO term的RichFactor值,背景网格线,每一格代表 0.1。B:z-score气泡图。上图横坐标为z-score值(上调与下调差异基因数目的差值占总差异基因的比例);下表为P值前20的GO term列表。不同的颜色代表不同的一级分类。表格中条目从上到下依次为:气体运输;酰基-[酰基载体蛋白]水解酶活性;脂肪酸合酶活性;自噬调控;细胞分解代谢过程的调控;免疫效应器过程;四吡咯结合;硫酯水解酶活性;分解代谢过程的调节;自噬;免疫效应器过程的调控;线粒体;线粒体成分;蛋白代谢过程负调控;线粒体包膜;线粒体膜;氧化还原酶活性,作用于CH-NH基团;氧化还原酶活性,作用于CH-NH基团,NAD或NADP作为受体;细胞外区域成分;微绒毛膜。A: Enrichment circle diagram. From the outside to the inside, the first circle shows the top 20 GO sub-terms, with the coordinate ruler of the number of DEGs outside the circle. Different colors represented different primary classifications. The second circle represents the number and P-value of differentially expressed genes in the GO term, and the more genes, the longer the bar, and the smaller the P-value, the redder the color. The third circle represented the number of the DEGs (red, up-regulated; blue, down-regulated). The fourth circle was the RichFactor of each GO term. Each case of the background grid line represented 0.1. B: z-score bubble chart. The horizontal axis of the figure above was the z-score value (the proportion of the difference between the number of up-regulated and down-regulated DEGs to the total DEGs). The table below shows a list of the top 20 GO terms ranked by P-value. Different colors represented different primary classifications. Table terms from top to bottom: Gas transport; Acyl-[acyl-carrier-protein] hydrolase activity; Fatty acid synthase activity; Regulation of autophagy; Regulation of cellular catabolic process; Immune effector process; Tetrapyrrole binding; Thiolester hydrolase activity; Regulation of catabolic process; Autophagy; Regulation of immune effector process; Mitochondrion; Mitochondrial part; Negative regulation of protein metabolic process; Mitochondrial envelope; Mitochondrial membrane; Oxidoreductase activity, acting on the CH-NH group of donors; Oxidoreductase activity, acting on the CH-NH group of donors, NAD or NADP as acceptor; Extracellular region part; Microvillus membrane. ) 图 3 差异表达基因的GO富集分析 Fig. 3 GO enrichment analysis of differentially expressed genes |
对620个差异表达基因进行KEGG富集分析显示,差异基因被富集到新陈代谢、疾病、有机体系统、基因信息处理、细胞过程和环境信息处理六个大类中(见图 4)。在新陈代谢类别中,除了全局和整体图谱,脂质代谢是代谢类别中注释最多的通路;在疾病类别中,差异表达基因大多注释到与感染类疾病相关的通路上;在有机体系统类别中,内分泌系统通路的注释基因数量最多,其次是免疫系统通路;在基因信息处理和细胞过程这两大类中,注释最多的通路分别为翻译和运输分解代谢,不过总体而言,注释到信号转导通路的基因数量最多。图 5A和图 5B列出了显著富集的前20个KEGG通路,大部分与疾病相关,包括金黄色葡萄球菌感染(ko05150)、哮喘(ko05310)、同种异体移植物排斥反应(ko05330)、系统性红斑狼疮(ko05322)、炎症性肠病(ko05321)、Ⅰ型糖尿病(ko04940)、肥厚性心肌病(ko05410)和移植物抗宿主病(ko05332)等。其中,金黄色葡萄球菌感染通路与细菌感染相关,Ⅰ型糖尿病和肥厚性心肌病分别与内分泌代谢和心血管疾病相关,其余通路均与免疫疾病相关。此外,直接与机体免疫相关的补体和凝血级联通路(ko04610)、与细胞分子相互作用相关的细胞黏附分子(ko04514)、ECM受体相互作用(ko04512)等通路也发生显著富集。与代谢相关的脂肪酸生物合成(ko00061)、亚油酸代谢(ko00591)、α-亚麻酸(ko00592)、类固醇生物合成(ko00100)、组氨酸代谢(ko00340)和不饱和脂肪酸合成(ko01040)等也显著富集。
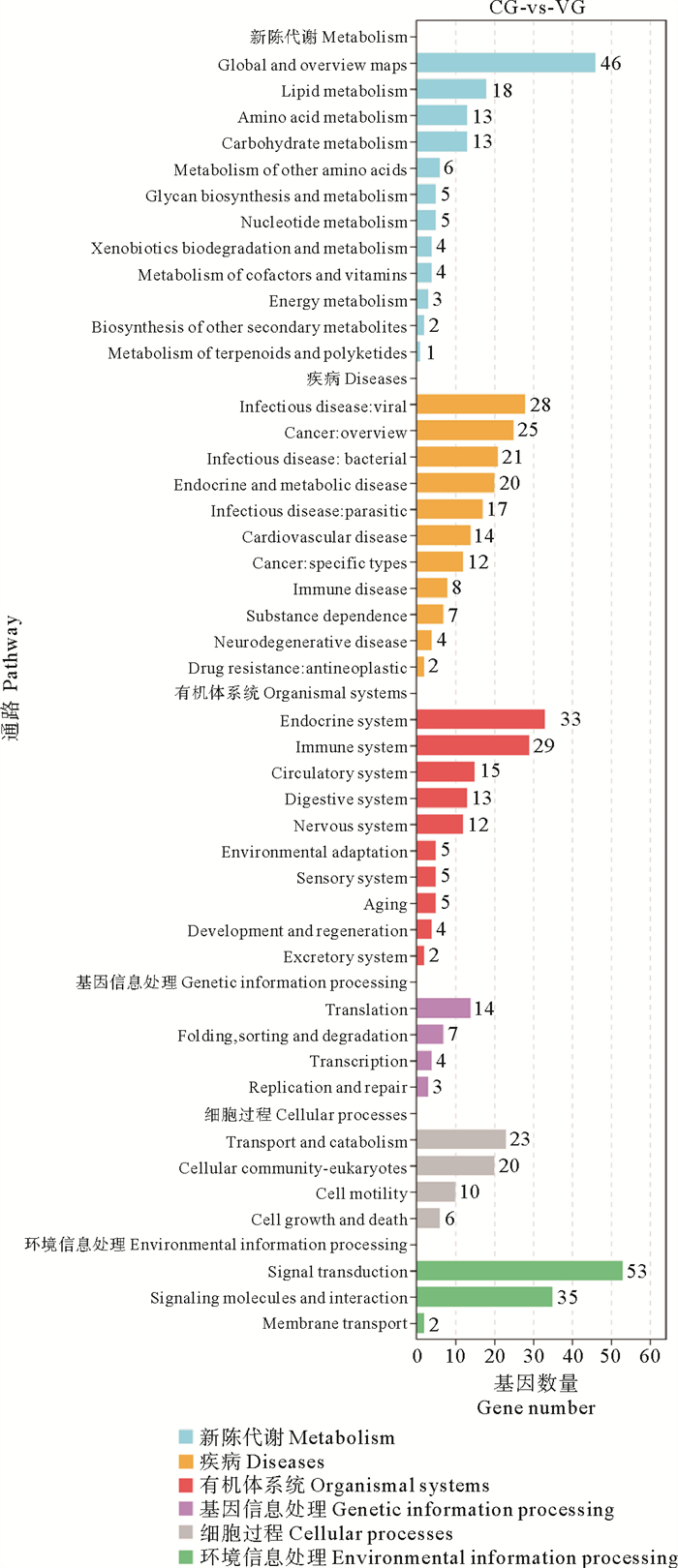
|
( 新陈代谢、疾病、有机体系统、基因信息处理、细胞过程、环境信息处理分别为KEGG的A层级分类信息,纵坐标不同颜色表示KEGG的B层级分类信息通路,横坐标表示差异基因中富集到各通路中的差异基因数目。通路从上到下依次为:新陈代谢:代谢、全局与概述图谱、脂质代谢、氨基酸代谢、碳水化合物代谢、其他氨基酸代谢、聚糖生物合成和代谢、核苷酸代谢、异生素生物降解和代谢、辅因子和维生素代谢、能量代谢、其他次生代谢物的生物合成、萜类化合物和聚酮类化合物的代谢。疾病:传染病:病毒、癌症:概述、传染病:细菌、内分泌代谢疾病、传染病:寄生虫、心血管疾病、癌症:特定类型、免疫疾病、药物依赖、神经变性疾病、耐药性:抗肿瘤。有机体系统:内分泌系统、免疫系统、循环系统、消化系统、神经系统、环境适应、感觉系统、衰老、发育与再生、排泄系统。基因信息处理:翻译、折叠,分类和降解、转录、复制和修复。细胞过程:运输和分解代谢、细胞群落-真核生物、细胞运动、细胞生长和死亡。环境信息处理:信号转导、信号分子相互作用、膜运输。Metabolism, diseases, organismal systems, genetic information processing, cellular processes, and environmental information processing were the A-level classification information of KEGG. Different colors on the y-axis represented B-level classification information (pathways) of KEGG, and the x-axis represented the number of differentially expressed genes enriched in each pathway. Pathways from top to bottom: Metabolism: global and overview maps, lipid metabolism, amino acid metabolism, carbohydrate metabolism, metabolism of other amino acids, glycan biosynthesis and metabolism, nucleotide metabolism, xenobiotics biodegradation and metabolism, metabolism of cofactors and vitamins, energy metabolism, biosynthesis of other secondary metabolites, metabolism of terpenoids and polyketides. Diseases: infectious disease: viral, cancer: overview, infectious disease: bacterial, endocrine and metabolic disease, infectious disease: parasitic, cardiovascular disease, cancer: specific types, immune disease, substance dependence, neurodegenerative disease, drug resistance: antineoplastic. Organismal systems: endocrine system, immune system, circulatory system, digestive system, nervous system, environmental adaptation, sensory system, aging, development and regeneration, excretory system. Genetic information processing: translation, folding, sorting and degradation, transcription, replication and repair. Cellular processes: transport and catabolism, cellular community-eukaryotes, cell motility, cell growth and death. Environmental information processing: signal transduction, signaling molecules and interaction, membrane transport. ) 图 4 KEGG数目统计图 Fig. 4 KEGG number statistics chart |
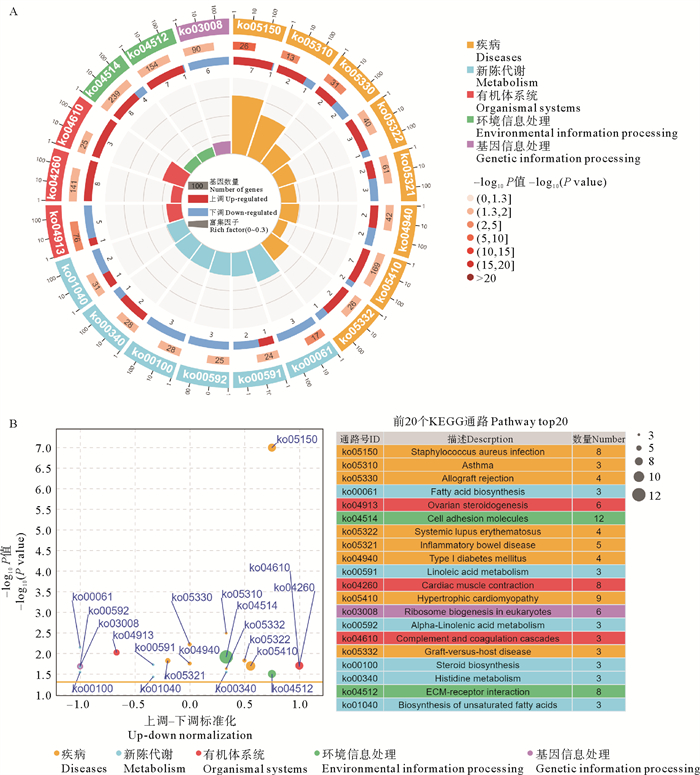
|
( A:富集圈图。B:z-score气泡图。注解同图 3。表格中通路从上到下依次为:黄色葡萄球菌感染、哮喘、同种异体移植物排斥反应、脂肪酸生物合成、卵巢类固醇合成、细胞黏附分子、系统性红斑狼疮、炎症性肠病、Ⅰ型糖尿病、亚油酸代谢、心肌收缩、肥厚型心肌病、真核生物核糖体的生物合成、α-亚麻酸、补体和凝血级联、移植物抗宿主病、类固醇生物合成、组氨酸代谢、ECM受体相互作用、不饱和脂肪酸合成。A: Enrichment circle diagram. B: z-score bubble chart. Annotation was the same as Figure 3. Table pathways from top to bottom: Staphylococcus aureus infection, Asthma, Allograft rejection, Fatty acid biosynthesis, Ovarian steroidogenesis, Cell adhesion molecules, Systemic lupus erythematosus, Inflammatory bowel disease, Type Ⅰ diabetes mellitus, Linoleic acid metabolism, Cardiac muscle contraction, Hypertrophic cardiomyopathy, Ribosome biogenesis in eukaryotes, Alpha-Linolenic acid metabolism, Complement and coagulation cascades, Graft-versus-host disease, Steroid biosynthesis, Histidine metabolism, ECM-receptor interaction, Biosynthesis of unsaturated fatty acids. ) 图 5 差异表达基因的KEGG富集分析 Fig. 5 KEGG enrichment analysis of differentially expressed genes |
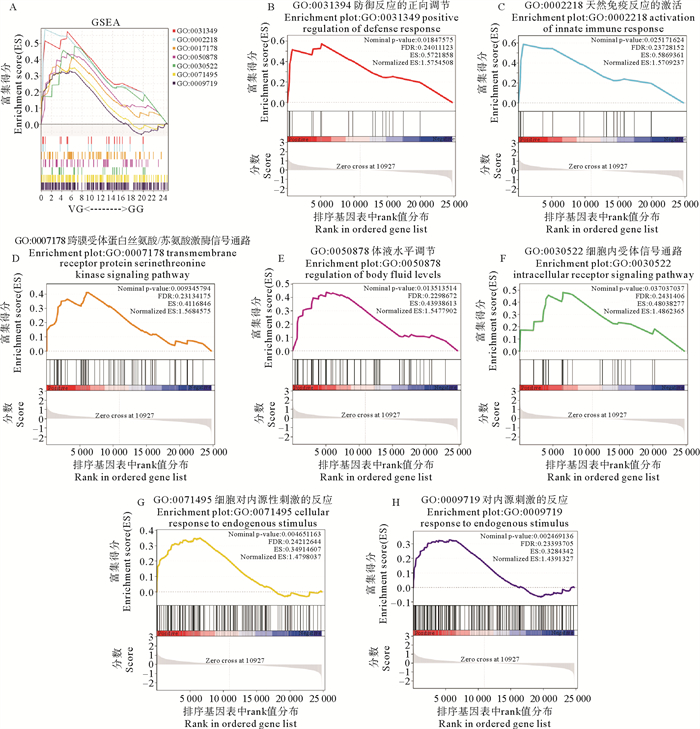
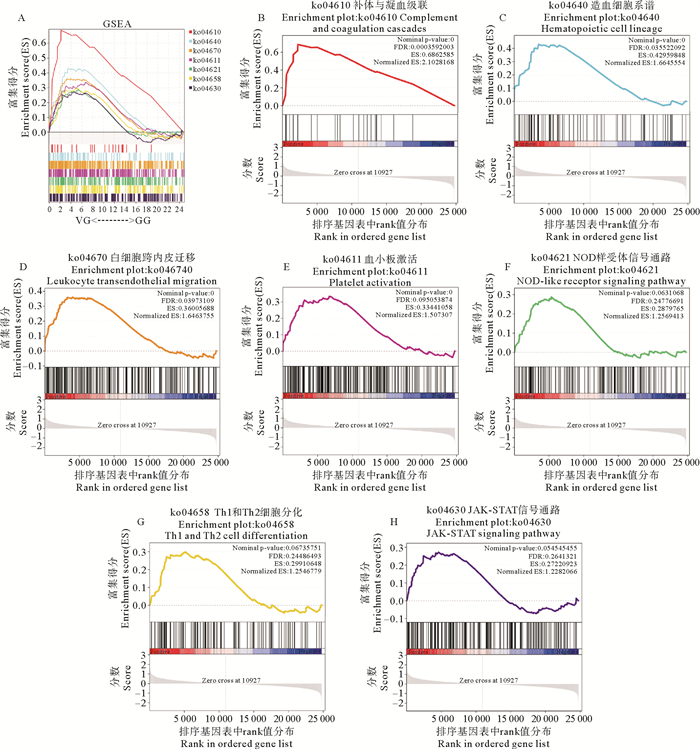
通过对两个组牙鲆鳃组织转录组数据进行GSEA分析,发现在实验组中与机体免疫和防御相关的GO条目发生显著上调(见图 6A),包括防御反应的正向调节(GO: 0031394)、天然免疫反应的激活(GO: 0002218)、跨膜受体蛋白丝氨酸/苏氨酸激酶信号通路(GO: 0007178)、体液水平调节(GO: 0050878)、细胞内受体信号通路(GO: 0030522)、细胞对内源性刺激的反应(GO: 0071495),以及对内源性刺激的反应(GO: 0009719)(见图 6B—H)。与免疫相关的KEGG通路也发生显著上调(见图 7A),包括补体与凝血级联(ko04610)、造血细胞系谱(ko04640)、白细胞跨内皮迁移(ko04670)、血小板激活(ko04611)、NOD样受体信号通路(ko04621)、Th1和Th2细胞分化(ko04658)和JAK-STAT信号通路(ko04630)(见图 7B—H)。
|
( A:总图。B—H:分别为与免疫相关的各GO term的详细GSEA图,均在VG组呈现整体上调趋势(判断标准为P<0.05,|NES|>1)。B:GO: 0031349防御反应的正向调节;C:GO: 0002218天然免疫反应的激活;D:GO: 0007178跨膜受体蛋白丝氨酸/苏氨酸激酶信号通路;E:GO: 0050878体液水平调节;F:GO: 0030522细胞内受体信号通路;G:GO: 0071495细胞对内源性刺激的反应;H:GO: 0009719对内源性刺激的反应。A: Overview of the GSEA enrichment analysis. B—H: Detailed GSEA maps of various GO terms related to immunity, all showing an overall upregulation trend in the VG group (P < 0.05, | NES |>1). B: GO: 0031349 positive regulation of defense response; C: GO: 0002218 activation of innate immune response; D: GO: 0007178 transmembrane receptor protein serine/threonine kinase signaling pathway; E: GO: 0050878 regulation of body fluid levels; F: GO: 0030522 intracellular receptor signaling pathway; G: GO: 0071495 cellular response to endogenous stimulus; H: GO: 0009719 response to endogenous stimulus. ) 图 6 GO的GSEA富集分析 Fig. 6 GSEA enrichment analysis of GO terms |
|
( A:总图。B—H:分别为与免疫相关的各KEGG pathway的详细GSEA图,均在VG组呈现整体上调趋势(判断标准为P<0.05,|NES|>1)。B:ko04610补体和凝血级联;C:ko04640造血细胞系谱;D:ko04670白细胞跨内皮迁移;E:ko04611血小板激活;F:ko04621 NOD样受体信号通路;G:ko04658 Th1和Th2细胞分化;H:ko04630 JAK-STAT信号通路。A: Overview of the GSEA enrichment analysis. B—H: Detailed GSEA maps of various KEGG pathways related to immunity, all showing an overall upregulation trend in the VG group (P < 0.05, | NES |>1). B: ko04610 complement and coagulation cascades; C: ko04640 hematopoietic cell lineage; D: ko04670 leukocyte transendothelial migration; E: ko04611 platelet activation; F: ko04621 NOD-like receptor signaling pathway; G: ko04658 Th1 and Th2 cell differentiation; H: ko04630 JAK-STAT signaling pathway. ) 图 7 KEGG的GSEA富集分析 Fig. 7 GSEA enrichment analysis of KEGG pathway |
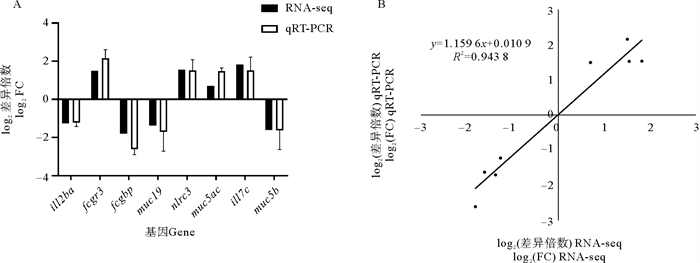
为验证RNA-seq的准确性和可重复性,选取8个免疫相关差异表达基因(il12ba、fcgr3、fcgbp、muc19、nlrc3、muc5ac、il17c和muc5b)进行qRT-PCR分析(见图 8)。结果显示,虽然所选基因在表达变化幅度上存在一定差异,但qRT-PCR定量结果与RNA-seq测序结果中的基因表达趋势基本一致(见图 8A),相关性分析显示R2=0.943 8(见图 8B),表明测序获得的转录组数据以及生物信息学分析结果准确可靠。
|
( A:qRT-PCR验证牙鲆鳃组织RNA-seq中的8个免疫相关DEGs的表达情况。B:RNA-seq结果和qRT-PCR分析基因表达的相关性。黑点表示RNA-seq和qRT-PCR的log2差异倍数。A: qRT-PCR validation of eight differentially expressed genes generated from RNA-seq results in the gills of flounder. B: The correlation between RNA-seq results and qRT-PCR analysis of gene expression. The black dots represented the log2(FC) results of RNA-seq and qRT-PCR. ) 图 8 DEGs的qRT-PCR验证 Fig. 8 qRT-PCR validation of DEGs |
鳃组织是鱼类与水环境交流进行气体交换的重要器官,已有研究证实在病原体攻击鱼体后,鳃组织通过补体活化、应激反应、抗原加工和呈递等途径参与机体防御[28],同时生成多种免疫因子,如白介素、干扰素、趋化因子、细胞因子等,在免疫防御中发挥重要作用[29]。
对牙鲆鳃转录数据进行GO和GSEA富集分析,发现鳗弧菌感染鱼体一天后GO条目显著富集于免疫效应器过程、细胞对刺激的反应和免疫防御反应激活及调节相关的条目,说明鳗弧菌感染鱼体后,鳃组织中的免疫细胞与入侵病原菌接触后受到刺激,从而激活下游免疫信号,引发机体免疫防御反应。有研究证明自噬能协助机体清除病原菌并抑制其生长和增殖,是通过自噬体和溶酶体结合分解自身需要被降解的成分达到维持细胞内环境稳定的细胞死亡方式,也是对抗外界病原体入侵的重要保护机制[30]。本研究中,鳗弧菌感染后DEGs显著富集于自噬相关条目,表明在鱼类鳃黏膜免疫中,自噬同样发挥着清除病原菌、抵御感染的作用。
鳗弧菌感染后牙鲆鳃组织中补体和凝血级联信号通路发生显著上调,表明补体系统和凝血级联反应可能是牙鲆抵御病原菌入侵和维持机体免疫健康的重要防御机制。文献[31]显示凝血级联反应和补体系统能通过相互交织和紧密联系共同发挥防止失血、免疫调节、调理吞噬和黏附并清除入侵病原体的作用。鳗弧菌感染半滑舌鳎(Cynoglossus semilaevis)后,补体和凝血级联通路显著差异表达,补体蛋白和凝血因子显著上调,作者推测在鱼类免疫系统中,凝血同样可以捕捉入侵的病原微生物,并通过补体系统促进固有免疫应答的发生[32]。这与我们的研究结果一致,表明补体和凝血级联通路在防御病原菌感染方面起重要的作用,但对其具体的应答机制仍需进一步研究。刺激隐核虫(Cryptocaryon irritans)感染大黄鱼(Larimichthys crocea)后,免疫相关DEGs显著富集于补体和凝血级联通路、血小板活化、造血细胞谱系、白细胞跨内皮迁移通路[33],这与本文GSEA富集分析结果一致,我们在VG组中也观察到上述通路的激活和显著上调表达。在牙鲆弹状病毒感染后发现,血小板与白细胞会相互作用促进白细胞募集渗出至炎症部位并释放促炎性介质,引发吞噬作用,同时刺激淋巴细胞通过血管内皮迁移至感染部位并驱动其免疫功能[34]。
本研究中,炎症性肠病、NOD样受体(NLRs)、JAK-STAT信号通路等显著富集,以及nlrc3、il17c、il12ba等因子显著表达,说明鳗弧菌感染后在鳃组织内引发炎症反应,结合上述结果可以推测血小板和白细胞共同作用激活针对入侵病原体的免疫反应。NOD样受体信号通路主要由模式识别受体NLR及其下游信号因子caspase-1、ASC等构成,能识别入侵病原体,启动机体免疫防御反应,作为先天免疫的关键组成部分发挥重要的免疫防御作用。我们前期对牙鲆MALTs中NLRs基因家族的研究证明NLRs参与鱼类免疫反应和炎症调节[35]。本研究发现鳗弧菌感染后,NOD样受体信号通路整体呈上调趋势,模式识别受体nlrc3显著上调表达,说明nlrc3被入侵病原体激活并通过信号通路启动机体免疫,进一步证明NLRs参与鱼类黏膜组织中的免疫应答。作为众多细胞因子受体的下游,JAK-STAT信号通路是传递和调节细胞因子信号的主要通路,除了能介导细胞炎症反应,在启动先天免疫、协调适应性免疫机制的机体免疫应答中也发挥重要作用[36]。对JAK-STAT信号通路的免疫作用研究主要集中于抗病毒感染方面,但近期关于其抗菌感染能力的研究也有了进展,在感染鳗弧菌的圆鳍鱼(Cyclopterus lumpus L.)中,JAK-STAT信号通路相关分子显著上调表达,参与了鱼体抗菌免疫过程[37],在对感染鳗弧菌的牙鲆脾脏研究中同样发现JAK-STAT信号通路的显著富集,进一步说明其在细菌感染后的免疫反应中发挥重要作用[38]。本研究结果也显示,在牙鲆鳃组织中,JAK-STAT信号通路同样参与了牙鲆针对鳗弧菌感染的免疫反应,说明其在黏膜组织的抗菌免疫中发挥重要功能,结合上述文献更全面地说明了JAK-STAT信号通路在免疫防御病原体感染中的重要性。此外,还有研究证明JAK-STAT信号通路对Th1和Th2的发育也至关重要,IL-12能通过调节STAT信号通路参与Th1细胞的发育和分化、增强T细胞增殖以及增加CD8 T细胞的密度;IL-4和IL-17通过参与STAT信号通路分别起到促进Th2细胞分化和调节Th17细胞与Treg细胞平衡的作用[39]。我们发现il12ba显著下调表达,Th1和Th2细胞分化通路整体呈现上调趋势,可能是il12ba分子被分解并激活了Th1细胞分化途径,参与了机体早期免疫应答过程,而il17c显著上调表达也表明在牙鲆鳃组织中细胞免疫在抵御病原菌感染方面发挥重要作用。
识别入侵病原体是机体免疫反应发生的基础。本研究中,鳗弧菌感染牙鲆后引发了强烈的宿主防御反应,模式识别受体tlr13和nlrc3显著上调表达且作为核心基因在基因互作中起关键作用。TLR和NLR具有识别微生物并激活机体产生非特异性和特异性免疫应答的能力。不同病原菌侵染鱼体后TLR和NLR会上调表达[40-42],尤其在鱼类黏膜组织中变化更加剧烈。例如:感染了鳗弧菌和杀鲑气单胞菌(Aeromonas salmonicida)的大菱鲆(Scophthalmus maximus)黏膜组织中,NLR的基因表达变化显著[43];而TLR的基因在感染了嗜水气单胞菌(Aeromonas hydrophila)的鲫(Carassius auratus)黏膜组织中也变化显著[28]。这些结果表明模式识别受体tlr13和nlrc3在鱼类黏膜组织对病原菌识别、诱导免疫应答和抗菌反应中发挥关键调控作用。
此外,具有细胞内病原体识别功能的凝集素表现出不同的调控模式,如cd209e、cd209c、masp1、sell、clec11A、clec10A、lgals3和mrc1上调表达,而ladd、colec10和lgals1下调表达。凝集素是一类不具有酶催化活性的糖蛋白或糖结合蛋白,能特异性识别、结合微生物表面的糖蛋白或糖基复合物。鱼类体表黏液凝集素分为L-鼠李糖凝集素、C型凝集素、半乳糖凝集素和Lily型凝集素4类,能通过应对外界病原侵染参与鱼类机体免疫防御[43]。CD209是C型凝集素家族的一员,可介导细胞相互作用并识别多种病原体,参与先天性和适应性免疫的调节[44],但目前的研究主要集中在哺乳动物中,在鱼类中研究较少。嗜水气单胞菌刺激斑马鱼(Danio rerio)后,发现干扰CD209的表达会抑制细菌免疫保护作用、T细胞活化和IgM的产生[45]。嗜水气单胞菌感染团头鲂(Megalobrama amblycephala)后,CD209表达显著增加,且表现出细菌结合和凝集活性[46]。迟缓爱德华氏菌(Edwardsiella tarda)感染半滑舌鳎后,CD209蛋白能显著促进白细胞对细菌的吞噬作用[47]。同样,我们发现鳗弧菌感染后牙鲆鳃组织中cd209e和cd209c表达升高,且cd209e是基因互作网络中的核心基因,cd209c也处于互作网络中心位置,说明二者在鱼体免疫中发挥关键调控作用。甘露糖结合凝集素(MBL)也是一种C型凝集素,能在MBL相关丝氨酸蛋白酶等因子的参与下通过凝集素途径激活补体系统,发挥抗感染作用[48]。在草鱼(Ctenopharyngodon idella)感染嗜水气单胞菌[49]和尼罗罗非鱼(Oreochromis niloticus)感染链球菌(Streptococcus iniae)[50]后,巨噬细胞甘露糖受体1(MR1)显著上调表达。本研究中,masp1和mrc1同样显著上调表达,结合补体和凝血级联信号通路显著上调,推测牙鲆受到鳗弧菌侵袭后,甘露糖结合凝集素捕捉结合病原菌,在相关丝氨酸蛋白酶参与下激活补体系统,发挥抵御入侵病原体的作用。同样属于C型凝集素家族的sell、clec11A和clec10A的上调表达,也说明C型凝集素在牙鲆鳃组织抗菌感染应答中发挥重要作用。lgals3是半乳糖凝集素家族的一员,在大多数细胞类型和组织中表达。鱼类免疫中,半乳糖凝集素介导的免疫识别和效应机制能有效地预防并对抗病毒、细菌和寄生虫感染[51]。在感染鳗弧菌的大菱鲆中galectin参与黏膜免疫应答[52]。在大西洋鲑[53]和尼罗罗非鱼[54]中,galectin-3通过识别和凝集病原体参与抗菌免疫应答。在哺乳动物的研究中也发现galectin-3在黏膜上皮中高表达[55]。结合本文结果,说明galectin-3在鱼类黏膜组织对病原微生物的免疫反应中发挥作用。因此,凝集素能保护鱼体免受病原体侵袭,在黏膜组织抵御病原菌感染的免疫应答过程中发挥识别病原体、启动免疫反应、激活下游信号通路、吞噬等作用,在鱼类黏膜免疫防御中起重要作用。
作为最常见且最古老的细胞死亡形式之一,吞噬是宿主抵御入侵病原体不可或缺的机制。宿主白细胞或中性粒细胞能吞噬入侵的病原菌,导致细菌细胞死亡[56]。本研究发现,吞噬作用相关基因表达量发生变化,除了前面提到的cd209e、cd209c、masp1和mrc1外,lyg也同样显著上调表达。lyg作为溶酶体相关基因,表达量增加可促进细菌的裂解。Cytosolic phospholipase A2在小龙虾(Procambarus clarkii)中被证明能通过调节抗菌肽参与细菌清除,并在巨噬细胞的吞噬过程中形成吞噬体,从而参与吞噬过程[57]。本研究中,pla2g4c表达量显著下调,推测其下调趋势可能是免疫反应激活后动态调节的表现,但其在鱼类抗感染免疫中的作用还有待进一步研究。另外,参与凋亡途径基因的表达也发生显著变化。细胞凋亡是第一个被确定的细胞死亡形式,能通过清除体内受损细胞维持机体内环境稳定,作为机体应对病原体的防御机制发挥免疫作用[58]。Bcl-2蛋白家族是调控细胞凋亡的重要因子,boka和bcl2l15都是Bcl-2蛋白家族中的一员,前者是凋亡调节因子,作为促凋亡蛋白激活细胞凋亡响应机体免疫,后者在凋亡中的作用还缺乏研究,但结合有关鱼类Bcl-2家族成员的研究,推测其可能同时具有抗凋亡及促凋亡双重作用,但还需进一步研究证实。本研究中上述凋亡相关因子的变化说明鳗弧菌感染牙鲆后引发了鳃组织的细胞凋亡,结合GO富集分析结果可知,吞噬作用、细胞凋亡和自噬等细胞死亡形式在抵御病原菌感染的鱼类黏膜免疫中发挥重要作用。
鱼类MHC分为MHC-Ⅰ、MHC-Ⅱ和MHC-Ⅲ3类,其中MHC-Ⅰ和MHC-Ⅱ研究较多,一般认为它们分别呈递内源性和外源性抗原[59],以二者为核心的抗原加工和呈递通路在免疫反应中起核心作用。本研究发现抗原呈递途径被快速激活,hla-dqb2和h2-ab1显著上调表达,说明MHC-Ⅱ类分子参与牙鲆鳃组织免疫应答。但cd5、tcrcb2、tcrd、ptprc、cd22、igll1等T、B淋巴细胞相关分子下调表达,暂未观察到适应性免疫的积极响应,推测细菌感染早期可能抑制T、B淋巴细胞的活动。这一结果与感染无乳链球菌的尼罗罗非鱼的响应一致[60],在感染鳃痘病毒的鲑鱼鳃组织中也有类似的结果[6]。前期研究表明,在黏膜组织中,相比于细胞免疫,体液免疫在免疫反应中起主要作用[61]。然而,ly6d、fcgr3等T淋巴细胞分化标志分子及免疫球蛋白相关分子的上调表达,说明已有适应性免疫发生。这可能是由于本研究的时间点较局限,具体原因还需进一步证实。
鳃作为鱼类重要的MALTs之一,其分泌的黏液是鱼体抵御病原入侵、参与机体免疫防御的重要组成部分。黏蛋白是黏液的主要成分,本研究发现黏蛋白基因也产生了不同程度的响应,muc5ac上调表达,而muc5b、muc19下调表达。Muc5ac、Muc5b和Muc19都属于分泌型黏蛋白。我们实验室近期研究发现,Muc5ac在牙鲆鳃和皮肤中表达,灭活爱德华氏菌免疫后其呈现显著上调表达,同时还能够清除黏液中的病原菌,在维持黏液屏障、参与黏膜免疫防御方面发挥重要作用[11]。但目前关于鱼类Muc5b和Muc19的研究匮乏。本研究中,二者呈现出不同于Muc5ac的表达变化趋势,对其在鳃黏膜免疫响应中的作用值得深入研究。
此外,还检测到牙鲆鳃中细胞黏附相关因子cldn3d和esama的上调表达。细胞黏附是维持组织间结构稳定、调控正常生理功能的基础,也是黏膜屏障的主要成分[62],cldn3d作为紧密连接蛋白在其中扮演重要角色,可为阻止有害物质和抗原进入机体提供有效保障。结合KEGG富集结果,发现cldn3d和esama在细胞黏附分子和白细胞跨内皮迁移信号通路中显著富集,推测细胞黏附也在牙鲆鳃组织抗感染中发挥作用。
4 结语本文采用RNA-seq和生物信息学分析方法,研究了感染鳗弧菌后牙鲆鳃黏膜相关淋巴组织的转录组数据,获得了620个差异表达基因,鉴定到大量抗感染免疫相关通路和基因,发现补体和凝血级联通路、白细胞跨内皮迁移、NOD样受体、JAK-STAT等免疫相关通路被激活,与疾病、细胞黏附相关的通路也显著富集;并且凝集素、模式识别受体、凋亡、吞噬、黏蛋白、黏膜防御屏障、T/B淋巴细胞等相关基因显著应答,体现出鱼体通过增强鳃黏膜固有免疫和获得性免疫应答,协同抵抗病原入侵。研究结果揭示了病原菌感染引发的鱼体免疫信号和抗感染机制,为深入理解鱼类黏膜免疫系统的抗菌免疫防御机制提供了新视角。
| [1] |
Peatman E, Beck B H. Mucosal Health in Aquaculture[M]. San Diego: Academic Press, 2015: 1-2.
(  0) 0) |
| [2] |
Salinas I, Zhang Y A, Sunyer J O. Mucosal immunoglobulins and B cells of teleost fish[J]. Developmental and Comparative Immunology, 2011, 35: 1346-1365. DOI:10.1016/j.dci.2011.11.009 (  0) 0) |
| [3] |
Caipang C M, Lazado C C, Brinchmann M F, et al. Infection-induced changes in expression of antibacterial and cytokine genes in the gill epithelial cells of Atlantic cod, Gadus morhua during incubation with bacterial pathogens[J]. Comparative Biochemistry & Physiology Part B: Biochemical and Molecular Biology, 2010, 156(4): 319-325. (  0) 0) |
| [4] |
Meng X Z, Shen Y B, Wang S T, et al. Complement component 3 (C3): An important role in grass carp (Ctenopharyngodon idella) experimentally exposed to Aeromonas hydrophila[J]. Fish and Shellfish Immunology, 2019, 88: 189-197. DOI:10.1016/j.fsi.2019.02.061 (  0) 0) |
| [5] |
Deng J J, Xu S, Li Y W, et al. Role of major histocompatibility complex Ⅱ antigen-presentation pathway genes in orange-spotted grouper infected with Cryptocaryon irritans[J]. Journal of Fish Diseases, 2020, 43(12): 1541-1552. DOI:10.1111/jfd.13256 (  0) 0) |
| [6] |
Gjessing M C, Krasnov A, Timmerhaus G, et al. The Atlantic salmon gill transcriptome response in a natural outbreak of salmon gill pox virus infection reveals new biomarkers of gill pathology and suppression of mucosal defense[J]. Frontiers in Immunology, 2020, 11: 2154. DOI:10.3389/fimmu.2020.02154 (  0) 0) |
| [7] |
Xie X, Jiang Y Y, Miao R J, et al. The gill transcriptome reveals unique antimicrobial features that protect Nibea albiflora from Cryptocaryon irritans infection[J]. Journal of Fish Diseases, 2021, 44(8): 1215-1227. DOI:10.1111/jfd.13382 (  0) 0) |
| [8] |
Ding Y Y, Zhang Y M, Shen Y B, et al. Aggregation and proliferation of B cells and T cells in MALTs upon Cryptocaryon irritans infection in large yellow croaker Larimichthys crocea[J]. Fish and Shellfish Immunology, 2024, 149: 109635. (  0) 0) |
| [9] |
Bu X L, Zhao W S, Zou H, et al. Immune response and apoptosis of gibel carp (Carassius auratus gibelio) gills to Chilodonella hexasticha infection: Insights from histopathological and multi-omics analyses[J]. Fish and Shellfish Immunology, 2024, 147: 109429. DOI:10.1016/j.fsi.2024.109429 (  0) 0) |
| [10] |
Liang C C, Sheng X Z, Tang X Q, et al. Structural characteristics and mucosal immune response of the interbranchial lymphoid tissue in the gills of flounder (Paralichthys olivaceus)[J]. Fish and Shellfish Immunology, 2022, 123: 388-398. DOI:10.1016/j.fsi.2022.03.022 (  0) 0) |
| [11] |
Wang J C, Gao J L, Sheng X Z, et al. Teleost Muc2 and Muc5ac: Key guardians of mucosal immunity in flounder (Paralichthys olivaceus)[J]. International Journal of Biological Macromolecules, 2024, 277: 134127. DOI:10.1016/j.ijbiomac.2024.134127 (  0) 0) |
| [12] |
Wang Z, Gerstein M, Snyder M. RNA-Seq: A revolutionary tool for transcriptomics[J]. Nature Reviews Genetics, 2009, 10(1): 57-63. DOI:10.1038/nrg2484 (  0) 0) |
| [13] |
Wu Q, Ning X H, Jiang S, et al. Transcriptome analysis reveals seven key immune pathways of Japanese flounder (Paralichthys olivaceus) involved in megalocytivirus infection[J]. Fish and Shellfish Immunology, 2020, 103: 150-158. DOI:10.1016/j.fsi.2020.05.011 (  0) 0) |
| [14] |
Mo Z L, Tan X G, Xu Y L, et al. A Vibrio anguillarum strain associated with skin ulcer on cultured flounder, Paralichthys olivaceus[J]. Chinese Journal of Oceanology and Limnology, 2001, 19: 319-326. DOI:10.1007/BF02850735 (  0) 0) |
| [15] |
李淑芳, 唐磊, 李杰, 等. 3株O3血清型鳗弧菌灭活疫苗的免疫原性和免疫保护效果[J]. 渔业科学进展, 2018, 39(1): 137-143. Li S F, Tang L, Li J, et al. Immunogenicity and protective efficacy of three inactivated Vibrio anguillarum serotype O3 vaccine preparations[J]. Progress in Fishery Sciences, 2018, 39(1): 137-143. (  0) 0) |
| [16] |
Li Z Y, Sun Y G, Tan R M, et al. Identification, characterization and complete genome analysis of a Vibrio anguillarum isolated from Sebastes schlegelii[J]. Microbial Pathogenesis, 2024, 190: 106611. DOI:10.1016/j.micpath.2024.106611 (  0) 0) |
| [17] |
Xing J, Xu H S, Wang Y, et al. Protective efficacy of six immunogenic recombinant proteins of Vibrio anguillarum and evaluation them as vaccine candidate for flounder (Paralichthys olivaceus)[J]. Microbial Pathogenesis, 2017, 107: 155-163. DOI:10.1016/j.micpath.2017.03.027 (  0) 0) |
| [18] |
Zhou X J, Xing J, Tang X Q, et al. Evaluation of bivalent vaccines candidates among VAA, OmpK and OmpR from Vibrio anguillarum in flounder (Paralichthys olivaceus)[J]. Developmental and Comparative Immunology, 2018, 85: 1-9. DOI:10.1016/j.dci.2018.03.014 (  0) 0) |
| [19] |
Chen S F, Zhou Y Q, Chen Y R, et al. Fastp: An ultra-fast all-in-one FASTQ preprocessor[J]. Bioinformatics, 2018, 34: 884-890. DOI:10.1093/bioinformatics/btx692 (  0) 0) |
| [20] |
Kim D, Langmead B, Salzberg S L. HISAT: A fast spliced aligner with low memory requirements[J]. Nature Methods, 2015, 12(4): 357. DOI:10.1038/nmeth.3317 (  0) 0) |
| [21] |
Langmead B, Salzberg S L. Fast gapped-read alignment with Bowtie2[J]. Nature Methods, 2012, 9(4): 357-359. DOI:10.1038/nmeth.1923 (  0) 0) |
| [22] |
Pertea M, Pertea G M, Antonescu C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J]. Nature Biotechnology, 2015, 33(3): 290. DOI:10.1038/nbt.3122 (  0) 0) |
| [23] |
Pertea M, Kim D, Pertea G M, et al. Transcript-level expression analysis of RNA-seq experiments with HISAT, StringTie and Ballgown[J]. Nature Protocols, 2016, 11(9): 1650. DOI:10.1038/nprot.2016.095 (  0) 0) |
| [24] |
Li B, Dewey CN. RSEM: Accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 2011, 12: 323. DOI:10.1186/1471-2105-12-323 (  0) 0) |
| [25] |
Love M I, Huber W, Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014, 15(12): 550. DOI:10.1186/s13059-014-0550-8 (  0) 0) |
| [26] |
Gao Y N, Wang Z W, Yang X, et al. Aflatoxin M1 and ochratoxin A induce a competitive endogenous RNA regulatory network of intestinal immunosuppression by whole-transcriptome analysis[J]. Science of the Total Environment, 2023, 854: 158777. DOI:10.1016/j.scitotenv.2022.158777 (  0) 0) |
| [27] |
Zhang L, Chen S K, Zeng X X, et al. Revealing the pathogenic changes of PAH based on multiomics characteristics[J]. Journal of Translational Medicine, 2019, 17(1): 231. DOI:10.1186/s12967-019-1981-5 (  0) 0) |
| [28] |
李雪. 嗜水气单胞菌诱导鲫皮肤和鳃免疫相关基因差异表达研究[D]. 徐州: 江苏师范大学, 2016. Li X. Differential Expression of Immune-Related Genes in the Skin and Gills of Carassius auratus Infected with Aeromonas hydrophil[D]. Xuzhou: Jiangsu Normal University, 2016. (  0) 0) |
| [29] |
Gomez D, Sunyer J O, Salinas I. The mucosal immune system of fish: The evolution of tolerating commensals while fighting pathogens[J]. Fish and Shellfish Immunology, 2013, 35(6): 1729-1739. DOI:10.1016/j.fsi.2013.09.032 (  0) 0) |
| [30] |
Glick D, Barth S, Macleod K F. Autophagy: Cellular and molecular mechanisms[J]. The Journal of Pathology, 2010, 221(1): 3-12. DOI:10.1002/path.2697 (  0) 0) |
| [31] |
Krem M M, Cera E D. Evolution of enzyme cascades from embryonic development to blood coagulation[J]. Trends in Biochemical Sciences, 2002, 27(2): 67-74. DOI:10.1016/S0968-0004(01)02007-2 (  0) 0) |
| [32] |
Zhang X, Wang S L, Chen S L, et al. Transcriptome analysis revealed changes of multiple genes involved in immunity in Cynoglossus semilaevis during Vibrio anguillarum infection[J]. Fish and Shellfish Immunology, 2015, 43(1): 209-218. DOI:10.1016/j.fsi.2014.11.018 (  0) 0) |
| [33] |
Yin F, Gao Q X, Tang B J, et al. Transcriptome and analysis on the complement and coagulation cascades pathway of large yellow croaker (Larimichthys crocea) to ciliate ectoparasite Cryptocaryon irritans infection[J]. Fish and Shellfish Immunology, 2016, 50: 127-141. DOI:10.1016/j.fsi.2016.01.022 (  0) 0) |
| [34] |
Tang X Q, Ma X B, Cao J, et al. The influence of temperature on the antiviral response of mIgM+ B lymphocytes against hirame Novirhabdovirus in flounder (Paralichthys olivaceus)[J]. Frontiers in Immunology, 2022, 13: 802638. DOI:10.3389/fimmu.2022.802638 (  0) 0) |
| [35] |
Liu Y Q, Sheng X Z, Tang X Q, et al. Genome-wide identification, phylogenetic relationships and expression patterns of the NOD-like receptor (NLR) gene family in flounder (Paralichthys olivaceus)[J]. Fish and Shellfish Immunology, 2023, 141: 109083. DOI:10.1016/j.fsi.2023.109083 (  0) 0) |
| [36] |
Xin P, Xu X Y, Deng C J, et al. The role of JAK/STAT signaling pathway and its inhibitors in diseases[J]. International Immunopharmacology, 2020, 80: 106210. DOI:10.1016/j.intimp.2020.106210 (  0) 0) |
| [37] |
Rao S S, Nelson P A, Lunde H S, et al. Evolutionary, comparative, and functional analyses of STATs and regulation of the JAK-STAT pathway in lumpfish upon bacterial and poly(I: C)exposure[J]. Frontiers in Cellular and Infection Microbiology, 2023, 13: 1252744. DOI:10.3389/fcimb.2023.1252744 (  0) 0) |
| [38] |
Ning X H, Sun L. Gene network analysis reveals a core set of genes involved in the immune response of Japanese flounder (Paralichthys olivaceus) against Vibrio anguillarum infection[J]. Fish and Shellfish Immunology, 2020, 98: 800-809. DOI:10.1016/j.fsi.2019.11.033 (  0) 0) |
| [39] |
Hu Q, Bian Q H, Rong D C, et al. JAK/STAT pathway: Extracellular signals, diseases, immunity, and therapeutic regimens[J]. Frontiers in Bioengineering and Biotechnology, 2023, 11: 1110765. DOI:10.3389/fbioe.2023.1110765 (  0) 0) |
| [40] |
Rauta P R, Samanta M, Dash H R, et al. Toll-like receptors (TLRs) in aquatic animals: Signaling pathways, expressions and immune responses[J]. Immunology Letters, 2014, 158(1-2): 14-24. DOI:10.1016/j.imlet.2013.11.013 (  0) 0) |
| [41] |
Zhang L, Gao Z Y, Yu L, et al. Nucleotide-binding and oligomerization domain (NOD)-like receptors in teleost fish: Current knowledge and future perspectives[J]. Journal of Fish Diseases, 2018, 41(9): 1317-1330. DOI:10.1111/jfd.12841 (  0) 0) |
| [42] |
Zhang L, Cao M, Li Q, et al. Genome-wide identification of NOD-like receptors and their expression profiling in mucosal tissues of turbot (Scophthalmus maximus L.) upon bacteria challenge[J]. Molecular Immunology, 2021, 134: 48-61. DOI:10.1016/j.molimm.2021.02.003 (  0) 0) |
| [43] |
刘晓晨. 鲟鱼体表黏液及其凝集素分离、鉴定的研究[D]. 大连: 大连海洋大学, 2022. Liu X C. Isolation and Characterization of Acipenser schrenki Brandt Skin Mucus and Its Lectins[D]. Dalian: Dalian Ocean University, 2022. (  0) 0) |
| [44] |
Rahimi N. C-type lectin CD209L/L-SIGN and CD209/DC-SIGN: Cell adhesion molecules turned to pathogen recognition receptors[J]. Biology (Basel), 2020, 10(1): 1. (  0) 0) |
| [45] |
Lin A F, Xiang L X, Wang Q L, et al. The DC-SIGN of zebrafish: Insights into the existence of a CD209 homologue in a lower vertebrate and its involvement in adaptive immunity[J]. Journal of Immunology, 2009, 183(11): 7398-7410. DOI:10.4049/jimmunol.0803955 (  0) 0) |
| [46] |
Cui H J, Shen X X, Zheng Y C, et al. Identification, expression patterns, evolutionary characteristics and recombinant protein activities analysis of CD209 gene from Megalobrama amblycephala[J]. Fish and Shellfish Immunology, 2022, 126: 47-56. DOI:10.1016/j.fsi.2022.04.043 (  0) 0) |
| [47] |
Jiang S, Sun L. Tongue sole CD209: A pattern-recognition receptor that binds a broad range of microbes and promotes phagocytosis[J]. International Journal of Molecular Sciences, 2017, 18(9): 1848. DOI:10.3390/ijms18091848 (  0) 0) |
| [48] |
Ali Y M, Lynch N J, Haleem K S, et al. The lectin pathway of complement activation is a critical component of the innate immune response to pneumococcal infection[J]. PLoS Pathogens, 2012, 8(7): e1002793. DOI:10.1371/journal.ppat.1002793 (  0) 0) |
| [49] |
Wang L, Liu L C, Zhou Y, et al. Molecular cloning and expression analysis of mannose receptor C type 1 in grass carp (Ctenopharyngodon idella)[J]. Developmental and Comparative Immunology, 2014, 43(1): 54-58. DOI:10.1016/j.dci.2013.10.006 (  0) 0) |
| [50] |
Zhu J J, Li C, Ao Q W, et al. Trancriptomic profiling revealed the signatures of acute immune response in tilapia (Oreochromis niloticus) following Streptococcus iniae challenge[J]. Fish and Shellfish Immunology, 2015, 46(2): 346-353. DOI:10.1016/j.fsi.2015.06.027 (  0) 0) |
| [51] |
Liu F T, Stowell S R. The role of galectins in immunity and infection[J]. Nature Reviews Immunology, 2023, 23(8): 479-494. DOI:10.1038/s41577-022-00829-7 (  0) 0) |
| [52] |
Tian M Y, Xu D X, Fu Q, et al. Galectins in turbot (Scophthalmus maximus L.): Characterization and expression profiling in mucosal tissues[J]. Fish and Shellfish Immunology, Elsevier, 2021, 109: 71-81. DOI:10.1016/j.fsi.2020.12.004 (  0) 0) |
| [53] |
Chen M K, Liu X, Zhou J, et al. Molecular characterization and expression analysis of galectins in Japanese pufferfish (Takifugu rubripes) in response to Vibrio harveyi infection[J]. Fish and Shellfish Immunology, Elsevier, 2019, 86: 347-354. DOI:10.1016/j.fsi.2018.11.054 (  0) 0) |
| [54] |
Niu J Z, Huang Y, Liu X C, et al. Functional characterization of galectin-3 from Nile tilapia (Oreochromis niloticus) and its regulatory role on monocytes/macrophages[J]. Fish and Shellfish Immunology, 2019, 95: 268-276. DOI:10.1016/j.fsi.2019.10.043 (  0) 0) |
| [55] |
Lippert E, Falk W, Bataille F, et al. Soluble galectin-3 is a strong, colonic epithelial-cell-derived, lamina propria fibroblast-stimulating factor[J]. Gut, 2007, 56(1): 43-51. DOI:10.1136/gut.2005.081646 (  0) 0) |
| [56] |
Brown G C. Cell death by phagocytosis[J]. Nature Reviews Immunology, 2024, 24(2): 91-102. DOI:10.1038/s41577-023-00921-6 (  0) 0) |
| [57] |
Lin C Y, Xu W B, Li B Z, et al. Identification and functional analysis of cytosolic phospholipase A2 (cPLA2) from the red swamp crayfish Procambarus clarkii: The first evidence of cPLA2 involved in immunity in invertebrates[J]. Fish and Shellfish Immunology, 2023, 140: 108944. DOI:10.1016/j.fsi.2023.108944 (  0) 0) |
| [58] |
Ai Y W, Meng Y T, Yan B, et al. The biochemical pathways of apoptotic, necroptotic, pyroptotic, and ferroptotic cell death[J]. Molecular Cell, 2024, 84(1): 170-179. DOI:10.1016/j.molcel.2023.11.040 (  0) 0) |
| [59] |
Wilson A B. MHC and adaptive immunity in teleost fishes[J]. Immunogenetics, 2017, 69(8-9): 521-528. DOI:10.1007/s00251-017-1009-3 (  0) 0) |
| [60] |
Linden S K, Sutton P, Karlsson N G, et al. Mucins in the mucosal barrier to infection[J]. Mucosal Immunology, 2008, 1(3): 183-197. DOI:10.1038/mi.2008.5 (  0) 0) |
| [61] |
Zhang R, Zhang L L, Ye X, et al. Transcriptome profiling and digital gene expression analysis of Nile tilapia (Oreochromis niloticus) infected by Streptococcus agalactiae[J]. Molecular Biology Reports, 2013, 40(10): 5657-5668. DOI:10.1007/s11033-013-2667-3 (  0) 0) |
| [62] |
杨倩. 黏膜免疫及其疫苗设计[M]. 北京: 科学出版社, 2016. Yang Q. Mucosal Immunology and Vaccine Design[M]. Beijing: Science Press, 2016. (  0) 0) |
 2025, Vol. 55
2025, Vol. 55


