2. 青岛海洋科技中心海洋渔业科学与食物产出过程功能实验室,山东 青岛 266237
病毒性出血性败血症(Viral hemorrhagic septicemia,VHS)是世界动物卫生组织(WOAH)要求通报的鱼类病毒性疾病,由病毒性出血性败血症病毒(Viral hemorrhagic septicemia virus,VHSV)感染引起[1]。VHSV隶属于弹状病毒科(Rabdoviridae)粒外弹状病毒属(Novirhabdovirus),基因大小为11~12 kb,由6个基因按3′ N-P-M-G-NV-L 5′顺序排列,并分别编码核蛋白(N)、磷蛋白(P)、质蛋白(M)、糖蛋白(G)和聚合酶蛋白(L)五种结构蛋白和一个非结构蛋白NV[2]。VHSV首次在欧洲养殖虹鳟中被检测到,随后在韩国、日本、加拿大等多个国家流行,可感染100多种海淡水鱼类,给世界鱼类养殖业造成了巨大威胁[3-4]。根据G基因的核酸序列差异,可将VHSV不同分离株归为四种主要基因型(Ⅰ、Ⅱ、Ⅲ和Ⅳ),其中Ⅰ和Ⅳ型分别具有5种(Ⅰa~Ⅰe)和4种亚型(Ⅳa~Ⅳd),不同基因型在流行区域、宿主毒力方面具有明显的差异[5-7],甚至相同基因型不同分离株间的毒力也会存在较大差异[8-9]。Ⅳa型毒株主要在东亚地区与北美洲流行,系统进化分析显示亚洲地区流行的Ⅳa型毒株与北美地区流行株仍存在一定的差异[10-11]。VHSV Ⅳa基因型可感染多种海淡水鱼类,其中在中国、日本及韩国养殖牙鲆(Paralichthys olivaceus)为该毒株的重要感染宿主[12-14]。为了预防和控制该病的暴发与流行,多种类型的VHSV候选疫苗被开发[15-17],但目前疫苗还未商业化,无法大规模的使用。因此,开发一种快速、灵敏、便捷、适用于现场的VHSV检测技术,对于VHS疫病的防控具有重要价值。
针对鱼类病毒病的诊断,细胞培养法仍是WOAH作为VHS疾病诊断的金标准而推荐使用[18],但因其耗时长,需要专业细胞培养设备,同时需要经验丰富的技术人员,影响其广泛应用。为此,多种基于分子生物学与免疫学技术的VHSV检测方法相继建立,主要包括一步法RT-PCR[19]、实时RT-PCR[10]、RPA[20]、原位杂交[21]、宏阵列分析[22]及胶体金免疫层析技术[23]等,这些方法大大提升了检测的效率与便捷性。但是,这些检测方法难以同时满足检测的便捷性与高灵敏度,且部分技术对仪器设备与操作人员具有较高的要求。随着分子诊断技术的发展,基于RPA和CRISPR技术开发的疾病诊断技术快速得到大家的认可与重视,该诊断体系兼有RPA快速高效的核酸扩增及CRISPR对基因序列精准识别的技术优势,能有效实现更短的检测时间、更高的检测准确性和灵敏度[24-25]。同时,该检测可以结合层析试纸和荧光观察通过肉眼对结果进行判读,从而使其摆脱了对仪器设备和专业人员操作的依赖,极大地适应了现场快速诊断的需求[26]。目前,RPA-CRISPR/Cas12a技术已用于检测多种水产病原体,如鳞片脱落病毒[27]、传染性造血器官坏死病毒(Infectious hematopoietic necrosis virus,IHNV)[28]、罗非鱼湖病毒[29]等。然而,到目前为止,基于RPA和CRISPR/Cas12a的VHSV(Ⅳa)检测方法仍未建立。
为了实现对VHSV的便捷、高灵敏度检测,本研究开发了一种基于RPA和CRISPR/Cas12a的检测方法,检测结果可通过试纸条和荧光法实现肉眼可判断(见图 1)。通过筛选RPA引物和crRNA、优化RPA反应温度和时间,使整个检测过程可在1 h内完成,具有较高的灵敏度和特异性,可用于VHSV感染的早期现场诊断,对及时有效地防控VHS暴发与流行具有重要意义。
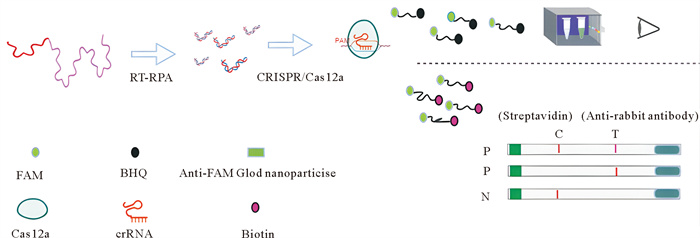
|
( 利用RT-RPA扩增目标序列,Cas12a-crRNA复合体识别到目的基因后激活Cas12a的旁切活性,从而对报告分子进行切割,然后根据不同的报告分子,分别通过紫外光激发或利用试纸条显色进行结果判读。Amplifying the target sequence by RT-RPA, then the Cas12a-crRNA complex activates the collateral cleavage activity of Cas12a after recognizing the target gene, thereby cleaving the reporter molecule. The detection results can then be interpreted under UV light excitation or through color change on lateral flow test strips, depending on the different reporter molecules. ) 图 1 靶向VHSV的RPA-CRISPR/Cas12a检测流程示意图 Fig. 1 Schematic workflow of RPA-CRISPR/Cas12a assay for VHSV detection |
VHSV N基因片段的制备:提取VHSV感染的牙鲆脾脏总RNA,逆转录成cDNA后,利用靶向病毒N基因的引物对F5/R5(见表 1)扩增目标序列。扩增产物经凝胶电泳检测确认后,利用胶回收试剂盒回收目的序列并连接至pClone007载体(TsingKe,南京)。重组载体经测序确认后,使用含有T7启动子的引物F4和R4(见表 1)对N基因片段进行PCR扩增,扩增产物经胶回收后获得线性N基因片段,以作为crRNA筛选和体外转录的模板。
|
|
表 1 用于VHSV检测的引物核酸序列表 Table 1 The primers and oligonucleotides used in VHSV detection |
标准RNA模板的制备:根据体外转录试剂盒(Vazyme)的使用说明,以纯化的带T7启动子序列的N基因扩增产物作为体外转录(IVT)的DNA模板进行体外转录,结束后以Trizol法提取转录的RNA产物。使用NanoDrop 2000分光光度仪(Thermo Scientific,美国)测定纯化的IVT产物浓度,并基于测定浓度参考以下公式计算N基因转录产物的拷贝数:RNA拷贝数=(M×6.022×1023)/(n×109×330),其中M代表RNA的质量(以ng为单位),n是RNA的碱基长度,设定碱基的平均分子量为330 Da。
1.2 RT-RPA引物及crRNA筛选基于实验室保存的VHSV毒株G基因序列比对分析结果,该牙鲆感染毒株为Ⅳa基因型。鉴于此,我们从NCBI上获取所有Ⅳa基因型的VHSV病毒株N基因序列,通过Bioedit进行保守序列分析,然后根据RPA引物设计的要求(https://www.twistdx.co.uk/en/support/manuals/twistamp-manuals)针对N基因保守序列设计RPA扩增的三条正向(F1、F2、F3)与反向引物(R1、R2、R3)(见表 1)。提取感染VHSV牙鲆脾脏的总RNA,以此为模板进行RT-RPA扩增,RPA扩增体系包括:缓冲液29.5 μL、10 μmol/L正向引物与反向引物各2.4 μL、ddH2O 8.2 μL、RNA模板4 μL、逆转录酶(M-MuLV)0.5 μL。RPA反应液轻微震荡至全部溶解,短暂离心后加入2.5 μL的MgOAc混匀,总体积为50.5 μL,于40 ℃反应32 min。扩增产物进行2.5%琼脂糖凝胶电泳,然后在化学发光仪下观察拍照,根据扩增效果优选引物组合。
基于优选获得的RPA引物组合,根据其扩增序列,设计3条CRISPR反应的crRNA序列(见表 1),由上海生工生物公司合成。CRISPR/Cas12a反应体系包括:ssDNA探针(1 μmol/L FQ-reporter)2 μL、10×缓冲液2 μL、crRNA(1 μmol/L)2 μL、Cas12a蛋白2 μL、N基因片段(30 ng/μL)4 μL、ddH2O 8 μL,总体积20 μL,于37 ℃反应15 min,并以ddH2O作为阴性对照。反应结束后,将反应产物置于化学发光仪中拍照,并吸取10 μL反应液加入到90 μL超纯水中混匀,然后用酶标仪对荧光值进行测定。最终,根据反应产物的荧光显色和荧光强度值,筛选可高效介导CRISPR体系切割活性的crRNA。
1.3 RPA-CRISPR/Cas12a反应时间和温度的优化以病毒感染的脾脏RNA为模板,根据反应温度的预实验结果,设计合适的温度范围和温度梯度,用筛选到的最佳引物在合适的温度梯度下进行RT-RPA反应,反应结束后用DNA快速提取液提取RT-RPA产物,经琼脂糖凝胶电泳判断最佳反应温度。在获得最适反应温度后,设置反应时间梯度进行RT-RPA反应,扩增产物同样经琼脂糖凝胶电泳,确定最佳反应时间。
1.4 RPA-CRISPR/Cas12a的灵敏度和特异性检测将体外转录的VHSV-N基因RNA稀释为9×104、9×103、9×102、9×101、9×100 copies/μL作为灵敏度检测模板,以ddH2O作为阴性对照。RPA-CRISPR/Cas12a在优选确定的最佳反应条件下对各稀释样本进行检测,反应产物分别以荧光法(探针:FQ-reporter)与侧向层析试纸(探针:FB-reporter)进行结果判读,以确定检测灵敏度。为了验证RPA-CRISPR/Cas12a检测的特异性,从牙鲆弹状病毒(Hirame novirhabdovirus,HIRRV)、IHNV和VHSV感染的细胞浓缩液中提取RNA作为检测模板,分别进行RPA-CRISPR/Cas12a检测,以ddH2O作为阴性对照,通过荧光法与试纸法判读检测的特异性。
1.5 临床样本检测将反应体系,检测样本和相关检测用品封闭好放在实验台上,然后用核酸清除剂喷实验台和实验用具,等待10 min,然后用浸泡在75%的酒精棉擦拭掉表面的核酸清除液,然后进行检测。检测中避免不同样本添加时越过其他样本检测管上方,并正确使用移液枪,防止气溶胶污染。为了评估RPA-CRISPR/Cas12a的诊断性能,根据OIE《水生动物诊断手册》1.2章的“感染性疫病诊断分析验证的原理和方法”,在95%以上置信区间、允许误差5%范围内对本检测方法的52个检测反应进行DSp和DSe测试。对经OIE推荐的RT-PCR方法验证为VHSV阳性的37份自然感染的组织样品以及验证为VHSV阴性的15份组织样品进行检测,评估本研究构建检测方法的DSp和DSe。
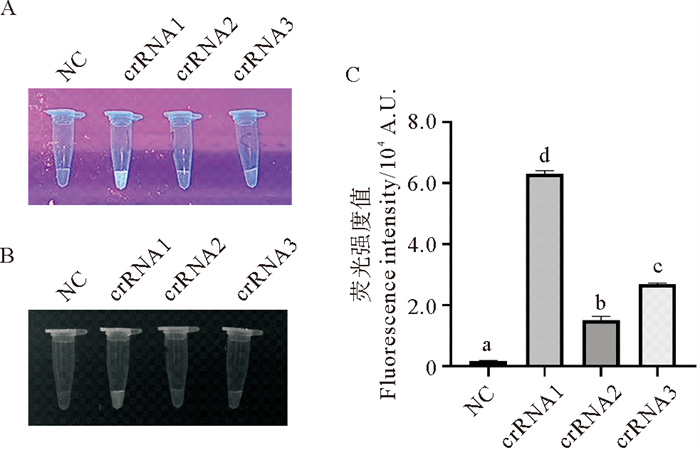
2 结果 2.1 RT-RPA引物和crRNA的筛选crRNA是影响RPA-CRISPR/Cas12a检测反应的关键要素,为了获得最佳的用于VHSV检测的Cas12a系统的crRNA,对NCBI上所有VHSV N基因的核酸序列进行对比。对比结果显示,VHSV N基因的稳定区内没有适合设计crRNA识别的序列。将本实验室VHSV N基因的测序结果与其他VHSV基因序列进行对比,我们发现,本实验室VHSV属于Ⅳa型菌株,为亚洲地区流行毒株。然后对NCBI上划分为VHSV(Ⅳa)型的核酸序列进行对比,在N基因稳定区设计3个crRNA识别位点应用于Cas12a检测,并基于荧光法筛选最佳crRNA。如图 2所示,crRNA1展现出最强的荧光信号(见图 2A和2B),通过酶标仪对CRISPR/Cas12a反应15 min的荧光强度值进行检测,结果显示,Cas12a-crRNA1切割产生的荧光强度值远高于其他Cas12a-crRNA(见图 2C),因此,我们选择了crRNA1应用于后续的检测实验。
|
( A:含3种不同crRNA的CRISPR/Cas12a反应体系在UV光下的显色结果(手机拍摄);B:含3种不同crRNA的CRISPR/Cas12a反应体系在化学发光仪中的显色结果;C: 酶标仪测定的3种CRISPR/Cas12a反应体系的荧光值,不同字母代表组间具有显著差异(p<0.05)。NC: 阴性对照。A: Visualization of CRISPR/Cas12a reactions containing 3 different crRNAs under UV light (captured by a smartphone camera); B: Visualization of CRISPR/Cas12a reactions containing 3 different crRNAs in an Imager System; C: Fluorescence values of 3 CRISPR/Cas12a reactions determined by microplate reader, with different letters representing significant differences between groups (p<0.05). NC: Negative control. ) 图 2 基于RPA-CRISPR/Cas12a反应体系筛选crRNA Fig. 2 crRNA screening based on the RPA-CRISPR/Cas12a assay |
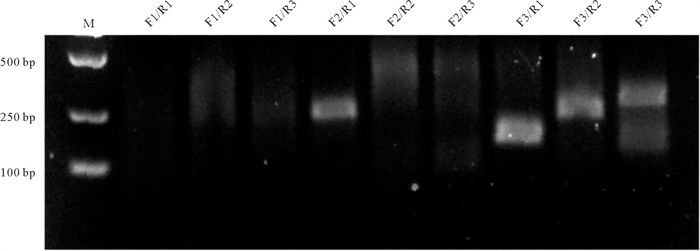
除了Cas12a的剪切效率外,RT-RPA扩增效率对RPA-CRISPR/Cas12a检测也十分关键。基于crRNA识别位点,我们设计了3个上游引物和3个下游引物,形成了9种引物组合,将它们应用于RT-RPA扩增。通过RT-RPA产物的凝胶电泳结果显示,F3和R1产生的扩增条带最为明亮特异(见图 3)。因此,F3、R1和crRNA1被应用于接下来的实验。
|
( F1~F3: VHSV正向引物;R1~R3: VHSV反向引物,引物组合标注在泳道上方。M:DNA分子量标准,条带大小标注在图片左侧。F1~F3: Forward primers of VHSV; R1~R3: Reverse primers of VHSV, the primer combinations are indicated above the lanes. M: DNA marker, the sizes of the DNA marker bands are indicated on the left side of the image. ) 图 3 VHSV RT-RPA检测引物筛选图 Fig. 3 Screening of primer pairs for VHSV RT-RPA assay |
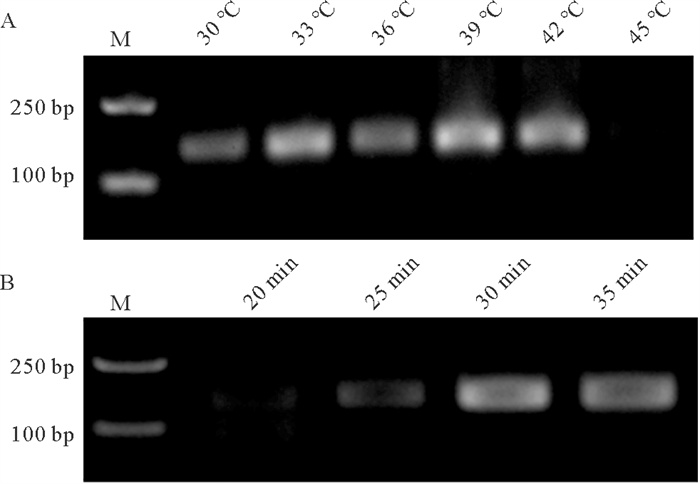
检测反应的温度是提高RT-RPA扩增效率的重要因素,而RT-RPA扩增的温度和时间与加入的逆转录酶、引物和RPA反应体系有关。本实验通过简并引物对VHSV进行检测,并基于预实验结果,设置了6个反应温度(30、33、36、39、42和45 ℃)进行RT-RPA反应,通过产物凝胶电泳结果确定RT-RPA反应的温度范围和最适反应温度。凝胶电泳结果显示,RT-RPA可在30~42 ℃温度范围内进行扩增反应,在39 ℃产生的条带最亮,所以最适的扩增温度为39 ℃(见图 4A)。
|
( A:在不同的温度下RT-RPA扩增产物的凝胶结果图;B:在最适温度(39 ℃)下不同时间RT-RPA扩增产物的凝胶结果图。反应温度和时间标注在泳道上方。A: Agarose gel electrophoresis of RT-RPA products amplified under different temperatures; B: Agarose gel electrophoresis of RT-RPA products amplified for different times at optimal reaction temperature (39 ℃). Reaction temperature and time are indicated above the lanes. ) 图 4 VHSV N基因RT-RPA扩增反应温度和时间的优化 Fig. 4 Optimization of the reaction temperature and time for RT-RPA amplification of the VHSV N gene |
在最适的反应温度下,基于预实验结果设置合适的RT-RPA反应时间点来确定最佳的反应时间。通过RT-RPA产物的凝胶电泳结果显示,随着反应时间的增加,凝胶电泳的条带更加明亮清晰,差异逐渐变小,在反应30 min时反应结果条带最亮,所以最佳的反应时间为30 min(见图 4B)。因此,RT-RPA反应在39 ℃下反应30 min为最适的RT-RPA反应条件。
2.3 RPA-CRISPR/Cas12a检测的灵敏性RPA-CRISPR/Cas12a检测的灵敏度是通过检测梯度稀释体外转录的VHSV(Ⅳa)N基因片段(9×100~9×104 copies/μL)确定的。如图 5所示,RT-RPA产物凝胶电泳图显示RT-RPA可以在9 copies/μL时扩增出明显的条带(见图 5A),且RPA-CRISPR/Cas12a荧光法检测结果在紫外线下可以产生明显的荧光信号(见图 5B和5C),通过对荧光信号强度值的检测结果分析,实验组的荧光值显著区别于对照组(见图 5D)。RPA-CRISPR/Cas12a试纸条的检测结果显示,RPA-CRISPR/Cas12a可以在9 copies/μL时检测出阳性信号,这与荧光法的结果一致(见图 5E)。
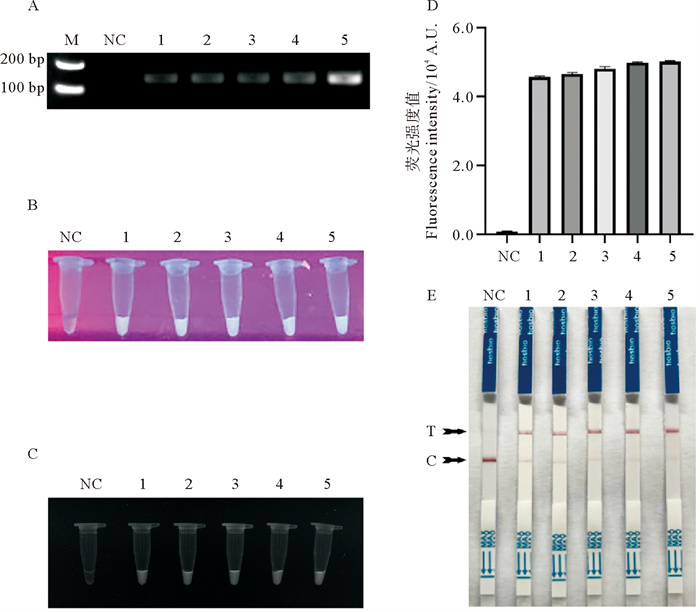
|
( A:RT-RPA扩增产物凝胶电泳结果;B:UV光激发下手机拍摄;C:UV光激发下化学发光仪拍摄;D:不同浓度梯度荧光法的荧光值通过酶标仪检测,每列表示一式三份荧光值的平均值±标准差;E:RPA-CRISPR/Cas12a试纸条灵敏度显色结果。1—5:检测样本浓度分别为9×100、9×101、9×102、9×103、9×104 copies/μL; M:DNA分子量标准, 条带大小标注在图片左侧;NC:阴性对照。A: Visualization of RT-RPA products by agarose gel electrophoresis; B: Sensitivity images were captured under UV light using a smartphone camera; C: Sensitivity images were imaged under UV light using an imager system; D: The fluorescent intensities were examined by microplate reader, each column indicates the mean of the triplicate fluorescence values±SD; E: Sensitivity examination by RPA-CRISPR/Cas12a lateral flow dipstick assay. 1—5: Template concentration is 9×100、9×101、9×102、9×103、9×104 copies/μL. M: DNA marker, the sizes of the DNA ladder bands are shown on the left. NC: Negative control. ) 图 5 VHSV RPA-CRISPR/Cas12a灵敏度检测 Fig. 5 Sensitivity analysis of RPA-CRISPR/Cas12a assay for VHSV detection |
为了验证RPA-CRISPR/Cas12a检测的特异性,从HIRRV、IHNV和VHSV感染的细胞培养液中提取核酸作为检测模板进行RPA-CRISPR/Cas12a检测。如图 6所示,RPA-CRISPR/Cas12a的荧光法和试纸条法检测结果显示,只有VHSV感染的细胞浓缩液提取的核酸样本检测结果为阳性。因此,RPA-CRISPR/Cas12a检测具有较高的特异性,且对粒外弹状病毒属的HIRRV和IHNV没有交叉反应。
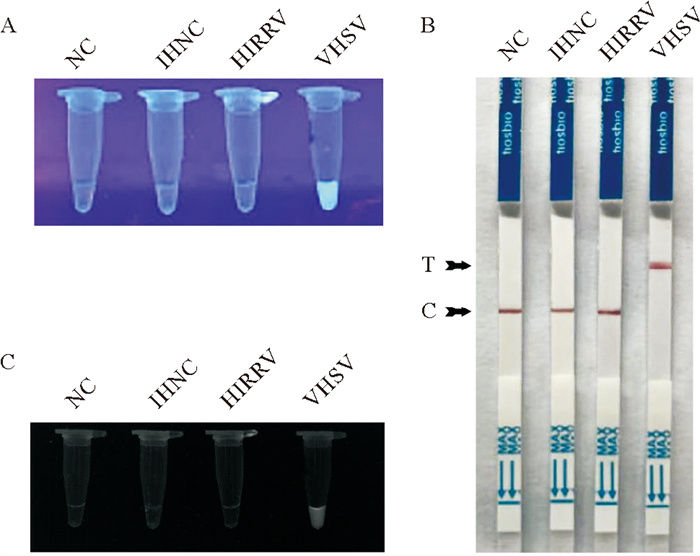
|
( A:荧光法的特异性检测通过UV光用智能手机拍摄;B:荧光法的特异性检测通过UV光用化学发光仪拍摄;C:试纸条法特异性检测结果。NC:阴性对照。A: Specificity examination was imaged under UV light using a smartphone camera; B: Specificity examination was imaged under UV light using an Imager System; C: Specificity analysis using lateral flow strip. NC: Negative control. ) 图 6 VHSV RPA-CRISPR/Cas12的特异性检测 Fig. 6 Specificity analysis of RPA-CRISPR/Cas12a assay for VHSV detection |
为了评估RPA-CRISPR/Cas12a临床检测的实用性,从临床样本中提取核酸,然后分别进行RT-PCR检测和RPA-CRISPR/Cas12a检测。如表 2所示,RPA-CRISPR/Cas12a检测出37个VHSV阳性样本和15个VHSV阴性样本,检测结果与RT-PCR检测结果一致,该检测的DSp为100%,DSe为100%(见表 2)。
|
|
表 2 临床样本VHSV的RPA-CRISPR/Cas12a检测与RT-PCR检测的对比 Table 2 The clinical performance of RPA-CRISPR/Cas12a assay for VHSV detection compared with RT-PCR |
感染VHSV的鱼体根据其发病进程与临床症状特征可分为急性、慢性和潜伏感染,急性感染通常伴随着快速发病的高死亡率,慢性感染死亡率相对较低,但会在一个较长的时间周期内发生。潜伏感染鱼体可携带病毒而不表现出VHS的任何临床症状,但很容易成为VHSV传播的载体,对鱼类养殖业产生巨大威胁[30]。目前,由于VHSV商品化疫苗以及有效治疗措施的缺失,给VHS疫病的防控带来了更大的挑战。本文基于RPA与CRISPR/Cas12a技术针对Ⅳa基因型VHSV毒株构建了一种先进的分子生物学检测方法,该方法具有较高的准确性与灵敏度,整个检测流程可于1 h以内完成,且检测结果肉眼可判。因此,本研究构建的VHSV快速、灵敏、便捷的诊断方法摆脱了对仪器设备和专业操作的依赖,可以应用于产业一线,对该病的防控具有重要价值与意义。
目的基因和靶器官的选择是病毒分子检测的首要环节。VHSV的基因型较多,不同基因型毒株在基因组序列上存在一定差异,且同基因型不同分离株仍存在序列突变差异[3]。通过对NCBI上现有VHSV毒株的N基因序列进行多序列对比,未能筛选到适配所有VHSV毒株的crRNA序列靶点。考虑到实验室保存的VHSV毒株为Ⅳa基因型,为亚洲地区的流行毒株。通过分析现有Ⅳa型毒株N基因序列的保守特征,可设计出适当简并度的引物应用于该基因型VHSV毒株的鉴定。VHSV基因组由6个基因构成,基因在转录时通过“停止-启动”机制顺序转录,显示N基因的mRNA水平最高[31],因此最适作为检测靶基因,可提升检测灵敏度。在选择取样靶器官时,我们充分考虑VHSV在感染动态及其在不同组织中的病毒载量水平,前期研究显示,VHSV感染后鱼体脾脏、鳃和肌肉组织比肝、肠道和胃组织可以更快检测出VHSV,且脾脏作为鱼体重要的免疫器官含有更高的病毒载量,因此选择脾脏、鳃和肌肉组织作为病原检测的靶器官,可在感染早期更快检测出病毒阳性[32]。
检测方法的灵敏度和特异性对于疾病诊断十分关键。目前已经建立多种针对VHSV的检测技术,其中PCR技术可检测出82 copies/μL的VHSV[33],RT-LAMP技术可检测出20 fg的VHSV[34]。本文通过RPA-CRISPR/Cas12a技术可以检测到9.0 copies/μL的VHSV(Ⅳa),相对于普通PCR检测极大地提升了检测的灵敏度。由于RPA-CRISPR/Cas12a技术检测灵敏度主要依赖于RPA的高灵敏扩增[35],所以相比基于RPA技术构建的VHSV检测方法具有相近的灵敏度[20]。同时,RPA-CRISPR/Cas12a技术通过RPA引物的互补配对和crRNA对扩增产物的特异性识别实现对检测过程的双重把关,使其检测特异性与准确性大大提高。另外,当仅有少量RPA扩增子产生时,其与Cas12a-crRNA复合物结合,仍可以激活Cas12a旁切活性切割报告分子,实现对RPA低量扩增结果的放大,使检测结果更加灵敏[36]。因此,相对于没有基因测序和探针识别的常规PCR、RPA和LAMP技术,RPA-CRISPR/Cas12a技术不仅可以有效地避免因引物非特异性扩增导致的假阳性,还可以保证在低扩增水平下的检测灵敏度与特异性[37]。
检测操作的简便性对检测方法在产业一线应用尤为重要,因为在养殖一线往往缺乏专业仪器设备,难以提供稳定的反应条件,且缺乏专业的检测人员,难以开展专业诊断。本研究构建的VHSV检测方法体系中,RT-RPA可以在30~42 ℃温度范围内进行扩增反应,反应温度跨度大,无需严格的温度调控,在条件不足时可以由体温加热实现核酸扩增。因此,RPA-CRISPR/Cas12a技术极大地降低了对大型仪器和操作专业性的依赖[38]。在检测结果判断时,RPA-CRISPR/Cas12a检测技术可通过试纸和荧光两种方式呈现,使检测结果肉眼可判。在检测时间方面,RPA-CRISPR/Cas12a技术从取样到结果判读仅需1 h左右,虽然其检测时长相对于免疫学检测试纸无明显优势[39],但RPA-CRISPR/Cas12a技术的检测灵敏度却远高于常规免疫学检测试纸,可以在疾病感染早期测出病原,从而达到及时预防和控制疾病发生的目的。另外,为了更加适应于现场使用,本研究将RNA快速提取试剂盒整合应用于RPA-CRISPR/Cas12a检测方法,临床样本检测结果显示,提取试剂盒的应用并未对检测结果造成明显负面影响,极大地提高了本检测方法的检测效率与便捷性。
4 结语本研究构建了针对Ⅳa基因型VHSV的RPA-CRISPR/Cas12a检测方法体系,该方法具有较高的检测灵敏度、特异性、准确性和稳定性。该方法可通过荧光显色和试纸条两种方式实现检测结果肉眼可判,极大地提升了VHSV检测的便捷性与实用性。综上,本文建立的VHSV检测方法,可以实现VHS早期现场诊断,对疾病的及时防控具有重要意义。
| [1] |
Crane M, Hyatt A. Viruses of fish: An overview of significant pathogens[J]. Viruses, 2011, 3(11): 2025-2046. DOI:10.3390/v3112025 (  0) 0) |
| [2] |
Dietzgen R G, Kondo H, Goodin M M, et al. The family Rhabdoviridae: mono- and bipartite negative-sense RNA viruses with diverse genome organization and common evolutionary origins[J]. Virus Research, 2017, 227: 158-170. DOI:10.1016/j.virusres.2016.10.010 (  0) 0) |
| [3] |
Pereiro P, Figueras A, Novoa B. Turbot (Scophthalmus maximus) vs. VHSV (viral hemorrhagic septicemia virus): A review[J]. Frontiers in Physiology, 2016, 7: 192. (  0) 0) |
| [4] |
Escobar L E, Escobar-Dodero J, Phelps N B D, et al. Infectious disease in fish: Global risk of viral hemorrhagic septicemia virus[J]. Reviews in Fish Biology and Fisheries, 2018, 28: 637-655. DOI:10.1007/s11160-018-9524-3 (  0) 0) |
| [5] |
Emmenegger E J, Moon C H, Hershberger P K, et al. Virulence of viral hemorrhagic septicemia virus (VHSV) genotypes Ⅰa, Ⅳa, Ⅳb, and Ⅳc in five fish species[J]. Diseases of Aquatic Organisms, 2013, 107(2): 99-111. DOI:10.3354/dao02671 (  0) 0) |
| [6] |
Vennerström P, Maunula L, Välimäki E, et al. Presence of viral haemorrhagic septicaemia virus (VHSV) in the environment of virus-contaminated fish farms and processing plants[J]. Diseases of Aquatic Organisms, 2020, 138: 145-154. DOI:10.3354/dao03454 (  0) 0) |
| [7] |
Escobar L E, Kurath G, Escobar-Dodero J, et al. Potential distribution of the viral haemorrhagic septicaemia virus in the Great Lakes region[J]. Journal of Fish Diseases, 2017, 40: 11-28. DOI:10.1111/jfd.12490 (  0) 0) |
| [8] |
Panzarin V, Cuenca A, Gastaldelli M, et al. VHSV single amino acid polymorphisms (SAPs) associated with virulence in rainbow trout[J]. Frontiers in Microbiology, 2020, 11: 1984. DOI:10.3389/fmicb.2020.01984 (  0) 0) |
| [9] |
Cho M Y, Lee U H, Moon C H, et al. Genetically similar VHSV isolates are differentially virulent in olive flounder Paralichthys olivaceus[J]. Diseases of Aquatic Organisms, 2012, 101(2): 105-114. DOI:10.3354/dao02503 (  0) 0) |
| [10] |
Kim J O, Kim W S, Kim S W, et al. Development and application of quantitative detection method for viral hemorrhagic septicemia virus (VHSV) genogroup Ⅳa[J]. Viruses, 2014, 6(5): 2204-2213. DOI:10.3390/v6052204 (  0) 0) |
| [11] |
Kim W S, Jung S J, Kim J O, et al. Genetic positioning of Korean viral hemorrhagic septicemia virus (VHSV) from cultured and wild marine fishes[J]. Journal of Fish Pathology, 2011, 24(1): 1-9. DOI:10.7847/jfp.2011.24.1.001 (  0) 0) |
| [12] |
Zhu R L, Zhang Q Y. Determination and analysis of the complete genome sequence of Paralichthys olivaceus rhabdovirus (porv)[J]. Archives of Virology, 2014, 159(4): 817-820. DOI:10.1007/s00705-013-1716-5 (  0) 0) |
| [13] |
Byon J Y, Takano T, Hirono I, et al. Genome analysis of viral hemorrhagic septicemia virus isolated from Japanese flounder Paralichthys olivaceus in Japan[J]. Fisheries Science, 2006, 72(4): 906-908. DOI:10.1111/j.1444-2906.2006.01235.x (  0) 0) |
| [14] |
Kim W S, Kim S R, Kim D, et al. An outbreak of VHSV (viral hemorrhagic septicemia virus) infection in farmed olive flounder Paralichthys olivaceus in Korea[J]. Aquaculture, 2009, 296(1-2): 165-168. DOI:10.1016/j.aquaculture.2009.07.019 (  0) 0) |
| [15] |
Kim S Y, Lee K M, Kim K H. Differences between DNA vaccine and single-cycle viral vaccine in the ability of cross-protection against viral hemorrhagic septicemia virus (VHSV) and infectious hematopoietic necrosis virus (IHNV)[J]. Vaccine, 2023, 41(38): 5580-5586. DOI:10.1016/j.vaccine.2023.07.058 (  0) 0) |
| [16] |
Kole S, Qadiri S S N, Shin S M, et al. Nanoencapsulation of inactivated-viral vaccine using chitosan nanoparticles: Evaluation of its protective efficacy and immune modulatory effects in olive flounder (Paralichthys olivaceus) against viral haemorrhagic septicaemia virus (VHSV) infection[J]. Fish and Shellfish Immunology, 2019, 91: 136-147. DOI:10.1016/j.fsi.2019.05.017 (  0) 0) |
| [17] |
Kim M S, Kim D S, Kim K H. Oral immunization of olive flounder (Paralichthys olivaceus) with recombinant live viral hemorrhagic septicemia virus (VHSV) induces protection against VHSV infection[J]. Fish and Shellfish Immunology, 2011, 31(2): 212-216. DOI:10.1016/j.fsi.2011.05.003 (  0) 0) |
| [18] |
World Organisation for Animal Health. Infection with viral haemorrhagic septicaemia viru[EB/OL]. (2021-05-01)[2024-04-05]. https://www.woah.org/fileadmin/Home/eng/Health_standards/aahm/current/2.3.10_VHS.pdf.
(  0) 0) |
| [19] |
Kim H J, Cuenca A, Olesen N J. Validation of a novel one-step reverse transcription polymerase chainreaction method for detecting viral haemorrhagic septicaemia virus[J]. Aquaculture, 2018, 492: 170-183. DOI:10.1016/j.aquaculture.2018.03.047 (  0) 0) |
| [20] |
Yin W, Sun T, Huang W, et al. Establishment of recombinase polymerase amplification technology for detecting viral hemorrhagic septicemia virus (VHSV) in cultured aquatic animals[J]. Aquaculture, 2021, 535: 736338. DOI:10.1016/j.aquaculture.2020.736338 (  0) 0) |
| [21] |
Al-Hussinee L, Lumsden J S. Detection of VHSV Ⅳb within the gonads of Great Lakes fish using in situ hybridization[J]. Diseases of Aquatic Organisms, 2011, 95(1): 81-86. DOI:10.3354/dao02338 (  0) 0) |
| [22] |
López-Vázquez C, Bandín I, Dopazo C P. Design and evaluation of a macroarray for detection, identification, and typing of viral hemorrhagic septicemia virus (VHSV)[J]. Animals, 2021, 11(3): 841. DOI:10.3390/ani11030841 (  0) 0) |
| [23] |
Kong K H, Jeong H N, Shyam K U, et al. Development and validation of a lateral flow immunochromatographic assay for specific detection of viral hemorrhagic septicemia virus (VHSV, genotype Ⅳa) in olive flounder (Paralichthys olivaceus)[J]. Aquaculture, 2021, 537: 736491. DOI:10.1016/j.aquaculture.2021.736491 (  0) 0) |
| [24] |
Li S Y, Cheng Q X, Liu J K, et al. CRISPR-Cas12a has both cis- and trans-cleavage activities on single-stranded DNA[J]. Cell Research, 2018, 28(4): 491-493. DOI:10.1038/s41422-018-0022-x (  0) 0) |
| [25] |
Li J, Macdonald J, Von Stetten F. A comprehensive summary of a decade development of the recombinase polymerase amplification[J]. Analyst, 2019, 144(1): 31-67. DOI:10.1039/C8AN01621F (  0) 0) |
| [26] |
Lee R A, Puig H, Nguyen P Q, et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(41): 25722-25731. (  0) 0) |
| [27] |
Sukonta T, Senapin S, Meemetta W, et al. CRISPR-based platform for rapid, sensitive and field-deployable detection of scale drop disease virus in Asian sea bass (Lates calcarifer)[J]. Journal of Fish Diseases, 2022, 45(1): 107-120. DOI:10.1111/jfd.13541 (  0) 0) |
| [28] |
Rong F, Wang H, Tang X, et al. The development of RT-RPA and CRISPR-Cas12a based assay for sensitive detection of infectious hematopoietic necrosis virus (IHNV)[J]. Journal of Virological Methods, 2024, 326: 114892. DOI:10.1016/j.jviromet.2024.114892 (  0) 0) |
| [29] |
Sukonta T, Senapin S, Taengphu S, et al. An RT-RPA-Cas12a platform for rapid and sensitive detection of tilapia lake virus[J]. Aquaculture, 2022, 560: 738538. DOI:10.1016/j.aquaculture.2022.738538 (  0) 0) |
| [30] |
Sharifnia Z, Kazemi B. Diagnosis of viral haemorrhagic septicaemia (VHS) in Iranian rainbow trout aquaculture by pathology and molecular techniques[J]. Bulletin of the European Association of Fish Pathologists, 2008, 28(5): 170. (  0) 0) |
| [31] |
Banerjee A K, Barik S. Gene expression of vesicular stomatitis virus genome RNA[J]. Virology, 1992, 188(2): 417-428. DOI:10.1016/0042-6822(92)90495-B (  0) 0) |
| [32] |
Kim H, Baek K W, Ko E J, et al. Genome based quantification of VHSV in multiple organs of infected olive flounder (Paralichthys olivaceus) using real-time PCR[J]. Genes and Genomics, 2020, 42(7): 773-777. DOI:10.1007/s13258-020-00951-7 (  0) 0) |
| [33] |
Weili Y, Yao L, Wei H, et al. Establishment of pyrosequencing technology for detecting viral hemorrhagic septicemia virus (VHSV)[J]. Animal Husbandry and Feed Science, 2017, 9(6): 4. DOI:10.3969/j.issn.1672-5190.2017.06.003 (  0) 0) |
| [34] |
安元龙, 吴斌, 林长军, 等. 荧光环介导逆转录等温扩增(RT-LAMP)技术在病毒性出血性败血症(VHS)诊断中的应用[J]. 中国动物检疫, 2012, 29(12): 23-38. An Y L, Wu B, Lin C J, et al. Application of fluorescent real-time reverse transcription loop-mediated isothermal amplification for detection of haemorrhagic septicaemia[J]. China Animal Health Inspection, 2012, 29(12): 23-38. DOI:10.3969/j.issn.1005-944X.2012.12.015 (  0) 0) |
| [35] |
Tang G, Zhang Z, Tan W, et al. RT-RPA-Cas12a-based assay facilitates the discrimination of SARS-CoV-2 variants of concern[J]. Sensors and Actuators B: Chemical, 2023, 381: 133433. DOI:10.1016/j.snb.2023.133433 (  0) 0) |
| [36] |
Xiong D, Dai W, Gong J, et al. Rapid detection of SARS-CoV-2 with CRISPR-Cas12a[J]. PLoS Biology, 2020, 18(12): e3000978. DOI:10.1371/journal.pbio.3000978 (  0) 0) |
| [37] |
Li S Y, Cheng Q X, Wang J M, et al. CRISPR-Cas12a-assisted nucleic acid detection[J]. Cell Discovery, 2018, 4: 20. (  0) 0) |
| [38] |
Hu J J, Liu D, Cai M Z, et al. One-pot assay for rapid detection of benzimidazole resistance in Venturia carpophila by combining RPA and CRISPR/Cas12a[J]. Journal of Agricultural and Food Chemistry, 2023, 71(3): 1381-1390. DOI:10.1021/acs.jafc.2c06549 (  0) 0) |
| [39] |
孙涛, 高瑞刚, 孙明君, 等. 鱼类病毒性出血性败血症病毒诊断胶体金免疫层析方法的建立[J]. 中国动物检疫, 2017, 34(12): 6. Sun T, Gao R G, Sun M J, et al. Development of a colloidal gold immunochromatographic assay for antigen detection of viral hemorrhagic septicemia virus[J]. China Animal Health Inspection, 2017, 34(12): 6. (  0) 0) |
2. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao Marine Science and Technology Center, Qingdao 266237, China
 2025, Vol. 55
2025, Vol. 55


