2. 青岛海洋科技中心海洋生态与环境科学功能实验室,山东 青岛 266237
抗生素是预防和治疗疾病感染的重要抗菌药物,被誉为20世纪现代医学的最伟大发现,为保障人类健康和畜牧业发展做出了巨大贡献[1]。然而,近年来抗生素滥用导致了抗生素抗性细菌(antibiotic resistant bacteria, ARB)和抗生素抗性基因(antibiotic resistance genes, ARGs)在各种环境介质中的存留与传播,对临床治疗造成了严重挑战[2]。如果不采取有效措施,预计到2050年,每年将有1 000万人死于细菌耐药污染[3]。目前,ARGs污染已被联合国环境规划署列为全球新兴六大环境问题之一[4],引发了更广泛关注。
大气颗粒物(particulate matter, PM)是环境空气中各种固态和液态颗粒状物质的总称[5],由于能够提供大量的附着位点而成为ARGs和ARB迁移扩散的重要载体[6]。在全球不同地区的大气颗粒物中已检测到丰富的ARGs,如Gao等[7]在横跨4个大洲的392个大气颗粒物宏基因组样本中发现了1 893种ARGs,Li等[8]在19个国家和地区的城市大气颗粒物样本中检出7种抗生素的30余种ARGs亚型,Zhao等[9]在大学校园的大气颗粒物中检测到总丰度高达106 copies/m3的46种ARGs。相较于水和土壤等环境介质,赋存于大气颗粒物中的ARGs极易通过呼吸作用直接进入人体,具有更大的人体健康潜在危害性。
ARGs不仅可以通过垂直转移在亲代与子代之间传递,还能借助可移动遗传元件(mobile genetic elements, MGEs)通过水平转移在不同菌株间传递。ARGs所具有的自我复制能力和扩散传播能力以及在日常环境中的广泛分布,使其比传统的化学污染物更难以控制[12]。因此,不仅要从源头上减少抗生素使用,研究ARGs的去除手段也是至关重要的。不同于对水和土壤等环境介质中ARGs的控制,去除大气颗粒物中ARGs的有效方法仍然缺乏[13]。紫外消毒作为常见的杀菌手段,能在短时间内破坏微生物的遗传物质使其死亡,具有高效、简便、无二次污染等优点,已被广泛应用于环境空气中细菌的清除[14]。在紫外线中,254 nm的短波紫外线对DNA的破坏能力最强,杀菌效果最好[15]。相关研究表明,紫外消毒在杀灭水体中细菌的同时也能一定程度上削减ARGs丰度[16-17],但目前尚不了解紫外消毒对大气颗粒物中ARGs的去除效果。因此,本研究旨在探究紫外消毒对大气颗粒物中ARGs的去除作用,为控制大气环境中的细菌耐药传播提供新思路。
本研究对采集的大气颗粒物样品进行了紫外消毒实验,基于培养法检测了消毒前后可培养细菌的数量,采用qPCR技术定量评估了10种典型ARGs、2种MGEs和16S rRNA的丰度变化,以探究在常规杀菌剂量下,紫外消毒对大气颗粒物中细菌和ARGs的去除率。此外,还分析了ARGs及MGEs之间的相关关系,并探究了紫外消毒前后ARGs的组成变化及潜在转移风险,以综合评估紫外消毒控制大气颗粒物中ARGs污染的可行性。
1 方法 1.1 样品采集与预处理2022年11月—2023年9月,使用2031型大流量环境空气颗粒物采样器(青岛崂应环境科技有限公司)在青岛市崂山实验室东区(36.333°N,120.722°E)办公楼顶采得大气总悬浮颗粒物(total suspended particulate matter, TSP)样品24套。每个TSP样品以1.05 m3/min流量采集约24 h,采样膜为Whatman石英滤膜((8×10) inch)。采样前将石英滤膜于450 ℃马弗炉灼烧5 h,采样时用75%酒精擦拭采样器、滤料夹和镊子进行灭菌。每套样品采集后,随即转移至超净台内将其均匀裁剪为12等份,并均分为4组(每组约103.5 cm2),再将各组样品分别放于无菌培养皿中进行后续实验。
1.2 紫外消毒实验参照文献[18-19],自主搭建了一个实验室规模的紫外消毒装置,并将其固定于恒温恒湿培养箱内部。该装置主要由4个功率相同、主波长为254 nm的低压紫外灯管组成,为使其功率保持恒定,实验前先将紫外灯预热20 min, 将放有大气颗粒物样品的培养皿置于平行光束中心,并使用紫外线辐射强度测量仪(林上科技有限公司)测定辐照强度。根据《消毒技术规范》(2002年版)[20]中的相关规定,紫外消毒实验在避光条件下进行,设置环境温度为(25±2) ℃,相对湿度为70%。通过调整辐照时间设定辐照剂量(0、30、60和120 mJ/cm2)。由于缺乏环境空气消毒的剂量标准,本研究中辐照剂量设置参考了中国水处理领域对ARGs的消毒标准[21]。紫外照射后的样品用于下一步的细菌培养与DNA提取。
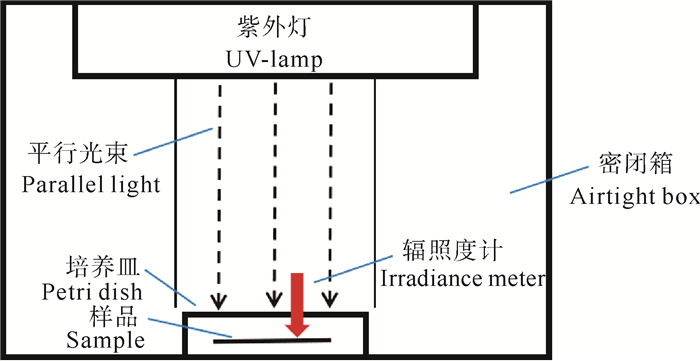
|
图 1 紫外消毒装置图 Fig. 1 Ultraviolet disinfection device diagram |
于超净台内裁取约18 cm2紫外处理后的样品膜,加入20 mL 1×PBS缓冲液,37 ℃恒温振荡2 h后短暂涡旋,取100 μL菌悬液用无菌涂布棒均匀涂抹于LB固体培养基表面,每组三个平行。将涂布好的细菌平板于37 ℃恒温培养箱中倒置培养24 h后进行菌落计数,并以每立方米菌落形成单位(CFU/m3)计算,用空气采样体积归一化处理[22-23]。
| $ C=\frac{a \times v_2}{v_1 \times V} \text { 。} $ | (1) |
式中:C为可培养细菌数量(CFU/m3);a为菌落数(CFU/皿);v1为涂布菌液体积(mL);v2为菌悬液总体积(mL);V为每次采样时记录的采样体积(m3)。
1.4 DNA提取在超净台内将上一步剩余样品(约85.5 cm2)卷起放入50 mL无菌无酶离心管中,加入50 mL 1×PBS缓冲液,4 ℃ 300g离心3 h,经0.2 μm PES滤膜过滤,采用Powersoil土壤DNA提取试剂盒(QIAGEN, Germany)和AMpure XP磁珠(Beckman, USA)提取过滤得到颗粒物中的DNA[24]。使用琼脂糖凝胶电泳仪(Bio-rad, USA)和超微量紫外分光光度计(NanoDrop, USA)对DNA的纯度及浓度进行检测,将合格的样品置于-20 ℃避光保存。
1.5 实时荧光定量PCR采用PCR仪对选定的5类10种ARGs(sul1、sul2、sul3、tetA、tetL、tetW、ermB、ermC、strB、blaTEM-1)、2种MGEs(intI1、tnpA2)和16S rRNA基因进行定性检测。将PCR产物克隆到PMD19-T质粒中,并通过DH5α大肠杆菌扩大培养,提取其中的质粒进行测序。将包含目的基因的质粒作为qPCR标准品并进行梯度稀释,以建立标准曲线。采用qPCR仪(Bio-rad CFX96, USA)进行实时荧光定量检测,总反应系统包括10 μL FastStart SYBR Greeen Master (Rox), 0.6 μL Primer-F(10 mmol/L), 0.6 μL Primer-R(10 mmol/L), 1 μL DNA和7.8 μL ddH2O。反应程序为:95 ℃预变性5 min,然后进入40个循环的扩增过程(94 ℃变性30 s,退火30 s,72 ℃延伸30 s),最后再72 ℃延伸7 min,熔解曲线按仪器原始设定进行。各目的基因引物序列、扩增片段长度及退火温度见表 1。
|
|
表 1 目的基因的引物信息 Table 1 Primer information of target genes |
采用SPSS 26.0软件对实验结果进行统计学分析(以95%作为置信区间),使用graphpad prism 9.5软件的单因素方差分析(one way ANOVA)比较不同处理组样品间的差异,采用ORIGIN 2022软件绘图。
2 结果与讨论 2.1 紫外消毒对细菌的去除效果实验结果表明,紫外消毒能有效削减大气颗粒物中细菌的数量,且消毒效果随紫外照射剂量的增加而增强。如图 2(a)所示,紫外消毒前大气颗粒物样品中的可培养细菌数量高达(1 056±448) CFU/m3,经过30和60 mJ/cm2的紫外照射后,其去除率分别为58.86%和89.4%,而当紫外剂量达到120 mJ/cm2时,99%以上的可培养细菌都已去除(见图 2(c))。随紫外照射剂量的增加,可培养细菌的灭活率得到显著提升(p<0.01),在120 mJ/cm2紫外剂量下的对数灭活率达到2个对数级。由于大气颗粒物中仍存在大量不可培养的细菌,而16S rRNA的丰度通常用于表征样品中的总细菌量,因此本研究结合分子生物学技术,定量评估了16S rRNA的丰度,以进一步分析紫外消毒对大气颗粒物中细菌数量的影响。如图 2(b)所示,未经紫外照射时,大气颗粒物样品中16S rRNA的平均丰度高达3.63×105 copies/m3,经低剂量(30 mJ/cm2)紫外消毒后仍有大量细菌存在,16S rRNA的丰度高于1.21×105 copies/m3。而在中等剂量(60 mJ/cm2)和高剂量(120 mJ/cm2)的紫外照射后,16S rRNA的丰度分别下降至6.92×104和2.82×104 copies/m3。值得注意的是,120 mJ/cm2的紫外剂量下,16S rRNA的丰度下降1.09个对数级,去除率达到91.6%,这表明紫外消毒能同时削减大气颗粒物中可培养和不可培养细菌的数量,从而降低总细菌的含量。
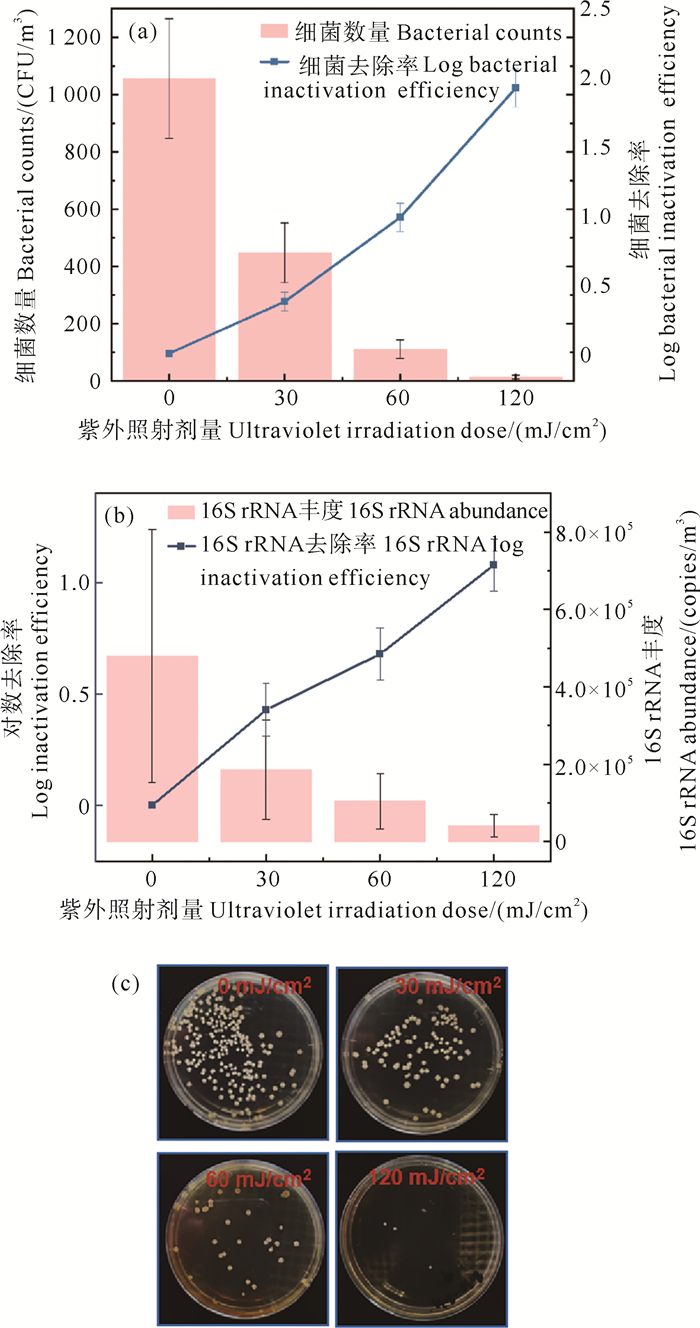
|
图 2 (a) 不同紫外剂量下的可培养细菌数量及去除率、(b)不同紫外剂量下的16S rRNA丰度及去除率及(c)不同紫外剂量下的细菌平板培养图 Fig. 2 (a) Bacterial counts and inactivation efficiency at different UV doses, (b) abundance of 16S rRNA and inactivation efficiency at different UV doses and (c) plates of bacteria at different UV doses |
此前也有研究报道紫外消毒对空气中常见致病菌的杀灭效果,如Yin等[25]发现75 mJ/cm2的紫外剂量可使O157∶H7型大肠杆菌数量降低1.95个对数级,而这一致病菌曾在1982年导致中国超2万人死于感染性腹泻[26];Singh等[27]发现21.6 mJ/cm2的紫外剂量能够杀灭2个对数级的沙门氏菌,极大降低了霍乱等疾病的传染风险。由此可见,紫外消毒能有效降低空气中细菌的人类暴露风险,从而有助于遏制ARGs的传播风险,这是因为ARGs在不同菌群间主要通过接合、转导、转化这三种途径传播扩散,其中,接合转移需要在活的供体菌和受体菌中进行[28]。Guo等[29]的实验也发现,10 mJ/cm2的紫外照射能显著降低水体中存活细菌间ARGs水平转移的频率。因此,紫外消毒有望通过杀灭细菌而降低ARGs在环境中的传播风险。
2.2 紫外消毒对大气颗粒物中ARGs和MGEs的去除作用紫外消毒对细菌及其DNA的强大破坏作用意味着在一定程度上能够去除ARGs和MGEs。然而,胞内物质的遮挡可能会降低ARGs暴露于紫外光的几率,从而降低其去除效率[18]。尽管高剂量(120 mJ/cm2)的紫外消毒能有效去除细菌,但仍可能有部分胞内ARGs存留下来,并随细胞膜通透性增大或细胞死亡降解而进入环境中,产生新的威胁[30]。因此,本研究通过qPCR技术定量评估了不同紫外照射剂量下10种ARGs和2种MGEs的丰度变化。
未经紫外消毒时,大气颗粒物样品中存在丰富的ARGs和MGEs,总ARGs的平均丰度高达2.12×104 copies/m3,两种MGEs的平均丰度分别为1.74×103 copies/m3(intI1)和7.85×102 copies/m3(tnpA2)。10种ARGs中,tetL的平均丰度最高,达到3.73×103 copies/m3,其次为strB和blaTEM-1,平均丰度分别为2.28×103和3.13×103 copies/m3。此外,sul1、tetW和ermC的丰度也均超过了103 copies/m3,仅sul3的丰度在102 copies/m3以下。图 3显示了样品中各ARGs和MGEs在不同紫外照射剂量下的绝对丰度变化。可以看出,在低剂量(30 mJ/cm2)的紫外照射下,总ARGs去除率为47.7%,10种ARGs的绝对丰度降低了0.09~0.30个对数级。然而,经高剂量(120 mJ/cm2)的紫外照射后,总ARGs去除率达到65.4%,10种ARGs的绝对丰度降低了0.36~0.69个对数级,但tetL、ermB和blaTEM-1的绝对丰度仍在103 copies/m3以上。其中,sul1的去除率最高,达到79.61%;其次为sul2,去除率为77.17%,最低的是tetW,其去除率只有55.50%。此外,2种MGEs在紫外消毒后也出现了不同程度的减少,在30~120 mJ/cm2的紫外照射下,intI1和tnpA2的丰度分别降低了0.21~0.71和0.24~0.80个对数级,且在120 mJ/cm2紫外消毒后的丰度均下降至102 copies/m3。Liu等[31]曾对实验室制备的模拟气溶胶进行紫外消毒,发现在135.3 mJ/cm2的照射剂量下,16S rRNA和sul2的绝对丰度分别下降了1.41和0.91个对数级,相比本研究有更高的去除率。这可能是由于实际大气颗粒物中的复杂组分干扰及大颗粒对紫外光线的遮挡导致的,这些作用降低了紫外消毒对真实大气颗粒物的净化效果。
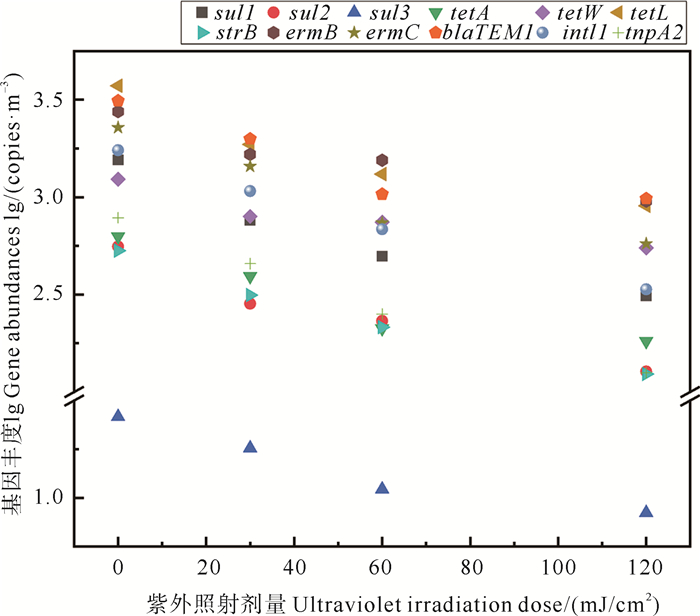
|
图 3 不同紫外剂量下ARGs、MGEs的绝对丰度 Fig. 3 Absolute abundance of ARGs, MGEs at different UV doses |
总体而言,随着紫外照射剂量的增加,ARGs的绝对丰度呈现下降趋势。然而,在同一紫外剂量下,ARGs的削减效果远低于细菌,总ARGs与可培养细菌的去除率相差33.6%。需要强调的是,去除不同种类的ARGs亚型往往需要不同的紫外剂量。例如,尽管同属于磺胺类抗性基因,sul1、sul2、sul3在120 mJ/cm2紫外剂量下的绝对丰度分别降低了0.69、0.64和0.49个对数级,可见,相比于sul1和sul2,sul3抵抗紫外线和光化学损伤的能力更强。此外,ARGs在大气颗粒物中的初始丰度可能不会影响紫外去除效果。例如,未消毒时tetW比sul3的丰度高两个数量级,但后者的对数去除率反而比前者高0.14个对数级,这可能是因为DNA的结构和碱基排列是决定紫外线损伤程度的主要因素。由于较长的基因片段通常存在更多的二聚体形成位点(CC, TT, CT, TC),因而更容易受到光化学损伤[18],并且不同种类ARGs的去除效率与其DNA片段上潜在胸腺嘧啶二聚体形成位点的数量也密切相关[15]。
紫外线作为一种选择压力可能会改变ARGs耐药菌在大气颗粒物中的占比。ARGs的富集程度通常利用其相对于16S rRNA的丰度加以表征[32]。如图 4所示,ARGs的相对丰度随紫外照射剂量的增大反而增加。对比图 3和4可以发现,10种ARGs的绝对丰度在120 mJ/cm2的紫外剂量下均有所下降,但相对丰度却增加了0.39~0.74个对数级, 其中,sul3的相对丰度增加最高(0.74个对数级),在总ARGs中的占比上升了4%。此外,对于MGEs,intI1和tnpA2的相对丰度也分别增加了0.37和0.29个对数级。整体而言,经过高剂量(120 mJ/cm2)的紫外照射后,ARGs总绝对丰度下降了0.54个对数级,但总相对丰度增加了0.92个对数级。这意味着,一方面,携带ARGs的细菌可能对紫外线具有更高的耐受能力,紫外去除过程中,这些具有高耐受能力的耐药菌将会存留下来,进一步危害人类健康;另一方面,一些细菌在紫外消毒后死亡,但其DNA可能并未被完全破坏,一部分存留于质粒等载体上的ARGs被保留下来,将会随细胞的死亡流出胞外,造成潜在转移风险。同时,在紫外消毒的选择压力下,存活的细菌可能会摄取游离在环境中的ARGs,导致耐药菌的富集。也有研究发现, 紫外消毒会导致多重抗性基因(如mexB)富集,并使水体中ARGs总相对丰度显著升高[33],这意味着紫外消毒的筛选压力下,耐药性比例可能会放大,更需要关注其在环境中的转移风险。总体而言,尽管消毒后残存细菌中耐药菌的占比略有增加,但总ARGs丰度明显下降,因此紫外消毒对大气颗粒物中的ARGs具有一定的去除作用。
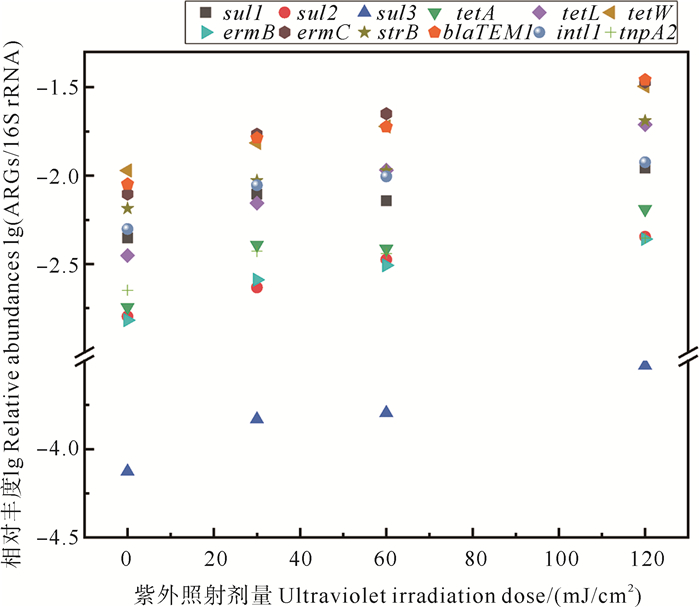
|
图 4 不同紫外剂量下ARGs、MGEs的相对丰度 Fig. 4 Relative abundance of ARGs, MGEs at different UV doses |
紫外消毒对不同类型ARGs的去除效率存在一定差异,因此很可能导致环境中抗生素抗性组的变化,即在一定程度上改变ARGs的组成结构和主导类型。为探究不同紫外照射剂量下大气颗粒物中ARGs的组成差异,本研究利用主成分分析(PCA)对数据进行降维(见图 5)。整体来看,对照组与处理组的样品之间存在一定的重叠区域,说明ARGs的整体组成具有相似性。然而,对照组(0 mJ/cm2)的大部分样品同三个处理组有明显区分,但三个处理组(30、60和120 mJ/cm2)之间的差异则很小。由此可见,紫外消毒很可能作为一种选择压力影响大气颗粒物中ARGs的组成结构,使一些对紫外线耐受能力强的ARGs成为主导亚型,在环境中占据优势,并且,这种选择机制主要受到不同种类ARGs紫外耐受性的影响。此前也有研究发现,紫外消毒导致废水中气单胞菌和盐单胞菌等优势属几乎完全消失,而假单胞菌和芽孢杆菌等一些次要属却成为主导类型[34]。同时,菌落结构的变化通常会影响ARGs的组成,该研究进一步发现四环素类抗性基因多存在于一些不耐受紫外照射的种属中,所以紫外消毒后四环素耐药菌在环境中的占比下降[34]。
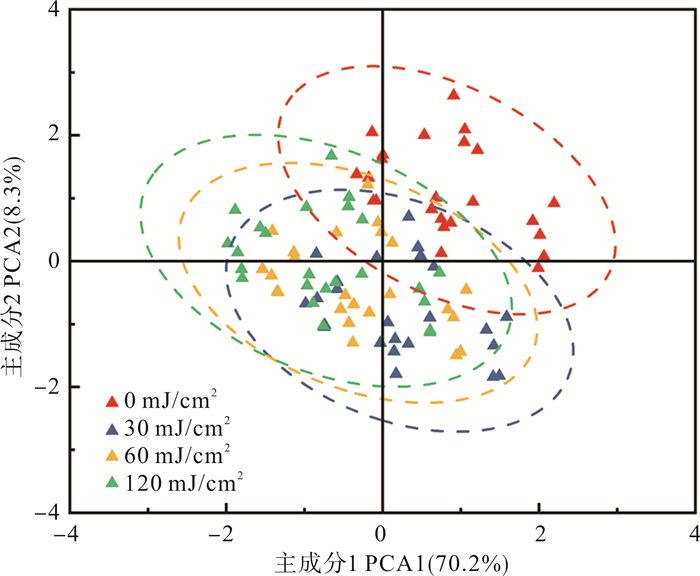
|
( 每个椭圆中是相关性较强的样品。In each ellipse are the more highly correlated samples. ) 图 5 不同紫外照射剂量组大气颗粒物ARGs丰度的PCA分析 Fig. 5 PCA analysis of the abundance of atmospheric particulate ARGs in different UV irradiation dose groups |
除此之外,本研究还对三种紫外剂量下不同类型ARGs的去除率分别进行了单因素方差分析(one way ANOVA)。结果表明,即便在同一剂量的紫外照射下,针对不同抗生素的抗性基因,或是针对同一抗生素的不同抗性基因亚型,其去除效果也存在一定差异,并且这种差异随紫外剂量的增加而更加显著。具体来看,30 mJ/cm2的紫外剂量下,sul1、sul2、sul3、tetA之间均存在显著性差异(p<0.05)(见图 6(a)),而在60 mJ/cm2的紫外剂量下,ARGs去除率梯度趋势发生改变,如sul1、sul2、tetA和ermC之间均存在显著性差异(p<0.05)(见图 6(b))。当紫外剂量持续加大到120 mJ/cm2时,ARGs的去除率更加分散化,呈现更为复杂的梯度趋势,除sul2、tetL、ermB和strB之间,以及tetA、sul3、ermC和blaTEM-1之间无明显差异外,其余各ARGs的去除率互相之间均存在显著性差异(p<0.05)(见图 6(c))。随着紫外照射剂量的增大,ARGs的去除效率很可能在更大程度上受到其种类的影响,因而一些对紫外线抵抗力强的ARGs及其宿主菌将会在环境中占据优势。因此,对更多种类型ARGs的紫外去除率进行定量评估非常必要,并建立相关的动力学模型,以更好地评价其去除所需要达到的紫外消毒剂量。
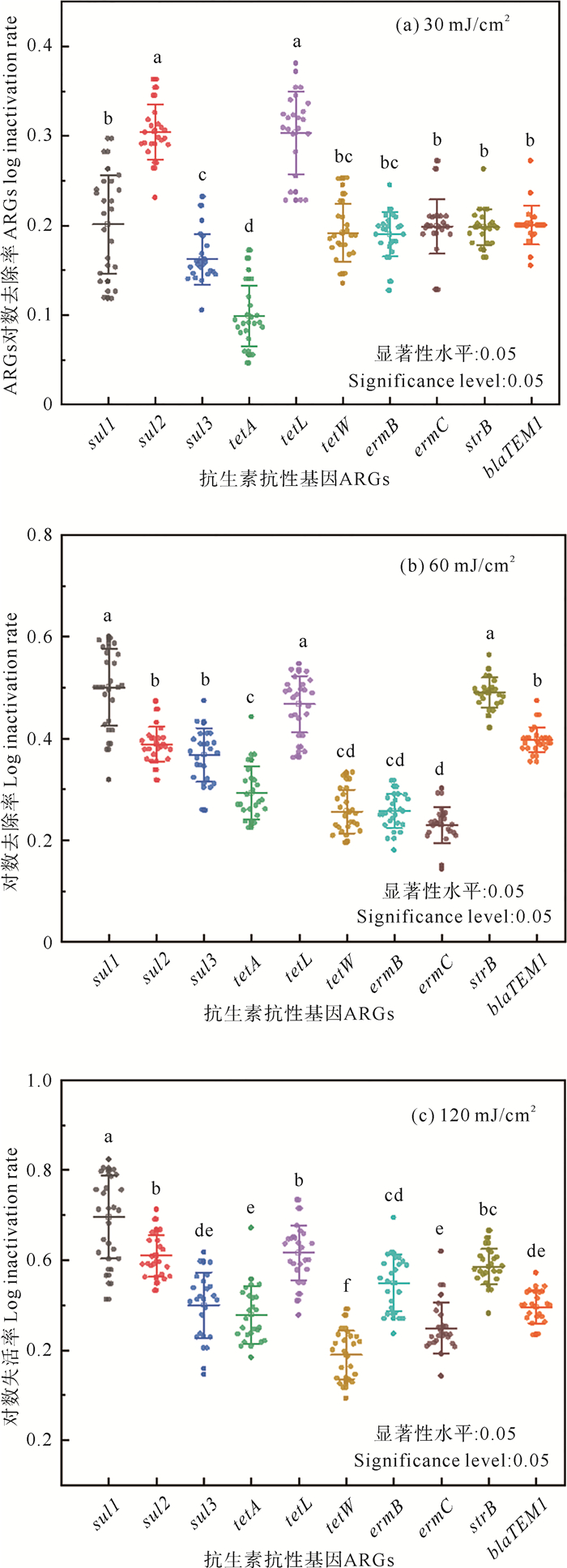
|
( 图中不同的字母代表不同的组之间存在显著性差异,含有相同字母的组之间不存在显著性差异。Different letters in the graph represent significant differences between different groups. There are no significant differences between groups containing the same letter. ) 图 6 不同类型ARGs在同一紫外照射剂量下的显著性差异 Fig. 6 Significant differences between different types of ARGs at the same UV irradiation dose |
研究表明,环境介质中的ARGs和MGEs普遍存在共现关系,这对于ARGs的传播扩散具有重要意义[35]。为进一步探究紫外消毒过程中不同类型ARGs及其与MGEs间的相关性,本研究基于去除率对各ARGs及MGEs进行了Spearman相关性分析(见图 7)。
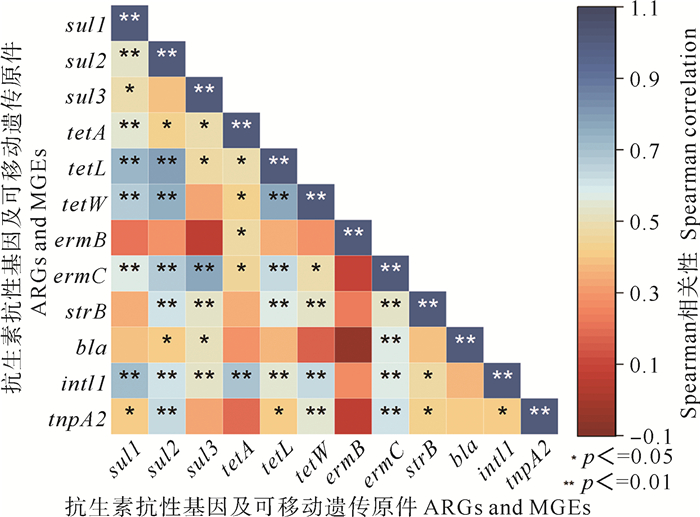
|
( *表示存在相对强的显著相关, 0.01≤p<0.05, **表示存在强显著相关, p<0.01。*Indicated relatively strong correlation, 0.01≤p < 0.05, **Indicated strong correlation, p < 0.01. ) 图 7 紫外消毒过程中不同ARGs及MGEs之间的Spearman相关性 Fig. 7 Spearman′s correlation between different ARGs and MGEs during UV sterilization process |
根据相关性可以将10种ARGs分为三组(每组至少两个,且满足p<0.05、r>0.50)。第一组:sul1、sul2、tetW和tetL;第二组:sul3、strB和ermC;第三组:tetA和blaTEM-1。可以发现,每组内的ARGs存在较强关联,可能在紫外消毒过程中有着相似的行为特性,这些ARGs有一定概率存在于相同的基因组或质粒上,因而具有相近的去除效率。例如,Wang等[36]在施用粪肥土壤中发现,磺胺类抗性基因sul1和sul2通常共存于同一基因组或质粒上,但并未发现sul1和sul3的类似组合。还有研究发现tetA和blaTEM-1共存于同一个IncFIB质粒上[37],而氨基糖苷类和四环素类抗性基因常同时存在于ESBL抗性菌内[38]。这些ARGs的共存无疑会促进多重耐药菌的产生,并很可能在环境中不断积累和扩散,因此有必要进一步关注紫外消毒后ARGs的潜在转移风险。
对于MGEs,intI1的去除率与第一组内的ARGs均存在较强相关性(p<0.05、r>0.50),说明intI1很可能是这些ARGs在环境中传播的载体。作为最常见的MGEs,intI1在多种临床耐药菌中广泛存在,尤其是与人类肠道感染密切相关的革兰氏阴性菌,并且这些革兰氏阴性菌对紫外线的抵抗力比革兰氏阳性菌弱[33]。随紫外照射剂量的增大,intI1和tnpA2的丰度均呈现递减规律(见图 3),在高剂量(120 mJ/cm2)紫外照射下的对数去除率分别为0.71和0.80个对数级,尤其是intI1,其平均丰度从1.74×103 copies/m3下降至3.36×102 copies/m3,大大降低了介导ARGs转移的可能性。此前也有研究分析了紫外消毒对废水中ARGs及其载体的去除效果,发现即使1 mJ/cm2的紫外照射也能直接损坏携带ARGs的质粒,从而极大程度地抑制ARGs在不同菌群间的水平转移[39]。
此外,本研究发现,紫外消毒过程中,总抗性基因丰度与intI1和tnpA2均显著相关(p<0.01)(见图 8),相关系数R2分别为0.99和0.95。同时,sul1、tetL与intI1之间显著相关(p<0.01),表明这两种ARGs很可能同时位于intI1的基因盒中;而sul2、tetW与tnpA2之间显著相关(p<0.05),说明二者可能共存于同一转座子上,因此在紫外线的筛选压力下出现了共选择机制。这与前文的分组结果相吻合,也解释了不同种类抗生素的抗性基因在紫外消毒的选择压力下为何会有相似的去除特性。
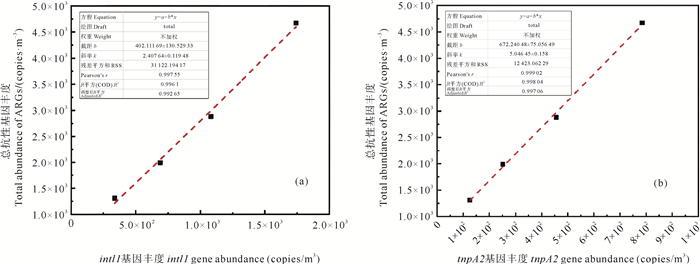
|
( 红色虚线为线性拟合,R2为拟合优度。The red dotted line is a normal fitting line. R2 is the goodness of fit. ) 图 8 总抗性基因丰度与(a)intI1和(b)tnpA2间的相关性 Fig. 8 Correlation between total resistance gene abundance and number of (a) intI1 and (b) tnpA2 |
由此可见,紫外消毒对MGEs的削减将有助于降低ARGs在环境中的潜在转移风险,这一点也在其他领域的研究中得到证实[40]。Zhu等[41]曾采用结构模型评估高温堆肥去除ARGs过程的多重驱动因素,发现MGEs的减少通常是决定环境中ARGs去除作用的关键所在。因此,紫外消毒能够在一定程度上控制大气颗粒物介导的ARGs污染在大气环境中的传播,从而有效缓解环境空气中的细菌耐药污染问题。
3 结语本研究基于传统培养方法与分子生物学技术,评估了紫外消毒法对大气颗粒物中细菌和ARGs的去除作用,并分析了紫外消毒对ARGs组成和潜在转移风险的影响。结果表明,紫外消毒对大气颗粒物中的细菌有较好的去除效果,并能在一定程度上降低ARGs与MGEs的丰度,削弱ARGs的传播风险,但去除ARGs比灭活细菌更难。同时,紫外消毒可能导致ARGs在残存细菌胞内选择性富集,从而使其总相对丰度上升。由于不同种类ARGs的去除率之间存在显著差异(p<0.01),紫外消毒在一定程度上改变了大气颗粒物中的抗生素抗性组,导致对紫外线耐受能力强的ARGs及其宿主菌成为优势类型。作为ARGs的载体,MGEs的丰度与总ARGs丰度呈显著正相关(p<0.01),紫外消毒后MGEs丰度的降低有助于减弱ARGs在大气环境中的转移风险。此外,位于同一基因元件上的不同ARGs间通常存在较强的相关性,这将促进环境中多重抗生素抗性菌的传播扩散,在消毒过程中应当重点关注。总体而言,紫外消毒具有控制大气颗粒物中ARGs污染的应用潜力,未来可以通过与过滤、氧化消毒等方法的联用,进一步提高对ARGs的去除效率。
| [1] |
Wright G D. The antibiotic resistome: The nexus of chemical and genetic diversity[J]. Nature Reviews Microbiology, 2007, 5(3): 175-186. DOI:10.1038/nrmicro1614 (  0) 0) |
| [2] |
Fournier P E, Vallenet D, Barbe V, et al. Comparative genomics of multidrug resistance in Acinetobacter baumannii[J]. PLoS Genetics, 2006, 2(1): e7. DOI:10.1371/journal.pgen.0020007 (  0) 0) |
| [3] |
Sutradhar I, Ching C, Desai D, et al. Computational model to quantify the growth of antibiotic-resistant bacteria in wastewater[J]. Msystems, 2021, 6(3): 10-21. (  0) 0) |
| [4] |
Tao Y, Yue Y, Wang J. Abundance and diversity of antibiotic resistance genes possibly released to ambient air by experiments in biology laboratories[J]. Science of the Total Environment, 2021, 797: 149147. DOI:10.1016/j.scitotenv.2021.149147 (  0) 0) |
| [5] |
王明星. 大气化学[M]. 北京: 气象出版社, 1999. Wang M X. Atmospheric Chemistry[M]. Beijing: China Meteorological Press, 1999. (  0) 0) |
| [6] |
Hu J, Zhao F, Zhang X X, et al. Metagenomic profiling of ARGs in airborne particulate matters during a severe smog event[J]. Science of the Total Environment, 2018, 615: 1332-1340. DOI:10.1016/j.scitotenv.2017.09.222 (  0) 0) |
| [7] |
Gao M, Zhang Q, Lei C, et al. Atmospheric antibiotic resistome driven by air pollutants[J]. Science of the Total Environment, 2023, 902: 165942. DOI:10.1016/j.scitotenv.2023.165942 (  0) 0) |
| [8] |
Li J, Cao J, Zhu Y, et al. Global survey of antibiotic resistance genes in air[J]. Environmental Science & Technology, 2018, 52(19): 10975-10984. (  0) 0) |
| [9] |
Zhao Y, Wang Q, Chen Z, et al. Significant higher airborne antibiotic resistance genes and the associated inhalation risk in the indoor than the outdoor[J]. Environmental Pollution, 2021, 268: 115620. DOI:10.1016/j.envpol.2020.115620 (  0) 0) |
| [10] |
Xie J, Jin L, He T, et al. Bacteria and antibiotic resistance genes (ARGs) in PM2.5 from China: Implications for human exposure[J]. Environmental Science & Technology, 2018, 53(2): 963-972. (  0) 0) |
| [11] |
Liang Z, Yu Y, Ye Z, et al. Pollution profiles of antibiotic resistance genes associated with airborne opportunistic pathogens from typical area, Pearl River Estuary and their exposure risk to human[J]. Environment International, 2020, 143: 105934. DOI:10.1016/j.envint.2020.105934 (  0) 0) |
| [12] |
Huijbers P M C, Blaak H, de Jong M C M, et al. Role of the environment in the transmission of antimicrobial resistance to humans: A review[J]. Environmental Science & Technology, 2015, 49(20): 11993-12004. (  0) 0) |
| [13] |
李中浤, 薛媚, 李顺江, 等. 气溶胶中抗生素抗性基因研究进展[J]. 生物工程学报, 2022, 38(7): 2423-2432. Li Z H, Xue M, Li S J, et al. Progress in the study of antibiotic resistance genes in aerosols[J]. Chinese Journal of Biotechnology, 2022, 38(7): 2423-2432. (  0) 0) |
| [14] |
Pereira A R, Braga D F O, Vassal M, et al. Ultraviolet C irradiation: A promising approach for the disinfection of public spaces?[J]. Science of the Total Environment, 2023, 879: 163007. DOI:10.1016/j.scitotenv.2023.163007 (  0) 0) |
| [15] |
Chang P H, Juhrend B, Olson T M, et al. Degradation of extracellular antibiotic resistance genes with UV254 treatment[J]. Environmental Science & Technology, 2017, 51(11): 6185-6192. (  0) 0) |
| [16] |
Lin W, Li S, Zhang S, et al. Reduction in horizontal transfer of conjugative plasmid by UV irradiation and low-level chlorination[J]. Water Research, 2016, 91: 331-338. DOI:10.1016/j.watres.2016.01.020 (  0) 0) |
| [17] |
Laghari A A, Liu L, Kalhoro D H, et al. Mechanism for reducing the horizontal transfer risk of the airborne antibiotic-resistant genes of Escherichia coli species through microwave or UV irradiation[J]. International Journal of Environmental Research and Public Health, 2022, 19(7): 4332-4345. DOI:10.3390/ijerph19074332 (  0) 0) |
| [18] |
McKinney C W, Pruden A. Ultraviolet disinfection of antibiotic resistant bacteria and their antibiotic resistance genes in water and wastewater[J]. Environmental Science & Technology, 2012, 46(24): 13393-13400. (  0) 0) |
| [19] |
Beck S E, Rodriguez R A, Linden K G, et al. Wavelength dependent UV inactivation and DNA damage of adenovirus as measured by cell culture infectivity and long range quantitative PCR[J]. Environmental Science & Technology, 2014, 48(1): 591-598. (  0) 0) |
| [20] |
中华人民共和国卫生部. 消毒技术规范[S]. 北京: 中华人民共和国卫生部, 2002. Ministry of Health of the People's Republic of China. Technical Standard for Disinfection[S]. Beijing: Ministry of Health of the People's Republic of China, 2002. (  0) 0) |
| [21] |
国家质量监督检验检疫总局. GB/T 19837—2005, 城市给排水紫外线消毒设备[S]. 北京: 中国标准出版社, 2005. PRC State Administration of Quality Supervision and Quarantine. GB/T 19837—2005, Ultraviolet (UV) Disinfection Equipment for Municipal Water and Wastewater Treatment[S]. Beijing: Standard Press of China, 2005. (  0) 0) |
| [22] |
Gou H, Lu J, Li S, et al. Assessment of microbial communities in PM1 and PM10 of Urumqi during winter[J]. Environmental Pollution, 2016, 214: 202-210. DOI:10.1016/j.envpol.2016.03.073 (  0) 0) |
| [23] |
Rodriguez A, Sesena S, Sanchez E, et al. Temporal variability measurements of PM2.5 and its associated metals and microorganisms on a suburban atmosphere in the central Iberian Peninsula[J]. Environmental Research, 2020, 191: 110220. DOI:10.1016/j.envres.2020.110220 (  0) 0) |
| [24] |
Jiang W J, Liang P, Wang B Y, et al. Optimized DNA extraction and metagenomic sequencing of airborne microbial communities[J]. Nature Protocols, 2015, 10(5): 768-779. DOI:10.1038/nprot.2015.046 (  0) 0) |
| [25] |
Yin F, Zhu Y, Koutchma T, et al. Inactivation and potential reactivation of pathogenic Escherichia coli O157∶H7 in apple juice following ultraviolet light exposure at three monochromatic wavelengths[J]. Food Microbiology, 2015, 46: 329-335. DOI:10.1016/j.fm.2014.08.015 (  0) 0) |
| [26] |
杨纯佳, 张娟, 周臣清, 等. 数字PCR在食源性致病微生物检测中的应用研究进展[J]. 食品安全质量检测学报, 2016, 7(12): 4726-4730. Yang C J, Zhang J, Zhou C Q, et al. Application progress on the digital in detection of foodborne pathogenic bacteria[J]. Journal of Food Safety and Quality Inspection, 2016, 7(12): 4726-4730. (  0) 0) |
| [27] |
Singh H, Bhardwaj S K, Khatri M, et al. UVC radiation for food safety: An emerging technology for the microbial disinfection of food products[J]. Chemical Engineering Journal, 2021, 417: 128084. DOI:10.1016/j.cej.2020.128084 (  0) 0) |
| [28] |
D'Costa V M, King C E, Kalan L, et al. Antibiotic resistance is ancient[J]. Nature, 2011, 477(7365): 457-461. DOI:10.1038/nature10388 (  0) 0) |
| [29] |
Guo M T, Kong C. Antibiotic resistant bacteria survived from UV disinfection: Safety concerns on genes dissemination[J]. Chemosphere, 2019, 224: 827-832. DOI:10.1016/j.chemosphere.2019.03.004 (  0) 0) |
| [30] |
郑吉, 周振超, 陈芳, 等. 3种常规消毒方法对磺胺类抗性基因削减效果的比较[J]. 环境科学, 2017, 38(4): 1497-1505. Zheng J, Zhou Z C, Chen F, et al. Comparison of the effects of three conventional sterilization methods on the reduction of sulfonamide resistance genes[J]. Environmental Science, 2017, 38(4): 1497-1505. (  0) 0) |
| [31] |
Liu L, Meng G, Laghari A A, et al. Reducing the risk of exposure of airborne antibiotic resistant bacteria and antibiotic resistance genes by dynamic continuous flow photocatalytic reactor[J]. Journal of Hazardous Materials, 2022, 429: 128311. DOI:10.1016/j.jhazmat.2022.128311 (  0) 0) |
| [32] |
Yin X, Chen X, Jiang X T, et al. Toward a universal unit for quantification of antibiotic resistance genes in environmental samples[J]. Environmental Science & Technology, 2023, 57(26): 9713-9721. (  0) 0) |
| [33] |
Zhang Z, Li B, Li N, et al. Effects of UV disinfection on phenotypes and genotypes of antibiotic-resistant bacteria in secondary effluent from a municipal wastewater treatment plant[J]. Water Research, 2019, 157: 546-554. DOI:10.1016/j.watres.2019.03.079 (  0) 0) |
| [34] |
Hu Q, Zhang X X, Jia S, et al. Metagenomic insights into ultraviolet disinfection effects on antibiotic resistome in biologically treated wastewater[J]. Water Research, 2016, 101: 309-317. DOI:10.1016/j.watres.2016.05.092 (  0) 0) |
| [35] |
Li B, Yang Y, Ma L, et al. Metagenomic and network analysis reveal wide distribution and co-occurrence of environmental antibiotic resistance genes[J]. The ISME Journal, 2015, 9(11): 2490-2502. DOI:10.1038/ismej.2015.59 (  0) 0) |
| [36] |
Wang N, Yang X, Jiao S, et al. Sulfonamide-resistant bacteria and their resistance genes in soils fertilized with manures from Jiangsu Province, Southeastern China[J]. PloS One, 2014, 9(11): e112626. DOI:10.1371/journal.pone.0112626 (  0) 0) |
| [37] |
Sun J, Li L, Liu B, et al. Development of aminoglycoside and β-lactamase resistance among intestinal microbiota of swine treated with lincomycin, chlortetracycline, and amoxicillin[J]. Frontiers in Microbiology, 2014, 5: 580. (  0) 0) |
| [38] |
Soni K, Kothamasi D, Chandra R. Municipal wastewater treatment plant showing a potential reservoir for clinically relevant MDR bacterial strains co-occurrence of ESBL genes and integron-integrase genes[J]. Journal of Environmental Management, 2024, 351: 119938. DOI:10.1016/j.jenvman.2023.119938 (  0) 0) |
| [39] |
Guo M T, Yuan Q B, Yang J. Distinguishing effects of ultraviolet exposure and chlorination on the horizontal transfer of antibiotic resistance genes in municipal wastewater[J]. Environmental Science & Technology, 2015, 49(9): 5771-5778. (  0) 0) |
| [40] |
Ye C, Chen Y, Feng L, et al. Effect of the ultraviolet/chlorine process on microbial community structure, typical pathogens, and antibiotic resistance genes in reclaimed water[J]. Frontiers of Environmental Science & Engineering, 2022, 16(8): 100. (  0) 0) |
| [41] |
Zhu N, Long Y, Kan Z, et al. Reduction of mobile genetic elements determines the removal of antibiotic resistance genes during pig manure composting after thermal pretreatment[J]. Bioresource Technology, 2023, 387: 129672. DOI:10.1016/j.biortech.2023.129672 (  0) 0) |
2. Laboratory for Marine Ecology and Environmental Science, Qingdao Marine Science and Technology Center, Qingdao 266237, China
 2025, Vol. 55
2025, Vol. 55


