2. 唐山海都水产食品有限公司,河北 唐山 063000
红鳍东方鲀(Takifugu rubripes)是中国北方特色海水养殖种类,其养殖主要分布在我国的山东、河北和辽宁省的沿海区域。目前红鳍东方鲀的池塘养殖方式还存在一定的不足,主要体现为由于其生物学特性导致的养殖产量较低和生产效率较低等,尤其是红鳍东方鲀单养易造成水体空间利用率低、饵料资源浪费和养殖环境污染等问题[1]。这些问题不仅可能对周边水域环境产生负面影响[2],还会增加养殖生物患病的风险,进而给养殖带来潜在的经济损失[3]。为了优化红鳍东方鲀养殖系统,在养殖系统中引入不同种类的养殖生物进行混养,不仅能够更充分地利用饵料资源,还有助于水质的有效调控,从而实现经济和生态效益的双重提升,促进红鳍东方鲀养殖业的可持续发展。
微生物群落是养殖系统的重要组成部分,对养殖动物的健康和生长起着至关重要的作用。在养殖过程中,残饵、粪便的积累以及养殖动物的排泄物等都会对水质和底质等环境因子产生影响,从而改变养殖系统环境微生物群落结构。与此同时,环境微生物也会对养殖动物的生存和生长产生影响[4]。肠道微生物与肠道上皮细胞存在紧密的共生关系,是细菌、古菌、真核生物和病毒等在肠腔内的集合[5-8]。肠道内的共生微生物群在长期的协同进化过程中,建立了错综复杂的平衡共存机制,形成了彼此依赖、相互制约的微生态系统,参与到宿主对营养的消化[9]、免疫、生理、病原体拮抗以及神经功能[10-12]等多个方面,对于动物的发育乃至生存都起着至关重要的作用[13-15]。水体微生物群落是水产养殖生态系统重要的组成部分之一,为水生动物提供了丰富多样的微生物种类,被视为水生动物体内微生物群落的储藏库之一[16-20]。正常的水体微生物群落不仅有助于维持养殖环境的生态平衡,还可为整个养殖系统提供必要的生态服务。水体微生物群。水体微生物群的组成是动态变化的,它随着水参数的物理化学因素的变化而变动[21-26]。沉积物也是水产养殖生态系统的重要组成部分,是养殖系统基本营养物质交换的重要场所[27-28]。研究表明,沉积物微生物群落在水产养殖生态系统中具有重要功能,对养殖生态系统元素的生物地球化学循环起着关键作用,并影响到水体各种理化因子的变化[29]。在养殖过程中,人们经常会人工投入大量的营养物质,如饲料和肥料等。然而,由于养殖系统和养殖生物对这些有机质的利用效率和能力限制,往往会导致有机质在系统中不断沉积和累积,并最终沉积在池塘底部。因此,在养殖实践中如何合理调控养殖系统微生物群落,以提高养殖系统物质循环效率,有效改善养殖环境,一直是养殖环境优化与调控技术研究的重要方向之一。
海水池塘养殖是红鳍东方鲀的主要养殖方式之一。然而,关于红鳍东方鲀池塘养殖系统的微生物多样性和结构特点,迄今尚少见报道。本研究通过对比研究四种不同红鳍东方鲀养殖系统的微生物群落结构,探究了不同养殖系统红鳍东方鲀肠道、水体和沉积物细菌群落多样性、以及组成和结构特征,旨在为红鳍东方鲀池塘养殖中微生物调控、益生菌筛选和水产疾病防治等提供理论参考。
1 材料与方法 1.1 养殖系统本研究于2022年5月—10月在唐山海都水产食品有限公司养殖基地(河北,唐山)池塘中进行。研究设置了四种不同的养殖系统:红鳍东方鲀单养系统(A系统),红鳍东方鲀-美洲帘蛤(Mercenaria mercenaria)二元混养系统(B系统),红鳍东方鲀-日本对虾(Penaeus japonicus)-美洲帘蛤三元混养系统(C系统),红鳍东方鲀(成鱼)-日本对虾传统系统(E系统)。其中,红鳍东方鲀(成鱼)-日本对虾养殖模式是唐山地区传统的二元混养方式。每种系统分别选择3个池塘,合计12个海水池塘。池塘均为泥沙质底,平均面积为3.33 hm2,研究期间水深保持在1.8~2.0 m。
1.2 养殖动物放养情况各系统红鳍东方鲀、美洲帘蛤和日本对虾放养情况见表 1。其中,红鳍东方鲀单养系统(A系统)投放质量在7~10 g之间的红鳍东方鲀幼鱼7 500尾/hm2;红鳍东方鲀-美洲帘蛤二元混养系统(B系统)投放质量在7~10 g之间的红鳍东方鲀幼鱼7 500尾/hm2,同时在池底投放90 kg/hm2规格在2 400粒/kg的美洲帘蛤苗种;红鳍东方鲀-日本对虾-美洲帘蛤三元混养系统(C系统)投放质量在7~10 g之间的红鳍东方鲀幼鱼7 500尾/hm2,同时投放90 000尾/hm2日本对虾苗和90 kg/hm2规格在2 400粒/kg的美洲帘蛤苗种;E系统中投放均重400~500 g左右的大规格红鳍东方鲀225尾/hm2,投放75 000尾/hm2日本对虾苗种。
|
|
表 1 不同红鳍东方鲀养殖系统放养和收获基本情况 Table 1 Comparison of stocking and yield of Takifugu rufugu in different culture systems |
各养殖系统红鳍东方鲀投喂人工饵料玉筋鱼每日投喂2~3次,投喂比例为总湿质量的3%~5%,日本对虾投喂人工饵料玉筋鱼、卤虫、蜢虾和兰蛤等。根据养殖动物生物量、鱼体大小、摄食情况、水温和天气条件适当调整投喂频率和投喂量。
每日定时巡塘,观察鱼虾生长情况,并根据池塘实际需求和水质变化适当换水。
1.4 样品采集与处理养殖中期,分别从四种养殖系统中采集红鳍东方鲀肠道、水体和沉积物微生物样品。其中,每个系统分别随机捕捞15尾红鳍东方鲀,肠道微生物样品装入无菌冻存管后,及时放入液氮中贮存。按照5点采样法,使用灭菌后的玻璃采水器从养殖系统水面下0.5 m处分别采集水样,对不同系统采集到的水样分别充分混合并立即放在冰上运回实验室。使用0.22 μm聚合纤维滤膜对水样进行过滤,然后将滤膜置于无菌冻存管并立即保存在液氮中,剩余的水样则用于检测水体理化参数。池塘沉积物样品也根据5点法从表层(0~5 cm)底泥中获取,对采集的沉积物分系统充分混合后放入无菌冻存管并储存在液氮中。
1.5 DNA提取和16S rRNA测序利用MagPure Soil DNA LQ Kit试剂盒(美基生物,广州,中国)对样本中的DNA进行提取,利用琼脂糖凝胶电泳和NanoDrop2000仪器(NanoDrop Technologies, Wilmington, 美国)检测抽提DNA的质量和浓度。随后以提取的DNA为模板,根据选择的测序区域,使用带barcode的特异引物进行PCR扩增。使用通用引物343F (5′-TACGGRAGGCAGCAG-3′) 和798R (5′-AGGGTATCTAATCCT-3′) 对16S rRNA基因中的V3—V4高变区进行特异扩增。第一轮扩增产物通过琼脂糖凝胶电泳进行检测和分离,并使用Agencourt AMPure XP beads (Beckman Coulter Inc., Brea, CA, 美国)进行纯化。纯化后的扩增产物随即进行第二轮PCR以添加测序接头。PCR体系组成以及具体反应流程参见文献[30]。
二轮扩增产物经过再次纯化后,使用Qubit dsDNA assay kit (Life Technologies, Carlsbad, CA, 美国)检测浓度并构建等量的文库。高通量测序在美吉生物技术有限公司的Illumina Miseq平台上进行。最终,从测序平台上获得原始的双端序列数据(Paired-end sequence)。
1.6 生物信息分析原始数据通过barcode序列拆分后生成独立的Fastq格式文件,然后在QIIME2开源平台上进行菌群组成和多样性分析[31]。使用DADA2算法对原始的双端序列执行减除低质量碱基、降噪、合并以及去除嵌合体等以获得有效的特征表和代表序列[32]。为了便于下游分析,根据最小的样品序列数作为稀释深度对特征表进行均一化处理。利用QIIME2的“phylogeny”插件构建有根系统发育树,用于后续微生物多样性分析。
使用QIIME2的“diversity”插件计算Simpson指数、Chao1指数和Shannon均匀度指数等,以评估细菌群落的α多样性。通过基于Bray-Curtis距离的主坐标分析(PCoA)来评估细菌群落在整体结构上的差异,并在R(version 4.1.0)上使用“Vegan”包来执行PERMANOVA分析,以检验群落结构差异的显著性[33]。构建基于Unweighted unifrac距离的层级样品聚类分析,以探究不同样品间的系统发育关系。利用LEfSe(Linear discriminant analysis Effect Size)分析来鉴别群落间的差异分类群[34],|LDA score|>2且P<0.05被设置为筛选的阈值。Venn分析在R语言上使用“VennDiagram”包进行,相似性分析在R语言上使用“Vegan”包进行[35]。
在本研究中,α多样性数值通过Kruskal-Wallis检验进行组间比较,取P<0.05为差异显著。使用单因素方差分析(one-way ANOVA)和Duncan’s法进行多组间比较,取P<0.05为差异显著。
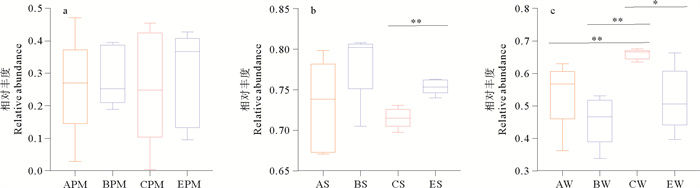
2 结果 2.1 不同养殖系统微生物群落多样性分析 2.1.1 不同养殖系统微生物群落α多样性分析对四种养殖系统的红鳍东方鲀肠道、沉积物和水体微生物进行α多样性分析,分析结果见表 2。可以看出,四种养殖模式红鳍东方鲀肠道微生物多样性指数未见显著差异,E系统沉积物微生物群落物种丰富度显著高于A、C系统(P<0.05),E系统沉积物微生物多样性显著高于C系统(P<0.05),C系统水体微生物多样性显著高于A、B系统(P<0.05)。在群落均匀度方面,四种养殖模式红鳍东方鲀肠道微生物均匀度未见显著差异(见图 1a),E系统沉积物微生物显著高于C系统(P<0.05)(见图 1b),C系统水体微生物均匀度显著高于A、B、E系统(P<0.05)(见图 1c)。
|
|
表 2 不同养殖模式下微生物群落α多样性指数 Table 2 Alpha-diversity of microbial community in different culture systems |
|
( a: 红鳍东方鲀肠道样品; b: 底泥样品; c: 水体样品。APM、AS、AW分别代表A系统红鳍东方鲀肠道、沉积物、水体微生物样品,BPM、BS、BW分别代表B系统红鳍东方鲀肠道、沉积物、水体微生物样品,CPM、CS、CW分别代表C系统红鳍东方鲀肠道、沉积物、水体微生物样品,EPM、ES、EW分别代表E系统红鳍东方鲀肠道、沉积物、水体微生物样品,下图同。星号表示显著性水平, * : P<0.05, ** : P<0.01。a: Takifugu rubripes intestine samples; b: Sediment samples; c: Water samples. APM, AS and AW represent Takifugu rubripes intestine, sediment and water microbial samples of system A, BPM, BS and BW represent T. rubripes intestine, sediment and water microbial samples of system B, CPM, CS and CW represent T. rubripes intestine, sediment and water microbial samples of system C, respectively, EPM, ES and EW represent T. rubripes intestine, sediment and water microbial samples of system E, the same below. Asterisks indicates significance levels, * : P<0.05, ** : P<0.01. ) 图 1 不同养殖模式Shannoneven指数差异分析 Fig. 1 The differentiation analysis of Shannoneven index from different culture patterns |
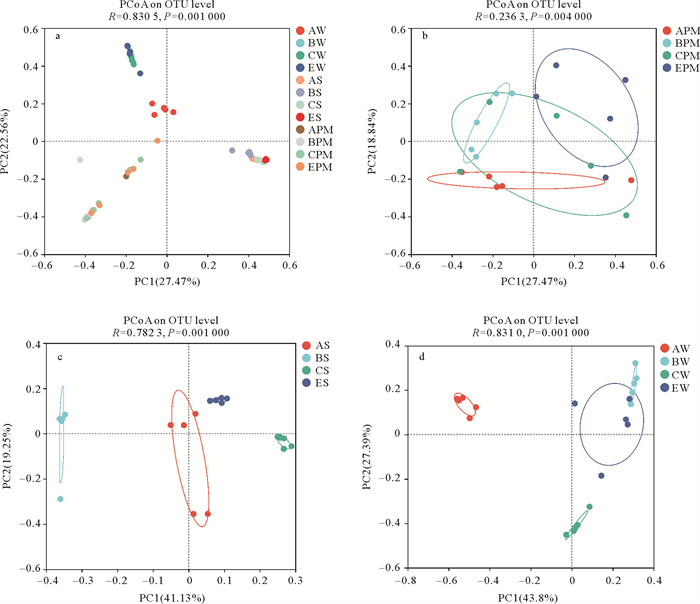
对不同养殖系统红鳍东方鲀肠道、沉积物和水体微生物样品进行基于Bary-Curtis距离的主坐标分析(PCoA),结果如图 2a所示。可以看出,第一主坐标轴对所有样本差异的贡献度为22.56%,第二主坐标轴对所有样本差异的贡献度为27.47%,代表红鳍东方鲀肠道微生物的点在第三象限聚集,代表沉积物微生物的点在第四象限聚集,代表水体微生物的点在第一、二象限聚集,表明水体、沉积物和红鳍东方鲀肠道的微生物存在明显差异。
|
( a: 所有样品; b: 红鳍东方鲀肠道样品; c: 底泥样品; d: 水体样品。a: All samples; b: Takifugu rubripes intestine samples; c: Sediment samples; d: Water samples. ) 图 2 不同养殖模式PCoA聚类分析 Fig. 2 PCoA cluster analysis from different culture patterns |
不同养殖系统红鳍东方鲀肠道菌群在结构上存在显著差异(P<0.05),其中分组因素可解释总差异的50.85%。不同系统比较,A系统红鳍东方鲀肠道菌群与E系统距离较远,表明红鳍东方鲀单养系统红鳍东方鲀肠道菌群与传统二元混养系统差异较大(见图 2b)。
四种养殖系统沉积物微生物群落结构具有显著差异(P<0.05),其中分组因素可解释总差异的60.38%(见图 2c)。
四种红鳍东方鲀不同养殖系统的微生物群落结构具有显著差异(P<0.05),其中分组因素可解释总差异的71.19%。不同系统比较,A系统水体微生物样品与另外三个系统距离较远,表明红鳍东方鲀单养系统水体微生物样品与其他三个系统差异较大(见图 2d)。
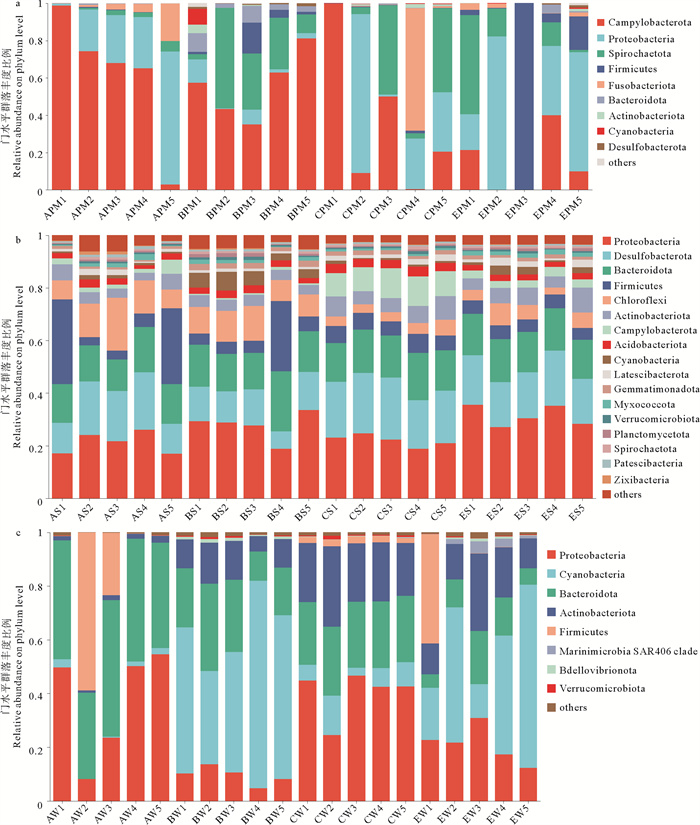
2.2 不同养殖系统微生物群落组成分析 2.2.1 基于门水平的微生物群落组成分析针对不同样本的微生物组成结构,分析了红鳍东方鲀肠道、沉积物、水体在门水平上的物种组成,仅统计丰度占比大于0.01%的种类,所有样本中未在该分类级别上注释的物种被合并到“other”组中,结果如图 3所示。
|
( a: 红鳍东方鲀肠道样品; b: 底泥样品; c: 水体样品。a: Takifugu rubripes intestine samples; b: Sediment samples; c: Water samples. ) 图 3 不同养殖系统基于门水平上的微生物组成与丰度 Fig. 3 Composition and relative abundance of microbiota from different culture patterns at phylum level |
由图 3a可以看出,A系统红鳍东方鲀肠道中的优势菌门主要(相对丰度大于5%)为弯曲杆菌门(Campilobacterota,61.85%)、变形菌门(Proteobacteria,29.58%)和梭杆菌门(Fusobacteriota,5.71%)。B系统红鳍东方鲀肠道中的优势菌门为弯曲杆菌门(56.08%)、螺旋菌门(Spirochaetota,24.87%)、拟杆菌门(Bacteroidota,5.43%)和变形菌门(5.05%)。C系统红鳍东方鲀肠道中的优势菌门为弯曲杆菌门(36.05%)、变形菌门(28.00%)、螺旋菌门(19.89%)和梭杆菌门(13.39%)。E系统中红鳍东方鲀肠道中的优势菌门为变形菌门(40.47%)、厚壁菌门(Firmicutes,24.10%)、螺旋菌门(16.44%)和弯曲杆菌门(14.31%)。
由图 3b可以看出,A系统沉积物中的优势菌门主要为变形菌门(21.23%)、脱硫杆菌门(Desulfobacterota,16.85%)、厚壁菌门(14.52%)、拟杆菌门(14.55%)和绿弯菌门(Chloroflexi,11.95%)。B系统沉积物中的优势菌门为变形菌门(27.67%)、拟杆菌门(16.33%)、脱硫杆菌门(11.97%)、绿弯菌门(10.30%)和厚壁菌门(9.12%)。C系统沉积物中的优势菌门为变形菌门(22.00%)、脱硫杆菌门(21.24%)、拟杆菌门(16.10%)、弯曲杆菌门(10.01%)、放线菌门(Actinobacteriota,6.70%)和厚壁菌门(6.23%)。E系统沉积物中的优势菌门为变形菌门(31.34%)、脱硫杆菌门(18.34%)、拟杆菌门(15.66%)、放线菌门(6.15%)和绿弯菌门(5.25%)。
由图 3c可以看出,A系统水体中的优势菌门主要为拟杆菌门(42.42%)、变形菌门(37.29%)和厚壁菌门(16.47%)。B系统水体中的优势菌门为蓝细菌门(Cyanobacteria,54.33%)、拟杆菌门(22.00%)、放线菌门(11.38%)和变形菌门(9.52%)。C系统水体中的优势菌门为变形菌门(40.27%)、拟杆菌门(24.63%)、放线菌门(23.05%)和蓝细菌门(7.89%)。E系统中水体中的优势菌门为蓝细菌门(38.96%)、变形菌门(21.05%)、放线菌门(16.67%)、拟杆菌门(11.08%)和厚壁菌门(8.19%)。
在肠道菌群的组成上,弯曲杆菌门、变形菌门、螺旋菌门和厚壁菌门占绝对优势,分别在A、B、C和E系统红鳍东方鲀肠道菌群占总丰度的93.98%、90.64%、85.36%、96.32%;在沉积物微生物群落的组成上,变形菌门、拟杆菌门、脱硫杆菌门,厚壁菌门和绿弯菌门占绝对优势,分别在A、B、C和E系统沉积物微生物群落中占总丰度的79.11%、75.58%、69.67%、75.53%;在水体微生物群落的组成上,蓝细菌门、拟杆菌门、变形菌门和放线菌门占绝对优势,分别在A、B、C和E系统水体微生物群落中占总丰度的82.96%、97.33%、95.85%、87.55%。
总体来看,A、B和C系统红鳍东方鲀肠道微生物占比较低的厚壁菌门在E系统红鳍东方鲀肠道微生物中为优势菌门,占比可达25.10%,四个系统的沉积物微生物组成和群落相对丰度相似。B、C和E系统水体微生物中为优势菌门的放线菌门在A系统水体微生物中丰度占比仅为1.77%。
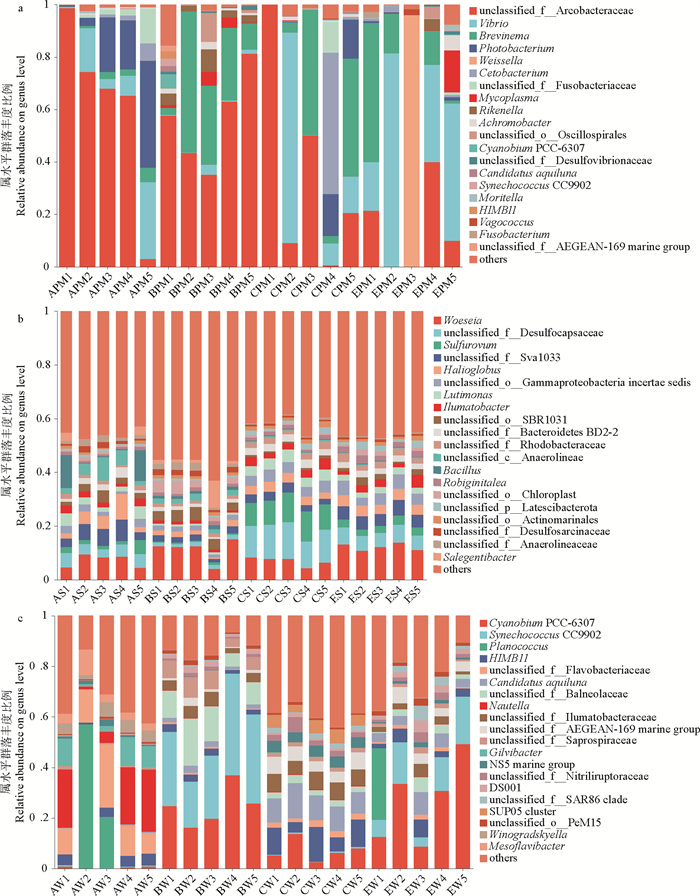
2.2.2 基于属水平的微生物群落组成分析分析了红鳍东方鲀肠道、沉积物、水体在属水平上的物种组成,仅统计丰度占比前20种的种类,所有样本中未在该分类级别上注释的物种被合并到“other”组中,结果如图 4所示。
|
( a: 红鳍东方鲀肠道样品; b: 底泥样品; c: 水体样品。a: Takifugu rubripes intestine samples; b: Sediment samples; c: Water samples. ) 图 4 不同养殖系统基于属水平上的微生物组成与丰度 Fig. 4 Composition and relative abundance of microbiota from different culture patterns at genus level |
由图 4a可以看出,在属水平上,A系统红鳍东方鲀肠道中的优势菌属(相对丰度大于5%)为未分类的弓形杆菌科(unclassified_f__Arcobacteraceae,61.85%)、发光杆菌属(Photobacterium,16.80%)和弧菌属(Vibrio,11.52%)。B系统红鳍东方鲀肠道中的优势菌属为未分类的弓形杆菌科(56.08%)和短螺旋菌属(Brevin-ema,24.87%)。C系统红鳍东方鲀肠道中的优势菌属为未分类的弓形杆菌科(36.05%)、弧菌属(20.54%)、短螺旋菌属(19.89%)、鲸杆菌属(Cetobacterium,10.99%)和发光杆菌属(6.22%)。E系统红鳍东方鲀肠道中的优势菌属为弧菌属(37.82%)、魏斯氏菌属(Weissella,19.20%)、短螺旋菌属(16.44%)和未分类的弓形杆菌科(14.28%)。
由图 4b可以看出,A系统沉积物中的优势菌属为海洋伍斯菌属(Woeseia,7.08%)和未分类的Sva1033 (unclassified_f__Sva1033,5.26%)。B系统沉积物中的优势菌属为海洋伍斯菌属(11.23%)。C系统沉积物中的优势菌属为未分类的脱硫杆菌科(unclassified_f__Desulfocapsaceae,12.06%)、硫卵菌属(Sulfurovum,9.87%)和海洋伍斯菌属(6.87%)。E系统沉积物中的优势菌属为海洋伍斯菌属(12.15%)和未分类的脱硫杆菌科(5.21%)。
由图 4c可以看出,A系统水体中的优势菌属为动性球菌属(Planococcus,15.47%)、小海员杆菌属(Nautella,15.02%)、未分类的黄杆菌科(norank_f__Flavobacteriaceae,13.73%)和Gilvibacter(6.68%)。B系统水体中的优势菌属为Synechococcus CC9902(29.64%)、Cyanobium PCC-6307(24.65%)、norank_f__Balneolaceae(11.44%)和未分类的腐败螺旋菌科(norank_f__Saprospiraceae,6.21%)。C系统水体中的优势菌属为Candidatus auiluna(10.28%)、HIMB11(9.03%)、Cyanobium PCC-6307(7.16%)、norank_f__llumatobacteraceae(6.42%)和norank_f__AEGEAN-169 marine group(5.54%)。E系统水体中的优势菌属为Cyanobium PCC-6307(26.97%)、Synechococcus CC9902(11.81%)和动性球菌属(5.66%)。
2.3 不同养殖系统微生物群落组成差异分析对不同系统红鳍东方鲀肠道、沉积物和水体分别进行了LEfSe多级物种差异分析,结果如图 5所示。可以看出,在门水平上,拟杆菌门和脱硫杆菌门在B系统红鳍东方鲀肠道中显著富集;梭杆菌门在C系统红鳍东方鲀肠道中显著富集;厚壁菌门在E系统红鳍东方鲀肠道中显著富集。绿弯菌门在A系统沉积物中显著富集;蓝细菌门在B系统沉积物中显著富集;脱硫杆菌门、弯曲杆菌门在C系统沉积物中显著富集;变形菌门在E系统沉积物中显著富集。拟杆菌门在A系统水体中显著富集;蓝细菌门在B系统水体中显著富集;变形菌门、放线菌门在C系统水体中显著富集;Marinimicrobia SAR406 clade在E系统水体中显著富集。
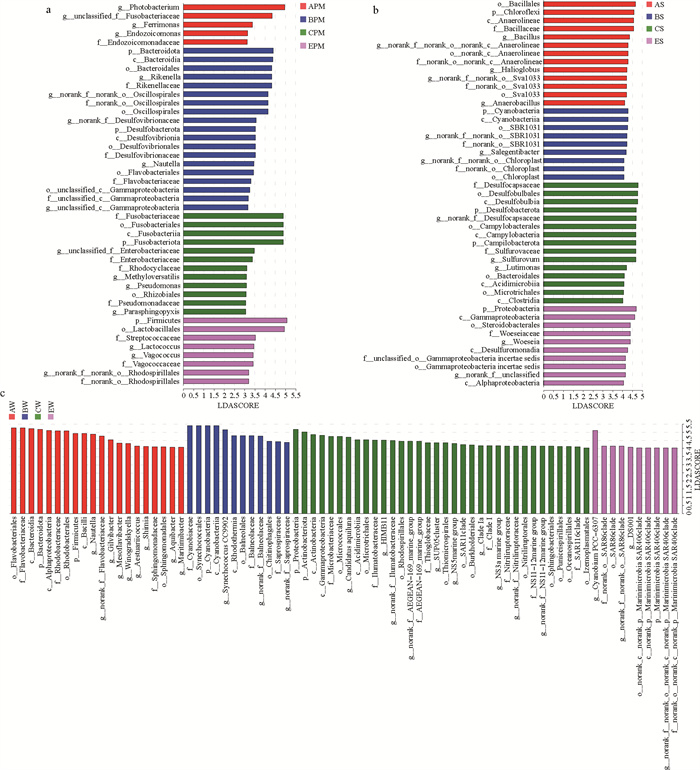
|
( a: 红鳍东方鲀肠道样品; b: 底泥样品; c: 水体样品。a: Takifugu rubripes intestine samples; b: Sediment samples; c: Water samples. ) 图 5 不同养殖系统微生物群落的LEfSe分析 Fig. 5 LEfSe analysis of microbial communities in different culture systems |
在目水平上,拟杆菌目(Bacteroidales)、颤螺菌目(Oscillospirales)、脱硫弧菌目(Desulfovibrionales)、黄杆菌目(Flavobacteriales)和未分类的γ-变形菌纲(unclassified_c__Gammaproteobacteria)在B系统红鳍东方鲀肠道中显著富集;梭杆菌目(Fusobacteriales)和根瘤菌目(Rhizobiales)在C系统红鳍东方鲀肠道中显著富集;乳杆菌目(Lactobacillales)和红螺菌目(Rhodospirillales)在E系统红鳍东方鲀肠道中显著富集。芽孢杆菌目(Bacillales)、Sva1033和未分类的厌氧线菌纲(norank_c__Anaerolineae)在A系统沉积物中显著富集;SBR1031和Chloroplast在B系统沉积物中显著富集;脱硫杆菌目(Desulfobulbales)、弯曲杆菌目(Campylobacterales)、拟杆菌目和Microtrichales在C系统沉积物中显著富集;Gammaproteobacteria incertae sedis和Steroidobacterales在E系统沉积物中显著富集。红杆菌目(Rhodobacterales)、黄杆菌目和鞘脂单胞菌目(Sphingomonadales)在A系统水体中显著富集;聚球藻目(Synechococcales)、Balneolales和噬几丁质菌目(Chitinophagales)在B系统水体中显著富集;Microtrichales、微球菌目(Micrococcales)、红螺菌目、Thiomicrospirales、SAR11 clade、伯克氏菌目(Burkholderiales)、解腈杆菌目(Nitriliruptorales)、鞘氨醇杆菌目(Sphingobacteriales)、Puniceispirillales、海洋螺杆菌目(Oceanospirillales)和Izemoplasmatales在C系统水体中显著富集;SAR406 clade和norank_c__norank_p__Marinimicrobia SAR406 clade在E系统水体中显著富集。
在属水平上,发光杆菌属、未分类的梭杆菌科(unclassified_f__Fusobacteriaceae)、铁还原单胞菌属(Ferrimonas)和动物内生单胞菌属(Endozoicomonas)在A系统红鳍东方鲀肠道内显著富集;理化所菌属(Rikenella)、未分类的颤螺菌目(norank_f__norank_o__Oscillospirales)、未分类的脱硫弧菌科(norank_f__Desulfovibrionaceae)、小海员杆菌属和未分类的γ-变形菌纲在B系统红鳍东方鲀肠道中显著富集;未分类的肠杆菌科(unclassified_f__Enterobacteriaceae)、甲基营养菌属(Methyloversatilis)、假单胞菌属(Pseudomonas)和副鞘氨醇盒菌属(Parasphingopyxis)在C系统红鳍东方鲀肠道中显著富集;乳球菌属(Lactococcus)、漫游球菌属(Vagococcus)和未分类的红螺菌目(norank_f__norank_o__Rhodospirillales)在E系统红鳍东方鲀肠道中显著富集。芽孢杆菌属(Bacillus)、未分类的厌氧线菌纲、海球形菌属(Halioglobus)、norank_f__norank_o__Sva1033和厌氧芽孢杆菌属(Anaerobacillus)在A系统沉积物中显著富集;嗜盐杆菌属(Salegentibacter)、norank_f__norank_o__SBR1031和norank_f__norank_o__Chloroplast在B系统沉积物中显著富集;硫卵菌属、绿脓杆菌属(Lutimonas)和未分类的脱硫杆菌科在C系统沉积物中显著富集;海洋伍斯菌属在E系统沉积物中显著富集。小海员杆菌属、未分类的黄杆菌科、黄色杆状样菌属(Gilvibacter)、中温黄杆菌属(Mesoflavibacter)、维诺格拉德斯基氏菌属(Winogradskyella)、泥滩球菌属(Aestuariicoccus)、沈氏菌属(Shimia)、沿海水杆菌属(Aquibacter)和海杆状菌属(Maritimibacter)在A系统水体中显著富集;Synechococcus CC9902、norank_f__Balneolaceae和未分类的腐败螺旋菌科在B系统水体中显著富集;Candidatus aquiluna、HIMB11、norank_f__Ilumatobacteraceae、norank_f__AEGEAN-169 marine group、SUP05 cluster、NS5 marine group、Clade Ia、Clade I、NS3a marine group、norank_f__Nitriliruptoraceae和norank_f__NS11-12 marine group在C系统水体中显著富集;Cyanobium PCC-6307、norank_f__norank_o__SAR86 clade、DS001和norank_f__norank_o__norank_c__norank_p__Marinimicrobia SAR406 clade在E系统水体中显著富集。
2.4 细菌群落相似性分析同组间不同样品共有及独有OTU数经加和后得到Venn图(见图 6)。所有样品共有的OTU共7个(见图 6a),A系统红鳍东方鲀肠道样品与水体和沉积物样品共有62和69个OTU,分别占红鳍东方鲀肠道总样品的42.8%和47.6%(见图 6b),B系统红鳍东方鲀肠道样品与水体和沉积物样品共有169和114个OTU,分别占红鳍东方鲀肠道总样品的51.7%和34.9%(见图 6c),C系统红鳍东方鲀肠道样品与水体和沉积物样品共有58和36个OTU,分别占红鳍东方鲀肠道总样品的29.7%和18.5%(见图 6d),E系统红鳍东方鲀肠道样品与水体和沉积物样品共有36和62个OTU,分别占红鳍东方鲀肠道总样品的17.0%和29.2%(见图 6e)。
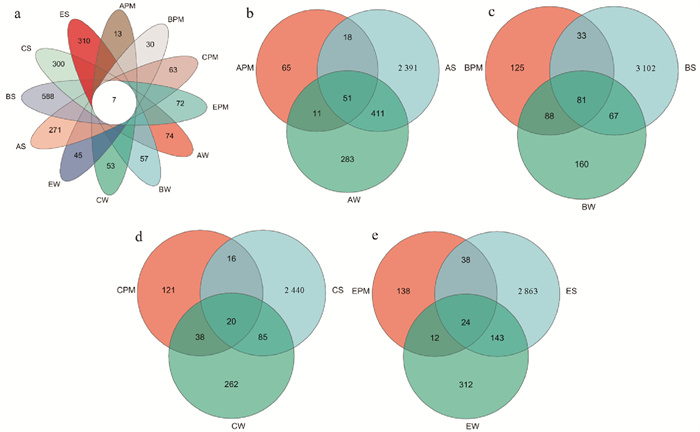
|
( a: 所有样品; b: A系统; c: B系统; d: C系统: e: E系统。a: All samples; b: System A; c: System B; d: System C; e: System E. ) 图 6 不同养殖模式样品OTU分布Venn图 Fig. 6 OTU Venn analysis of samples from different culture modes |
不同养殖系统细菌群落间的相似性分析结果如表 3所示,可以看出,四个系统红鳍东方鲀肠道微生物与环境微生物之间相似性很低,仅B系统肠道样品与沉积物样品平均相似度较高,为4.65%,这表明红鳍东方鲀肠道微生物组成也由宿主本身因素决定。
|
|
表 3 菌群相似性分析 Table 3 SIMPER analysis of bacterial communities |
不同养殖系统水体理化因子与水体微生物群落结构冗余分析如图 7所示,具体相关性和显著性参数如表 4所示。可以看出,盐度、氨氮、亚硝氮、硝氮、总氮与水体菌群群落结构显著相关(P<0.05),其中盐度、氨氮和硝氮浓度是影响水体菌群群落结构重要的影响因子,这些环境因子解释了四种养殖系统水体微生物群落结构差异的57.72%。
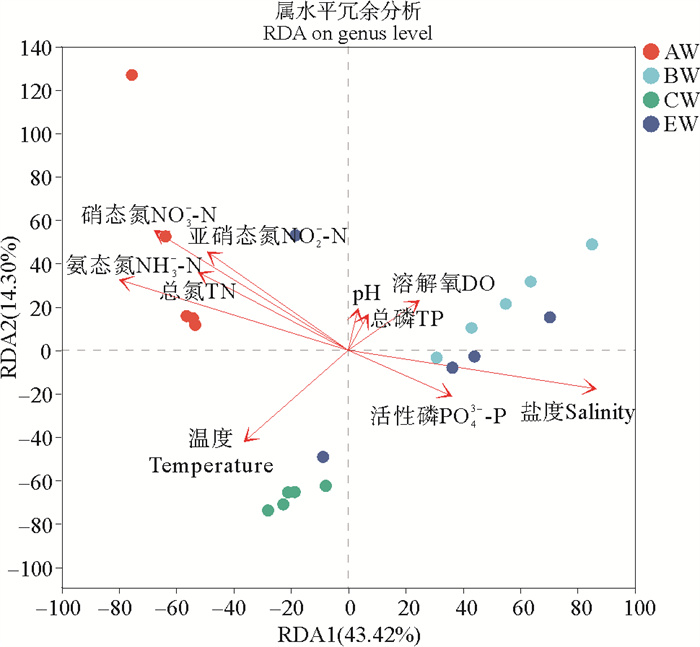
|
( RDA1:冗余分析1; RDA2: 冗余分析2; AW: A系统水体; BW: B系统水体; CW: C系统水体; EW: E系统水体。RDA1: Redundancy analysis 1; RDA2: Redundancy analysis 2; AW: Water in system A; BW: Water in system B; CW: Water in system C; EW: Water in system E. ) 图 7 水体理化因子与水体微生物群落冗余分析 Fig. 7 Redundancy analysis of physicochemical factors and microbial communities in water |
|
|
表 4 水体理化因子与水体微生物群落相关性分析 Table 4 Correlation analysis between physical and chemical factors and microbial communities in water |
不同养殖系统沉积物理化因子与沉积物微生物群落结构冗余分析结果如图 8所示,具体相关性和显著性参数如表 5所示。可以看出,氨氮、硝氮、总氮与沉积物菌群群落结构显著相关(P<0.05),其中氨氮是影响沉积物微生物群落结构最重要的影响因子。这些环境因子解释了四种不同养殖系统沉积物微生物群落结构总差异的48.01%。
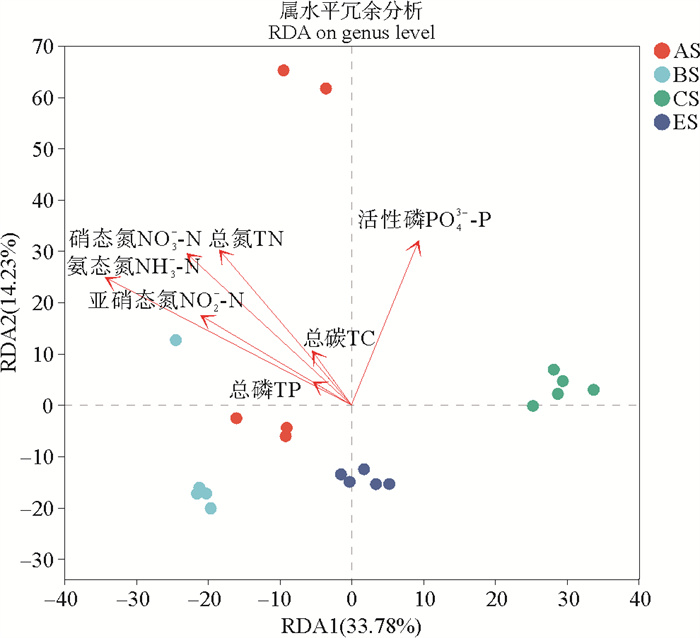
|
( RDA1:冗余分析1; RDA2: 冗余分析2; AS: A系统沉积物; BS: B系统沉积物; CS: C系统沉积物; ES: E系统沉积物。RDA1: Redundancy analysis 1; RDA2: Redundancy analysis 2; AS: Sediment in system A; BS: Sediment in system B; CS: Sediment in system C; ES: Sediment in system E. ) 图 8 沉积物理化因子与沉积物微生物群落冗余分析 Fig. 8 Redundancy analysis of sediment physicochemical factors and sediment microbial communities |
|
|
表 5 沉积物理化因子与沉积物微生物群落相关性分析 Table 5 Correlation analysis between sediment physical and chemical factors and sediment microbial community |
微生物群落是鱼类肠道微生态系统的重要组成部分,参与到宿主对营养的利用[9]、免疫、生理、病原体拮抗[10-12]等多个方面,对于动物生长发育、机体健康等都起着至关重要的作用[13-15]。研究表明,鱼类肠道中占优势的细菌类群主要为拟杆菌门、变形菌门、放线菌门、梭杆菌门、厚壁菌门和疣微菌门等[36]。其中,厚壁菌门、变形菌门、拟杆菌门在不同鱼类肠道中均有发现,一般认为其为核心微生物菌群[37]。在本研究中,红鳍东方鲀肠道中的菌群优势菌门主要包括弯曲杆菌门、变形菌门、梭杆菌门、螺旋菌门、拟杆菌门和厚壁菌门等,总体组成与上述研究类似。然而,本研究发现红鳍东方鲀肠道中弯曲杆菌门占绝对优势,相对丰度在14.31%~61.85%之间,这与其他鱼类显著不同。红鳍东方鲀肠道菌这一群特点及弯曲杆菌的功能值得进一步关注,这可能对于红鳍东方鲀饲料开发及益生菌筛选具有重要参考价值。
鱼类处在一个复杂多变的生态环境中,栖息生境、温度、饵料以及肠道结构等多种因素的变化均可能导致鱼类肠道菌群组成、结构和多样性发生改变[38]。本研究发现,尽管不同养殖系统下红鳍东方鲀肠道菌群α多样性未见显著差异,然而养殖模式明显改变了红鳍东方鲀肠道优势菌群的丰度。可以看出,单养系统(A系统)红鳍东方鲀肠道中的优势菌门主要为弯曲杆菌门(61.85%)、变形菌门(29.58%)和梭杆菌门(5.71%),红鳍东方鲀-美洲帘蛤二元混养系统(B系统)红鳍东方鲀肠道中的优势菌门为弯曲杆菌门(56.08%)、螺旋菌门(24.87%)和拟杆菌门(5.43%),而红鳍东方鲀-日本对虾-美洲帘蛤三元混养系统(C系统)红鳍东方鲀肠道中优势菌门则为弯曲杆菌门(36.05%)、变形菌门(28.00%)、螺旋菌门(19.89%)和梭杆菌门(13.39%)。相比较,传统二元混养系统(E系统)中红鳍东方鲀肠道中的优势菌门为变形菌门(40.47%)、厚壁菌门(24.10%)、螺旋菌门(16.44%)和弯曲杆菌门(14.31%)。总体上看,不同系统之间比较,弯曲杆菌门的丰度差异最大,在红鳍东方鲀单养系统肠道中占比高达61.85%,其他养殖系统丰度则均低于单养系统。而在厚壁菌门的丰度上,放养小规格红鳍东方鲀的A、B和C三个系统中厚壁菌门比例均较低,但在放养大规格红鳍东方鲀的传统养殖系统(E系统)则成为第一优势菌门。基于Bray-Curtis距离的PCoA分析进一步研究发现,四种红鳍东方鲀养殖系统红鳍东方鲀肠道细菌群落在结构上存在显著差异,表明养殖模式对红鳍东方鲀的肠道菌群产生了很大影响,红鳍东方鲀二元、三元混养系统与单养系统红鳍东方鲀肠道菌群结构差异显著。PCoA分析表明,养殖系统的差异可解释总差异的50.85%,表明红鳍东方鲀池塘混养美洲帘蛤、日本对虾以及红鳍东方鲀与二者组合的差异可显著影响红鳍东方鲀的肠道菌群结构,推测这可能与美洲帘蛤、日本对虾混养后导致养殖系统环境理化因子的改变有关。通常,肠道微生物群落被认为是一种可塑性的群体,其群落结构和功能可对环境变化作出不同响应[39]。目前,环境对养殖动物肠道微生物的影响已被广泛报道,例如栖息或者养殖在不同环境中的大西洋鲑(Salmo salar)[40]、尼罗罗非鱼(Oreochromis niloticus)[41]、鳊鱼(Parabramis pekinensis)[42]、凡纳对虾(Penaeus vannamei)[43]和克氏原螯虾(Procambarus clarkii)[44]肠道菌群的结构和多样性均发生了显著的变化。与此同时,鱼体不同生长阶段也是导致红鳍东方鲀肠道菌群产生显著差异的影响因素之一。多项研究表明,在鱼类生长发育过程中肠道菌群均会发生相应的变化[45],这可以部分解释放养小规格红鳍东方鲀的单养与混养模式中红鳍东方鲀肠道菌群与传统的放养红鳍东方鲀大规格混养模式存在显著差异的原因。值得注意的是,不同系统之间的比较显示,小规格红鳍东方鲀单养系统鱼体肠道菌群与传统红鳍东方鲀二元混养距离最远,这表明二者肠道菌群结构差异最大,由此可知养殖模式与生长阶段的差异可能共同影响了红鳍东方鲀肠道菌群结构。
水产养殖中的微生态系统由各种相互影响、相互作用的微生物组成,它们的稳定对鱼类的健康成长至关重要。水体和沉积物中的微生物是水产养殖微生态系统的重要组成部分[46],对其深入的了解可以帮助我们预防水产病害的发生,并创造一个健康的养殖环境。本研究发现,不同养殖系统沉积物中细菌群落优势菌门主要由变形菌门、脱硫杆菌门、拟杆菌门、厚壁菌门和绿弯菌门等组成,但不同养殖系统沉积物的优势物种组成存在一定差异。基于Bray-Curtis距离的PCoA分析发现,养殖系统的差异可解释总差异的60.38%。从水体微生物组成上看,不同养殖系统水体的优势菌群主要由拟杆菌门、变形菌门、蓝细菌门、放线菌门和厚壁菌门等组成,但不同模式之间存在明显差异。例如,单养系统显著富集了红杆菌科和黄杆菌科的细菌,这两个科的细菌已经被广泛证实具有降解有机物和去除氮磷等营养盐的能力,经常在一些在污水处理系统中被大量发现[47-48],这是否与单养系统的氨氮浓度显著高于其他三个系统有关值得进一步研究。基于Bray-Curtis距离的PCoA分析发现,四种红鳍东方鲀养殖系统水体微生物群落在结构上存在显著差异,养殖系统的差异可解释总差异的71.19%。上述结果表明,池塘中不同生态位的红鳍东方鲀、美洲帘蛤及日本对虾的不同组合的差异显著影响养殖系统环境菌群组成与结构。通过对不同养殖系统养殖环境理化因子与水体微生物群落结构进行冗余分析发现,氨氮、硝氮、总氮与沉积物菌群群落结构显著相关,其中氨氮是影响沉积物微生物群落结构最重要的影响因子,以上环境因子解释了总差异的48.01%。盐度、氨氮、亚硝氮、硝氮、总氮与水体菌群群落结构显著相关,其中盐度、氨氮和硝氮浓度是影响水体菌群群落结构最重要的影响因子,这些环境因子解释了四种养殖系统水体微生物群落结构差异的57.72%。值得注意的是,不同系统之间比较,红鳍东方鲀单养系统水体微生物群落与各混养系统水体微生物群落距离较远,表明单养系统水体微生物群落结构与混养系统结构差异明显,推测可能是红鳍东方鲀单养系统水体氨氮、亚硝氮等环境理化指标与混养系统差异较大所导致的。上述结果进一步表明,不同混养模式显著影响红鳍东方鲀肠道及环境微生物群落结构,且养殖模式对水体微生物的影响要大于沉积物,养殖模式不同导致的环境理化因子变化可能是导致环境微生物群落结构差异重要原因。
众所周知,宿主和食物对肠道微生物群的形成起着重要作用,但环境因素也可影响肠道微生物群落[49-50]。其中,水和沉积物微生物组成是影响鱼类肠道微生物群落的生物因素之一[25, 51-53]。不过,水和沉积物微生物对鱼类肠道微生物群落的相对贡献因鱼类种类而异。例如,Sheng等[54]研究了三种杂食性鱼类的水、沉积物和肠道中的细菌群落。结果表明,海南似鱎(Toxabramis houdemeri)和(Hemiculter leucisculus)的肠道微生物群落更接近于水中的微生物群落,而莫桑比克罗非鱼(O. mossambicus)的肠道微生物群落更接近于沉积物中微生物群落,这与莫桑比克罗非鱼的底栖习性紧密相关。本研究发现,各养殖系统红鳍东方鲀肠道微生物与环境微生物之间相似性均较低,表明尽管红鳍东方鲀为底栖习性,但其肠道微生物组成受环境微生物的影响较小,而更可能主要由宿主决定。这一结果与Smith等[55]研究相似,野生三刺棘鱼(Gasterosteus aculeatus) 肠道内的微生物组成在不同种群间存在差异,宿主基因型对肠道微生物群落的选择压力超过了栖息地类型的影响。而Li等对养殖在同一环境下的鲢(Hypophthalmichthys molitrix)、草鱼(Ctenopharyngodon idella)、鳙(Aristichthys nobilis)和团头鲂(Megalobrama amblycephala)的相关研究也发现,这些鱼类在肠道菌群组成上存在显著差异主要取决于宿主的特异性[56]。此外,大西洋鲑的肠道微生物显示出生命周期对群落结构的影响,而不是地理位置的影响[57]。同时,无论是在实验室条件下还是在网箱养殖条件下,大西洋鲑肠道中都存在一组共同的核心微生物,这表明宿主生理状态在肠道微生物群落构建方面发挥着重要作用[58]。上述研究表明,在鱼类肠道菌群构建过程中,宿主、食物和环境因子影响的相对重要性在不同物种之间存在较大差异,值得进一步研究。
4 结语本研究利用16S rRNA高通量测序技术对四种红鳍东方鲀养殖系统微生物群落结构进行了比较研究。研究表明,弯曲杆菌门、变形菌门、螺旋菌门和厚壁菌门为红鳍东方鲀肠道优势菌门,弯曲杆菌门可能是红鳍东方鲀肠道的特有优势菌门。四种红鳍东方鲀养殖系统的沉积物优势细菌群落主要包括变形菌门、脱硫杆菌门、拟杆菌门、厚壁菌门和绿弯菌门等,而拟杆菌门、变形菌门、蓝细菌门、放线菌门和厚壁菌门等则是各系统水体优势菌门。不同系统红鳍东方鲀肠道微生物与环境微生物之间相似性较低,表明红鳍东方鲀肠道微生物组成受环境微生物的影响较小。不同养殖模式对红鳍东方鲀肠道、沉积物和水体微生物群落结构影响显著,且对水体微生物的影响大于沉积物微生物。养殖模式不同导致的环境理化因子变化可能是导致环境微生物群落结构差异的重要原因。
| [1] |
刘松岩, 熊彦辉. 水产养殖对水域环境的影响及其治理措施[J]. 安徽农业科学, 2007, 35(23): 7258-7259. Liu S Y, Xiong Y H. Effect of Aquaculture on water way environment and its control measure[J]. Journal of Anhui Agricultural Sciences, 2007, 35(23): 7258-7529. DOI:10.3969/j.issn.0517-6611.2007.23.104 (  0) 0) |
| [2] |
陈应华, 杨宇峰, 焦念志. 海水养殖对浮游生物群落和水环境的影响[J]. 海洋科学, 2001, 25(10): 20-22. Chen Y H, Yang Y F, Jiao N Z. Effects of mariculture on the planktonic community and water environments: A review[J]. Marine Sciences, 2001, 25(10): 20-22. (  0) 0) |
| [3] |
何悦强, 郑庆华, 温伟英, 等. 大亚湾海水网箱养殖与海洋环境相互影响研究[J]. 热带海洋, 1996, 15(2): 22-27. He Y Q, Zheng Q H, Wen W Y, et al. Study on the interaction between Marine cage culture and Marine environment in Daya Bay[J]. Tropic Oceanology, 1996, 15(2): 22-27. (  0) 0) |
| [4] |
Callahan B J, Mcmurdie P J, Rosen M J, et al. DADA2: High-resolution sample inference from Illumina amplicon data[J]. Nature Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869 (  0) 0) |
| [5] |
Sundh H, Sundell K S. Environmental impacts on fish mucosa[M]// Beck B, Peatman E. Mucosal Health in Aquaculture. Cambridge: Academic Press, 2015: 171-197.
(  0) 0) |
| [6] |
Jandhyala S M, Talukdar R, Subramanyam C, et al. Role of the normal gut microbiota[J]. World Journal of Gastroenterology, 2015, 21: 8787. DOI:10.3748/wjg.v21.i29.8787 (  0) 0) |
| [7] |
Sekirov I, Russell S L, Antunes L C M, et al. Gut microbiota in health and disease[J]. Physiological Reviews, 2010, 90: 859-904. DOI:10.1152/physrev.00045.2009 (  0) 0) |
| [8] |
Merrifield D L, Rodiles A. The fish microbiome and its interactions with mucosal tissues[M]// Beck B, Peatman E. Mucosal Health in Aquaculture. Cambridge: Academic Press, 2015: 273-295.
(  0) 0) |
| [9] |
Llewellyn M S, Boutin S, Hoseinifar S H, et al. Teleost microbiomes: The state of the art in their characterization, manipulation and importance in aquaculture and fisheries[J]. Frontiers in Microbiology, 2014, 5: 207. (  0) 0) |
| [10] |
Egerton S, Culloty S, Whooley J, et al. The gut microbiota of marine fish[J]. Frontiers in Microbiology, 2018, 9: 873. DOI:10.3389/fmicb.2018.00873 (  0) 0) |
| [11] |
Flint H J, Scott K P, Louis P, et al. The role of the gut microbiota in nutrition and health[J]. Nature Reviews Gastroenterology Hepatology, 2012, 9: 577. DOI:10.1038/nrgastro.2012.156 (  0) 0) |
| [12] |
Lee W J, Hase K. Gut microbiota-generated metabolites in animal health and disease[J]. Nature Chemical Biology, 2014, 10: 416-424. DOI:10.1038/nchembio.1535 (  0) 0) |
| [13] |
Read M N, Holmes A J. Towards an integrative understanding of diet-host-gut microbiome interactions[J]. Frontiers in Immunology, 2017, 8: 538. DOI:10.3389/fimmu.2017.00538 (  0) 0) |
| [14] |
Strand M, Jing Y, Sandve S R, et al. Transkingdom network analysis provides insight into hostmicrobiome interactions in Atlantic salmon[J]. Computational and Structural Biotechnology Journal, 2021, 19: 1028-1034. DOI:10.1016/j.csbj.2021.01.038 (  0) 0) |
| [15] |
徐静静, 冯广朋, 张涛, 等. 基于高通量测序的长江口中华绒螯蟹雌蟹食性分析[J]. 水产科学, 2023, 42(1): 1-10. Xu J J, Feng G P, Zhang T, et al. Diet composition and feeding habits of female Chinese mitten crab Eriocheir sinensis in the Yangtze River Estuary based on high-throughput sequencing[J]. Fisheries Science, 2023, 42(1): 1-10. DOI:10.16378/j.cnki.1003-1111.20164 (  0) 0) |
| [16] |
Grisez L, Reyniers J, Verdonck L, et al. Dominant intestinal microflora of sea bream and sea bass larvae, from two hatcheries, during larval development[J]. Aquaculture, 1997, 155: 387-399. DOI:10.1016/S0044-8486(97)00113-0 (  0) 0) |
| [17] |
Wang A R, Ran C, Ringø E, et al. Progress in fish gastrointestinal microbiota research[J]. Reviews in Aquaculture, 2018, 10: 626-640. DOI:10.1111/raq.12191 (  0) 0) |
| [18] |
Giatsis C, Sipkema D, Smidt H, et al. The impact of rearing environment on the development of gut microbiota in tilapia larvae[J]. Scientific Reports, 2015, 5: 18206. DOI:10.1038/srep18206 (  0) 0) |
| [19] |
Al-Harbi A H, Uddi M N. Seasonal variation in the intestinal bacterial flora of hybrid tilapia (Oreochromis niloticus×Oreochromis aureus) cultured in earthen ponds in Saudi Arabia[J]. Aquaculture, 2004, 229: 37-44. DOI:10.1016/S0044-8486(03)00388-0 (  0) 0) |
| [20] |
Nayak S K. Role of gastrointestinal microbiota in fish[J]. Aquaculture Research, 2010, 41: 1553-1573. DOI:10.1111/j.1365-2109.2010.02546.x (  0) 0) |
| [21] |
Butt R L, Volkoff H. Gut microbiota and energy homeostasis in fish[J]. Frontiers in Endocrinology, 2019, 10: 9. DOI:10.3389/fendo.2019.00009 (  0) 0) |
| [22] |
Fu J, Yang D, Jin M, et al. Aquatic animals promote antibiotic resistance gene dissemination in water via conjugation: Role of different regions within the zebra fish intestinal tract, and impact on fish intestinal microbiota[J]. Molecular Ecology, 2017, 26: 5318-5333. DOI:10.1111/mec.14255 (  0) 0) |
| [23] |
Huyben D, Sun L, Moccia R, et al. Dietary live yeast and increased water temperature influence the gut microbiota of rainbow trout[J]. Journal of Applied Microbiology, 2018, 124: 1377-1392. DOI:10.1111/jam.13738 (  0) 0) |
| [24] |
Mekuchi M, Asakura T, Sakata K, et al. Intestinal microbiota composition is altered according to nutritional biorhythms in the leopard coral grouper (Plectropomus leopardus)[J]. PLoS One, 2018, 13(6): e0197256. DOI:10.1371/journal.pone.0197256 (  0) 0) |
| [25] |
Sylvain F E, 'Cheaib B, Llewellyn M, et al. pH drop impacts differentially skin and gut microbiota of the Amazonian fish tambaqui (Colossoma macropomum)[J]. Scientific Reports, 2016, 6: 1-10. DOI:10.1038/s41598-016-0001-8 (  0) 0) |
| [26] |
Tigchelaar M, Leape J, Micheli F, et al. The vital roles of blue foods in the global food system[J]. Global Food Security-Agriculture Policy Economics and Environment, 2022, 33: 100637. (  0) 0) |
| [27] |
Lin G, Lin X. Bait input altered microbial community structure and increased greenhouse gases production in coastal wetland sediment[J]. Water Research, 2012, 218: 118520. (  0) 0) |
| [28] |
Nealson K H. Sediment bacteria: Who's there, what are they doing, and what's new?[J]. Annual Review of Earth Planet Sciences, 1997, 25: 403-434. DOI:10.1146/annurev.earth.25.1.403 (  0) 0) |
| [29] |
Xu M, Xu R Z, Shen X X, et al. The response of sediment microbial communities to temporal and site-specific variations of pollution in interconnected aquaculture pond and ditch systems[J]. Science of the Total Environment, 2022, 806: 150498. DOI:10.1016/j.scitotenv.2021.150498 (  0) 0) |
| [30] |
泮淼军, 王明阳, 王龙斌, 等. 海水池塘跑道养殖系统中鱼类肠道及养殖环境的细菌群落研究[J]. 中国海洋大学学报(自然科学版), 2023, 53(5): 47-60. Pan M J, Wang M Y, Wang L B, et al. Studies on bacterial community in fish gut and environment of a marine in-pond raceway pond system[J]. Periodical of Ocean University of China, 2023, 53(5): 47-60. DOI:10.16441/j.cnki.hdxb.20220195 (  0) 0) |
| [31] |
Bolyen E, Rideout J R, Dillon M R, et al. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2[J]. Nature Biotechnology, 2019, 37(8): 852-857. DOI:10.1038/s41587-019-0209-9 (  0) 0) |
| [32] |
Oksanen J, Blanchet F G, Kindt R, et al. Vegan: Community ecology package[CP/OL]. (2020-11-28)[2023-11-13]. https://cran.r-project.org/web/packages/vegan/index.html.
(  0) 0) |
| [33] |
Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology, 2011, 12(6): 60. DOI:10.1186/gb-2011-12-6-r60 (  0) 0) |
| [34] |
Clarke K R. Non-parametric multivariate analyses of changes in community structure[J]. Australian Journal of Ecology, 1993, 18(1): 117-143. (  0) 0) |
| [35] |
Ghanbari M, Konrad J D, Wolfgang K. A new view of the fish gut microbiome: Advances from next-generation sequencing[J]. Aquaculture, 2015, 448: 464-475. (  0) 0) |
| [36] |
孟晓林, 李文均, 聂国兴. 鱼类肠道菌群影响因子研究进展[J]. 水产学报, 2019, 43(1): 143-155. Meng X L, Li W J, Nie G X. Effect of different factors on the fish intestinal microbiota[J]. Journal of Fisheries of China, 2019, 43(1): 143-155. (  0) 0) |
| [37] |
翟万营, 郭安宁. 鱼类肠道微生物研究进展[J]. 河南水产, 2016(4): 18-21. Zhai W Y, Guo A N. Research progress on the fish intestinal microbiota[J]. Henan Fisheries, 2016(4): 18-21. (  0) 0) |
| [38] |
Givens C E, Ransom B, Bano N, et al. Comparison of the gut microbiomes of 12 bony fish and 3 sharkspecies[J]. Marine Ecology Progress, 2015, 518(7): 209-223. (  0) 0) |
| [39] |
Candela M, Biagi E, Maccaferri S, et al. Intestinal microbiota is a plastic factor responding to environmental changes[J]. Trends in Microbiology, 2012, 20(8): 385-391. (  0) 0) |
| [40] |
Lavoie C, Courcelle M, Redivo B, et al. Structural and compositional mismatch between captive and wild Atlantic salmon (Salmo salar) parrs' gut microbiota highlights the relevance of integrating molecular ecology for management and conservation methods[J]. Evolutionary Applications, 2018, 11(9): 1671-1685. (  0) 0) |
| [41] |
Bereded N K, Abebe G B, Fanta S W, et al. The impact of sampling season and catching site (wild and aquaculture) on gut microbiota composition and diversity of Nile tilapia (Oreochromis niloticus)[J]. Biology, 2021, 10(3): 180. (  0) 0) |
| [42] |
Gu H L, Feng Y M, Zhang Y, et al. Differential study of the Parabramis pekinensis intestinal microbiota according to different habitats and different parts of the intestine[J]. Annals of Microbiology, 2021, 71(1): 1-11. (  0) 0) |
| [43] |
Landsman A, St-Pierre B, Rosales-Leija M, et al. Impact of aquaculture practices on intestinal bacterial profiles of Pacific whiteleg shrimp Litopenaeus vannamei[J]. Microorganisms, 2019, 7(4): 93. (  0) 0) |
| [44] |
Wei D D, Xing C G, Hou D W, et al. Distinct bacterial communities in the environmental water, sediment and intestine between two crayfish-plant coculture ecosystems[J]. Applied Microbiology and Biotechnology, 2021, 105(12): 5087-5101. (  0) 0) |
| [45] |
Yan Q, Gast C J, Yu Y. Bacterial community assembly and turnover within the intestines of developing zebrafish[J]. PLoS One, 2012, 7(1): e30603. (  0) 0) |
| [46] |
Sakami T, Fujioka Y, Shimoda T. Comparison of microbial community structures in intensive and extensive shrimp culture ponds and a mangrove area in Thailand[J]. Fisheries Science, 2008, 74(4): 889-898. (  0) 0) |
| [47] |
Cui Y W, Huang J L, Alam F. Fast granulation of halophilic activated sludge treating low-strength organic saline wastewater via addition of divalent cations[J]. Chemosphere, 2021, 264(1): 128396. (  0) 0) |
| [48] |
Gui X W, Li Z L, Wang Z J. Kitchen waste hydrolysate enhances sewage treatment efficiency with different biological process compared with glucose[J]. Bioresource Technology, 2021, 341: 125904. (  0) 0) |
| [49] |
Li X, Zhou L, Yu Y, et al. Composition of gut microbiota in the gibel carp (Carassius auratus gibelio) varies with host development[J]. Microbial Ecology, 2017, 74(1): 239-249. (  0) 0) |
| [50] |
Talwar C, Nagar S, Lal R, et al. Fish gut microbiome: Current approaches and future perspectives[J]. Indian Journal of Microbiology, 2018, 58(4): 397-414. (  0) 0) |
| [51] |
Zeng A, Tan K, Gong P, et al. Correlation of microbiota in the gut of fish species and water[J]. 3 Biotech, 2020, 10(11): 472. (  0) 0) |
| [52] |
Vestrum R I, Attramadal K J K, Winge P, et al. Rearing water treatment induces microbial selection influencing the microbiota and pathogen associated transcripts of Cod (Gadus morhua) Larvae[J]. Frontiers in Microbiology, 2018, 9: 851. (  0) 0) |
| [53] |
Vestrum R I, Attramadal K J K, Vadstein O, et al. Bacterial community assembly in Atlantic cod larvae (Gadus morhua): Contributions of ecological processes and metacommunity structure[J]. FEMS Microbiology Ecology, 2020, 96(9): fiaa163. (  0) 0) |
| [54] |
Bi S, Lai H, Guo D, et al. The characteristics of intestinal bacterial community in three omnivorous fishes and their interaction with microbiota from habitats[J]. Microorganisms, 2021, 9(10): 2125. (  0) 0) |
| [55] |
Smith C C R, Snowberg L K, Caporaso J G, et al. Dietary input of microbes and host genetic variation shape among-population differences in stickleback gut microbiota[J]. ISME Journal, 2015, 9(11): 2515-2526. (  0) 0) |
| [56] |
Li X M, Yu Y H, Feng W S, et al. Host species as a strong determinant of the intestinal microbiota of fish larvae[J]. Journal of Microbiology, 2012, 50(1): 29-37. (  0) 0) |
| [57] |
Llewellyn M S, Mcginnity P, Dionne M, et al. The biogeography of the Atlantic salmon (Salmo salar) gut microbiome[J]. ISME Journal, 2016, 10(5): 1280-1284. (  0) 0) |
| [58] |
Dehler C E, Secombes C J, Martin S A M. Environmental and physiological factors shape the gut microbiota of Atlantic salmon parr (Salmo salar L.[J]. Aquaculture, 2017, 467: 149-157. (  0) 0) |
2. Tangshan Haidu Seafood Company Limited, Tangshan 063000, China
 2025, Vol. 55
2025, Vol. 55


