2. 青岛海洋科技中心海洋生态与环境科学功能实验室,山东 青岛 266237;
3. 中国海洋大学进化与海洋生物多样性研究所,山东 青岛 266003
栖盐田菌属(Salinicola)是2007年建立的细菌属[1],隶属于γ-变形菌纲大洋螺菌目盐单胞菌科,目前该属有14种,其中,关联栖盐田菌(Salinicola socius)是该属的模式种。该属细菌为中度嗜盐的需氧型或兼性厌氧型革兰氏阴性菌、形态为棒状且具有运动性,常分离自受石油污染、高盐等极端环境[2]。据报道,该属菌株已从海水、盐矿、太阳盐沼、红树林根际、深海沉积物等环境中分离获得[3-8]。比如,在盐沼植物体相关的环境中,先后发现了包括葡萄牙栖盐田菌(Salinicola lusitanus)和2020年分离获得的菌种Salinicola corii在内的5种栖盐田菌属菌株[7]。通过对Salinicola socius SMB35T和Salinicola sp. MH3R3-1的基因组草图进行分析,揭示了栖盐田菌属菌株耐受渗透胁迫和适应高盐环境的能力[9]。2023年有研究者通过流式细胞术分析了嗜盐菌Salinicola halophilus S28在不同NaCl浓度下的生理状态,结果显示该菌株在NaCl浓度高达24%时仍具有良好生理反应,为其较高的抗盐胁迫能力提供了有力的证据[10];相关研究还发现栖盐田菌属菌株W-Y11能够乳化原油,不仅可用于微生物采油,提高采油率,还可用于石油污染的治理和修复,具有很好的应用前景[11]。此外,Salinicola tamaricis F01被发现对锰、镍、铅、亚铁离子、铜离子等具有较高的耐受性[12]。由此可见,该属菌株具有较强的环境适应能力,也具有较高的研究意义和潜在的应用价值。目前,该属菌株大多分离自动植物内共生体、盐矿、珊瑚、红树林根际等,且对其功能的研究集中于耐盐、耐碱、乳化原油、抵抗重金属等方面,尚未见来自于极端深海热液区菌株的报道,且其对不同海洋或陆地环境的适应机制尚不清楚。
葡萄牙栖盐田菌CR50T于2019年由Catia Fidalgo首次报道[7],它分离自葡萄牙一株马齿苋(Halimione portulacoides)组织,属于兼性厌氧型菌株,温度生长范围为15~45 ℃,pH耐受范围为4.0~10.0,NaCl浓度耐受性范围为0~30%(W/V),具有过氧化氢酶和氧化酶活性,能够水解吐温20,产生吲哚-3-乙酸,但不产生H2S,主要脂肪酸为C16∶0,summed feature 8(C18∶1 ω7c/C18∶1 ω6c和cyclo-C19∶0 ω8c),主要呼吸醌类型为泛醌UQ-9。目前,该种只有菌株的分离鉴定方面的相关报道,并未有从比较基因组学角度对其代谢和环境适应机制进行深入研究。
深海热液区是地球上最极端和最活跃的环境之一,高温、低氧且富含大量重金属和有毒气体(H2S、NH4+和甲烷等)。由于热液区独特的地质构造和复杂的物理化学特征,导致硫和/或氢氧化、化能自养、嗜热细菌、古菌等在此广泛分布[13]。冲绳海槽位于西太平洋中北部,东起琉球群岛,西达东海大陆坡折处,北起日本九州西南诸岛,南至台湾本岛东北角,是中国与日本之间大陆架的天然分界线,是菲律宾海板块向欧亚板块俯冲形成的一个半深海盆地,在地质构造、地形地貌、沉积物性质等特征上和中国大陆连为一体,属于中国大陆自然延伸的一部分[14]。自1989年Halbach等首次发现位于冲绳海槽的JADE热液区以来,对该海域中热液区的研究日益增多,人们对其间微生物的代谢机理、生存极限、元素地球化学循环作用等方面的认识不断加深。尽管目前已对冲绳海槽热液区地质化学环境、热液流体、理化性质与其微生物多样性进行了许多研究,但对其微生物群落及其功能代谢机制的了解仍十分有限[15-18]。
本研究自冲绳海槽热液区分离获得了一株葡萄牙栖盐田菌LLJ914,为了了解其在极端环境的适应机制及其与同种陆源菌株CR50T的遗传特征及代谢潜力的差异,对菌株LLJ914进行了全基因组测序,并通过与其亲缘关系相近的菌株进行比较基因组分析,揭示该菌株与重金属抗性和耐盐、耐碱等特征相关的功能基因、代谢途径及环境适应特征,初步探究其对富含硫化物的热液环境的适应机制,促进对深海细菌适应极端环境的认识。
1 材料与方法 1.1 样品采集本研究中葡萄牙栖盐田菌LLJ914,分离自2016年5—6月的HOBAB4航次冲绳海槽第四与那国海丘热液区W10站点(122.70°E,24.86°N)所采集的1 235 m深处的海水样品。采集的海水样品依次通过3.00和0.22 μm的滤膜,将滤膜收集至冻存管并加入15%甘油保藏液,于-80 ℃低温保藏。菌株LLJ914已保藏至中国海洋大学海洋微生物菌种保藏中心(Marine Culture Collection,Ocean University of China,MCCO),保藏编号是MCCO11505。
1.2 培养基2216E培养基:称取1.00 g的酵母膏,5.00 g的蛋白胨,0.01 g的磷酸铁,溶于陈海水中并定容至1 L,调节pH至7.6。固体培养基中添加2%(W/V)琼脂粉。121 ℃灭菌20 min。
MMJS基础培养基配方见表 1[19],在此基础上加入2%琼脂即为固体培养基,121 ℃高温高压灭菌20 min。
|
|
表 1 MMJS基础培养基配方 Table 1 The ingredients of MMJS basic medium |
将冻存管保藏的0.22 μm海水滤膜剪取1/2,用0.85%的无菌生理盐水(0.85% NaCl,W/V)充分震荡将菌体冲洗下来,取5 mL接种于50 mL的MMJS培养基中,28 ℃下进行富集培养,且5~7 d转接一次,将转接第6次以后的培养物稀释为原本的1/100, 涂布于MMJS琼脂培养基上,28 ℃避光生长48 h,观察菌落不同的形态,挑取生长状态良好的菌落于2216E琼脂培养基上,使用平板划线培养法进行3次分离纯化后,用15%甘油保藏于-80 ℃冰箱。
1.4 菌株鉴定采用煮沸法[20]提取已分离纯化菌株LLJ914的DNA,并使用引物B8F(5′-AGAGTTTGATCCTGGCTCAG-3′)和B1510R(5′-GGTTACCTTGTTACGACTT-3′)进行16S rRNA基因扩增,PCR产物交由北京六合华大基因科技有限公司进行Sanger测序,使用Chromas对测序结果进行分析,并提交至EzBiocloud(https://www.ezbiocloud.net/)数据库[21]中进行16S rRNA基因序列相似性比对,即可初步判断菌株的分类地位。同时,选择与菌株LLJ914相似度较高菌株的16S rRNA基因序列, 采用MEGA 11.0[22]进行系统发育分析,使用MUSCLE[23]进行多序列比对,将所有序列截齐,然后使用邻接法(Neighbor-Joining,NJ)构建系统发育树,Bootstrap为1 000,从而得到分离菌株的进化关系。
1.5 全基因组测定与组装收集对数生长期的菌株LLJ914,经过4 ℃,12 000 r/min离心10 min,生理盐水重悬三次,将收集的菌体放入液氮速冻。菌株基因组交由武汉华大基因科技有限公司基于Illumina Hiseq和PacBio平台进行测序。对基因组DNA样品进行检测,样品合格后构建文库:首先把DNA样品随机打断,收集所需长度的DNA片段,然后通过3′端加碱基“A”,DNA片段能与3′端带有“T”碱基的特殊接头连接,最后,用合格的文库进行cluster制备和测序。利用Unicycler软件[24]将除杂后的clean reads进行拼接,采用Canu v1.1[25]组装全基因组,使用Pilon v1.16[26]校正测序错误位点,最终获得菌株全基因组完成图。菌株LLJ914的全基因组序列已上传至国家组学数据百科全书(National Omics Data Encyclopedia,NODE),项目编号为OEP005193。
1.6 系统发育与基因组相似性分析为了进一步确定菌株LLJ914的进化地位,从NCBI数据库(https://www.ncbi.nlm.nih.gov/)下载同属以及同科不同属菌株的基因组蛋白注释文件[27],然后使用Orthofinder获取单拷贝基因[28],MAFFT进行多重序列比对[29],使用TrimAl对比对后的序列进行修剪[30],去掉比对质量不佳的区域,用fastaConcat[31]连接所有核心基因后,使用iqtree 2.0软件构建系统发育树[32],自展值是1 000次重复抽样建树的结果,最后,基于网站iTOL(https://itol.embl.de/)进行系统发育树美化[33]。
通过平均核苷酸一致性(Average nucleotide identity,ANI)[34]和平均氨基酸一致性(Average amino acid identity,AAI)[35]的计算,研究基因组之间的进化距离,判断两个基因组在核苷酸和氨基酸水平上的亲缘关系。将菌株LLJ914及参考菌株的基因组序列分别上传至网站https://jspecies.ribohost.com/jspeciesws/和http://enve-omics.ce.gatech.edu/aai/,计算不同菌株基因组之间的ANI值和AAI值。
1.7 基因功能预测对菌株LLJ914及其参考菌株的基因组进行功能注释与分析。将该基因组序列上传至RAST(Rapid Annotation using Subsystem Technology server,https://rast.nmpdr.org/)数据库[36]进行注释,预测基因数目、种类及相应的蛋白功能等;将菌株LLJ914及其参考菌株基因组的蛋白序列上传至KAAS-KEGG Automatic Annotation Server(http://www.genome.jp/tools/kaas/)进行代谢通路的分类和注释[37],并利用KEGG-decoder[38]计算代谢通路的完整度。使用eggNOG-mapper(http://eggnog-mapper.embl.de/)对直系同源蛋白簇(COG,Clusters of Orthologous Groups)进行注释和分类[39];同时,将蛋白序列提交至碳水化合物活性酶(CAZy,Carbohydrate-Active Enzymes,http://www.cazy.org/)数据库[40]进行注释,分析菌株基因组中碳水化合物活性酶的种类及数目。使用Linux版本的BLAST(Basic Local Alignment Search Tool)工具[41]将菌株LLJ914和CR50T的蛋白氨基酸序列进行两两比对,筛选菌株LLJ914中的特有基因。
2 结果与分析 2.1 葡萄牙栖盐田菌LLJ914的基因组分析葡萄牙栖盐田菌LJJ914的基因组组装后获得1条contig序列,完整度达到98.92%,基因组大小为4 781 556 bp,GC含量为64.0%,共包含4 311个基因,编码4 229个蛋白,其中69个tRNA,12个rRNA基因(4个5S rRNA,4个16S rRNA,4个23S rRNA)(见表 2)。运用CGView Server绘出菌株LLJ914的基因组圈图(见图 1)。
|
|
表 2 葡萄牙栖盐田菌LLJ914基因组的一般特征 Table 2 General feature of genome of Salinicola lusitanus LLJ914 |
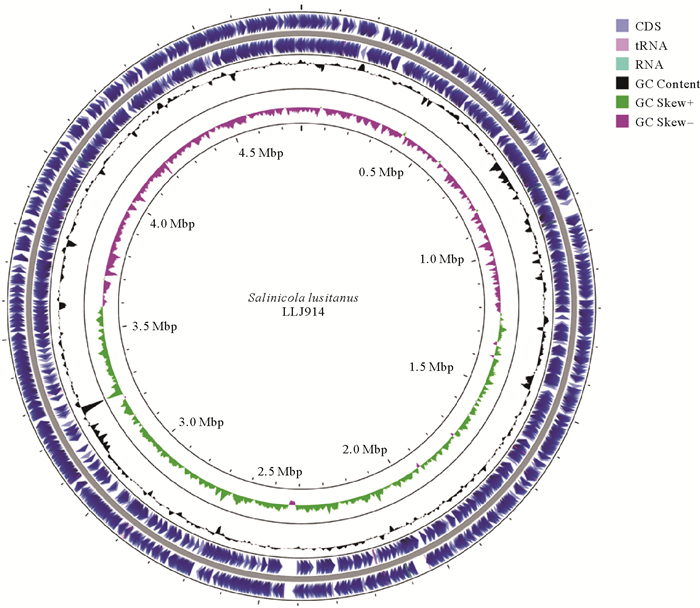
|
图 1 葡萄牙栖盐田菌LLJ914的全基因组环形图 Fig. 1 The genome map of Salinicola lusitanus LLJ914 |
从组装的全基因组数据中提取菌株LLJ914的16S rRNA基因序列,序列长度为1 535 bp,并将此序列上传至EzBioCloud网站进行比对,经过比对发现该菌株属于栖盐田菌属,与葡萄牙栖盐田菌CR50T的16S rRNA基因序列相似度为99.93%,初步判断二者为分离自不同环境的葡萄牙栖盐田菌的同种不同株(判断同种的阈值为16S rRNA基因相似性>98.65%)[42]。从NCBI基因组数据库中选取13个栖盐田菌属标准种的基因组与该菌株进行比较基因组分析(见表 3),发现14个栖盐田菌属细菌的基因组大小介于3.62~4.78 Mbp之间,G+C含量介于60.6%~65.8%之间。
|
|
表 3 葡萄牙栖盐田菌LLJ914及参考菌株的基因组特征 Table 3 Genome characteristics ofSalinicola lusitanus LLJ914 and reference strains |
通过EzBioCloud和NCBI数据库查找参考菌株的16S rRNA基因序列,基于邻接法(Neighbor-joining,NJ),对菌株LLJ914的16S rRNA基因构建系统发育树,将同科不同属的碱蓬拉森氏单胞菌(Larsenimonas suaedae)ST307T设为外群(见图 2A)。结果显示菌株LLJ914与栖盐田菌属聚为一簇,与葡萄牙栖盐田菌CR50T进化距离最近。

|
( A:基于16S rRNA基因;B:基于单拷贝核心基因。图中加粗字体为本研究分离获得的菌株。A: Based on 16S rRNA gene; B: Based on single-copy core genes. The bold fonts in the figure are the isolates obtained in this study. ) 图 2 葡萄牙栖盐田菌LLJ914及其相近菌株的系统发育进化树 Fig. 2 Phylogenetic tree of Salinicola lusitanus LLJ914 and its related strains |
将菌株LLJ914的基因序列与NCBI数据库中下载的参考基因组进行比对,基于最大似然法(Maximum-likelihood,ML),利用单拷贝核心基因构建系统发育树,将同科不同属的盐拉森氏单胞菌(Larsenimonas salina)CCM8464T设为外群,结果与基于16S rRNA基因序列构建的系统发育树一致(见图 2B)。
将菌株LLJ914与13个同属菌株的基因组进行比较基因组分析,发现ANI分析结果与基于16S rRNA基因序列和核心基因的系统发育分析结果一致,其与亲缘关系最近的菌株CR50T的ANI值最高,为98.28%(判断新种的阈值为ANI值<95%)[42],且AAI值为98.76%(判断新种的阈值为AAI值<90%)[43],表明菌株LLJ914为葡萄牙栖盐田菌。除此之外,菌株LLJ914与聚为一支的Salinicola acroporae S4-41、Salinicola acroporae LMG 28587和Salinicola corii L3的ANI值分别为93.52%、93.54%和88.43%。结合16S rRNA基因和单拷贝核心基因系统发育树的结果,发现以上4株相较其余参考菌株而言与LLJ914亲缘关系较近(见图 3)。
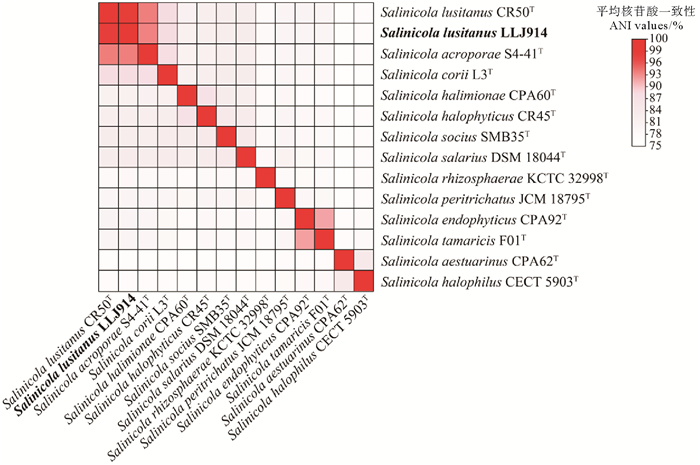
|
( 图中加粗字体为本研究分离获得的菌株。The bold fonts in the figure are the isolates obtained in this study. ) 图 3 葡萄牙栖盐田菌LLJ914以及参考基因组的ANI热图 Fig. 3 ANI heat map of Salinicola lusitanus LLJ914 and reference strains |
菌株LLJ914的直系同源蛋白簇注释到3 960个基因(占总基因组的91.86%),分布在22种类型中。注释结果显示与氨基酸运输和代谢(Amino acid transport and metabolism,E;10.9%)、转录(Transcription,K;8.2%)、碳水化合物运输和代谢(Carbohydrate transport and metabolism,G;7.0%)、能源生产和转换(Energy production and conversion,C;6.5%)、细胞壁/膜/被膜生物发生(Cell wall/membrane/envelope biogenesi,M;5.7%)、无机离子运输和代谢(Inorganic ion transport and metabolism,P;5.9%)相关的基因占比较高(见图 4A)。
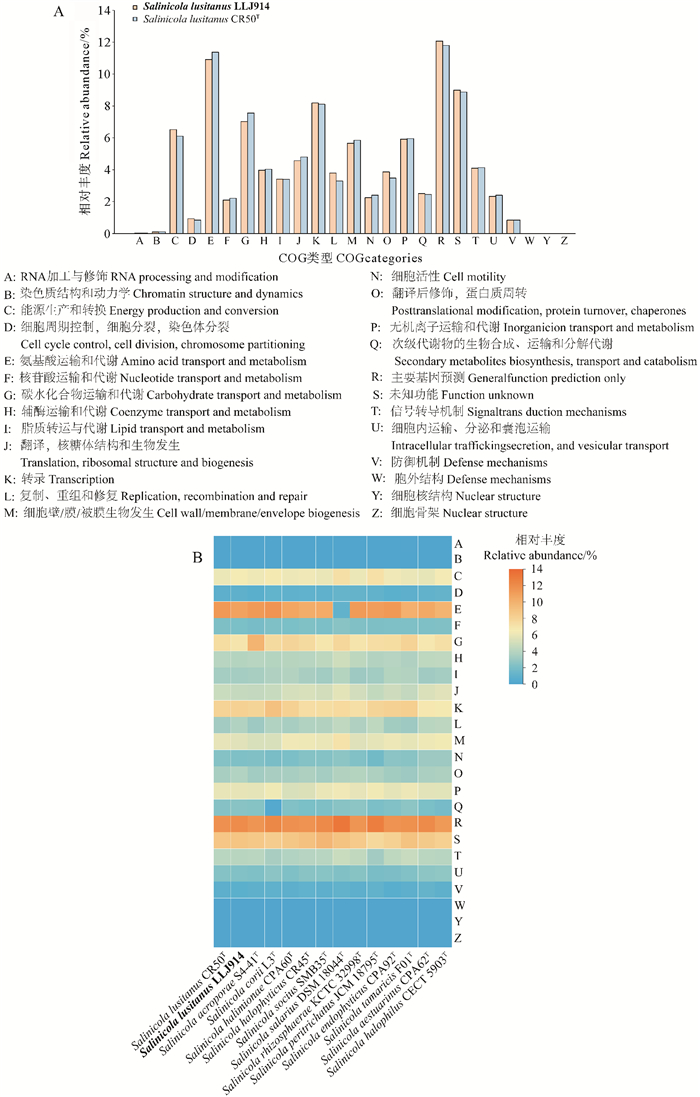
|
( A:菌株LLJ914和CR50T基因组中COG注释基因的比例;B:菌株LLJ914及参考菌株的COG注释基因相对丰度的热图。图中加粗字体为本研究分离获得的菌株。A: The proportion of COG-annotated genes in the genome of strains LLJ914 and CR50T; B: Heat map of the relative abundances of COG-annotated genes in strain LLJ914 and reference strain. The bold fonts in the figure are the isolates obtained in this study. ) 图 4 菌株LLJ914基因组中COG注释基因的比例 Fig. 4 Proportion of COG-annotated genes in the genome of strain LLJ914 |
从菌株LLJ914与同属其他菌株的COG注释结果可见,栖盐田菌属菌株基因组中各类别基因丰度并无显著差异(见图 4B)。菌株LLJ914注释结果与菌株CR50T注释到的3 798个基因相比,菌株LLJ914在能源生产和转换、细胞周期控制、细胞分裂、染色体分裂、转录、复制重组和修复、翻译后修饰蛋白周转、次级代谢物生物合成、运输和分解代谢方面比菌株CR50T略高,在其他方面略少。
2.3.2 葡萄牙栖盐田菌LLJ914及参考菌株基因组的KEGG注释结果菌株LLJ914及其参考菌株的基因组KEGG注释结果显示,菌株LLJ914与参考菌株的部分代谢通路存在一致性。栖盐田菌属菌株大多具有与呼吸代谢相关的F型三磷酸腺苷酶(F-type ATPase)、细胞色素o泛醌氧化酶(Cytochrome o ubiquinol oxidase)、NADH-醌氧化还原酶(NADH-quinone oxidoreductase)、CP裂解酶的编码基因phnJ以及核黄素生物合成(Riboflavin biosynthesis)和双向多磷酸盐生物合成(Bidirectional polyphosphate)途径(见图 5)。
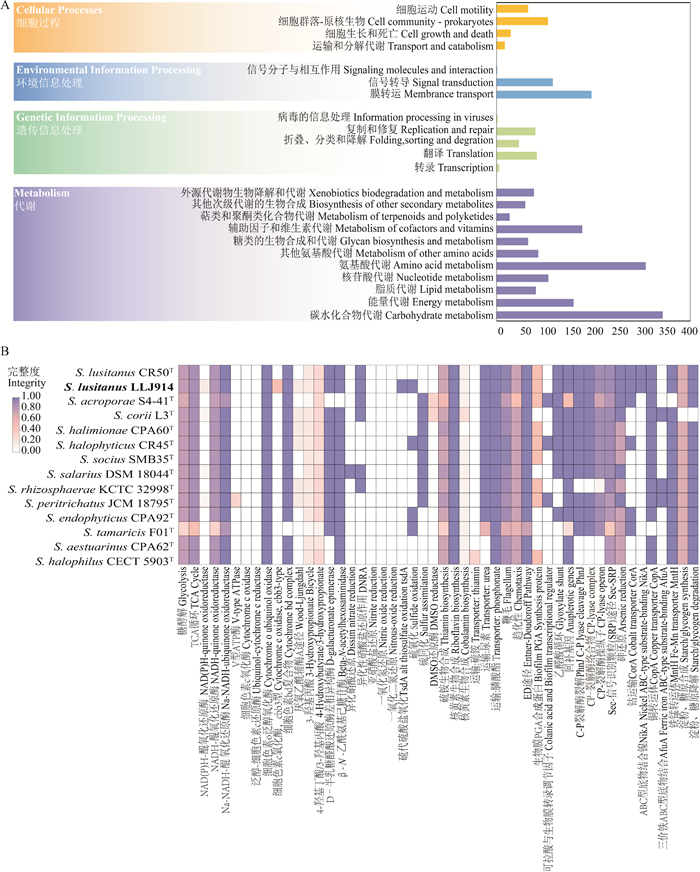
|
( A:菌株LLJ914基因组中KEGG注释基因的数目;B:菌株LLJ914及参考菌株注释的部分KEGG通路完整度。图中加粗字体为本研究分离获得的菌株。A: Numbers of genes involved in KEGG pathways in the genome of strain LLJ914; B: Completeness of partial KEGG pathways in strain LLJ914 and reference strains. The bold fonts in the figure are the isolates obtained in this study. ) 图 5 葡萄牙栖盐田菌LLJ914及其参考菌株的KEGG注释结果 Fig. 5 Results of KEGG annotation of strain LLJ914 and reference strains |
葡萄牙栖盐田菌株LLJ914与CR50T在呼吸代谢方面均拥有Na-NADH-醌氧化还原酶(Na-NADH-ubiquinone oxidoreductase)、细胞色素bd复合物(Cytochrome bd complex)。在碳水化合物代谢方面,两株菌均具有完整的乙醛酸循环(Glyoxylate shunt)、ED途径(Entner-Doudoroff pathway)、糖代谢相关的β-N-外切糖苷酶(β-N-acetylhexosaminidase)和D-半乳糖醛酸还原酶差相异构酶(D-galacturonate epimerase)、糖原/淀粉的合成和降解相关基因。在硫代谢通路中,二者均具有参与同化硫酸盐还原作用的腺苷三磷酸硫酸激酶(Adenylylsulfate kinase CysC)、双功能酶(Bifunctional enzyme CysN/CysC)、硫酸腺苷酰转移酶(Sulfate adenylyltransferase CysD)、3′/2′, 5′-双磷酸核苷酸酶(3′/2′, 5′-bisphosphate nucleotidase CysQ)和磷酸腺苷磷硫酸还原酶(Phosphoadenosine phosphosulfate reductase CysH)等,参与硫酸盐/硫代硫酸盐运输的结合蛋白CysP、CysU和CysW,以及结合ATP的CysA蛋白等。在氮代谢通路中,两株葡萄牙栖盐田菌均拥有同化硝酸盐还原途径中铁氧还蛋白-亚硝酸盐还原酶(Ferredoxin-nitrite reductase NirA)和异化硝酸盐还原途径中亚硝酸盐还原酶(Nitrite reductase NADH,NirB/NirD)的编码基因。在重金属抗性方面,葡萄牙栖盐田菌具有抵抗重金属的ABC型镍结合蛋白(Nickel ABC-type substrate-binding NikA)、钴转运蛋白CorA(Cobalt transporter CorA)、砷还原(Arsenic reduction)、铜转运体CopA(Copper transporter CopA)以及铁锰转运体MntH(Fe-Mn transporter MntH)相关编码基因。
根据注释结果发现,来自两种不同生活环境的葡萄牙栖盐田菌的代谢特征具有较高的相似性,但也存在一定的差异。分离自冲绳海槽热液区的菌株LLJ914额外拥有参与氮代谢的铁氧还蛋白-亚硝酸盐还原酶(Ferredoxin-nitrite reductase NirA)编码基因、参与硫代谢的磺酸盐转运系统ATP结合蛋白(Sulfonate transport system ATP-binding protein, SsuB)和硫化醌氧化还原酶(Sulfide quinone oxidoreductase, Sqr),缺乏菌株CR50T基因组中存在的亚硫酸盐还原酶血红蛋白β组分(Sulfite reductase NADPH hemoprotein β-component)。此外,基于菌株LLJ914基因组与CR50T基因组Blast比对结果可知,菌株LLJ914特有与重金属抗性相关的蓝铜氧化酶CueO(Blue copper oxidase, CueO)、锌转运蛋白ZupT(Zinc transporter, ZupT)、免疫防御系统相关的CRISPR(Clustered regularly interspaced short palindromic repeats)相关蛋白以及有氧呼吸链相关的cbb3型细胞色素c氧化酶(Cytochrome c oxidase, cbb3-type)编码基因。
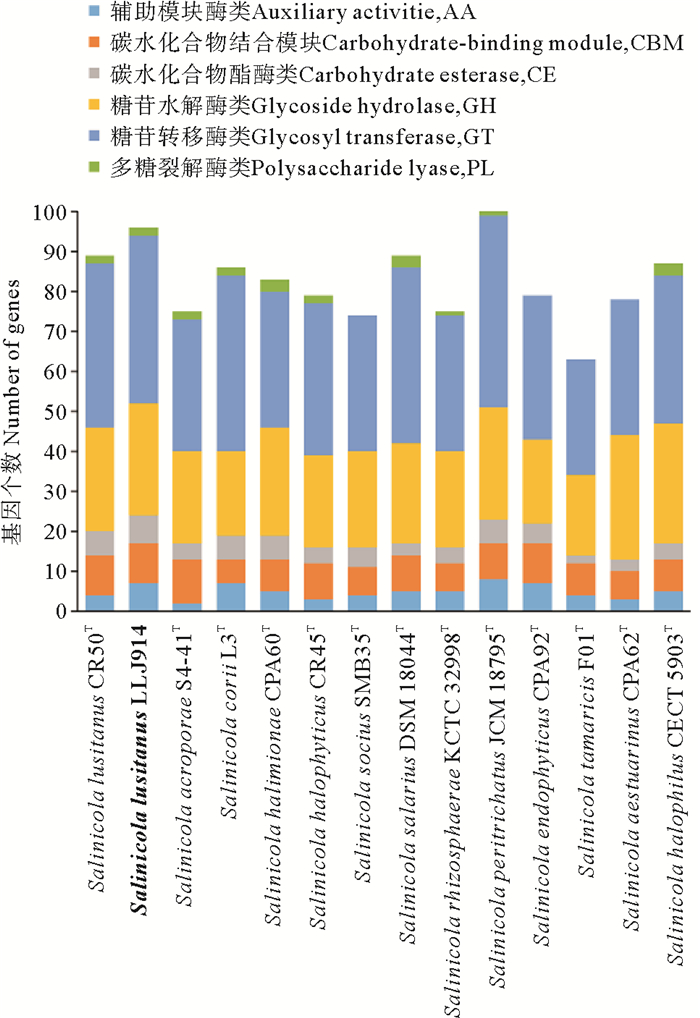
2.3.3 葡萄牙栖盐田菌LLJ914及参考菌株基因组的CAZy注释结果通过对菌株LLJ914的碳水化合物活性酶进行分析,发现其基因组中编码碳水化合物活性酶的基因有96个,可分为6类,其中糖苷转移酶类(Glycosyl transferases,GT)相关基因42个,糖苷水解酶类(Glycoside hydrolases,GH)相关基因28个,碳水化合物结合模块(Carbohydrate-binding modules,CBM)相关基因10个,碳水化合物酯酶类(Carbohydrate esterases,CE)相关基因7个,多糖裂解酶类(Polysaccharide lyases,PL)相关基因2个,辅助模块酶类(Auxiliary activities,AA)相关基因7个(见图 6)。
|
( 图中加粗字体为本研究分离获得的菌株。The bold fonts in the figure are the isolates obtained in this study. ) 图 6 葡萄牙栖盐田菌LLJ914及其参考菌株的CAZy注释结果 Fig. 6 Results of CAZy annotation of Salinicola lusitanus LLJ914 and reference strains |
菌株LLJ914与参考菌株碳水化合物活性酶的差异性分析,发现该属菌株中GH家族碳水化合物活性酶的编码基因占比较高,其次是GT家族,PL家族最少,甚至有些菌株未注释到PL基因。该属的菌株中碳水化合物活性酶的编码基因数量大多小于96个(菌株Salinicola peritrichatus JCM 18795除外)。相比而言,菌株LLJ914可能具有相对较强的碳水化合物利用潜力,它包含多个α-葡萄糖苷酶(EC 3.2.1.20)、β-N-乙酰氨基己糖苷酶(EC 3.2.1.52)、溶菌酶(EC 3.2.1.17)、α-海藻糖酶(EC 3.2.1.28)、纤维素酶(EC 3.2.1.4)和淀粉合成酶(EC 2.4.1.213)等,且在糖苷转移酶基因、糖苷水解酶基因和辅助模块酶类相关基因均略高于菌株CR50T。
3 讨论深海热液区作为地球上极端的海洋环境之一,具有独特的地质和理化环境特征,孕育了丰富且独特的微生物类群,并且不断挑战着人们对微生物的代谢机理、生存极限等方面的传统认识[16]。葡萄牙栖盐田菌LLJ914是一株分离自冲绳海槽热液区海水样品中的菌株,本文对其进行了全基因组测序,通过功能注释和比较基因组学分析了该菌株的基因组功能和代谢潜力。根据16S rRNA基因序列比对可知,菌株LLJ914与葡萄牙栖盐田菌CR50T的相似性最高,它们基因组的ANI和AAI值分别为98.28%和98.76%。结合基于16S rRNA基因和单拷贝核心基因构建的系统发育树,可确定菌株LLJ914为葡萄牙栖盐田菌。
本研究针对葡萄牙栖盐田菌LLJ914选取了13株同属菌株作为参考菌,这些菌株大多是分离自盐矿、珊瑚、红树林根际或者动植物内共生等,只有菌株LLJ914来自深海热液区极端环境[3-8]。功能注释结果显示,该属菌株的基因功能主要集中于能源生产和转换、氨基酸与碳水化合物运输和代谢、转录方面;菌株LLJ914在能源生产和转换、转录、复制重组方面注释到的基因数目略高于菌株CR50T,KEGG基因通路注释结果与以上注释结果相一致。因此,结合热液区极端环境特征,推测菌株LLJ914的这些遗传优势能够增强其抗逆性,更加适应深海热液的极端环境。
据报道,栖盐田菌属菌株为中度嗜盐的需氧或兼性厌氧型菌株,拥有耐盐、耐碱、乳化原油、抵抗重金属等代谢潜力[2]。为了适应高盐环境,该属菌株采取了一系列渗透压保护机制,如通过特殊的转运蛋白主动积累高浓度的盐分来平衡周围高盐环境、合成和积累一些有机溶质,如甜菜碱可减少细胞对水分的丧失,有助于保持细胞的渗透平衡,或者通过调整膜脂的成分来提高膜的稳定性[44]。该属菌株基因组中与氨基酸运输和代谢、细胞壁/膜/被膜生物发生相关基因占比较高,可能更有利于该属菌株调整细胞膜脂成分来维持渗透压平衡[45]。该属大部分菌株均有CP-裂解复合物和phn基因簇,该途径使菌株可以利用甲基膦酸生成甲烷,从有机磷化合物中释放磷,并将其转化为无机磷酸盐,然后这些无机磷酸盐可以再次被该属菌株或其他生物体吸收和利用[46]。通过对菌株LLJ914进行RAST注释,该菌株拥有phnCDEFGIJKLMNP基因簇,并且在产甲烷检测实验中,将菌株培养62 h后,检测甲烷产量约为4.31 nmol/(L·h)(数据未发表),验证了该菌株有低产甲烷的能力。
在碳水化合物利用方面,菌株CR50T和LLJ914除了拥有糖原/淀粉的合成与降解相关基因,还拥有完整的乙醛酸循环,该途径对TCA循环进行回补,可以将TCA循环中间代谢产物异柠檬酸和底物乙酰CoA转化为一分子琥珀酸和苹果酸,通常在氧化应激或以二碳化合物为碳源时被激活[47]。由此可见,该种菌株可以利用乙酸盐或脂肪酸作为唯一碳源获得能量。其次,发现该种菌株还可以借助ED途径进行碳水化合物的利用。ED途径与其他糖代谢途径相比,葡萄糖通过一系列反应直接生成丙酮酸和丙酮,产生ATP的同时,避免了在磷酸戊糖途径中产生丙酮酸的附加步骤,能够更高效地产生ATP[48]。同时,该途径的存在使菌株能够代谢多糖、酸等碳源,使得该种菌株在不同环境中能够更灵活地利用各种有机物。此外,由于在ED途径中不涉及氧敏感的酶,因此菌株在缺氧或低氧环境中更具竞争优势,在低氧条件下拥有高效能力[49]。除此之外,该种菌株拥有糖降解相关的β-N-外切糖苷酶和D-半乳糖醛酸还原酶差相异构酶,可能反映了该种菌株对糖类的降解能力,有助于其在复杂的生态系统中存活和繁衍。
碳水化合物亦称为糖类化合物,是分布最为广泛的一类有机物。通过碳水化合物活性酶注释,菌株LLJ914在GH家族中注释到有β-N-乙酰氨基己糖苷酶、溶菌酶和α-海藻糖酶,GT家族注释到α-葡萄糖苷酶、纤维素酶和淀粉合成酶,与陆源菌株CR50T的基因组相比,糖苷转移酶基因、糖苷水解酶基因的个数均略高。在生物代谢反应过程中,α-葡萄糖苷酶直接参与淀粉及糖原中α-1.4-糖苷键的水解,这在农业、食品行业、生物能源以及医药行业具有重要价值[50]。另外,海藻糖酶具有稳定的化学性质,对细菌适应高温、寒冷、高渗透和脱水抗性的环境发挥重要作用。有研究表明,大多数细菌处于极端环境时可通过碳水化合物活性酶对糖原物质进行代谢,以满足其生存条件,如处于极冷环境中的节杆菌属可以利用糖原和海藻糖代谢途径快速适应环境[51]。该研究结果表明菌株LLJ914对多种碳源具有较强的利用能力,保障了菌株在深海热液区生命活动的正常进行,且其碳水化合物活性酶具有潜在的应用价值。
深海热液生态系统作为海洋中硫元素的重要来源[52],包含微生物介导的活跃硫氧化还原过程。KEGG代谢通路分析显示,菌株LLJ914和CR50T拥有同化硫酸盐还原过程中的cysC、cysNC、cysD、cysQ和cysH等相关基因,能够将SO42-在ATP的参与下还原成腺苷-5′-磷酰硫酸(Adenosine-5-phosphosulfate,APS),APS被ATP分子磷酸化产生3′-磷酸腺苷-5′-磷酸硫酸盐(3′-phosphoadenosine-5′-phosphosulfate,PAPS)随后生成SO32-,再被还原为H2S后最终生成半胱氨酸[53]。因此,初步认为该种菌株具有将环境中无机硫化合物(如硫酸盐、亚硫酸盐、硫代硫酸盐及硫烷等)转化为含硫有机化合物,进而用作细胞合成所需硫源的潜力。同时,菌株CR50T和LLJ914基因组均存在铁氧还蛋白-亚硝酸盐还原酶NirA与亚硝酸盐还原酶NirB和NirD的编码基因,表明该种菌株同时存在同化和异化硝酸盐还原途径以利用环境中的硝酸盐[54]。分离自深海热液区的菌株LLJ914,可能更倾向于在厌氧条件下利用异化硝酸盐还原途径产生能量。该途径也是电子传递链的重要组成部分,说明葡萄牙栖盐田菌可以利用硝酸盐作为最终电子,对于产生ATP以及维持细胞内能量平衡至关重要[55]。此外,异化硝酸盐还原菌在环境修复中还具有潜在应用。可以利用其还原能力对硝酸盐或其他污染物进行降解和清除。相反,在氧气充足的区域,该种菌株具有通过同化硝酸盐还原途径利用硝酸盐中的氮源进行生物合成的潜力,从而也反映了菌株兼性厌氧生长的能力。两株葡萄牙栖盐田菌还拥有钴转运蛋白、砷还原酶和铜转运体等[56-58],通过对离子吸收、转运和摄取的调控以及参与一些酶的生物合成,确保该菌株在重金属含量相对较高的环境下更好地生长[59]。综上所述,该种菌株对复杂底物的代谢能力较强,能够利用多种有机物作为能源和碳源来合成细胞组分和维持生长,使其在各种复杂环境中具有竞争优势。
根据KEGG-decoder的注释结果,并结合菌株LLJ914与CR50T的基因组BLAST比对结果可知,菌株LLJ914缺乏菌株CR50T基因组中存在的亚硫酸盐还原酶血红蛋白β组分,但特有cbb3型细胞色素c氧化酶的编码基因。亚硫酸盐还原酶是微生物在缺氧环境中利用亚硫酸盐进行能量代谢的关键酶系,而缺乏其β组分可能意味着菌株LLJ914在热液环境中不能利用亚硫酸盐进行无氧呼吸,但其基因组中存在cbb3型细胞色素c氧化酶,有助于该菌株进行有氧呼吸,为其生长提供能量。然而也有研究发现cbb3型细胞色素c氧化酶可能是参与调节细胞在缺氧环境下呼吸代谢的关键酶[59]。当细胞处于缺氧条件下时,cbb3型细胞色素c氧化酶可诱导NO的积累,参与反硝化作用并影响菌株的生长和细胞形态,促进厌氧生物被膜的成熟,有助于菌株在许多胁迫环境下的生存[60]。菌株LLJ914的基因组中还包含sqr基因,sqr作为硫化氢生物氧化途径中的关键基因,能够将硫化物氧化与电子传递链中的辅酶Q10还原相偶联[61],使硫化氢氧化成过硫化物,阻止了H2S的积累,同时产生电子以供能量代谢使用。Sqr蛋白的活性和表达水平可能会受到硫化物浓度、氧气浓度等环境因素的调节,与细菌适应深海热液喷口环境紧密相关。
此外,菌株LLJ914特有重金属抗性相关蛋白的蓝铜氧化酶CueO和ZupT转运蛋白,这可能与菌株适应深海热液区富含重金属矿物的环境特征有关[62]。多种重金属转运蛋白的存在,通过对离子吸收、转运和摄取调控的综合作用,使得一些金属离子在菌株LLJ914体内达到平衡,为其更好地适应其分离环境的特殊性奠定了良好的基础。在实际应用中,多铜氧化酶已被用于纸浆脱木质素、纺织染料漂白、水或土壤解毒等。此外,研究发现ZupT蛋白可以同时转运Zn2+和Fe2+,对控制细胞中必需但有毒的Zn2+水平至关重要[63]。菌株LLJ914特有的CRISPR相关蛋白是大多数微生物不断进化适应的免疫防御机制,用来抵抗外源遗传物质的入侵,比如噬菌体病毒和外源质粒,该免疫系统可以识别出外源DNA,并将它们切断,沉默外源基因的表达,为菌株更好地占据生态位提供了保障[64]。由此可见,菌株LLJ914中特有基因的存在,为其更好地适应深海热液环境发挥着重要的作用。
4 结语本研究对分离自冲绳海槽热液区的一株葡萄牙栖盐田菌LLJ914进行了全基因组测序分析。系统发育分析显示,该菌株属于栖盐田菌属,与马齿苋内共生的葡萄牙栖盐田菌CR50T亲缘关系最近,是同种不同株菌。该属菌株耐盐且可以产生甲烷,通过功能注释发现,该属菌株的基因多与能源生产和转换、氨基酸与碳水化合物运输和代谢等功能相关。葡萄牙栖盐田菌拥有利用多种有机物作为能源和碳源的能力以及多种重金属抵抗能力。由于分离环境的不同,菌株LLJ914与菌株CR50T的代谢通路存在差异,菌株LLJ914的基因在能源生产和转换、转录、复制重组模块中基因数目略高。值得注意的是,菌株LLJ914拥有独特的重金属抗性基因、抵抗病毒的CRISPR相关蛋白以及呼吸链相关的cbb3型细胞色素c氧化酶编码基因,推测菌株LLJ914具有较强的适应复杂海洋环境甚至深海热液极端环境的能力。本研究为阐明栖盐田菌属及代表菌株葡萄牙栖盐田菌LLJ914的代谢特征及其适应深海热液环境的机制提供了理论基础,有利于对该属菌株的进一步开发利用。
| [1] |
Anan'ina L N, Plotnikova E G, Gavrish E Iu, et al. Salinicola socius gen. nov., sp. nov., a moderately halophilic bacterium from a naphthalene-utilizing microbial association[J]. Microbiology, 2007, 76(3): 324-330. DOI:10.1134/S0026261707030095 (  0) 0) |
| [2] |
Plotnikova E G, Anan'ina L N, Ariskina D V, et al. Salinicola[M]// Bergey's Manual of Systematics of Archaea and Bacteria, New York: Society of American Bacteriologists, 2020: 1-29.
(  0) 0) |
| [3] |
Cao L, Yan Q X, Ni H Y, et al. Salinicola zeshunii sp. nov., a moderately halophilic bacterium isolated from soil of a chicken farm[J]. Current Microbiology, 2013, 66(2): 192-196. DOI:10.1007/s00284-012-0259-4 (  0) 0) |
| [4] |
Raju K, Sekar J, Vaiyapuri R P. Salinicola rhizosphaerae sp. nov., isolated from the rhizosphere of the mangrove Avicennia marina L[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(2): 1074-1079. DOI:10.1099/ijsem.0.000837 (  0) 0) |
| [5] |
Li X G, Chen J, Gan L Z, et al. Description of Salinicola corii sp. nov., a halotolerant bacterium isolated from wetsalted hides[J]. Current Microbiology, 2020, 77: 1932-1938. DOI:10.1007/s00284-020-01989-2 (  0) 0) |
| [6] |
张晓华, 刘吉文, 于敏, 等. 海洋微生物学(第二版)[M]. 北京: 科学出版社, 2016. Zhang X H, Liu J W, Yu M, et al. Marine Microbiology[M]. Beijing: Science Press, 2016. (  0) 0) |
| [7] |
Catia F, Diogo N P, Paula V M, et al. The endosphere of the salt marsh plant Halimione portulacoides is a diversity hotspot for the genus Salinicola: description of five novel species Salinicola halimionae sp. nov., Salinicola aestuarinus sp. nov., Salinicola endophyticus sp. nov., Salinicola halophyticus sp. nov. and Salinicola lusitanus sp. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2019, 69(1): 46-62. DOI:10.1099/ijsem.0.003061 (  0) 0) |
| [8] |
Huo Y Y, Meng F X, Xu L, et al. Salinicola peritrichatus sp. nov. isolated from deep-sea sediment[J]. Antonie Van Leeuwenhoek International Journal of General & Molecular Microbiology, 2013, 104(1): 55-62. (  0) 0) |
| [9] |
Olsson B E, Korsakova E S, Anan'ina L N, et al. Draft genome sequences of strains Salinicola socius SMB35T, Salinicola sp. MH3R3-1 and Chromohalobacter sp. SMB17 from the Verkhnekamsk potash mining region of Russia[J]. Standards in Genomic Sciences, 2017, 12: 39. DOI:10.1186/s40793-017-0251-5 (  0) 0) |
| [10] |
Juárez-Jiménez B, Fenice M, Pasqualetti M, et al. Flow cytometric investigation of Salinicola halophilus S28 physiological response provides solid evidence for its uncommon and high ability to face salt-stress conditions[J]. Microbiology Research, 2023, 14(2): 454-465. DOI:10.3390/microbiolres14020034 (  0) 0) |
| [11] |
池昌桥. 一株耐盐栖盐田菌W-Y11及其应用: CN201811492029.0[P]. 2019-02-26. Chi C Q. A strain of salt-tolerant Salinicola W-Y11 and its application: CN201811492029.0[P]. 2019-02-26. (  0) 0) |
| [12] |
Zhao G Y, Zhao L Y, Xia Z J, et al. Salinicola tamaricis sp. nov. a heavy-metal-tolerant, endophytic bacterium isolated from the halophyte Tamarix chinensis Lour[J]. Vaccine, 2017, 35(31): 1813-1819. (  0) 0) |
| [13] |
Kawagucci S, Chiba H, Ishibashi J, et al. Hydrothermal fluid geochemistry at the Iheya North field in the mid-Okinawa Trough: Implication for origin of methane in subseafloor fluid circulation systems[J]. Geochemical Journal, 2011, 45(2): 109-124. DOI:10.2343/geochemj.1.0105 (  0) 0) |
| [14] |
郑翔. 冲绳海槽中部热液区及典型喷溢区地形地貌特征及成因分析[D]. 青岛: 中国科学院研究生院(海洋研究所), 2016. Zheng X. Topographic and Geomorphologic Characteristics and Genetic Analysis of the Hydrothermal Area in the Middle of Okinawa Trough and Typical Spillage Area[D]. Qingdao: Graduate School of Oceanology, Chinese Academy of Sciences, 2016. (  0) 0) |
| [15] |
Zhou K, Qian P Y, Zhang T, et al. Unique phage-bacterium interplay in sponge holobionts from the southern Okinawa Trough hydrothermal vent[J]. Environmental Microbiology Reports, 2021, 13(5): 675-683. DOI:10.1111/1758-2229.12979 (  0) 0) |
| [16] |
Miyazaki J, Kawagucci S, Makabe A, et al. Deepest and hottest hydrothermal activity in the Okinawa Trough: The Yokosuka site at Yaeyama Knoll[J]. Royal Society Open Science, 2017, 4(12): 171570. DOI:10.1098/rsos.171570 (  0) 0) |
| [17] |
Zhang X, Li L F, Du Z F, et al. Discovery of supercritical carbon dioxide in a hydrothermal system[J]. Science Bulletin, 2020, 65(11): 958-964. DOI:10.1016/j.scib.2020.03.023 (  0) 0) |
| [18] |
Inagaki F, Kuypers M M, Tsunogai U, et al. Microbial community in a sediment-hosted CO2 lake of the southern Okinawa Trough hydrothermal system[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(38): 14164-14169. (  0) 0) |
| [19] |
Wang S S, Shao Z Z, Lai Q L, et al. Sulfurimonas sediminis sp. nov., a novel hydrogen-and sulfur-oxidizing chemolithoautotroph isolated from a hydrothermal vent at the Longqi system, southwestern Indian ocean[J]. Antonie Van Leeuwenhoek, 2021, 114(6): 813-822. DOI:10.1007/s10482-021-01560-4 (  0) 0) |
| [20] |
Ahmed O B, Dablool A S. Quality improvement of the DNA extracted by boiling method in gram negative bacteria[J]. International Journal of Bioassays, 2017, 6(4): 5347-5349. DOI:10.21746/ijbio.2017.04.004 (  0) 0) |
| [21] |
Yoon S H, Ha S M, Kwon S, et al. Introducing EzBioCloud: A taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies[J]. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(5): 1613-1617. DOI:10.1099/ijsem.0.001755 (  0) 0) |
| [22] |
Tamura K, Stecher G, Kumar S. MEGA11: Molecular evolutionary genetics analysis version 11[J]. Molecular Biology and Evolution, 2021, 38(7): 3022-3027. DOI:10.1093/molbev/msab120 (  0) 0) |
| [23] |
Edgar R C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Research, 2004, 32(5): 1792-1797. DOI:10.1093/nar/gkh340 (  0) 0) |
| [24] |
Wick R R, Judd L M, Gorrie C L, et al. Unicycler: Resolving bacterial genome assemblies from short and long sequencing reads[J]. PLoS Computational Biology, 2017, 13(6): e1005595. DOI:10.1371/journal.pcbi.1005595 (  0) 0) |
| [25] |
Koren S, Walenz B P, Berlin K, et al. Canu: Scalable and accurate long-read assembly via adaptive k-mer weighting and repeat separation[J]. Genome Research, 2017, 27(5): 722-736. DOI:10.1101/gr.215087.116 (  0) 0) |
| [26] |
Walker B J, Abeel T, Shea T, et al. Pilon: An integrated tool for comprehensive microbial variant detection and genome assembly improvement[J]. Public Library of Science ONE, 2014, 9(11): e112963. (  0) 0) |
| [27] |
Schoch C L, Ciufo S, Domrachev M, et al. NCBI Taxonomy: A comprehensive update on curation, resources and tools[J]. Database, 2020, baaa062. (  0) 0) |
| [28] |
Emms D M, Kelly S. OrthoFinder: Phylogenetic orthology inference for comparative genomics[J]. Genome Biology, 2019, 20(1): 238. DOI:10.1186/s13059-019-1832-y (  0) 0) |
| [29] |
Katoh K, Standley D M. MAFFT multiple sequence alignment software version 7: Improvements in performance and usability[J]. Molecular Biology and Evolution, 2013, 30(4): 772-780. DOI:10.1093/molbev/mst010 (  0) 0) |
| [30] |
Capella-Gutiérrez S, Silla-Martínez J M, Gabaldón T. TrimAl: A tool for automated alignment trimming in large-scale phylogenetic analyses[J]. Bioinformatics, 2009, 25(15): 1972-1973. DOI:10.1093/bioinformatics/btp348 (  0) 0) |
| [31] |
Kück P, Longo G C. FASconCAT-G: Extensive functions for multiple sequence alignment preparations concerning phylogenetic studies[J]. Frontiers in Zoology, 2014, 11(1): 81. DOI:10.1186/s12983-014-0081-x (  0) 0) |
| [32] |
Nguyen L T, Schmidt H A, Von H A, et al. IQ-TREE: A fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J]. Molecular Biology and Evolution, 2015, 32(1): 268-274. DOI:10.1093/molbev/msu300 (  0) 0) |
| [33] |
Letunic I, Bork P. Interactive Tree Of Life (iTOL) v5: An online tool for phylogenetic tree display and annotation[J]. Nucleic Acids Research, 2021, 49: 293-296. DOI:10.1093/nar/gkab301 (  0) 0) |
| [34] |
Yoon S H, Ha S M, Lim J, et al. A large-scale evaluation of algorithms to calculate average nucleotide identity[J]. Antonie Van Leeuwenhoek, 2017, 110(10): 1281-1286. DOI:10.1007/s10482-017-0844-4 (  0) 0) |
| [35] |
Kim D, Park S, Chun J. Introducing EzAAI: A pipeline for high throughput calculations of prokaryotic average amino acid identity[J]. Microbiol, 2021, 59(5): 476-480. (  0) 0) |
| [36] |
Overbeek R, Olson R, Pusch G D, et al. The SEED and the Rapid Annotation of microbial genomes using Subsystems Technology(RAST)[J]. Nucleic Acids Research, 2014, 42: 6-14. DOI:10.1093/nar/gkt913 (  0) 0) |
| [37] |
Kanehisa M, Sato Y, Morishima K. BlastKOALA and GhostKOALA: KEGG tools for functional characterization of genome and metagenome sequences[J]. Journal of Molecular Biology, 2016, 428(4): 726-731. DOI:10.1016/j.jmb.2015.11.006 (  0) 0) |
| [38] |
Kanehisa M, Sato Y. KEGG mapper for inferring cellular functions from protein sequences[J]. Protein Science, 2020, 29(1): 28-35. DOI:10.1002/pro.3711 (  0) 0) |
| [39] |
Galperin M Y, Kristensen D M, Makarova K S, et al. Microbial genome analysis: The COG approach[J]. Brief Bioinform, 2019, 20(4): 1063-1070. DOI:10.1093/bib/bbx117 (  0) 0) |
| [40] |
刘庆, 辛玉华, 东秀珠. 基于16S rRNA基因和基因组序列对细菌物种的初步鉴定[J]. Bio-Protocol, 2021(1): 2003932. Liu Q, Xin Y H, Dong X Z. Preliminary identification of bacterial species based on 16S rRNA gene and genomic sequence[J]. Bio-Protocol, 2021(1): 2003932. DOI:10.21769/BioProtoc.2003932 (  0) 0) |
| [41] |
Mount D W. "Using the basic local alignment search tool (BLAST)."[J]. Cold Spring Harbor Protocols, 2007, 7(1): 17. (  0) 0) |
| [42] |
Jain C, Rodriguez L M, Phillippy A M, et al. High throughput ANI analysis of 90K prokaryotic genomes reveals clear speciesboundaries[J]. Nature Communications, 2018, 9(1): 5114. DOI:10.1038/s41467-018-07641-9 (  0) 0) |
| [43] |
Thompson C C, Chimetto L, Edwards R A, et al. Microbial genomic taxonomy[J]. BMC Medical Genomics, 2013, 14: 913. DOI:10.1186/1471-2164-14-913 (  0) 0) |
| [44] |
王慧敏, 姚倩倩, 李月月, 等. 渗透压冲击下中度嗜盐菌Halomonas sp. Y四氢嘧啶类相容性溶质的合成与释放[J]. 微生物学通报, 2018, 45(4): 744-752. Wang H M, Yao Q Q, Li Y Y, et al. Synthesis and release of ectoines in a moderate halophile Halomonas sp. Y subjected to osmotic shocks[J]. Microbiology China, 2018, 45(4): 744-752. (  0) 0) |
| [45] |
Guo H, Ji M, Du T, et al. Salt stress altered anaerobic microbial community and carbon metabolism characteristics: The trade-off between methanogenesis and chain elongation[J]. Environ Manage, 2023, 9(1): 341: 118111. (  0) 0) |
| [46] |
Stosiek N, Talma M, Klimek-Ochab M. Carbon-Phosphorus Lyase—the state of the art[J]. Applied Biochemistry and Biotechnology, 2020, 190(4): 1525-1552. DOI:10.1007/s12010-019-03161-4 (  0) 0) |
| [47] |
Ahn S, Jung J, Jang I A, et al. Role of glyoxylate shunt in oxidative stress response[J]. Journal of Biological Chemistry, 2016, 291(22): 11928-11938. DOI:10.1074/jbc.M115.708149 (  0) 0) |
| [48] |
Conway T. The Entner-Doudoroff pathway: History, physiology and molecular biology[J]. FEMS Microbiology Reviews, 1992, 9(1): 1-27. (  0) 0) |
| [49] |
Pastor J M, Borges N, Pagán J P, et al. Fructose metabolism in Chromohalobacter salexigens: Interplay between the Embden-Meyerhof-Parnas and Entner-Doudoroff pathways[J]. Microbial Cell Factories, 2019, 18(1): 134. DOI:10.1186/s12934-019-1178-x (  0) 0) |
| [50] |
Waldron K J, Robinson N J. How do bacterial cells ensure that metalloproteins get the correct metal[J]. Nature Reviews Microbiology, 2009, 7(1): 25-35. DOI:10.1038/nrmicro2057 (  0) 0) |
| [51] |
王帅, 陈冠军, 张怀强, 等. 碳水化合物活性酶数据库(CAZy)及其研究趋势[J]. 生物加工过程, 2014, 12(1): 102-108. Wang S, Chen G J, Zhang H Q, et al. Carbohydrate active enzyme database (CAZy) and its research trend[J]. Chinese Journal of Bioprocess Engineering, 2014, 12(1): 102-108. (  0) 0) |
| [52] |
杜瑞, 于敏, 程景广, 等. 冲绳海槽热液区可培养硫氧化细菌多样性及其硫氧化特性[J]. 微生物学报, 2019, 59(6): 1036-1049. Du R, Yu M, Cheng J G, et al. Diversity and sulfur oxidation characteristics of cultivable sulfur oxidizing bacteria in hydrothermal fields of Okinawa Trough[J]. Acta Microbiologica Sinica, 2019, 59(6): 1036-1049. (  0) 0) |
| [53] |
王亚鑫, 吴玉, 张洪琳, 等. 微生物硫代谢及其驱动下建立的生物生态关系[J]. 微生物学报, 2022, 62(3): 930-948. Wang Y X, Wu Y, Zhang H L, et al. Microbial sulfur metabolism and the bioecological relationships driven by sulfur metabolism[J]. Acta Microbiologica Sinica, 2022, 62(3): 930-948. (  0) 0) |
| [54] |
Huang X J, Weisener C G, Ni J P, et al. Nitrate assimilation, dissimilatory nitrate reduction to ammonium, and denitrification coexist in Pseudomonas putida Y-9 under aerobic conditions[J]. Bioresource Technology, 2020, 312: 123597. (  0) 0) |
| [55] |
Nie S, Zhang Z, Mo S, et al. Desulfobacterales stimulates nitrate reduction in the mangrove ecosystem of a subtropical gulf[J]. Science of the Total Environment, 2021, 15(769): 144562. (  0) 0) |
| [56] |
Tufano K J, Reyes C, Saltikov C W, et al. Reductive processes controlling arsenic retention: Revealing the relative importance of iron and arsenic reduction[J]. Environmental Science & Technology, 2008, 42(22): 8283-8289. (  0) 0) |
| [57] |
Rensing C, Fan B, Sharma R, et al. CopA: An Escherichia coli Cu(Ⅰ)-translocating P-type ATPase[J]. Proceedings of the National Academy of Sciences, 2000, 97(2): 652-656. (  0) 0) |
| [58] |
Heddle J, Scott D J, Unzai S, et al. Crystal structures of the liganded and unliganded nickel-binding protein NikA from Escherichia coli[J]. Journal of Biological Chemistry, 2003, 278(50): 50322-50329. (  0) 0) |
| [59] |
Masakaze H, Masanori T, Tomoki M, et al. Cbb3-type cytochrome c oxidases, aerobic respiratory enzymes, impact the anaerobic life of Pseudomonas aeruginosa PAO1[J]. Journal of Bacteriology, 2014, 196(22): 3881-3889. (  0) 0) |
| [60] |
Takeshi S, Kunishige K. Basic and applied features of multicopper oxidases, CueO, bilirubin oxidase, and laccase[J]. The Chemical Record, 2007, 7(4): 220-229. (  0) 0) |
| [61] |
Xia Y Z, Lü C, Hou N K, et al. Sulfide production and oxidation by heterotrophic bacteria under aerobic conditions[J]. The International Society for Microbial Ecology, 2017, 11(12): 2754-2766. (  0) 0) |
| [62] |
Cameron S, Roberts, Fei N. The zinc and iron binuclear transport center of ZupT, a ZIP transporter from escherichia coli[J]. Bharati Mitra Biochemistry, 2021, 60(48): 3738-3752. (  0) 0) |
| [63] |
郭鑫瑶, 季晶焱, 程敏, 等. 41株戈登菌的系统发育学分析及其基因组功能的挖掘[J]. 贵州医科大学学报, 2023, 48(9): 1003-1012. Guo X Y, Ji J Y, Cheng M, et al. Phylogenetic analysis and genomic function mining of 41 strains of genus Gordonia[J]. Journal of Guizhou Medical University, 2023, 48(9): 1003-1012. (  0) 0) |
| [64] |
Cong L, Ran F K, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. (  0) 0) |
2. Laboratory for Marine Ecology and Environmental Science, Qingdao Marine Science and Technology Center, Qingdao 266237, China;
3. Institute of Evolution & Marine Biodiversity, Ocean University of China, Qingdao 266003, China
 2025, Vol. 55
2025, Vol. 55


