2. 成都医学院第一附属医院检验科, 四川 成都 610500
2. Department of Laboratory Medicine, The First Affiliated Hospital of Chengdu Medical College, Chengdu 610500, China
普罗威登斯菌属广泛分布于环境,属于条件致病菌,常引起腹泻和肠道外感染,如尿路感染、脑膜炎和败血症等[1]。雷氏普罗威登斯菌属于普罗威登斯菌属,常在食品加工运输中大量繁殖从而引起食物中毒等[2]。近年来,雷氏普罗威登斯菌导致的感染不断发生,多重耐药菌的检出率不断升高,耐碳青霉烯雷氏普罗威登斯菌引起的感染不断出现[3],已成为引起医院感染的病原菌之一,给临床疾病诊治带来严峻挑战。研究显示,金属β-内酰胺酶相关耐药基因(如blaNDM、blaIMP、blaVIM)是耐碳青霉烯类药物革兰阴性菌携带的主要耐药基因[4]。本研究拟通过分析某院雷氏普罗威登斯菌的耐药性,测定耐碳青霉烯类菌株全基因组,阐明其遗传特征,为临床抗菌药物治疗选择及医院感染防控提供依据。
1 对象与方法 1.1 研究对象本研究严格按照病原微生物标本采集规范,收集2017年1月—2019年12月临床送检标本,排除同一患者相同部位重复送检标本。
1.2 细菌鉴定和药敏试验待检标本经细菌培养分纯后采用法国梅里埃全自动微生物鉴定仪进行菌株鉴定,质控菌株为铜绿假单胞菌ATCC 27853、大肠埃希菌ATCC 25922、肺炎克雷伯菌ATCC 70603,均购自国家卫生健康委临床检验中心。同时采用配套的药敏分析仪进行抗菌药物敏感性检测,药敏结果参照美国临床实验室标准化协会标准[5]。
1.3 细菌全基因组测序选取碳青霉烯类耐药菌,使用细菌基因组提取试剂盒提取其所选取菌株全基因组DNA,提取的DNA送诺禾致源进行illumina PE150建库和全基因组测定,获得序列进行质控、组装、注释,并使用生物信息学软件进行分析。
1.4 数据分析应用WHONER 5.6软件对数据进行处理和分析,计数资料采用构成比和率表示,采用生物信息软件对测序结果进行分析。
2 结果 2.1 菌株来源某院2017年1月—2019年12月临床分离鉴定雷氏普罗威登斯菌25株,分布在12个临床科室,其中神经外科检出最多(7株,占28.00%),其次为重症医学科(5株,占20.00%),感染科、泌尿外科和烧伤整形科各2株(占8.00%),肝胆外科、骨外科一区、康复医学科、老年医学院科、神经内科、神经外科重症和消化内科各1株(占4.00%)。
25株雷氏普罗威登斯菌标本来源包括6类,其中检出最多的标本为尿(13株,52.00%),其次为伤口分泌物和脑脊液,分别为4株(16.00%)、3株(12.00%),全血和痰各2株(8.00%),胸腔积液1株(4.00%)。
2.2 耐药情况25株菌对β-内酰胺类(氨苄西林、阿莫西林)均耐药,耐药率为100.0%;对碳青霉烯类(亚胺培南、厄他培南、美洛培南)耐药率分别为36.00%、40.00%、28.00%。见表 1。
| 表 1 25株雷氏普罗威登斯菌对常见抗菌药物的药敏结果 Table 1 Susceptibility testing results of 25 strains of P. rettgeri to commonly used antimicrobial agents |
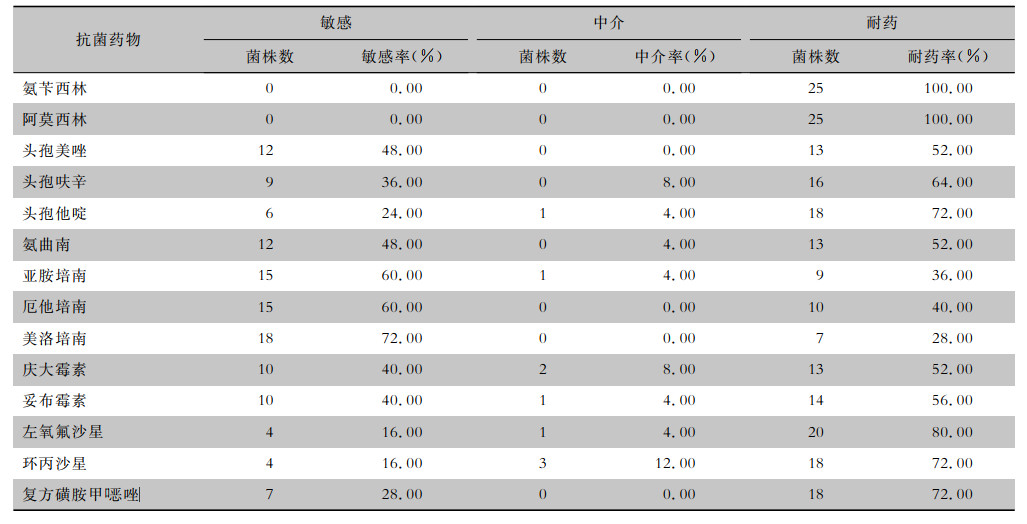
|
对3株耐碳青霉烯类雷氏普罗威登斯菌进行全基因组测序,结果显示A1菌株基因组最大,为4 725 540 bp;A1菌株开放阅读框数最多,为4 601个;A3菌株G+C含量最多,为40.86%;A3菌携带的RNAs数和耐药基因个数最多,分别为83个和26个。见表 2。
| 表 2 耐碳青霉烯类雷氏普罗威登斯菌基因组基本特性 Table 2 Basic characteristics of genomes of carbapenem-resistant P. rettgeri |
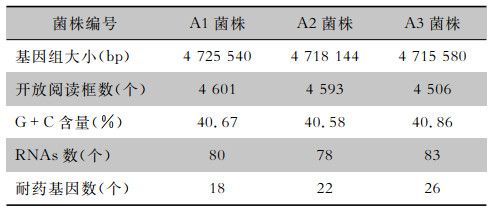
|
3株耐碳青霉烯类雷氏普罗威登斯菌全基因组测序,结果显示,均携带β-内酰胺类(blaNDM-1、blaOXA-10和blaTEM-1B)、大环内酯类[mph(A)、mph(E)、msr(E)]、甲氧苄啶(dfrA1)、氨基糖苷类[aadA1、aadA5、aph(3″)-Ib、aph(4)-Ia、acc(6′)-Ib3、aadB]、磺胺类(sul1)和氯霉素类(catB8)6类耐药基因型,碳青霉烯类耐药基因型均携带blaNDM-1、blaOXA-10。此外A3菌株还携带氟喹诺酮类基因型,见表 3。
| 表 3 耐碳青霉烯类雷氏普罗威登斯菌耐药基因筛选结果 Table 3 Screening results of antimicrobial resistance genes of carbapenem-resistant P. rettgeri |
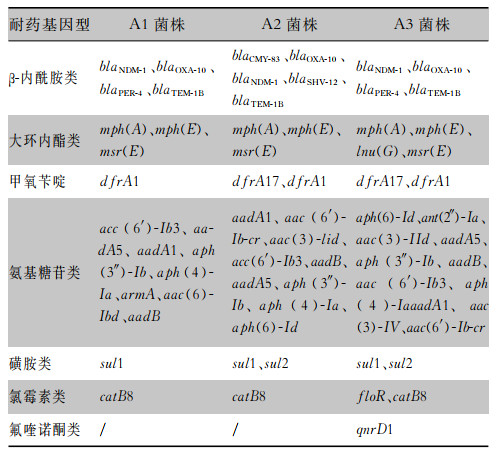
|
3株菌携带blaNDM-1耐药基因型,遗传结构为IS30-blaNDM-1-bleMBL-trpF-dsbC-orf-groES-groEL-IS91。与参考序列肺炎克雷伯菌质粒pC2343-2(序列号:CPO39824)、大肠埃希菌质粒pJ53(序列号:MN604268)相比较,与pC2343-2相似,在blaNDM-1的上游有插入序列IS30,下游有插入序列IS91。但不同于pJ53,上游为插入序列ISAba125。见图 1。
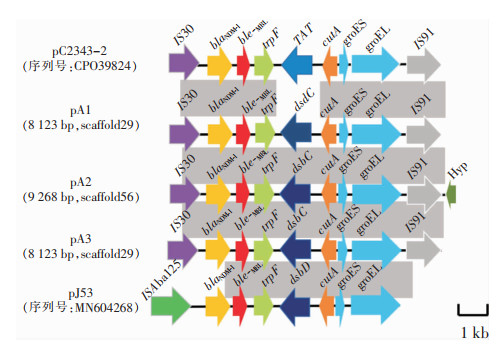 |
| 图 1 耐碳青霉烯类雷氏普罗威登斯菌blaNDM-1基因环境 Figure 1 blaNDM-1 gene environment of carbapenem-resistant P. rettgeri |
3株菌均携带blaOXA-10,遗传结构均为:aac(6’)-Ib3-dfrA1-aadA1-blaOXA10-catB8-aadB。与参考序列海氏普罗威登菌质粒p99101(序列号:CPO28385)相比较基因结构高度相似;与普通变形杆菌质粒pVsc3(序列号:CPO34667)相比较,下游基因不同,pVsc3携带的是磺胺类耐药基因型sul1,而3株菌携带的是甲氧苄啶耐药基因型dfrA1和氨基糖苷类耐药基因型aadA1、aac(6’)-Ib3。见图 2。
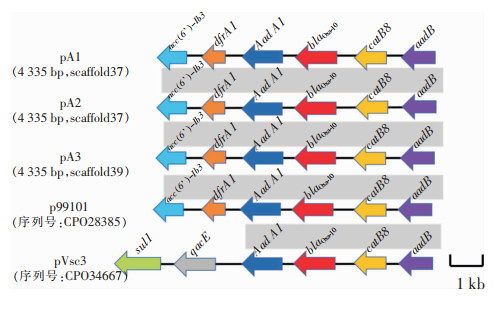 |
| 图 2 耐碳青霉烯类雷氏普罗威登斯菌blaOXA-10基因环境 Figure 2 blaOXA-10 gene environment of carbapenem-resistant P. rettgeri |
首株普罗威登斯菌1904年由Relttger分离,Kauffmam在1951年首次提议命名,1971年第一株耐药菌株被报道,而第一株完整的基因测序则是在2017年被报道,来源于一株产耐药基因blaNDM-1的临床菌株[6-8]。
普罗威登斯菌与变形杆菌和摩根菌种极为类似,不同于其他肠杆菌,虽然临床少见但普遍存于在环境中,有潜在致病性,是一类罕见的医院获得性和社区获得性感染病原体[1-2]。雷氏普罗威登斯菌可引起尿路感染和菌血症,也可引起心内膜炎、腹腔感染、脑膜炎等[1, 9]。近年来,不同地区相继报道雷氏普罗威登斯菌感染,从患者尿标本中分离出该菌[10-12]。该菌易存活于尿液中,本研究中雷氏普罗威登斯菌的主要标本来源也为尿。此外雷氏普罗威登斯菌感染还可引起脑膜炎,因Tanmoy Kamar Maiti[1]对雷氏普罗威登斯菌引起脑膜炎感染的案例进行报告,因此神经外科送检率高。本研究中该菌对氨苄西林和阿莫西林均耐药,耐药率为100.00%;对碳青霉烯类药物亚胺培南、厄他培南、美洛培南耐药分别为36.00%、40.00%、28.00%。目前,国内外文献对单株雷氏普罗威登斯菌的耐药性进行分析较多,而未大量的、系统的分析雷氏普罗威登斯菌耐药性,因此缺乏可比性。
全基因组测序的优点在于,不仅可以检测出细菌的耐药基因型,还可以发现可移动元件,从而分析出耐药基因型所处的基因环境[13]。引起细菌产生耐药的基因种类众多,本研究在3株耐碳青霉烯类雷氏普罗威登斯菌菌中发现7类抗菌药物耐药基因,包括β-内酰胺类、大环内酯类、甲氧苄啶、氨基糖苷类、磺胺类、氯霉素类和氟喹诺酮类。其中3株菌均同时携带β-内酰胺类耐药基因blaNDM-1、blaOXA-10和blaTEM-1B。与俞凤等[14]仅发现同时携带blaNDM-1和blaPER-4耐药基因型的菌株,贾琴妹等[15]仅发现携带blaKPC-2的菌株结果不一致,可能原因是菌株存在地区差异性,不同医院临床医生用药习惯不同。
细菌耐药性是通过存在于染色体或质粒中的耐药基因发生水平转移或自身突变而获得[16-17]。耐药基因在各个菌株间水平转移需借助可移动元件(插入序列、转座子和整合子等)的转化、转导、接合等形式完成复制[18]。本研究中3株菌均携带blaNDM-1和blaOXA-10耐药基因型,遗传结构分别为IS30-blaNDM-1-bleMBL-trpF-dsbC-orf-groES-groEL-IS91、aac(6’)-Ib3-dfrA1-aadA1-blaOXA10-catB8-aadB。耐药基因型blaNDM-1遗传结构分析表明,其与已报道的肺炎克雷伯菌质粒pC2343-2(序列号:CPO39824)的遗传结果高度一致,位于耐药基因型blaNDM-1两侧的片段,下游通常为trpF、groES、groEL基因和insE基因的一部分,在上游发现插入序列IS30。插入序列IS30可介导耐药基因的水平转移。耐药基因型blaOXA10的遗传结构与已报道的海氏普罗威登菌质粒p99101(序列号:CPO28385)高度相似,在耐药基因型blaOXA10的两侧均为其他类抗菌药物耐药基因型,但其上下游是否存在插入序列或转座子参与耐药基因的构成值得进一步研究。
综上所述,既往对普罗威登斯菌耐药情况缺乏系统性研究,对菌株分布,常见抗菌药物耐药率,尤其碳青霉烯耐药基因型和遗传特征均未见报道。本研究首次系统性地对普罗威登斯菌的耐药性及碳青霉烯类耐药菌株进行全基因组测序分析,具有一定的创新性。但仍存在测序菌株数量较少,耐药基因型和耐药基因环境分析不够完善,有待进一步扩大对多种抗菌药物的耐药基因及基因环境的检测,为临床抗菌药物规范化用药提供指导。
通过对某院雷氏普罗威登斯菌耐药性及碳青霉烯类耐药菌株的全基因组测序分析,耐药性结果显示出多重耐药现象,耐药基因筛选结果显示共有3株菌均携带β-内酰胺类、大环内酯类、甲氧芐啶、氨基糖苷类、磺胺类和氯霉素类耐药基因型,碳青霉烯类均携带blaNDM-1和blaOXA-10耐药基因型。为防止多重耐药菌株的出现,阻断耐药基因在病原菌和患者之间的传播,在临床治疗过程中应及早进行药敏试验和耐药基因分析,合理选择抗菌药物及联合用药。
| [1] |
Maiti TK, Singh VK, Pandey P. Providencia rettgeri: an unusual cause of central nervous system infections[J]. Am J Med Sci, 2013, 346(2): 158-159. DOI:10.1097/MAJ.0b013e318294f998 |
| [2] |
路娟, 赵占春. 普罗威登斯菌属属内分类和新种[J]. 哈尔滨医科大学学报, 1989, 23(5): 375-377. |
| [3] |
程阔, 何京, 冯军花, 等. 碳青霉烯类抑制法用于检测产碳青霉烯酶菌株的评价[J]. 中华检验医学杂志, 2017, 40(1): 64-66. DOI:10.3760/cma.j.issn.1009-9158.2017.01.014 |
| [4] |
Pedersen T, Sekyere JO, Govinden U, et al. Spread of plasmid-encoded NDM-1 and GES-5 carbapenemases among extensively drug-resistant and pandrug-resistant clinical Enterobacteriaceae in Durban, South Africa[J]. Antimicrob Agents Chemother, 2018, 62(5): e02178-17. |
| [5] |
Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing[S]. CLSl supplement M100, 27th ed. Wayne, PA: CLSI, 2017.
|
| [6] |
O'Hara CM, Brenner FW, Miller JM. Classification, identification, and clinical significance of Proteus, Providencia, and Morganella[J]. Clin Microbiol Rev, 2000, 13(4): 534-546. DOI:10.1128/CMR.13.4.534 |
| [7] |
Washington MA, Barnhill J, Griffin JM. A case of wound infection with Providencia rettgeri and coincident gout in a patient from Guam[J]. Hawaii J Med Public Health, 2015, 74(11): 375-377. |
| [8] |
Marquez-Ortiz RA, Haggerty L, Sim EM, et al. First complete Providencia rettgeri genome sequence, the NDM-1-producing clinical strain RB151[J]. Genome Announc, 2017, 5(3): e01472-16. |
| [9] |
Sharma D, Sharma P, Soni P. First case report of Providencia rettgeri neonatal sepsis[J]. BMC Res Notes, 2017, 10(1): 536. DOI:10.1186/s13104-017-2866-4 |
| [10] |
Tada T, Miyoshi-Akiyama T, Dahal RK, et al. NDM-1 meta-llo-β-lactamase and ArmA 16S rRNA methylase producing Providencia rettgeri clinical isolates in Nepal[J]. BMC Infect Dis, 2014, 14: 56. DOI:10.1186/1471-2334-14-56 |
| [11] |
Sagar S, Narasimhaswamy N, D'souza J. Providencia rettgeri: an emerging nosocomial uropathogen in an indwelling urinary catheterised patient[J]. J Clin Diagn Res, 2017, 11(6): DD01-DD02. |
| [12] |
宋嘉言, 张孟孝, 范宁. 多重耐药雷氏普罗威登斯菌医院感染一例[J]. 中华临床感染病杂志, 2018, 11(5): 391-393. DOI:10.3760/cma.j.issn.1674-2397.2018.05.013 |
| [13] |
谷美, 刘慧敏, 孟璐, 等. 全基因组测序技术在细菌耐药性检测中的研究进展[J]. 中国乳品工业, 2019, 47(5): 26-31. DOI:10.3969/j.issn.1001-2230.2019.05.007 |
| [14] |
俞凤, 胡龙华, 陈艳慧, 等. 两株泛耐药雷氏普罗威登斯菌耐药机制及临床感染特征研究[J]. 国际检验医学杂志, 2018, 39(A01): 97-101. |
| [15] |
贾琴妹, 孙乐, 满宝华. 耐碳青霉烯类雷氏普罗威登斯菌耐药机制研究[J]. 分子诊断与治疗杂志, 2018, 10(5): 332-336. DOI:10.3969/j.issn.1674-6929.2018.05.009 |
| [16] |
Mergeay M, Van Houdt R. Plasmids as secondary chromosomes[M]//Wells RD, Bond JS, Klinman J, et al. Molecular life sciences: an encyclopedic reference. New York: Springer New York, 2018: 961-964.
|
| [17] |
Rozwandowicz M, Brouwer MSM, Fischer J, et al. Plasmids carrying antimicrobial resistance genes in Enterobacteriaceae[J]. J Antimicrob Chemother, 2018, 73(5): 1121-1137. DOI:10.1093/jac/dkx488 |
| [18] |
Xu ZB, Xie JH, Yang L, et al. Complete sequence of pCY-CTX, a plasmid carrying a phage-like region and an ISEcp1-mediated Tn2 element from Enterobacter cloacae[J]. Microb Drug Resist, 2018, 24(3): 307-313. DOI:10.1089/mdr.2017.0146 |



