2. 青海大学包虫病重点实验室
棘球蚴病(echinococcosis)又称包虫病,是一种危害严重、呈世界性分布的人兽共患性寄生虫病,中国属高发地区之一。目前对于包虫病的免疫学诊断所用抗原是通过粗提原头节、囊液蛋白而获得,虽具有较高的敏感性和特异性[1],但抗原成分复杂,易发生过敏反应,且抗原来源困难,不利于大规模生产,限制其在临床应用。从棘球蚴原头节、囊液中筛选出具有特异性的抗原,通过基因工程技术大规模合成有望成为今后的发展方向。而抗原表位是抗原分子抗原性的基础,因此预测抗原分子的抗原表位,是寻找特异性抗原分子有效的方法。确定抗原表位常用的技术有X射线衍射与核磁共振分析、实验技术、生物信息学技术[2-3],前两者具有很高的准确性但成本昂贵,技术要求高,而通过生物信息学技术预测抗原表位,不仅可节省大量经费及时间,其预测准确性也随着生物信息学的发展而不断提高。有研究发现,延伸因子1(elongation factor 1,EF-1) 是一种具有潜在诊断价值的抗原[4-5],本研究通过生物信息学技术对其进行分析,旨在了解EF-1在细粒棘球绦虫的免疫学诊断价值。
1 材料与方法 1.1 基因及氨基酸序列查询GenBank并经BLAST比对确定EF-1基因序列,采用网站自动翻译的氨基酸序列用来后续分析。
1.2 理化性质推测EF-1的理化性质预测使用Expasy系统的protparam数据库(http://www.expasy.org/tools/protparam.html)。使用DNAStar软件Kyte-Doolittle法,预测EF-1亲水性;Karplus-Schulz法预测EF-1柔性区域;Jameson-Wolf法预测EF-1抗原性指数;Plot-Emini法预测EF-1表面可及性。
1.3 B、T细胞表位预测利用ABCpred在线软件(http://www.imtech.res.in/raghava/abcpred/)及IEDB软件(http://tools.immuneepitope.org/main/html/tcell_tools.html)综合预测EF-1 B表位,选取2者重叠部分中可能形成B细胞表位的部分。为提高预测准确性,ABCpred只保留得分≥0.8的结果,IEDB软件预测结果中只保留长度 > 7的结果。应用DNAStar软件AMPHI法预测Th细胞抗原表位、Rothbard-Taylor预测含特定基序(motif)的T淋巴细胞抗原表位,综合2种预测结果推测优势性T细胞表位。
1.4 二、三级结构预测二级结构预测使用SOPMA在线软件(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_ automat.pl?page=/NPSA/npsa_sopma.html),三级结构预测运用SWISS-MODEL网站(http://swissmodel.expasy.org/)同源建模方法,进行自动建模。
1.5 EF-1氨基酸序列同源性分析从GenBank中下载部分绦虫EF-1的氨基酸序列、使用MEGA软件选择neighbor joining法构建系统发育树。
2 结果 2.1 EF-1基因及氨基酸序列EF-1基因序列(No.AB306934) 全长1 412 bp,含有2个内含子(1~144、745~776),网站自动翻译的氨基酸序列(No.BAF63675) 由448个氨基酸残基组成。通过protparam数据库预测得到EF-1相对分子量为48 856.7 kDa,等电点pI为9.23,半衰期为30 h(mammalian)、 > 20 h(yeast)、 > 10 h(E.coli),不稳定指数为28.32(为稳定蛋白)。
2.2 EF-1蛋白亲水性、柔性区域、抗原指数及表面可及性(图 1)
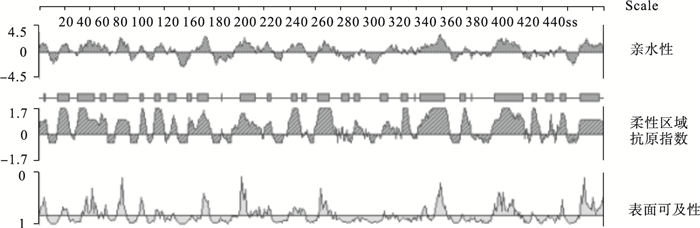
|
图 1 DNAStar软件预测的EF-1蛋白亲水性、柔性区域、抗原指数及表面可及性 |
Kyte-Doolittle法预测的EF-1亲水性得分较高的区域为35~45、52~70、126~140、158~183、301~322、357~380、424~448;Karplus-Schulz法预测的柔性区域散在分布,较大的几个区域为26~45、54~71、162~176、300~318、357~381、432~448;Jameson-Wolf法预测的抗原性指数得分较高的区域分布与Karplus-Schulz法预测的柔性区域一致;Plot-Emini法预测的表面可及性得分较高的区域较少,主要分布在39~54、62~76、128~132、161~170、220~234、315~329、354~372、424~448。
2.3 B细胞表位预测(图 2)
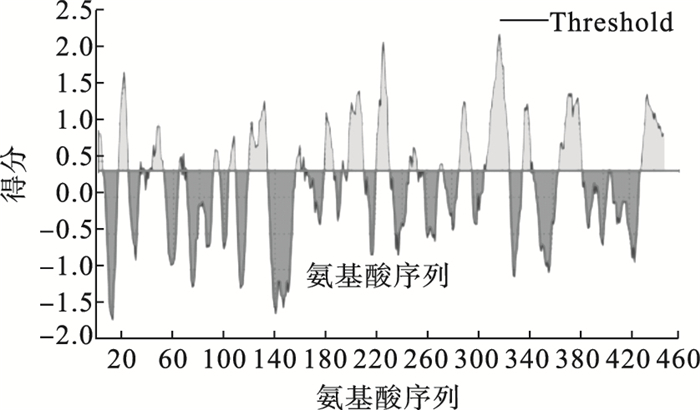
|
图 2 IEDB软件预测的EF-1 B细胞表位 |
通过ABCpred软件,得到的可能形成B细胞线性表位的氨基酸序列区域为87~103、114~130、146~162、187~203、253~269、279~295、304~320、330~346、336~352、362~378、377~393,IEDB软件预测的区域为17~24、41~52、120~134、198~210、221~231、286~296、306~325、365~383,两者重叠区域中可能形成B细胞线性表位的氨基酸序列为:120~130、286~295、306~320、365~378。
2.4 T细胞表位预测AMPHI法预测可能形成T细胞表位的区域为15~22、34~49、82~92、97~106、163~175、212~251、258~266、280~290、367~378、397~408;Rothbard-Taylor法预测可能的T淋巴细胞抗原表位为36~51、94~101、155~162、172~179、181~188、217~224、236~242、281~291、321~330、349~355、356~362、398~405、420~428,取两者重叠部分,得到可能的优势性T细胞表位区域为34~48、164~177、222~244、280~290、321~330、396~407。
2.5 二、三级结构(图 3)
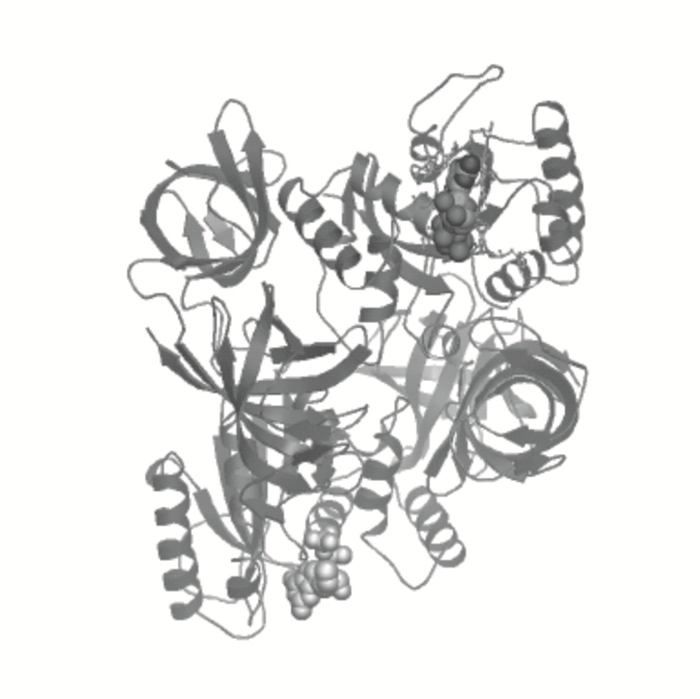
|
图 3 SWISS-MODEL网站构建的EF-1三级结构 |
SOPMA在线软件预测的EF-1二级结构中α螺旋占32.81%、β折叠占22.54%、β转角占10.94%、无规则卷曲占33.71%,SWISS-MODEL网站预测的EF-1的三级结构见图 3。
2.6 EF-1基因同源性分析(图 4)

|
图 4 MEGA软件构建的EF-1的neighbor joining系统发育树 |
从构建的系统发育树来看,EF-1具有较高的保守性,除多房棘球绦虫外,其他聚于一枝,并且在这枝内部形成梳齿状分枝,人作为外类群单独处于一枝。
3 讨论EF-1是一种多聚体核糖体蛋白质,在细胞内普遍存在且大量表达,真核细胞可能有α、β、γ、δ 4个亚基。其中α亚基属G蛋白家族,与鸟苷酸结合调节蛋白(guanine nucleotide-binding regulatoryprotein,GTP)、氨基酰-tRNA组成复合体,促进氨基酰-tRNA进入氨基酰位,EF-1β具有GTP酶活性,在EF-1α从核糖体脱落构象由GDP变为GTP准备结合新氨基酰-tRNA的过程中起催化作用,γ亚基常与β亚基形成复合物,可提高β亚基的GTP酶活性[6-7]。有研究发现,在脊椎动物中还有δ亚基,其C端与β亚基具有同源性,但N端没有,提示,它们可能执行着不同功能[8]。
生物信息学是由生物学、计算机和信息论等融合而成的一门学科,组织和存储了海量由基因遗传学、分子生物学和生物技术推动的生物信息[9]。已在药物设计、蛋白结构预测,表位疫苗预测等方面得到广泛应用。随着理论和技术的积累,大量蛋白、抗原表位数据库得以建立,如MHC配体和多肽基序的数据库SYFPEITHI、欧洲蛋白序列数据库(Swiss-Prot Protein Sequence Database,SWISS-PROT)、免疫表位数据库(immune epitope database,IEDB)、B细胞表位预测数据库(prediction of linear B-cell epitopes,Bcepred)等,预测结果精确度不断提高,使得通过生物信息学技术预测蛋白结构、抗原表位成为及时高效的手段。
抗原表位是指免疫应答过程中被特异的效应分子或B、T淋巴细胞识别的抗原分子中具有特殊结构与免疫活性的化学基团,根据所结合细胞的不同,分为B细胞表位和T细胞表位[10-11]。B细胞表位多为亲水性氨基酸或单糖残基,常位于抗原分子表面,而T细胞表位常为隐藏于抗原内部的疏水性肽段[2]。抗原表位决定着抗原分子的抗原性,研究抗原分子的抗原表位,对诊断试剂的制备、多肽疫苗的合成、单克隆抗体的筛选具有重要意义[12]。
对于B细胞线性表位的预测主要通过蛋白亲水性、柔性区域、抗原指数、表面可及性、二级结构综合来推测可能形成结合抗原的区域[13-16],亲水性区域位于抗原表面与B细胞表位关系密切。本研究结果显示,EF-1蛋白亲水性区域占50%以上,柔性区域的分布与抗原指数的分布区域一致,占60%以上,这为EF-1形成B细胞抗原表位提供了有利条件;表面可及性分布的区域较少提示,EF-1也可能形成构象表位。结合ABCpred和IEDB软件分析的结果得到可能形成B细胞抗原表位区域为:120~130、286~295、306~320、365~378,与抗原指数得分较高的区域分布有较大的重叠区域。研究表明二级结构中,α螺旋、β折叠常位于抗原内部,形态固定,结构规则,不利于抗体结合,而β转角和无规则卷曲位于抗原的表面,利于抗体结合,易于形成抗原表位[17]。SOPMA在线软件预测的EF-1二级结构中β转角占10.94%、无规则卷曲33.71%,为形成B细胞抗原表位提供了有利条件;EF-1三级结构中有较多的无规则卷曲,为形成B细胞线性表位提供了可能。
T细胞表位常位于抗原内部的疏水性区域。本研究结合AMPHI与Rothbard-Taylor的预测结果,得到EF-1可能的优势性T细胞表位区域为34~48、164~177、222~244、280~290、321~330、396~407,由亲水性预测结果来看,这些区域多分布在得分为负值的疏水性区域,结果较为准确。从构建的系统发育树来看,EF-1具有较高的保守性,除多房棘球绦虫外,其他均聚在一枝,并在这枝内部呈现梳齿状分枝。提示,EF-1在棘球绦虫与其他绦虫之间存在种属差异,利用这种差异,寻找细粒棘球绦虫EF-1特有表位,可为设计表位抗原疫苗提供新的选择[18-19]。
| [1] | Zhang W, Wang S, McManus DP. Echinococcus granulosus genomics:a new dawn for improved diagnosis, treatment, and control of echinococcosis[J]. Parasite, 2014, 21: 66. DOI:10.1051/parasite/2014066 |
| [2] | 宋帅, 李春玲, 贾爱卿, 等. 抗原表位研究方法进展[J]. 动物医学进展, 2010, 12: 87–91. DOI:10.3969/j.issn.1007-5038.2010.01.021 |
| [3] | Dall′Antonia F, Pavkov-Keller T, Zangger K, et al. Structure of allergens and structure based epitope predictions[J]. Methods, 2014, 66(1): 3–21. DOI:10.1016/j.ymeth.2013.07.024 |
| [4] | Matsubayashi M, Teramoto-Kimata I, Uni S, et al. Elongation factor-1α is a novel protein associated with host cell invasion and a potential protective antigen of Cryptosporidium parvum[J]. J Biol Chem, 2013, 288(47): 34111–34120. DOI:10.1074/jbc.M113.515544 |
| [5] | 刘玉江, 戴佳琳, 黄江, 等. 猪带绦虫成虫EF-1基因生物信息分析及原核表达[J]. 中国公共卫生, 2011, 27(1): 83–84. DOI:10.11847/zgggws2011-27-01-40 |
| [6] | 庞建新, 黄江, 胡旭初, 等. 亚洲带绦虫成虫延伸因子-1基因的克隆和分析[J]. 热带医学杂志, 2007, 06: 513–515. DOI:10.3969/j.issn.1672-3619.2007.06.003 |
| [7] | Sasikumar AN, Perez WB, Kinzy TG. The many roles of the eukaryotic elongation factor 1 complex[J]. Wiley Interdiscip Rev RNA, 2012, 3(4): 543–555. DOI:10.1002/wrna.1118 |
| [8] | Cao Y, Portela M, Janikiewicz J, et al. Characterisation of translation elongation factor eEF1B subunit expression in mammalian cells and tissues and colocalisation with eEF1A2[J]. PLoS One, 2014, 9(12): e114117. DOI:10.1371/journal.pone.0114117 |
| [9] | Soria-Guerra RE, Nieto-Gomez R, Govea-Alonso DO, et al. An overview of bioinformatics tools for epitope prediction:implications on vaccine development[J]. J Biomed Inform, 2015, 53: 405–414. DOI:10.1016/j.jbi.2014.11.003 |
| [10] | Zhang C, Li Y, Tang W, et al. The relationship between B-cell epitope and mimotope sequences[J]. Protein Pept Lett, 2016, 23(2): 132–141. DOI:10.2174/0929866523666151230124538 |
| [11] | Backert L, Kohlbacher O. Immunoinformatics and epitope prediction in the age of genomic medicine[J]. Genome Med, 2015, 7: 119. DOI:10.1186/s13073-015-0245-0 |
| [12] | Saravanan V, Gautham N. Harnessing computational biology for exact linear B-cell epitope prediction:a novel amino acid composition-based feature descriptor[J]. OMICS, 2015, 19(10): 648–658. DOI:10.1089/omi.2015.0095 |
| [13] | 梁瑾, 王靖飞. B细胞表位预测方法研究进展[J]. 生命科学, 2009, 2: 320–323. |
| [14] | Lian Y, Ge M, Pan XM. EPMLR:sequence-based linear B-cell epitope prediction method using multiple linear regression[J]. BMC Bioinformatics, 2014, 15: 414. DOI:10.1186/s12859-014-0414-y |
| [15] | Hu YJ, Lin SC, Lin YL, et al. A meta-learning approach for B-cell conformational epitope prediction[J]. BMC Bioinformatics, 2014, 15: 378. DOI:10.1186/s12859-014-0378-y |
| [16] | Huang JH, Wen M, Tang LJ, et al. Using random forest to classify linear B-cell epitopes based on amino acid properties and molecularfeatures[J]. Biochimie, 2014, 103: 1–6. DOI:10.1016/j.biochi.2014.03.016 |
| [17] | Nasrul Islam M, Iqbal S, Katebi AR, et al. A balanced secondary structure predictor[J]. J Theor Biol, 2016, 389: 60–71. DOI:10.1016/j.jtbi.2015.10.015 |
| [18] | 彭传林, 魏川川, 吴建伟, 等. 家蝇溶菌酶MDLZM基因克隆、表达和序列分析[J]. 中国公共卫生, 2015, 31(6): 764–766. DOI:10.11847/zgggws2015-31-06-20 |
| [19] | 赵学军, 国果, 修江帆, 等. 家蝇伴侣蛋白CCTη基因序列分析与表达模式[J]. 中国公共卫生, 2015, 31(8): 1043–1046. DOI:10.11847/zgggws2015-31-08-18 |
 2017, Vol. 33
2017, Vol. 33


