2. 山东省医疗机械产品质量检验中心
肠道病毒71型(entervirus 71,EV71 )是引起手足口病(hand-foot-and-mouth disease, HFMD)的主要病原体之一。EV71感染的患儿临床表现并不完全一致[1],轻者仅表现为发热和疱疹等;重者可引起神经系统并发症。EV71基因组是单股正链RNA,含1个开放阅读框(open reading frame,ORF)[2],编码4个结构蛋白(VP1-VP4) 和7个非结构蛋白(2A-2C和3A-3D),3C与3D蛋白对病毒的复制至关重要。本研究利用反向遗传技术重组EV71强毒株(SDLY107) 与弱毒株(SDLY1) ,将SDLY1株的3C和3CD蛋白编码区与SDLY107株置换,在横纹肌肉瘤细胞中拯救获得重组病毒107(1-3C)和107(1-3CD),为研究3C与3D蛋白在EV71致病机制中的作用奠定基础。
1 材料与方法 1.1 实验材料人横纹肌肉瘤细胞(rhabdomyosarcoma cell,RD)(山东省疾病控制中心陶泽新医师惠赠)。病毒SDLY107和SDLY1株分离自临沂市人民医院的患儿[3],SDLY107 株分离自1 例HFMD 合并脑炎的死亡患儿,SDLY1株分离自单纯HFMD患儿。pMD19-T-107全长质粒(山东大学公共卫生学院病毒学研究室保存)。RD细胞生长至对数生长期时接种病毒,待细胞病变至80%左右时,-40 ℃和室温之间反复冻融3次后4 ℃ 10 000 g离心10 min收获,储存于-80 ℃冰箱中。
1.2 主要试剂与仪器病毒RNA提取试剂盒(中国OMEGA公司)、DNA胶回收试剂盒和质粒提取试剂盒(中国OMEGA公司);逆转录试剂盒(日本TOYOBO公司);体外转录试剂盒(美国Promega公司);高效保真酶PrimeSTAR GXL DNA Polymerase、pMD19-T(中国TaKaRa公司);限制性内切酶、转染试剂(美国Thermo公司);细胞培养基、血清(美国HyClone公司);引物(上海生工生物工程股份有限公司合成);测序(上海博尚生物技术公司完成)。PCR仪(德国Eppendorf公司)、PCR电泳仪(美国BIORAD公司)、多功能数字图像分析仪(美国ProteinSimple公司)、细胞培养箱(美国Thermo公司)和光学倒置显微镜(日本Olympus公司)。
1.3 方法 1.3.1 扩增SDLY107与SDLY1株病毒的cDNA 使用OMEGA公司的Viral RNA kit 试剂盒提取SDLY107与SDLY1株病毒的RNA,使用TOYOBO公司的ReverTra Ace qPCR Kit 逆转录生成cDNA。分别在pMD19-T-107全长质粒的基础上进行重组病毒SDLY107(1-3C)和SDLY107(1-3CD)株的拯救。酶切位点ApaⅠ为SDLY107株和SDLY1株病毒共同自身含有;XbaⅠ为引入位点;HindⅢ为pMD19-T含有的位点。引物见表 1。使用引物1、2,以SDLY1株cDNA为模板,扩增3C蛋白编码区;使用引物3、4,以SDLY107株cDNA为模板,扩增3D+3′UTR编码区;使用引物1、4,以扩增获得的SDLY1株3C与SDLY107株3D+3′UTR为模板进行重叠PCR,获得重组PCR产物3C(1) -3D-3′UTR(107) 。引物1的5′UTR上游引入3个3C蛋白编码序列上游的碱基CAG;引物2和引物3的5′UTR上游含有互补片段;引物4的5′UTR上游引物XbaⅠ的酶切位点。使用引物1、5,以SDLY1株cDNA为模板,扩增3CD蛋白编码区;使用引物6、4,以SDLY107株cDNA为模板,扩增3′UTR编码区;使用引物1、4,以扩增获得的SDLY1株3CD与SDLY107株3′UTR为模板进行重叠PCR,获得重组PCR产物3CD(1) -3′UTR(107) 。引物5和引物6的5′UTR为完全互补片段。
| 表 1 引物序列 |
1.3.2 pMD19-T-107(1-3C)与pMD19-T-107(1-3CD)的构建
分别将PCR产物3C(1) -3D-3′UTR(107) 和3CD(1) -3′UTR(107) T-A克隆连接入pMD19-T载体,形成pMD19-T-3C(1) -3D-3′UTR(107) 与pMD19-T-3CD(1) -3′UTR(107) 。分别使用限制性内切酶ApaⅠ和XbaⅠ对pMD19-T-3C(1) -3D-3′UTR(107) 、pMD19-T-3CD(1) -3′UTR(107) 和pMD19-T-107进行双酶切,并分别回收3C(1) -3D-3′UTR(107) 、3CD(1) -3′UTR(107) 和pMD19-T-107-(107-为去除3CD-3′UTR编码区的剩余片段)。使用T4连接酶分别连接pMD19-T-107-与3C(1) -3D-3′UTR(107) 、3CD(1) -3′UTR(107) ,得到pMD19-T-107(1-3C)与pMD19-T-107(1-3CD)。
1.3.3 重组107(1-3C)和107(1-3CD)全长的获得及体外转录使用XbaⅠ和HindⅢ对pMD19-T-107(1-3C)、pMD19-T-107(1-3CD)进行双酶切,使其线性化,并回收107(1-3C)、107(1-3CD)。使用RiboMAX Ⅲ Large Scale RNA Production Systems-T7试剂盒进行体外转录,RNA提取试剂盒对RNA进行提取。
1.3.4 转染与拯救病毒分别将1 μg的107(1-3C)、107(1-3CD)全长RNA转染入刚铺满单层的RD细胞,约72 h小时后收获细胞,置-40 ℃和室温冻融3次后,接种至刚铺满单层的RD细胞,每孔200 μL。培养72 h后收获细胞并冻融3次,接种至刚铺满单层的RD细胞。全程观察细胞病变情况,并设置阴性及SDLY107、SDLY1株作为对照。
1.3.5 重组病毒的鉴定分别提取拯救病毒107(1-3C)、107(1-3CD)的RNA,逆转录生成cDNA,使用引物1、4对3CD编码区进行扩增后回收,测序鉴定。
2 结果 2.1 PCR结果(图 1)3C(1) 、3D-3′UTR(107) 和3C(1) -3D-3′UTR(107) ;3CD(1) 、3′UTR(107) 和3CD(1) -3′UTR(107) PCR扩增产物长度分别约为550、1 500、2 100、2 000、130、2 100 bp(A~F)。
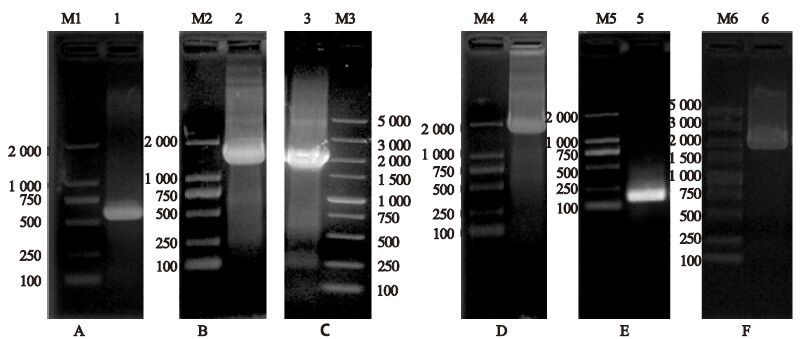
|
注: A:SDLY1株3C 扩增产物,maker1为DL2, 000(bp); B:SDLY107株3D-3′UTR扩增产物,maker2为DL2, 000(bp); C: 融合PCR产物3C(1) -3D-3′UTR,make3r为DL5, 000(bp); D: SDLY1株3CD扩增产物,maker4为DL2, 000(bp);E: SDLY107株3′UTR扩增产物,maker5为DL2, 000(bp); F:融合PCR产物3CD(1) -3′UTR(107) ,maker6为DL5, 000(bp)。 图 1 PCR扩增产物 |
2.2 pMD19-T-107、pMD19-T-3C(1) -3D-3′UTR和pMD19-T-3CD(1) -3′UTR(107) 双酶切结果
(图 2) 使用限制性内切酶ApaⅠ和XbaⅠ对pMD19-T-3C(1) -3D-3′UTR(107) 、pMD19-T-3CD(1) -3′UTR(107) 和pMD19-T-107进行双酶切,质粒分别被酶切为约8 000和2 100 bp、2 100和2 700 bp、2 100和2 700 bp的片段。箭头所指为需回收产物。
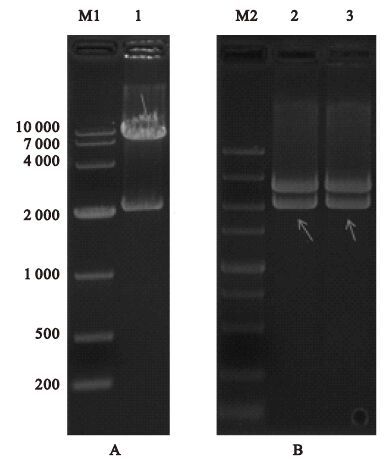
|
注:Maker1为DL10, 000(bp);maker2为DL5, 000 (bp);1号为pMD19-T-107双酶切产物(8000和2100 bp);2号为pMD19-T-3C(1) -3D-3′UTR(107) 双酶切产物(2100和2700 bp); 3号为pMD19-T-3CD(1) -3′UTR(107) 双酶切产物(2100和2700 bp)。 图 2 A:pMD19-T-107双酶切产物,;B:pMD19-T-3C(1) -3D-3′UTR(107) 和pMD19-T-3CD(1)-3′UTR(107) 双酶切。 |
2.3 pMD19-T-107(1-3C)和pMD19-T-107(1-3CD)双酶切产物(图 3)
使用XbaⅠ和Hind Ⅲ对pMD19-T-107(1-3C)、pMD19-T-107(1-3CD)进行双酶切,分别得到约为7 400和2 700、7 400和2 700 bp的片段,并回收7 400 bp片段。
|
注:Maker为DL10, 000(bp);1号为pMD19-T-107(1-3C)双酶切产物(7400、2700bp); 2号为pMD19-T-107(1-3CD)双酶切产物(7 400、2 700 bp)。 图 3 pMD19-T-07(1-3C)和pMD19-T-107(1-3CD)双酶切产物 |
2.4 重组病毒的细胞病变
将体外转录的RNA转染入RD细胞后48 h即出现明显的细胞病变,主要表现为细胞皱缩、变圆,72 h后-40 ℃反复冻融3次后收获1代病毒;将1代病毒接种至RD细胞,72 h内未出现明显的细胞病变,再次将细胞-40 ℃反复冻融3次后收获2代病毒;2代病毒接种至RD细胞,36 h后SDLY107(1-3C)与SDLY107(1-3CD)组细胞出现病变,48 h时病变可达80%。光学显微镜下观察到,重组病毒SDLY107(1-3C)、SDLY107(1-3CD)和病毒SDLY107病变没有明显区别,但3株病毒的致细胞病变能力强于SDLY1株。阴性对照组细胞没有病变。
2.5 重组病毒的鉴定将收获的重组病毒提取RNA后,进行逆转录为cDNA,对3CD编码区进行扩增回收后送测序公司进行测序,测序结果表明重组病毒拯救成功,分别命名为SDLY107(1-3C)和SDLY107(1-3CD)。
3 讨论肠道病毒71型作为手足口病的主要病原体之一,主要感染<5岁的婴幼儿。EV71自1969年首次分离出后,陆续在世界各地具有爆发[4-8]。EV71感染引起的临床表现也不尽相同,轻者仅表现为发烧、疱疹等,具有自限性;而重者会引起神经系统的并发症,甚至死亡。近年来,EV71引起的HFMD在中国多地流行[3, 9-11]。目前,关于EV71的致病机制尚未阐明。反向遗传技术的发展为在生物分子水平上研究EV71的致病机制提供了新的思路与途径。
研究发现,EV71 强毒株与弱毒株在ICR小鼠体内可以导致不同的病理改变;而强弱毒株在RD细胞的复制能力也存在着一定的差别,而这可能是导致EV71强弱毒株致病力不同的原因之一[1]。3C蛋白在病毒的复制以及宿主细胞的凋亡过程中发挥重要的作用[12],它可以抑制宿主细胞的转录[13];3D蛋白是一种RNA 依赖的RNA 聚合酶,在病毒的复制过程中主要完成RNA 链的延伸,对病毒的复制具有极其重要的作用[14]。同时对强弱毒株的序列进行比对,发现在EV71的3C和3D蛋白编码区可能存在着影响病毒毒力的位点[15-16]。
本研究发现直接转染RNA的RD细胞虽然可以产生cytopathic effect(CPE),但在收获一代病毒接种至RD细胞后并未产生明显的CPE。推测RD细胞在第1代产生病变可能是由于转染试剂的毒性及第1代病毒共同作用导致的。而第1代病毒接种入RD细胞产生的第2代病毒可能由于病毒滴度较低,所以并未对RD细胞产生明显的CPE。第2代病毒在接种入RD细胞36 h后即产生CPE,48h后产生明显的CPE。同时发现重组病毒产生的CPE与SDLY107较为接近。
本实验成功将弱毒株SDLY1株的3C、3CD蛋白编码区替换到强毒株SDLY107的3C、3CD蛋白编码区,成功构建了重组的EV71反向遗传系统,拯救了重组病毒SDLY107(1-3C)和SDLY107(1-3CD),这为我们研究3C、3D蛋白在EV71致病机制中发挥的作用奠定了良好的基础。
| [1] | Sun LL, Wang JK, Cui XQ, et al. Association of viral replication capacity with the pathogenicity of enterovirus 71[J]. Virus Res , 2014, 189 : 1–7. DOI:10.1016/j.virusres.2014.04.014 |
| [2] | McMinn PC. An overview of the evolution of enterovirus 71 and its clinical and public health significance[J]. FEMS Microbiol Rev , 2002, 26 (1) : 91–107. DOI:10.1111/j.1574-6976.2002.tb00601.x |
| [3] | 温红玲, 郝树彬, 高峰, 等. 肠道病毒71型山东临沂分离株全基因组序列分析[J]. 中华微生物学和免疫学杂志 , 2011, 31 (7) : 603–608. |
| [4] | Ho M, Chen ER, Hsu KH, et al. An epidemic of enterovirus 71 infection in Taiwan[J]. N Engl J Med , 1999, 341 (13) : 929–935. DOI:10.1056/NEJM199909233411301 |
| [5] | Mao LX, Wu B, Bao WX, et al. Epidemiology of hand,foot,and mouth disease and genotype characterization of enterovirus 71 in Jiangsu,China[J]. J Clin Virol , 2010, 49 (2) : 100–104. DOI:10.1016/j.jcv.2010.07.009 |
| [6] | AbuBakar S, Chee HY, Al-Kobaisi MF, et al. Identification of enterovirus 71 isolates from an outbreak of hand,foot and mouth disease(HFMD)with fatal cases of encephalomyelitis in Malaysia[J]. Virus Res , 1999, 61 (1) : 1–9. DOI:10.1016/S0168-1702(99)00019-2 |
| [7] | McMinn P, Lindsay K, Perera D, et al. Phylogenetic analysis of enterovirus 71 strains isolated during linked epidemics in Malaysia,Singapore,and Western Australia[J]. J Virol , 2001, 75 (16) : 7732–7738. DOI:10.1128/JVI.75.16.7732-7738.2001 |
| [8] | Kehle J, Roth B, Metzger C, et al. Molecular characterization of an enterovirus 71 causing neurological disease in Germany[J]. J Neurovirol , 2003, 9 (1) : 126–128. DOI:10.1080/13550280390173364 |
| [9] | 吴家兵, 方益荣, 王建军, 等. 安徽省2008年手足口病流行病学分析[J]. 安徽预防医学杂志 , 2010, 16 (2) : 96–98. |
| [10] | 刘威龙, 韦清, 杨桂林, 等. 深圳5例肠道病毒71型手足口病病毒株全基因组测序分析[J]. 中华微生物学和免疫学杂志 , 2010, 30 (5) : 409. |
| [11] | 郭淑珍, 孙渡, 王淑萍, 等. 哈尔滨市手足口病重症病例212例分析[J]. 中国公共卫生 , 2011, 27 (10) : 1353. |
| [12] | Barco A, Feduchi E, Carrasco L. Poliovirus protease 3C(pro)kills cells by apoptosis[J]. Virology , 2000, 266 (2) : 352–360. DOI:10.1006/viro.1999.0043 |
| [13] | Weidman MK, Yalamanchili P, Ng B, et al. Poliovirus 3C protease-mediated degradation of transcriptional activator p53 requires a cellular activity[J]. Virology , 2001, 291 (2) : 260–271. DOI:10.1006/viro.2001.1215 |
| [14] | Ferrer-Orta C, Arias A, Escarmis C, et al. A comparison of viral RNA-dependent RNA polymerases[J]. Curr Opin Struct Biol , 2006, 16 (1) : 27–34. DOI:10.1016/j.sbi.2005.12.002 |
| [15] | Wen HL, Si LY, Yuan XJ, et al. Complete genome sequencing and analysis of six enterovirus 71 strains with different clinical phenotypes[J]. Virol J , 2013, 10 (1) : 115–125. DOI:10.1186/1743-422X-10-115 |
| [16] | 温红玲, 郝树彬, 高峰, 等. 肠道病毒71型山东临沂分离株3D区遗传进化分析[J]. 中国公共卫生 , 2012, 28 (10) : 1283–1286. |
 2016, Vol. 32
2016, Vol. 32


