2. 河北师范大学
单核细胞增生李斯特氏菌(Listeria monocytogenes,Lm)是一种革兰阳性无芽孢兼性厌氧杆菌[1],对外界环境耐受性较强,可在较高的盐浓度(10% NaCl)、宽泛的pH范围(pH 4.5~9)和温度范围(0~45 ℃)内生长[2, 3]。当人或动物食用被Lm污染的食物后,可能引起以脑膜炎、脑炎、败血症、发热性胃肠炎或流产为症状的李斯特菌病[4]。李斯特菌病的发病率低,但是死亡率高(25%~30%),感染的潜伏期通常为7~60 d,因此在溯源上比较困难[5, 6, 7]。多位点序列分型(multi-locus sequence typing,MLST)技术基于管家基因的测序分析,分辨率高、重复性好,便于不同实验室间的数据比较,并可基于MLST网站进行世界范围内的比对,有利于进行全球范围内的分子流行病调查,对病原菌的溯源分析有重要意义。本研究采用MLST对分离自食品的69株Lm进行分型研究,了解河北省的主要基因序列型(sequence type,ST),为Lm分子流行病学研究提供资料。
1 材料与方法 1.1 菌株本研究检测的69株食源性Lm,均于2010—2012年间分离自生禽肉、生畜肉、水产品、速冻米面制品及凉拌色拉,均经API Listeria试剂条确认。其中30株Lm分离株由石家庄市疾病预防控制中心检出,其余39株Lm由河北省疾病预防控制中心馈赠。
1.2 主要仪器与试剂9700型PCR仪(美国ABI公司),QIAXCEL全自动毛细管电泳仪(美国QIAGEN公司),PCR扩增试剂盒(美国Promega公司GoTaq® Colorless Master Mix,批号00000390447)。
1.3 方法 1.3.1 PCR模板制备将菌株接种血平板37 ℃培养24 h,挑取纯培养的菌落至400 μL TE缓冲液(pH 8.0)中,震荡混匀,煮沸10 min,-20 ℃保存备用。
1.3.2 PCR扩增7个管家基因(abcZ、bglA、cat、dapE、dat、ldh、lhkA)的引物序列参考http://www.pasteur.fr/recherche/genopole/PF8/mlst/primers_Lmono.html网站,由大连TAKARA公司合成。PCR体系:2×Gotaq® Colorless Master Mix 25 μL,上、下游引物(10 μmol/L)各2 μL,DNA模板2 μL,加水补足体积至50 μL。PCR条件95 ℃ 4 min;94 ℃ 30 s,52 ℃ 30 s(bglA基因扩增的退火温度为45 ℃),72 ℃ 2 min,35个循环;72 ℃ 10 min。扩增片段分别为537、399、486、462、471、453和480 bp 扩增产物经全自动毛细管电泳仪检测后,委托上海生工生物工程有限公司进行双向测序。
1.3.3 测序结果处理与分析测序后的序列采用Sequencer软件进行比对拼接,截取目的片段,将7个管家基因序列上传至http://www.pasteur.fr/cgi-bin/genopole/PF8/mlstdbnet.pl?file=Lmono_isolates.xml网站获得每个序列的等位基因编号,以及对应菌株的ST型。利用Start2软件计算序列的G/C含量、非同义突变/同义突变(dN/dS)的比值、多态位点数。采用BURST对16个ST型进行亲缘分析,利用在线软件绘制SplitsTree进化树。采用辛普森指数(Simposons index of diversity,DI)[8]作为计算分辨力的方法。
2 结果 2.1 7个管家基因扩增序列分析(表 1)| 表 1 7个管家基因核酸序列分析 |
各管家基因的等位基因数分布于5~11之间,其中dat基因的等位基因数最少(5个),而cat基因的等位基因数最多(11个)。多肽位点数目最多的基因为dat基因,占序列长度的12.1%,lhkA基因的多态性位点数最少,占序列长度的3.8%;7个管家基因的G/C含量分布于36.48%~43.44%,dN/dS值分布于0.002 6~0.064 7之间。
2.2 MLST分型分析(表 2)| 表 2 69株Lm的MLST分析结果 |
本研究对河北地区的69 株分离自食品的Lm进行了MLST分型,通过对7个管家基因目的序列的扩增和测序比对,可将69株Lm分为16个ST型。7个基因的DI值介于72.12%~88.62%之间,将7个基因核酸序列整合后的DI值为89.98%。本研究确定了16个ST型,其中有3株Lm的ldh基因核酸序列经网站管理员(listeriaMLST@pasteur.fr)确认为新的等位基因型259,并确定了该3株菌的基因型为新型ST705 型(7-10-16-7-5-259-1)。在16种ST型中,ST8和ST87所占比例较高(ST8为14/69,ST87为12/69),其次为ST2(7/69)、ST1(6/69)、ST3(6/69)和ST9(6/69),其余各菌株的ST型出现较少,仅有3株或1株菌。采用BURST对16个ST型进行亲缘分析,ST155和ST705属于克隆群155(Group 155),ST9和ST122属于克隆群9(Group 9);其余ST型均为独特型(singleton)。而结合网站(http://pubmlst.org/data/profiles/lmonocytogenes.txt)上已公布的649个ST型进行亲缘分析,16个ST型被划分为14个克隆复合物(Clonal complexes CC)。
2.3 16个ST型的进化树分析(图 1)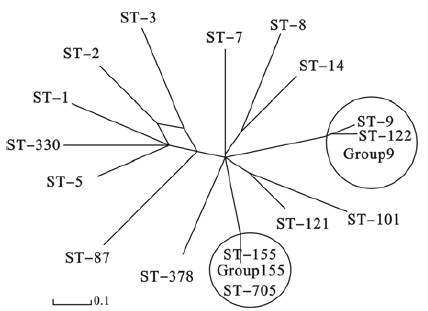 | 图 1 16种ST型的Splits Tree分析结果 |
利用Splits Tree建立的进化树分析菌株的亲缘相关性。ST155和ST705亲缘关系较近,ST9和ST122的亲缘关系较近,分别属于克隆群155和克隆群9。ST1和ST5,ST2和ST3,ST5和ST330,ST8和ST14,ST101和ST121这5组数据,每组均有3个相同的等位基因,具有较近的亲缘关系。
2.4 不同年份Lm分离株的MLST分型分析2010—2012年不同年份检出的Lm的主要ST型不同,2010年的14株Lm的主要ST型为ST9(4/14)和ST87(4/14);2011年的42株Lm 的主要ST型为ST8(12/42)、ST2(7/42)、ST1(6/42)和ST87(6/42);2012年13株Lm的主要ST型为ST3(6/13)和ST101(3/13)。同一种ST型在不同年份间分布亦存在差异。一些ST型集中出现在某一年,如ST1和ST2仅见于2011年;ST3仅出现于2012年,且占2012年检出菌株总数的46.15%;14株ST8型菌株,仅2011年就占12株,其余2株见于2010年。还有一些ST型,如ST9和ST87则普遍分布于2010—2012年3年中。
2.5 不同地区Lm分离株的MLST分型分析不同ST型在河北省不同地区的分布有差异,如ST1仅见于石家庄市,ST2仅见于沧州市,6株ST3秦皇岛占5株,而ST8、ST9和ST87却主要集中于秦皇岛市、承德市和石家庄市。不同ST型在不同城市所占比重不同,承德市仅有的7株Lm分离株均为ST8型,石家庄市的主要ST型为ST87和ST1,沧州主要ST型为ST2。同一ST型的地理分布还存在不同,ST8、ST87和ST121这3个ST型的Lm菌株分布范围较广,如ST8在承德市、张家口市、廊坊市、石家庄市和邯郸市均有分布;ST87则在秦皇岛市、保定市、石家庄市和邯郸市有分布;ST121在张家口市、廊坊市、石家庄市有分布。
3 讨论目前虽然有较多的分型方法可应用于食源性病原菌分型,但是传统分型方法,如血清型分型、噬菌体分型、多位点酶电泳分型等分型方法分辨力有限,检测结果易受操作人员经验等外界条件影响,尤其在突发公共卫生事件需进行流行病学调查和溯源时,很难满足准确、快速溯源的需要。随着分子生物学的发展,目前已有多种分子分型方法(如脉冲场凝胶电泳、扩增片段长度多态性、随机扩增多态性DNA等)可供选择,虽然这些方法具有较高的分辨力,但是它对仪器、试剂的要求较高,且需要专业的操作技能,实验室间结果差异较大,较难实现资源共享,目前还不能作为一种标准化的方法来应用[9, 10]。MLST选取一组管家基因作为研究对象,序列比较保守,在漫长的进化过程中能够保持一定的稳定性,能更好的表现不同菌株间的系统发育关系,因此,MLST是进行微生物流行病学调查的有力工具[11, 12]。本研究采用MLST方法对河北地区分离的69 株Lm进行分型检测,得到16个ST 型,DI值为89.98%,分辨力较高,可满足实验室对李斯特菌病溯源检测以及常规对Lm分子分型监测,从而建立分子流行病学数据库的需求。Wang等[13]在其研究中指出中国比较常见的ST型有ST9、ST8和ST87,而本研究发现河北省优势ST型为ST8、ST87、ST2、ST1、ST3和ST9,占全部菌株的73.9%,提示河北省优势的ST型相对较多,各ST型分布较分散,遗传具有多样性,这也与本地区的独特型Lm菌株占大多数相符合。本研究新发现的ST705与ST155具有较近的亲缘关系,形成了同一个克隆群CC38,提示ST705菌株可能由ST155菌株进化而来。在对网站全部649个ST型的分析可见,本研究的16个ST型分别属于14个CC,其中属于CC8、CC30、CC2、CC1、CC3、CC9的菌株占全部菌株的73.9%。有文献指出CC1和CC2分离株中,来源于病人的比率是来源于食品的2.60:1与2.77:1,强调该2种CC与临床致病菌关系较为密切[14],而本研究中发现的CC1和CC2的菌株有13株,占18.8%,比例相对较高。另外Wang等[13]在其研究中指出ST1、ST2、ST3、ST5、ST6、ST8和ST9是能够引起新生儿感染或暴发的ST型,对此开展监测,可以为预防李斯特菌病提供重要信息。通过分析pf8-bioinfo@pasteur.frpf8-bioinfo@pasteur.fr网站www.pasteur.fr/mls的Lm MLST数据库发现,在国际上引起暴发的ST型别主要有ST1(1989年法国和1995年瑞典)、ST63(1992年法国)、ST67(1993年法国)和ST4(1995年和2000年法国);对动物和人致病的ST型别有ST2、ST7、ST19、ST63、ST101、ST340、ST353和ST354。由此可见,引起人和动物致病的Lm的ST型别较多,在河北地区有可引起李斯特菌病的ST型存在。Lm在食品中的分离率较高[15],在国外Lm已成为除沙门氏菌和弯曲杆菌外的一种主要的食源性致病菌[16],而中国目前临床对李斯特菌病的诊断病例较少,除一方面中国特有的吃熟食的饮食习惯外,也与临床工作中对此病的重视程度不够有关。本研究结果提示临床工作者应提高对李斯特菌病的关注度,开展相关的临床检测工作。同时本研究还发现,虽然本研究所采用的菌株数目较少,但已能明显显示出Lm的优势ST型在不同时间和不同地区分布特点不同,提示各地区均应加强本地区Lm的长期分子流行病学监测,从而揭示Lm的流行规律和特点,为今后进行暴发疫情的流行病学调查和溯源提供基础资料。
| [1] | 崔焕忠,乔立桥,王义冲.单核细胞增生性李斯特菌的主要毒力因子及其致病机理[J].中国畜牧兽医,2010,37(1):128-133. |
| [2] | 刘桂华,黄鑫,王云立,等.吉林省食品中单增李斯特菌污染监测结果分析[J].中国公共卫生,2010,26(11):1461-1462. |
| [3] | 刘桂华,乔凤,黄鑫,等.食品中单增李斯特菌PCR检测方法建立与评价[J].中国公共卫生,2007,23(1):60-61. |
| [4] | Rocourt J,Hogue A,Toyofuku H,et al.Listeria and listeriosis:risk assessment as a new tool to unravel a multifaceted problem[J].American Journal of Infection Control,2001,29(4):225-227. |
| [5] | Chen BY,Pyla R,Kim TJ,et al.Prevalence and contamination patterns of Listeria monocytogenes in catfish processing environment and fresh fillets[J].Food Microbiology,2010,27(5):645-652. |
| [6] | Chen Y,ZhangW,Knabel SJ.Multi-virulence-locus sequence typing clarifies epidemiology of recent listeriosis outbreaks in the United States[J].Journal of Climinal Microbiology,2005,43(10):5291-5294. |
| [7] | 杨洋,刘秀梅.单核细胞增生性李斯特菌分子分型研究进展[J].中国食品卫生杂志,2008,20(2):158-162. |
| [8] | Hunter PR,Gaston MA.Numerical index of the discriminatory ability of typing systems:an application of Simpson's index of diversity[J].Journal of Climinal Microbiology,1988,26(11):2465-2466. |
| [9] | Jadhav S,Bhave M,Palombo EA.Methods used for the detection and subtyping of Listeria monocytogenes[J].Journal of Microbiological Methods,2012,88(3):327-341. |
| [10] | Zunabovic M,Domig KJ,Kneifel W.Practical relevance of methodologies for detecting and tracing of Listeria monocytogenes in ready-to-eat foods and manufacture environments-a review[J].Food Science and Technology,2011,44(2):351-362. |
| [11] | Parisi A,Latorre L,Normanno G.Amplied fragment length polymorphism and multi-locus sequence typing for high-resolution genotyping of Listeria monocytogenes rom foods and the environment[J].Food Microbiology,2010,27(1):101-108. |
| [12] | Salcedo C,Arreaza L,Alcala B,et al.Development of a multilocus sequence typing method for analysis of Listeria monocytogenes clones[J].Journal of Climinal Microbiology,2003,41(2):757-762. |
| [13] | Wang Y,Zhao AL,Zhu RF,et al.Genetic diversity and molecular typing of Listeria monocytogenes in China[J].BMC Microbiology,2012,12(1):1-9. |
| [14] | Chenal-Francisque V,Lopez J,Cantinelli T,et al.Worldwide distribution of major clones of Listeria monocytogenes[J].Emerging Infectious Diseases,2011,17(6):1110-1112. |
| [15] | Wang HH,Manuzon M,Lehman M,et al.Food commensal microbes as a potentially important avenue in transmitting antibiotic resistance genes[J].Federation of European Microbiological Societies.2005,254(2):226-231. |
| [16] | Renier S,Hébraud M,Desvaux M.Molecular biology of surface colonization by Listeria monocytogenes:an additional facet of an opportunistic Gram-positive foodborne pathogen[J].Environmental Microbiology,2010,13(4):835-850. |
 2016, Vol. 32
2016, Vol. 32


