针对遗传关联研究进行Meta分析已经成为一种探讨基因与疾病关联的常见研究方法。此方法可以综合不同研究结果从而获得较大把握度,而且可以发现不同研究之间潜在异质性[1, 2]。遗传关联研究Meta分析报告是否规范,直接影响其对以后研究和实践的指导作用。规范的报告,不仅可以条理清晰地展现整个分析过程和结果,还有助于编辑检查分析质量,读者准确理解分析内容。目前,适用于评价遗传关联研究Meta分析报告质量方面均有一定问题。如QUOROM(quality of reporting of meta-analyses)[3]及其修订版本PRISMA(preferred reporting items for systematic reviews and meta-analyses)[4]针对随机对照实验Meta分析;MOOSE(meta-analysis of observational studies in epidemiology)[5]针对观察性研究,但对具体领域针对性并不强,一些遗传关联研究的关键问题(如Hardy-Weinberg定律)难以涉及;STREGA(strengthening the reporting of genetic association studies)[6]为遗传关联研究报告规范,而非其Meta分析报告规范。由此可见,目前缺乏专门针对遗传关联研究Meta分析的报告规范。但相较而言,MOOSE规范得到了广泛认可,JAMA等[7]杂志要求观察性研究Meta分析均按此规范进行报告。国内研究者针对遗传关联研究进行了许多Meta分析,但这些Meta分析的报告质量尚不可知。故本研究采用MOOSE规范对发表于中文期刊的遗传关联研究Meta分析文章进行报告质量评价,不仅可以知晓此类文章的报告质量,还可以发现不足之处以待改善。为避免混淆,本文中,运用“研究”指代本研究,运用“文章”指代纳入的遗传关联研究Meta分析文章,运用“文献”指代Meta分析纳入的原始研究。 1 资料与方法 1.1 文章检索
由本研究研究者检索中国生物医学数据库、中国期刊全文专题数据库、万方数据资源和中国科技期刊数据库中收录的公开发表的中文文献,检索式为(“基因”或“遗传”或“变异”或“突变”或“位点”或“多态性”)和(“系统评价”或“Meta分析”或“系统综述”或“荟萃分析”),检索项目为题名或关键词,时间为数据库起始时间至2012年,最后1次检索日期为2013年1月23日。 1.2 文章纳入与排除标准
纳入标准:⑴公开发表于中文期刊的文章;⑵遗传关联研究Meta分析;⑶研究对象为人类;⑷纳入文献研究设计为病例对照研究、队列研究或全基因组遗传关联研究。排除标准:⑴外文文章或外文文章翻译;⑵无明确基因型;⑶研究暴露因素为基因表达或其产物;⑷重复发表文章,取最近发表的一篇;⑸文献类型为摘要、会议论文和学位论文。 1.3 文章筛选
分2步进行。首先,由2名研究者独立对检索到的文章进行初步筛选,通过阅读题目和摘要判断是否纳入本研究。然后,对初筛纳入的文章搜集全文,对于难以直接获得全文的文章,向原作者联系以取得文章,由2名研究者独立阅读全文进行再次筛选。以上2步中,均对2名研究者的意见进行交叉核对,不一致之处,参考第3位研究者意见进行判定。 1.4 报告质量评价工具
本研究采用MOOSE规范对纳入文章进行报告质量评价,规范的中文翻译参考相关文章[8, 9]。MOOSE规范关注观察性研究Meta分析的研究背景、文献检索策略、研究方法、研究结果、讨论和研究结论6个部分,要求文章必须报告各个部分中的相关内容。 1.5 文章信息提取及评价
运用Epi Data 3.1建立数据库,包括文章一般信息和MOOSE规范各条目内容。文章一般信息为文章题目、第一作者、单位、研究的基因、结局、研究人群等。由2名研究者独立进行信息提取及评价。所有研究者均熟知遗传流行病学和系统评价基本原理和方法。对提取的信息和评价进行交叉核对,不一致之处参考第3位研究者意见进行判定。 1.6 统计分析
运用SPSS 19.0软件进行统计分析。主要进行描述性分析,描述纳入文章中符合MOOSE规范各条目的比例。 2 结 果 2.1 文章筛选结果(图 1)
共检索到2003篇文章,去除重复文章914篇,通过阅读题目和摘要初步纳入488篇文章,通过阅读全文最终440篇文章被纳入本研究。
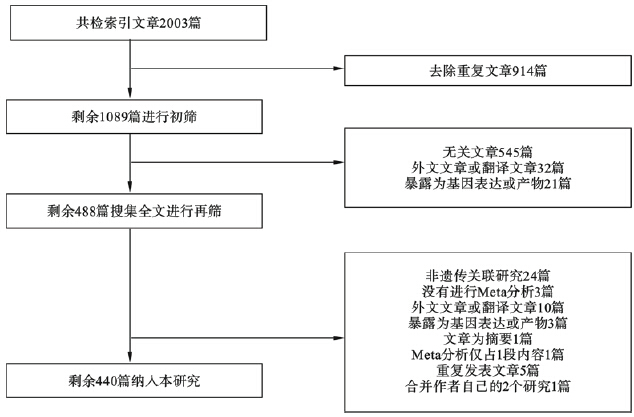 | 图 1 文章筛选流程图 |
纳入的440篇文章中,作者单位为大专院校占34.8%(153/440),医院占61.6%(271/440),疾病预防控制中心占1.8%(8/440),研究所占1.4%(6/440),其他单位占0.5%(2/440)。最早的中文遗传关联研究Meta分析文章发表于1997年[10];1998—2000年无文章发表;2008年之后,此类文章迅速增多。2012年有116篇文章。将纳入文章所关注结局按国际疾病分类第10版标准进行分类。440篇文章关注的结局前3位分别为肿瘤(如食管癌[11])、循环系统疾病(如冠心病[12])和内分泌营养代谢疾病(如糖尿病[13]),分别占34.3%(151/440)、20.2%(89/440)和12.7%(56/440)。440篇文章中,纳入文献数目中位数为10篇(范围3~61篇)。有150篇文章关注人群为中国人,占34.1%。有363篇文章有阳性结果,占82.5%。 2.3 MOOSE规范评价 2.3.1 研究背景部
分 440篇文章中,所有文章均报告了所研究问题,0.7%(3/440)的文章陈述了研究问题假设,所有文章均报告了研究结局、研究暴露及纳入文献类型,45.7%(201/440)的文章报告了研究人群。 2.3.2 文献检索部分
90.5%(398/440)的文章报告了文献检索时间范围,80.2%(353/440)的文章报告了使用的关键词,73.9%(325/440)的文章同时报告了文献检索时间范围和所用关键词。99.3%(437/440)的文章报告检索数据库,72.7%(320/440)的文章报告完整的检索策略(同时报告数据库、关键词及检索时间范围)。97.5%(429/440)的文章提供纳入文献清单,11.6%(51/440)的文章提供了排除文献清单,89.5%(394/440)的文章列出文献纳入标准,58.6%(258/440)的文章列出文献排除标准,6.4%(28/440)的文章同时列出纳入和排除的文献及判断标准。6.8%(30/440)的文章报告文献检索资格;5.5%(24/440)的文章报告了通过个人通信尽可能获得所有文献;0.7%(3/440)的文章报告了所采用的检索软件其及版本号;33.0%(145/440)的文章报告了手工检索补充文献;90.2%(397/440)的文章报告了处理不同语言文献的方法;15.2%(67/440)的文章报告了处理只有摘要和未发表文献的方法;0.2%(1/440)的文章介绍了个人通信情况,占所有进行过个人通信文章的比例为4.2%(1/24)。 2.3.3 研究方法部分
83.4%(367/440)的文章描述检索文献是否符合研究问题;35.5%(156/440)的文章报告数据整理和编码基本原则;47.3%(208/440)的文章报告文献筛选方法,38.6%(170/440)的文章至少有2名研究人员进行文献筛选并有解决不一致意见的方法;43.4%(191/440)的文章报告混杂的评估;32.7%(144/440)的文章评价了文献质量;93.4%(411/440)的文章评价了文献异质性;93.2%(410/440)的文章介绍了统计模型;88.0%(387/440)的文章提供了合适的统计图表。 2.3.4 研究结果部分
8.6%(38/440)的文章有流程图总结入选文献,92.5%(407/440)的文章列表描述入选各文献结果,61.1%(269/440)的文章报告了研究结果的敏感度分析,99.5%(438/440)的文章有统计学稳健性指标。 2.3.5 讨论部分
82.5%(363/440)的文章定量评价偏倚,12.5%(55/440)的文章解释了排除标准合理性,50.0%(220/440)的文章评价了入选文献质量。 2.2.6 结论部分
78.4%(345/440)的文章报告了导致结果的其他可能原因,53.6%(236/440)的文章对结论进行适当地外推,67.7%(298/440)的文章为以后该问题的研究提出指导意见,32.0%(141/440)的文章公布了研究资助来源。 3 讨 论
MOOSE作为观察性研究Meta分析报告规范,为研究者撰写Meta分析报告,编辑检查Meta分析报告质量,读者准确理解Meta分析报告内容提供了有力指导。按此规范撰写的观察性研究Meta分析报告,可以增加报告的条理性,并为编辑和读者评价文章质量提供完整信息。本文按此规范,对中文期刊中遗传关联研究Meta分析文章报告质量进行评价,发现存在一些问题。(1)研究背景:只有3篇文章报告研究假设。有研究者认为,研究假设报告与否并不影响文章质量[14]。但是,研究假设一个研究开展的基础。报告研究假设,可以方便读者对文章的目的和关注点一目了然,故笔者认为应该报告研究假设。(2)文献检索:文献检索欠广泛。本研究发现,>1/4的文章检索策略不全。完整的检索策略是广泛文献检索的基础,是防止Meta分析结果存在偏倚的重要措施。1/3的文章采用了手工检索对电子检索结果进行补充。电子数据库固然方便,但是由于种种原因,其覆盖范围不一定全面[15],为避免漏检,应该在电子检索的基础上,采用手工检索等方法进行补充。15.2%文章报告了处理只有摘要和未发表文献的方法。忽视灰色文献,可能会造成阳性结果选择偏倚,故应该尽可能搜集灰色文献,以确保结果真实性。由于观察性研究没有像随机对照实验那样的注册记录,故针对观察性研究的Meta分析查找灰色文献存在一定困难。(3)研究方法:文献
筛选和信息提取缺乏一定客观性。进行文献筛选和信息提取时,明确的操作原则,平行独立进行并核对最终形成统一意见是防止单个研究者主观作用对研究结果造成严重影响的重要措施。本研究发现,<1/2的文章报告了上述操作,说明中文期刊中,此类文章在此方面存在一定问题。文献质量评价情况有待提高。本研究发现,43.4%的文章报告了混杂的评估,32.7%的文章评价了文献质量。文献质量对Meta分析的影响存在一定争议[16],但是遗传关联研究有其特殊性,研究对象的人群分层、基因型实验室检测和结局测量等都会对文献研究质量和Meta分析结果造成影响,故笔者认为,遗传关联研究Meta分析必须注重文献的质量评价。目前,有许多观察性研究规范发表,Sanderson等[17]于2007年统计有86种,有些规范专门针对遗传关联研究[6, 18],相信采用这些规范进行文献质量评价,可以提高此类Meta分析的质量。(4)结论部分:所接受的资助情况不明确。本研究发现,68.0%的文章没有公布研究的资助来源,32.0%的文章只公布了资金资助来源而没有公布其他形式的资助。虽然与药物疗效Meta分析相比,遗传关联研究Meta分析中资金资助对结果结论的影响可能较小,但是不能忽视其他形式资助,如提供资料、进行外文文献翻译等,这些资助也可能会对研究结果结论造成影响。没有公布资金资助来源的文章可能没有得到资金资助,但并不代表没有接收其他形式资助。故此类研究应该将所接受的所有资助进行说明,以便读者对其结果客观性做出判断。(5)其他:本研究还发现,入选文章在提供文献筛选流程图,解释排除标准合理性方面也存在一定欠缺,这些问题妨碍了读者对文章整个研究过程的了解和结果科学性的判断。研究者应该重视此内容。
遗传关联研究是观察性研究的同时,又有其特殊之处,如Hardy-Weinberg平衡检验、合理选择遗传模型等,故此类研究Meta分析亦应考虑到此方面[8, 9]。但是,目前尚缺乏专门针对遗传关联研究Meta分析的规范。Meta分析质量分为报告质量和方法学质量,这两者并不等同[19, 20],中文遗传关联研究Meta分析文章方法学质量也有待探讨。MOOSE规范作为相较而言最适用的规范,已被许多外文期刊发表的遗传关联研究Meta分析文章所采用[21, 22, 23, 24];而且,一些国内作者发表于外文期刊的此类文章亦采用此规范进行报告[25, 26, 27, 28],但是中文期刊发表的此类文章尚未对此重视。
本研究采用MOOSE规范,对440篇中文遗传关联性Meta分析文章进行报告质量评价发现,此类文章存在一些欠缺和不足,主要集中在几方面:报告研究假设,文献检索,灰色文献处理,文献筛选方法,文献质量评价,提供文献筛选流程图和公布资助来源。相信今后研究者在进行遗传关联研究Meta分析时,参照MOOSE规范进行报告,不仅可以促进文章完整性和科学性,还可以帮助编辑和读者更容易对文章质量进行评价,从而促进中文遗传关联研究Meta分析文章质量的提高。
| [1] | Ioannidis J.Genetic associations:false or true?[J].Trends Mol Med, 2003, 9(4):135-138. |
| [2] | Ioannidis JP, Gwinn M, Little J, et al.A road map for efficient and reliable human genome epidemiology[J].Nat Genet, 2006, 38(1):3-5. |
| [3] | Moher D, Cook DJ, Eastwood S, et al.Improving the quality of reports of meta-analyses of randomised controlled trials:the QUOROM statement.Quality of Reporting of Meta-analyses[J].Lancet, 1999, 354(9193):1896-1900. |
| [4] | Moher D, Liberati A, Tetzlaff J, et al.Preferred reporting items for systematic reviews and meta-analyses:the PRISMA statement[J].PLoS Med 6(7):e1000097.doi:10.1371/journal.pmed.1000097. |
| [5] | Stroup DF, Berlin JA, Morton SC, et al.Meta-analysis of observational studies in epidemiology[J].JAMA, 2000, 283(15):2008-2012. |
| [6] | Little J, Higgins J, Ioannidis J, et al.Strengthening the reporting of genetic association studies(STREGA)—an extension of the STROBE statement[J].Eur J Clin Invest, 2009, 39(4):247-266. |
| [7] | JAMA Instructions for Authors[EB/OL].[2013-02-25].http://jama.jamanetwork.com/public/instructionsForAuthors.aspx#ManuscriptPreparationandSubmissionRequirements. |
| [8] | 詹思延.第一讲:如何报告系统综述和 Meta 分析—国际报告规范 QUOROM 和 MOOSE 解读[J].中国循证儿科杂志, 2010, 5(1):60-63. |
| [9] | 余灿清, 詹思延.如何撰写高质量的流行病学研究论文第二讲遗传关联性研究及其Meta分析的报告规范[J].中华流行病学杂志, 2006, 27(8):728-730. |
| [10] | 沈洪兵, 俞顺章.血管紧张素转换酶基因多态性与心肌梗塞关系的Meta分析[J].疾病控制杂志, 1997, 1(4):267-269. |
| [11] | 运玉霞, 代丽萍, 吴晓冰, 等.XRCC1基因多态性与食管癌发病风险Meta分析[J].中国公共卫生, 2010, 26(7):821-824. |
| [12] | 于慧会, 时景璞.汉族人群ApoA5基因多态性与冠心病关联Meta分析[J].中国公共卫生, 2011, 27(9):1195-1196. |
| [13] | 王长义, 王大鹏, 赵晓雯, 等.2型糖尿病与抵抗素基因-420位点多态性Meta分析[J].中国公共卫生, 2009, 25(12):1470-1472. |
| [14] | Attia J, Thakkinstian A, D'Este C.Meta-analyses of molecular association studies:methodologic lessons for genetic epidemiology[J].J Clin Epidemiol, 2003, 56(4):297-303. |
| [15] | Liberati A, Altman DG, Tetzlaff J, et al.The PRISMA statement for reporting systematic reviews and meta-analyses of studies that evaluate health care interventions:explanation and elaboration[J].PLoS Med, 2009, 6(7):e1000100.doi:10.1371/journal.pmed.1000100. |
| [16] | Herbison P, Hay-Smith J, Gillespie WJ.Adjustment of meta-analyses on the basis of quality scores should be abandoned.[J].J Clin Epidemiol, 2006, 59(12):1249-1256. |
| [17] | Sanderson S, Tatt ID, Higgins JP.Tools for assessing quality and susceptibility to bias in observational studies in epidemiology:a systematic review and annotated bibliography[J].Int J Epidemiol, 2007, 36(3):666-676. |
| [18] | Chanock SJ, Manolio T, Boehnke M, et al.Replicating genotype-phenotype associations[J].Nature, 2007, 447(7145):655-660. |
| [19] | Shea BJ, Grimshaw JM, Wells GA, et al.Development of AMSTAR:a measurement tool to assess the methodological quality of systematic reviews[J].BMC Med Res Methodol, 2007, 7(1):10. |
| [20] | Shea BJ, Bouter LM, Peterson J, et al.External validation of a measurement tool to assess systematic reviews(AMSTAR)[J].PloS One, 2007, 2(12):e1350.doi:10.1371/journal.pone.0001350. |
| [21] | Alfred T, Ben-Shlomo Y, Cooper R, et al.ACTN3 genotype, athletic status, and life course physical capability:meta-analysis of the published literature and findings from nine studies[J].Hum Mutat, 2011.doi:10.1002/humu.21526. |
| [22] | de Souza BM, Brondani LA, Boucas AP, et al.Associations between UCP1-3826A/G, UCP2-866G/A, Ala55Val and Ins/Del, and UCP3-55C/T polymorphisms and susceptibility to type 2 diabetes mellitus:case-control study and meta-analysis[J].PLoS One, 2013, 8(1):e54259.doi:10.1371/journal.pone.0054259. |
| [23] | Maxwell SS, Jackson CA, Paternoster L, et al.Genetic associations with brain microbleeds:systematic review and meta-analyses[J].Neurology, 2011, 77(2):158-167.doi:10.1212/WNL.0b013e318224afa3. |
| [24] | Clayden RC, Zaruk A, Meyre D, et al.The association of attempted suicide with genetic variants in the SLC6A4 and TPH genes depends on the definition of suicidal behavior:a systematic review and meta-analysis[J].Transl Psychiat, 2012, 2:e166.doi:10.1038/tp.2012.96. |
| [25] | Zhang H, Mo X, Hao Y, et al.Association between polymorphisms in the adiponectin gene and cardiovascular disease:a meta analysis[J].BMC Med Genet, 2012, 13:40.doi:10.1186/1471-2350-13-40. |
| [26] | Wang YQ, Qi XW, Wang F, et al.Association between TGFBR1 polymorphisms and cancer risk:a meta-analysis of 35 case-control studies[J].PLoS One, 2012, 7(8):e42899.doi:10.1371/journal.pone.0042899. |
| [27] | Chen H, Yu KD, Xu GZ.Association between variant Y402H in age-related macular degeneration(AMD)susceptibility gene CFH and treatment response of AMD:a meta-analysis[J].PLoS One, 2012, 7(8):e42464.doi:10.1371/journal.pone.0042464. |
| [28] | Liu F, Yuan D, Wei Y, et al.Systematic review and meta-analysis of the relationship between EPHX1 polymorphisms and colorectal cancer risk[J].PLoS One, 2012, 7(8):e43821.doi:10.1371/journal.pone.0043821. |
 2014, Vol. 30
2014, Vol. 30


