2. 河南省疾病预防控制中心免疫规划所
神经氨酸酶(neuraminidase,NA)是流感病毒的重要表面抗原之一,共有469个氨基酸残基组成,由病毒基因片段6编码〔1, 2〕;其功能主要是催化裂解唾液酸和糖脂、糖蛋白、寡聚糖间的α-糖苷键以利于子代病毒从宿主细胞表面释放,并阻止子代病毒的自我聚集,从而进一步感染其他细胞〔1, 3〕。NA是奥司它韦(克流感)[oseltamivir(tamiflu)]和扎那米伟(乐感清)[zanamivir(relenza)]等主要抗流感药物的作用位点〔4〕。对神经氨酸酶遗传进化特点分析研究,可及时发现其变异情况和不同变异的流行趋势,对流感病毒传播的防控有重要意义。
1 材料与方法 1.1 材料河南省各市(地)疾病预防控制中心和流感监测哨点医院采集流感患者咽拭样本,并经河南省疾病预防控制中心流感实验室,按照国家流感中心提供的标准操作规程〔5, 6〕分离鉴定并提供35例甲型H1N1病毒样本。
1.2 试剂与仪器犬肾细胞(MDCK细胞)MDCK传代细胞系,由国家流感中心提供并按照国家流感中心提供的标准操作规程培养细胞〔5〕;抗A(季节性H1N1)血清、抗A(季节性H3N2)血清,抗B(Victoria)血清,抗B(Yamagata)血清和抗A(甲型H1N1)血清(国家流感中心),RNeasy Mini Kit,Onestep RT-PCR试剂盒(德国QIAGEN公司);RNA Protecter(瑞士罗氏公司)。
1.3 方法 1.3.1 NA基因全长引物设计根据GenBank已经公布的甲型H1N1流感病毒的序列信息,采用中国国家流感中心提供的引物序列,对相应的引物库筛选,选择2对引物,序列如下:引物组1: NA-1: 5'-TGTAAAACGACGGCCAGTAGCAAAAGCAGGAGT-3',NA-2: 5'-CAGGAAACAGCTATGACCCGTATYTGAATGAAAACC-3';引物组2: NA-3: 5'-TGTAAAACGACGGCCAGTAATGGRCARGCCTCRTACAA-3';NA-4: 5'-CAGGAAACAGCTATGACCAGTAGAAACAAGGAG-3'。每条正反向引物前面分别加上-TGTAAAACGACGGCCAGT和-CAGGAAACAGCTATGACC的测序引物序列,由上海生物工程技术有限公司合成。预计扩增产物片段长度为:引物组1: 1100 bp左右,引物组2: 700 bp左右。
1.3.2 病毒RNA提取与cDNA制备采用RNeasy Mini Kit提取病毒总RNA,采用荧光定量PCR进行鉴定,采用onestep RT-PCR试剂盒一步扩增目的cDNA,RNA酶抑制剂采用RNA Protecter。RT-PCR扩增步骤严格按试剂盒操作说明进行操作。
1.3.3 目的基因测序和序列分析扩增出的cDNA产物由上海生物公司纯化测序,测序结果用Contigexpress软件进行序列拼接。扩增产物在NCBI中BLAST查重,进而与2009年的甲型H1N1流感进行比对分析选取国内外代表株,对其核酸和氨基酸序列进行比对。采用分子进化遗传分析软件(Mega4.1)绘制进化树。
2 结果 2.1 RT-PCR扩增结果(图 1)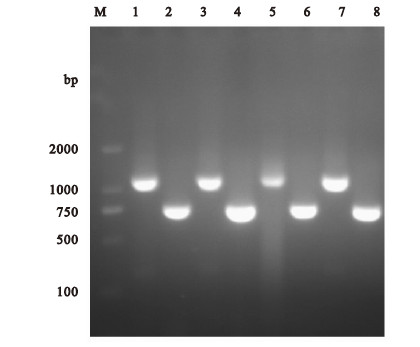 | 注:图中1、3、5、7为引物NA-1和NA-2扩增片段;2、4、6、8为引物NA-3和NA-4扩增片段。 图 1 RT-PCR扩增NA基因cDNA的部分扩增结果 |
河南省疾病预防控制中心流感实验室分离鉴定35例H1N1病毒样本,共扩增出20例,扩增结果与预期相符。
2.2 测序比对对每个分离株NA基因扩增出的2段序列进行正反双向测序,共测出17组全长序列。经Contigexpress软件序列拼接后得到1 410 bp的CDS全长序列,编码469个氨基酸。初步比对,有5对分离株序列结果完全相同,共得到12例不同的NA全长基因。本次河南分离株与2009年以后分离得到的甲型H1N1流感分离株同源性较高,均在99%以上。本研究得到17株分离株的NA核酸位点与A/California/07/2009(H1N1)(FJ 984 386)和A/Sichuan/1/2009(H1N1)(GQ 166 224)比较,有6处变异,包括: 316位G/A变异(17/17);408位A/G变异(3/17);742位A/G变异(17/17);945位A/G变异(14/17);1 305位A/G变异(4/17);1 338位T/C变异(11/17)。其中,本次分离株316位742位核酸,均发生突变。316位导致106位氨基酸的V/I变异,742位导致蛋白序列248位氨基酸的N/D变异。其他4处突变均为同义突变,对氨基酸序列无影响。
2.3 进化树分析将BLAST比对结果,结合序列地域关系和发布时间,分析此次分离株与国内外分离株的进化关系。进化树显示此次分离主要分为两大聚类:聚类Ⅰ中本次分离株含945位A/G和1 338位T/C突变的Subject-1(Genbank: JN036 561)和Subject-2(GenBank: JN 036 567)与国内部分地区[四川(HM 006 722)等]、俄罗斯赤塔地区(GU 234 175)、2010年泰国地区(CY 080 309)甲型H1N1流感以及国内猪流感亲缘较近,这一聚类均包含945位A/G和1 338位T/C突变。聚类Ⅱ中945位和1 338位未变异的Subject-3(GenBank: JN 036575)和2009-2011国际疫苗株〔7〕亲缘性更接近于欧美地区和2009年东南亚和中国大部分地区分离株。国际代表株A/California/07/2009(H1N1)(FJ 984 386)与A/Sichuan/1/2009(H1N1)(GQ 166 224)和2009年5月北京分离株A/Beijing/01/2009(H1N1)(GQ 225 375)为聚类Ⅱ中一小聚类,其为2009年甲型H1N1流感暴发初期得到的代表性序列,未发现蛋白序列106位V/I和148位N/D变异。进化树中除这一小聚类以外,其他毒株这两位点均发生变异,其中多株毒株为2009年6-8月间发布的毒株序列如:日本(GQ 365 427)、挪威(CY 046 953)、中国浙江(GQ 225 351)、中国上海(GQ225 359)等。
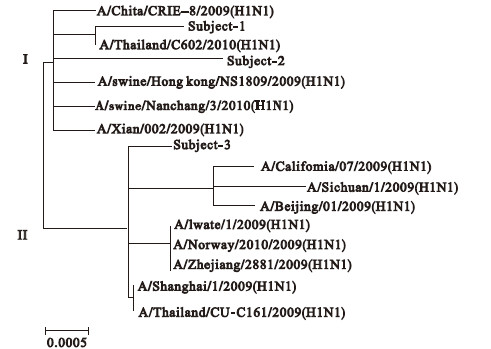 | 注: Sabject的为本次河南分离代表株 图 2 河南3株甲型H1N1流感病毒分离株与BLAST上其他毒株NA基因的进化关系 |
本次河南分离得到的病毒株NA蛋白序列无较大变异。106位V/I和248位N/D的变异相伴出现,并导致氨基酸序列中106位V/I和248位N/D发生变异。从Genbank下载的500余株2009、2010年NA序列中比对中发现,这2个位点变异于2009年6-8月间最早见于欧洲和亚洲(挪威(CY046 953),日本(GQ 365 427),浙江(GQ 225 351)等),且于2009年10月以后逐步成为优势变异株,分布世界各地。此次分离株均属于这类变异株。根据Sebastian Maurer-Stroh等人的研究推测的神经氨酸酶活性位点和抗原位点248位N/D不属于抗原区也不是NA蛋白活性位点〔2〕。106位V/I变异位点位于NA蛋白抗原结合区,因极性变异较小,结合郭永豪等〔6〕对河南地区甲型H1N1凝血因子HA的研究表明,现今疫苗在河南地区仍具较好保护效应。
自2009年5月中国发现第一例输入性病例〔8〕。同年8月,国内病例主要为能够明确判断传染源的输入性病例;2009年9月份开始,中国的本土病例开始大量出现,无法查证传染源,同年12月底甲型H1N1流感病毒流行达到高峰〔9〕。此次分离株测序发现的945位A/G和1338位T/C突变常相伴出现,在2009年10月以前Genbank发布的人源甲型H1N1中并未发现。国内含此变异毒株的比例按时间推移有增加趋势,在广东(CY 058 835等3株)、上海(AB 539 741)、西安(HM 006 722等6株)、山东青岛(CY 050 132等10株)陆续发现。故推测含这两个位点变异的为2009年10月份以后我国出现的本土病例。同时除我国外,变异的毒株分布集中于近邻我国的东北亚和东南亚地区(俄罗斯赤塔(HM 189 314等)、蒙古国(CY 065 997)、泰国(CY 080 309等),发现时间也均为2009年10月以后。
在序列比对中发现:这2个变异位点同样发现于2009年中国香港(CY 061 751等)和2010年南昌(JF 275 935等3株)猪流感中,且同源性达99.9%。这一现象表明,在中国2009年9月份以后,由输入型甲型H1N1流感病毒病例到以本土传染为主的流行时间内,中国有可能存在着猪-人之间相互传染。这一结果与郭永豪等〔6〕研究结果相同,可相互得到佐证。
| 〔1〕 | Venkatramani L,Bochkareva E,Lee JT,et al.An epidemiologically significant epitope of a 1998 human influenza virus neuraminidase forms a highly hydrated interface in the NA-antibody complex[J].Journal of Molecular Biology,2009,356:661-663. |
| 〔2〕 | Maurer-Stroh S,Ma J,Lee R,et al.Mapping the sequence mutations of the 2009 H1N1 influenza A virus neuraminidase relative to drug and antibody binding sites[EB/OL].Biomed central http://www.Biology-direct.com/content/4/1/18. |
| 〔3〕 | Russell1 RJ,Haire LF,Stevens DJ,et al.The structure of H5N1 avian influenza neuraminidase suggests new opportunities for drug design[J].Nature,2006:1-5. |
| 〔4〕 | Hurt AC,Ernest J,Deng YM,et al.Emergence and spread of oseltamivir-resistant A(H1N1) in fluenza virusesi n Oceania,South East Asia and South Africa[J].Antiviral Research,2009,83:90-93. |
| 〔5〕 | 卫生部办公厅.关于印发《甲型H1N1流感监测方案(第二版)》的通知[S].北京:卫生部,卫发明电(2009)184 号. |
| 〔6〕 | 郭永豪,张明瑜,蒋红丽,等.甲型H1N1流感病毒抗原HA基因进化分析[J].中国公共卫生,2011,27(1):28-30. |
| 〔7〕 | World Health Organization.World Health Organization Recommendation for composition of influenza vaccine[EB/OL].Influenza Research Database.http://www.fludb.org/brc/vaccineRecommend.do?Decorator=influenza. |
| 〔8〕 | 祝小平,方刚,李天舒,等.中国内地首例甲型 H1N1流感病例发现与处置[J].预防医学情报杂志,2009,25(7):504-507. |
| 〔9〕 | 莫建军.甲型H1N1流感流行病学研究进展[J].公共卫生与预防医学,2010,21(1):69-72. |
 2012, Vol. 28
2012, Vol. 28


