流感病毒外表面的血凝素蛋白(hem agg lutinin,HA)是流感病毒的重要抗原,也是流感疫苗主要的组成部分。在流感爆发的初期,如果对HA能预先免疫,会保护大约10%~40%的人群免于流感病毒侵袭〔1〕。因此,研究分析流感病毒HA基因的谱系进化关系,及时发现表面抗原突变株系,对于流感病毒疫苗的开发及及时有效地防控流感传播具有重要意义。为此,本研究对分离自河南省的51株甲型H 1N1流感病毒的HA编码基因序列进行了分析。结果报告如下。
1 材料与方法 1.1 材料(1)甲型H 1N1流感病毒:51株(河南省流感实验室);(2)试剂与仪器:犬肾细胞(MDCK细胞) MDCK传代细胞系,由国家流感中心提供并按照国家流感中心提供的标准操作规程培养细胞〔2〕;抗A (季节性H 1N1)血清、抗A (季节性H3N2)血清,抗B (V ictoria)血清,抗B (Yam aga ta)血清和抗A (甲型H 1N1)血清(国家流感中心);RN easyM in iK it,One step RT-PCR试剂盒(德国QIAGEN公司);RNA Protecter (瑞士罗氏公司);
1.2 方法 1.2.1 阳性样本收集由河南省各市(地)疾病预防控制中心和流感监测哨点医院采集流感患者咽拭样本,所有样本采集后-70 ℃冷冻保存,采用荧光定量PCR进行病毒核酸检测,检测结果为甲型H 1N1流感病毒核酸阳性的标本,每周1次专车专人送至河南省流感实验室。
1.2.2 病毒分离培养与分型咽拭样本接种MDCK细胞,富集培养3 d后镜检铺满培养瓶时,取25μL加于含有磷酸缓冲液(PBS)的血清板中,稀释3个数量级,然后用1。5%的O型血红细胞滴定做红细胞凝集实验,初步筛选甲型H 1N1流感病毒〔2〕。初筛阳性标本传接二代,富集病毒后,再进行1次红细胞凝集实验筛选,然后使用1%鸡红血球和亚型血清,分别进行红细胞凝集抑制实验(H I)检测,对分离毒株进行分型,最后鉴定分离到的新型H1N1流感病毒〔2〕保存备用。
1.2.3 引物根据GeneBank已经公布的甲型H 1N1流感病毒的序列信息,采用国家流感中心和世界卫生组织公布的引物序列〔3〕,对相应的引物库筛选,选择2对引物,序列如下:HA-1-F1:5'-tgt aaa acg acg gcc ag t a ta cga cta g ca aaa gca ggg g-3',HA-2-F1124:5'-tgt aaa acg acg gcc ag t tgg atg g ta ygg tta yca y ca-3',HA-1-R1204:5'-cag gaa aca g ct atg acc caa tgg crt ty t g tg tgc tc-3',HA-2-R1778:5'-cag gaa aca g ct atg acc g tg tca g ta gaa aca agg gtg ttt-3'。每条正反向引物前面分别加上-tgt aaa acg acg gcc agt和-cag g aa aca g ct atg acc的测序引物序列。引物由上海生工公司合成测序。
1.2.4 病毒RNA提取与cDNA制备采用RNeasy Mini Kit提取病毒总RNA,采用荧光定量PCR进行鉴定;采用One step RT-PCR试剂盒一步扩增目的cDNA,RNA酶抑制剂采用RNA Protecter。逆转录-取合酶键反应(RT-PCR)步骤:50 ℃逆转录30 m in;95 ℃失活逆转录酶15 m in,并同时启动热启动DNA聚合酶;94 ℃变性30 s,52 ℃退火45 s,72 ℃延伸1.2 min,共40个循环;然后72 ℃保持10 min。
1.2.5 目的基因测序PCR产物经1%琼脂糖电泳纯化,采用T iangen的凝胶回收试剂盒回收cDNA产物。然后将纯化好的cDNA送上海生工公司测序。随机选取2株突变的序列,将未经培养的咽拭样品直接抽提RNA,然后重复RT-PCR及测序流程,以排除病毒培养过程中可能发生的突变。
1.3 比对分析采用美国国立生物信息中心(NCB I)的核酸序列搜索工具(BLAST)〔4〕进行氨基酸序列比对,采用分子进化遗传分析软件Mega 4.1〔5〕绘制进化树。
2 结果 2.1 毒株分离与扩增片断(图 1)共分离到51株新型H 1N1流感病毒,传二代以后的H I滴度均> 640。随机选择20株,提取RNA,然后RT-PCR扩增HA编码基因的不同部分,成功扩增18株HA编码基因(GenBank编号:CY 064390~CY 064407)。图 1可见,扩增出的2部分基因片段分别为1 024和654 bp,与预期大小相符。
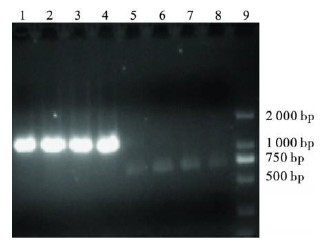 | 注:1~4:引物HA-1-F1和HA-1-R1204扩增的片段;5~8:引物HA- 2-F1124和HA-2-R1778扩增的片段;9:DNA ladder。 图 1 RT-PCR扩增HA 基因cDNA 的部分结果 |
分别对扩增的2个片段采取正反向测序,拼接后得到1 732-1 766 bp的片段。开放阅读框(ORF)全长均为1 701 bp (包含终止密码子),编码533个氨基酸。BLAST比对结果显示,与已经提交的其他甲型H 1N1流感病毒的核酸序列相比,同源性为99%。
2.3 氨基酸突变位点比对根据测序得到的cDNA序列分析,所有得到的HA基因编码序列均编码533个氨基酸。对分离株18个序列进行核酸和蛋白序列比对分析,结果显示,有14个序列发生了533位碱基T/C突变,对应着氨基酸序列145位S/P突变。
2.4 进化树分析(图 2)根据BLAST比对结果,结合序列提交的先后顺序和地域关系,随机选取533位T/C突变序列和无突变序列,分析与其他国家或地区分离株的进化关系。图 2可见,进化树共分为3个簇,第1簇包含本实验室分离到的无突变基因序列,和这个序列在一个进化分支簇的其他序列,均为未发生433位T/C碱基突变的序列,分布于世界各地,是最早起源于墨西哥的甲型H1N1流感病毒在全世界范围内流行的结果。中间1个进化分支簇包含来自中俄接壤的1个城市和美国加州的1个序列,该分支中来自俄罗斯哈巴洛夫斯克(H abarovsk)的序列中包含433位T/C突变位点,在进化关系上处于第1分支和第3分支的中间地带。在第3个进化分支簇中,包含本文中分离到的突变序列在内的所有序列,均包含有433位T/C碱基突变,对应氨基酸序列N端145 S/P的突变。
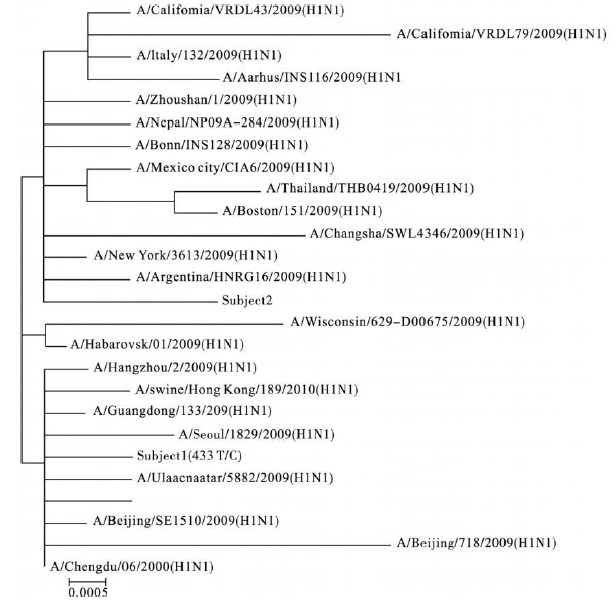 | 注:sub ject1为本实验室分离株433位碱基发生S/P突变序列(毒株编号CY064402);sub ject2为433位碱基无突变序列(毒株编号CY064400) 图 2 本实验室2 株甲型H1N1流感病毒分离株与BLA ST上其他毒株HA 基因的进化关系 |
BLAST比对结果显示,早期提交GeneB ank的HA基因序列中,未发现N端145位氨基酸为Pro的序列。2009年10月以后,S/P突变较早出现在编号为GU 057010~21的12个序列中,该序列的毒株来自于中国成都地区。将带有突变的氨基酸序列输入BLAST比对,结果显示,在统计出来的序列中,有45个序列发生了S/P的突变,其中有3条突变序列来自美国(gbACZ 17140.1,gbADE 28941,gbADE 28961),其余突变均来自于中国及与中国接壤的周边地区。2009年10月以后,这个位点的突变株在我国有逐渐扩大流行的趋势,先后在中国西安(ADD 84493)、北京(ADD 64208,ADD 64204)、成都(ACX 47370,ACX 47367)、广东(ADE 45499)、杭州(CY 047713)等地区,以及韩国首尔(ADF 80478)和中俄(ADB 82944)、中蒙(ACZ 18239)接壤地区的外方境内不断分离到该位点突变的毒株。而在同一时期的欧美等地区分离到的毒株,很少(仅3株)有N端145位氨基酸有明显的位点突变。
3 讨论2009年5月13日,中国发现第1例输入性病例以后,此后一直持续到2009年8月底,基本上以输入性病例为主〔6〕,能够明确判断传染源。自2009年9月份开始,我国开始出现大量的本土病例,无法查证传染源,至2009年12月底甲型H1N1流感病毒流行达到高峰。在2009年9月份以前提交的甲型H1N1流感病毒的HA基因序列中,未发现145位S/P的突变。本土病例呈现流行到爆发以后,S/P的突变序列不断增加。根据文献报道,甲型H 1N1流感病毒流感病毒血凝素上抗体结合位点A由开始于154位的PHAGAK组成7,所以145位S/P的突变没有影响抗体结合位点的保守性,145位的氨基酸位于重链突出的loop部分〔8〕,所以S/P的突变也不会改变氨基酸链折叠或者是螺旋的走向,以Ru iXu等解析的甲型H 1N1流感病毒的HA晶体结构为模板〔7〕,运用Swissmodel模拟建模以后〔8, 9〕,在Pymo l软件中比对分析,也证明HA的三维结构没有改变。总体分析,这一位点的突变没有改变甲型H 1N1流感病毒的抗原性,这一现象在H I检测中也得到了证实。
对比分析这一分支中的序列地域分布,发现中国大陆分布较多,其他周边国家和地区也有分布,其中来自中国香港的一个序列是分离自猪感染人甲型H 1N1流感病毒,并在猪体内得到重组以后的序列。从这一现象分析,甲型H 1N1流感病毒HA基因在猪体内也存在类似的进化关系。
| 〔1〕 | Skehel JJ, Wiley DC.Receptor binding and membrane fusion in virus entry:the in fluenza hemagglutin in[J], A nnu Rev Biochem, 2000, 69(45):531-569. |
| 〔2〕 | 卫生部办公厅.关于印发甲型H1N1流感监测方案(第二版)的通知(卫发明电2009 184号)[S].北京:卫生部, 2009. |
| 〔3〕 | WHO.Global A lert and Response(GAR)/PandemicH1N12009[EB/OL].[2010-02-20].http://www.who.int/csr/disease/swineflu/en/index.html. |
| 〔4〕 | National Center for Biotechnology Information.BLA STA ssem bled RefSeq Genomes[EB/OL].[2010-02-20].http://blast.ncbi.nml.nih.gov/Blast.cgi. |
| 〔5〕 | Sudhir K, Masatoshi N, Joel D, et al.MEGA:a biologist centric soft ware for evolution aryanalys is of DNA and protein sequences[J]. Briefings In Bioinfomratics, 2008, 9(4):299-306. |
| 〔6〕 | 中国网.卫生新闻/疾控中心流行病科学家:甲流本土病例开始占优势[OL].(2009-10-22)[2010-02-20].http://china.com.cn. |
| 〔7〕 | Xu R, Ekiert DC, Krause JC, et al.Structural basis of preexisting immunity to the 2009H1N1pandemic in fluenza virus[J].Science, 2010, 328(357):357-360. |
| 〔8〕 | Arnold K, Bordoli L, K opp J, et al, The SWISS MO DEL work space:a web based environment for protein structure homology modeling[J].Bioin formatics, 2006, 22(2):195-201. |
| 〔9〕 | Kiefer F, Arnold K, Knzli M, et al, The SW ISS M ODEL repository and associated resources[J].Nucleic Acids Research.2009, 37[Database issue] :D387-D392. |
 2011, Vol. 27
2011, Vol. 27


