2. 山东大学公共卫生学院
甲型流感病毒抗原变异率高,容易造成大流行。20世纪发生了4次流感大流行,均为甲型流感病毒抗原变异所致。2009年4月以后发生的流感大流行,是由甲型流感病毒变种-甲型H1N1流感病毒所致〔1〕。为了解该病毒抗原变异及进化特点,本研究收集2009年9月-2010年1月山东省流感监测网络实验室分离到的甲型H1N1流感病毒株,对其中神经氨酸酶(neuram in idase,NA)基因的变异特点进行了对比分析。现将结果报告如下。
1 材料与方法 1.1 材料(1)甲型H1N1流感病毒山东分离株:20株(山东省流感监测网络实验室分离保存),其中2009年9-12月分离株16株,2010年1月分离株4株。(2)参照比对毒株:山东省首例输入性甲型H1N1流感病毒株(编号:A.Shandong.1-2009);2009年四川省分离的中国第1株甲型H1N1流感病毒(编号:A.S ichuan.SWL1-2009);新型甲型H1N1流感病毒疫苗株[A.C aliforn ia.07-2009(H1N1)];2009年广东分离株(A.Guangdong.1102-2009)、澳大利亚分离株(A.Australia.82-2009)、意大利罗马分离株(A.Rome.630-2009)。(3)试剂:RNA easy minikit试剂盒(Cat NO 74104,德国Qiagen公司);QIAGEN One Step RT.PCR K it试剂盒(Cat NO 210212,德国QIAGEN公司)。
1.2 方法(1)病毒RNA提取:采用RNA easy minikit试剂盒(Cat NO 74104)提取甲型H1N1流感病毒RNA,具体操作方法参照使用说明书。(2)引物:网上下载部分甲型H1N1流感病毒NA基因序列进行比对,在NA基因的保守区域自行设计神经氨酸酶(NA)基因的扩增引物(SWH.NA1和SWH.NA2),引物序列由北京华大基因有限公司合成,其中正义引物SWH.NA1(28-45)为:5'CCA TTG GTT CGG TCT GTA 3';反义引物SWH .NA2(1380-1397)为:5'AAA TGG CAA CTC AGC ACC 3'。(3) RT.PCR反应:扩增反应体系按照QIAGEN One Step RT.PCR Kit试剂盒(Cat NO 210212,)操作,反应条件:60℃1 min,42℃10 min,50℃30 mim,95℃15 min,PCR循环如下:94℃40 s,62℃40 s,72℃2 m in,35次循环,72℃10 m in。(4)序列测定和分析:PCR产物经1%琼脂糖凝胶电泳后,送北京华大基因有限公司进行纯化并测序。
1.3 比对与序列分析用DNAStar 4.0软件(DNASTAR Inc.,M ad ison,W I,USA)对测得序列进行编辑,并用DNastar软件包中的Ed itSeq程序和M egA lign程序进行同源性分析,计算SequenceD isatances.根据各NA基因可读框(ORF)的碱基序列,用MEGA 4.0软件推导氨基酸序列,再用MEGA 4.0软件(Kumar,Tamura,Ne i2004),采用邻位相临法(N J)(Boot strap=1 000)绘制基因种系发生树;根据推导的蛋白序列,分析神经氨酸酶(NA)蛋白的抗原位点、酶活性中心及周围相关位点和糖基化位点的变异,并与2009年其他国家或地区分离株及新型甲型H1N1流感病毒疫苗株进行对比。
2 结果 2.1 同源性比较同源性比对结果可见,山东省2009年9月-2010年1月分离的20株甲型H1N1流感病毒NA基因的同源性均> 99.2%,其中有4株的同源性为100%;与山东省首例输入性甲型H1N1流感病毒分离株(A.Shandong.1-2009)以及广东分离株同源性较高,均> 99.5%,其中1株与广东株的同源性为100%;与疫苗株、四川分离株的同源性略低,分别为99.1%~99.5%和99.1%~99.6%;与罗马分离株、澳大利亚分离株分别为99.2%~99.7%和99.3%~99.8%。
2.2 系统进化树分析(图 1)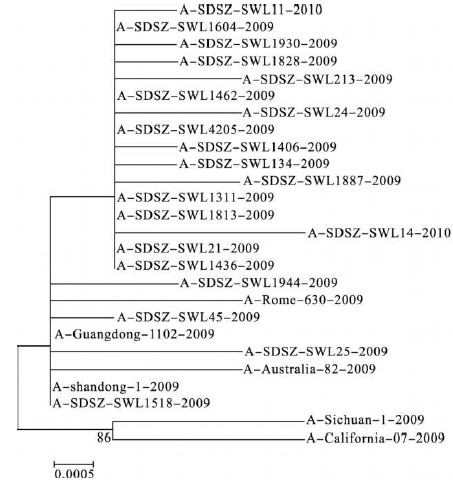 | 图 1 山东省2009-2010年甲型H1N1流感病毒NA 基因进化树 |
用M ega 4.0软件对山东省分离的20株甲型H1N1流感、其他国家或地区分离株、国内代表株及WHO推荐的甲型H1N1流感疫苗株NA基因构建系统进化树。结果显示,山东省20株甲型H1N1流感与其他国家或地区流行株之间的亲缘关系相对较近,共同构成了一个大的分支,与国内代表株及WHO-推荐疫苗株的分支相并列,这与同源性比较的结果一致。2009年分离的毒株与2010年分离毒株在进化分析上无明显差别,均分布在一个小的分支上。
2.3 NA蛋白位点氨基酸序列变异分析为进一步分析20株甲型H1N1流感病毒NA的遗传进化关系,本实验用Mega 4.0软件对所有NA基因的ORF编码氨基酸序列进行了推导。Mega 4.0对氨基酸序列进行进化分析未发现NA编码区存在氨基酸的丢失与插入。(1)抗原位点:与疫苗株A/Cali.fornia/07/2009和国内代表株A/S ichuan/SWL1/2009氨基酸序列比较,部分毒株的NA氨基酸序列在第21位点天冬酰胺-苏氨酸(N.T);第23位点异亮氨酸-精氨酸(I.R);第25位点谷氨酰胺-组氨酸(Q.H);第42位点天冬酰胺-丝氨酸(N.S);第43位点谷氨酰胺-脯氨酸(Q.P);第60位点苏氨酸-天冬酰胺(T.N);第76位点丙氨酸-天冬氨酸(A.D);第94位点缬氨酸-异亮氨酸(V.I);第106位点缬氨酸-异亮氨酸(V.I);第122位点异亮氨酸-缬氨酸(I.V);第248位点天冬酰胺-天冬氨酸(N.D);第307位点天冬酰胺-丝氨酸(N.S);第351位点酪氨酸-苯丙氨酸(Y.F)发生了变异,其中第106位、第248位和第351位在20株甲型H1N1流感病毒中均发生了变异。(2)酶活性中心及周围相关位点:序列分析结果显示,NA蛋白酶活性中心R118、D151、R152、R225、E277、R293、R368和Y402等催化部位的8个氨基酸位点,以及周围辅助位点E119、R156、W 179、S180、D/N199、I223、E228、H 275、E278、N295、E425等11个氨基酸位点〔2, 3, 4〕,均未出现氨基酸替换。(3)糖基化位点:测序比对结果显示,NA蛋白上第50、58、63、68、88、146、235、386位点等8个糖基化位点〔5〕未发生改变,序列为天冬酰胺(N)-任一氨基酸(X)-苏氨酸( T) /丝氨酸(S)。
3 讨论本研究主要对山东省分离的甲型H 1N1流感病毒的NA基因及氨基酸序列的同源性、抗原性位点、糖基化位点和进化树进行了分析,并与WHO-疫苗推荐株A/Ca lifornia/7/2009、国内代表株A/S ichuan/SWL1/2009及其他国家或地区的分离株进行了比较。
山东省甲型H 1N1流感病毒流行株的NA基因序列与疫苗株、国内代表株及其他国家、省市流行株进行比较,同源性> 99.1%,基因进化树显示山东省流行株与其他国家、省市所分离的流行株在一个大的分支上,亲缘关系比较近,与疫苗株、国内代表株处于另一分支上,亲缘关系较远,说明山东省流行株NA基因在流行期间保守型比较强,同源性较高,部分能达到100%的同源性,与Jandaghi等〔5〕报道一致。
抗原决定簇的改变可能导致传染性的改变,抗原决定簇大多存在于抗原的表面,但也有隐藏在抗原内部的,只有内部的抗原决定簇暴露出来才能发挥抗原决定簇的作用,据报道NA蛋白N端第140~157位氨基酸很有可能是抗原决定簇位点〔6, 7, 8〕。与疫苗株比较,NA蛋白的V106I、N248D、Y351F 3个位点在20株流行株中均发生了改变;N21T、I23R、Q25H、N42S、Q43P、T60N、A76D、V94 I、I122V和N307S在部分流行株中发生了改变,但这些位点均未导致抗原决定簇的改变。Po tdar等〔9〕同时也报道了印度分离的病毒NA蛋白的第106位(V106I)和第284位(N248D)发生了相关变异,但并未导致抗原性改变。
NA蛋白的作用是水解细胞膜上各种多糖受体末端的N-乙酰神经氨酸,清除病毒表面和感染细胞表面糖蛋白末端的唾液酸,防止子代病毒的自身凝集,促使病毒释放。本研究对NA蛋白的氨基酸变异分析表明,纳入本次研究的山东20株甲型H 1N1流感病毒的蛋白酶活性中心及周围辅助区的氨基酸组成均非常保守,未发生变异。由于NA蛋白在流感病毒复制和传播中发挥重要作用,并且酶活性中心的氨基酸组成高度保守,因此以NA为靶点开发选择性抑制剂具有非常重要的意义。目前针对甲型H 1N1流感有效的磷酸奥司他韦(oseltamivir phosphate,达菲)正是NA抑制剂,它能够与病毒的NA特异性结合,阻断该酶的活性,使病毒不能轻易地从感染细胞表面释放,促进病毒凝集,阻止病毒进一步扩散,从而发挥抗流感作用〔10〕。在分离的具有耐药性的流感毒株中,主要是NA蛋白的H 275Y发生改变,H 275Y突变株对奥司他韦的敏感性能降低1 466倍〔11〕。2007-2008年冬季,欧美国家出现携带H 275Y突变的奥司他韦耐药流感病毒流行,耐药株的出现与用药无关,其易感人群、引起的临床症状和患者预后都与奥司他韦敏感株相似,表现为普通季节性流感活动,并可能在人群中持续存在〔12, 13, 14〕。目前,已有丹麦、日本、中国香港和湖南省〔15〕相继报道奥司他韦耐药的新型甲型H1N1流感病例,这4株病毒的NA基因同源性达到99.9%,说明我国已经出现具有奥司他韦耐药特征性变异的新型甲型H 1N1流感病毒。山东省分离的20株流行株的第275位没有发生改变,因而仍对达菲具有敏感性。目前全世界发现的对奥司他韦耐药的新型甲型H 1N1流感病例还只是"个例",尚不会对公共卫生安全造成重大影响,但该耐药病毒株的出现提醒我们必须保持警惕,密切监测病毒变化,防止发生耐药株进一步传播扩散。
糖基化位点的增加或减少对病毒的抗原性及其生物学特征具有一定的影响,是决定流感病毒感染性和致病性的重要因素〔16, 17〕,山东省分离的20株流行株具有完整的8个潜在糖基化位点,没有发生改变。
综上所述,2009年新型甲型H 1N1流感病毒的NA基因在基因进化、NA蛋白潜在抗原位点和糖基化位点上均高度保守。另外,流感病毒NA蛋白的酶活性位点和周围相关位点高度保守,仍为抗流感药物研发的研究靶点。
(刘倜、林艺共同为第一作者)| 〔1〕 | Novel Swine origin In fluenza A(H1N1)Virus Investigation Team.Emergence of a novel swine origin influenza in humans[J]. N Engl J Med, 2009, 360(25):2605-2615. |
| 〔2〕 | Richard M, Del agea C, Barth l mya M, et al.Impact of influenza A virus nerram in idase muations on the stability, activity, and sensibility of the neuram in idase to neuram inidase inhibitors[J].J C lin Viro, l 2008, 41(1):20-24. |
| 〔3〕 | Chutin miitkul S, Chieochan sin T, Payungporn S, et al.Molecular characterization and phylogenetic analysis ofH1N1and H3N2 human in fluenza A virus among in fants and children in Thailand[J]. Virus Res, 2008, 132(1-2):122-131. |
| 〔4〕 | Yen HL, Ilyushina NA, S alomon R, et al.Neuram in idase inhibitor resistant recom binant A/Vietnam/1203/04(H5N 1)influenzaviru sesreta in their replication efficiency and pathogenicity in vitro and in vivo[J]. J Virol, 2007, 81(22):12418-12426. |
| 〔5〕 | Jandaghi NZ, A zad TM, N aseriM, et al.M olecular and genetic characteristics of hemagglutin in and neuram in idase in Iranian 2009 pademic in fluenza A(H1N1)viruses[J].Arch Virol, 2010, 155(5):717-721. |
| 〔6〕 | Russell R J, Haire LF, Stevens D, J et al.The structure of H5N1 avian influenza neuram in idase suggests newopportun it ies for drug design[J]. Nature, 2006, 443(7107):45-49. |
| 〔7〕 | 黄平, 俞守义, 柯昌文.逐步预测和统计学筛选人H5N1毒株神经氨酸酶蛋白B细胞表位[J]. 科学通报, 2008, 53(22):2748-2753. |
| 〔8〕 | Parida R, Shaila M S, M ukherjee S, et al.Computational analysis of proteome of H5N1 avian in fluenza virus to define Tcell epitopes with vaccine potential[J].Vaccine, 2007, 25(43):7530-7539. |
| 〔9〕 | Pordar VA, Chadha M S, Jadhav SM, et al.Genetic characterization of the in fluenza A pademic(H1N1)2009 virus isolates from India[J]. PLos One, 2010, 5(3):e9693. |
| 〔10〕 | G ubareva LV, K aiser L, H ayden FG.In fluenza virus neuram in idase inhibitors[J].Lancet, 2000, 355(9206):827-835. |
| 〔11〕 | Hurt AC, Ernest J, Deng YM, et al.Emergence and spread of oseltamivirres istant A(H1N1)in fluenza viruses in Oceania[J].South East Asia and South A frica1 Antiviral Res, 2009, 83(1):90. |
| 〔12〕 | Meijer A, Lackenby A, Hungnes O, et al.Oseltamivir resistant in fluenza virus A(H1N1), Europe, 2007-08 season[J] Emerg Infect Dis, 2009, 15(4):552. |
| 〔13〕 | G ooskens J, Jonges M, C laas EC, et al.Morbidity and mortality as sociated with nosocomial transmission of oseltamivir resistant influenza A(H1N1)virus[J]. JAMA, 2009, 301(10):1042. |
| 〔14〕 | D haran N, J G ubareva LV, M eyer JJ, et al.In fections with oseltami virres istant influenza A(H1N1)virus in the United States[J].JAM A, 2009, 301(10):1034. |
| 〔15〕 | World Health Organization.Pandemic(H1N1)2009 update 601[EB/OL].http://www.who.int/csr/don/2009_08 _04/en. |
| 〔16〕 | Abe Y, Takashita E, K anetsu S, et al.Effect of the addition of oligosaccharides on the biological activities and an tigenicity of in fluenza A/H3N2 virus hem aglut in in[J].Journal Virology, 2004, 78(18):9605-9611. |
| 〔17〕 | Liu M, Chen H, Luo FL, et al.Deletion of N glycosy lations ites of hepat it is Cvirus envelope protein E1 enhances specific cellular and humoral immune responses[J].Vaccine, 2007, 25(36):6572-6580 |
 2011, Vol. 27
2011, Vol. 27


