细菌性食物中毒是食源性疾病最主要的危险因素,其中沙门菌是常见的食源性疾病的首要病原菌之一〔1〕。肠炎沙门菌是沙门菌的一种常见血清型,也是引起食物中毒的重要病原菌。监测肠炎沙门菌的耐药性及其耐药基因的变异情况,对人类感染沙门菌的预防和控制具有重大意义。本研究对北京、新疆、广州3个地区的肠炎沙门菌临床分离株进行抗生素敏感性试验并对其耐药基因DNA促旋酶gy rA基因和拓扑异构酶parC基因进行PCR扩增和序列分析,旨在为临床治疗提供参考依据。
1 材料与方法 1.1 材料 1.1.1 菌株来源菌株分离自200份腹泻标本(北京地区 49份,广州43份,新疆108份),腹泻标本由3个地区的医院提供。
1.1.2 试剂LB肉汤增菌液、沙门/志贺菌选择性(Salmo-nella/Shigella,SS)琼脂、克氏双糖、营养琼脂、米勒-欣顿 (Mueller-Hinton,MH)琼脂(北京陆桥公司)、沙门菌属诊断血清(Murex,Dartford,United Kingdom)、0.85% NaCl、药敏纸片 (美国Oxiod公司)、细菌基因组DNA提取试剂盒(天根公司)、10 X ExTaq Buffer、ExTaq、dNTP、DL2000 DNA Marker (英国TAKARA公司)、引物(上海生工公司)。
1.1.3 主要仪器PCR仪、水浴锅、37℃温箱、37℃摇床、台式高速离心机、电泳仪、电泳槽。
1.2 方法 1.2.1 菌株的分离鉴定将3地区上送的菌液接种于LB肉汤增菌液37℃摇床培养过夜,次日,接种于沙门/志贺菌选择性SS平板37℃温箱过夜,然后挑取单克隆菌落接双糖斜面 37℃过夜,对疑似沙门菌的菌株用沙门菌属多价血清鉴定为沙门菌属后,再用沙门菌属诊断血清确认血清型。
1.2.2 抗生素敏感性试验采用K-B药敏纸片法对肠炎沙门菌进行抗生素敏感性试验。大肠埃希菌ATCC25922和金黄色葡萄球菌25923作质控,结果判读采用美国临床实验室标准化协会(CLSI)标准〔2〕。
1.2.3 gyrA、parC基因的PCR扩增用DNA提取试剂盒提取基因组做模板,模板浓度稀释至100 ng/μL,根据沙门菌耐药基因gyrA、parC的喹诺酮耐药决定区(quino loneresistance-determining reg ion,QRDR)查找引物,引物序列参考文献〔3〕,由上海生工生物技术有限公司合成。PCR反应体系如下:50 μL体系,10 × EXTaq buffer (Mg2+plus)5 μL,EXTaq 0.3 μL,dNTP 4 μL,模板2 μL,上下游引物各1.5 μL,加灭菌纯水至50 μL。反应条件:94℃5 min,94℃30s,55℃30 s,72℃1 min,30个循环,最后72℃延伸5 min。1%琼脂糖凝胶电泳检测PCR扩增结果。
1.2.4 测序及分析PCR产物送北京中科希林公司测序,采用双向测序,应用DNAStar软件整理序列后输入NCBI数据库中与野生敏感株Ty2进行blast分析。
2 结 果 2.1 菌株分离鉴定共分离鉴定出20株肠炎沙门菌新疆地区7株,广州地区8株,北京地区5株。
2.2 抗生素敏感性试验结果表明所有菌株均对环丙沙星、 庆大霉素、头孢他定和亚胺培南敏感,95%(19/20)的菌株对氟哌酸、氯霉素和左氧氟沙星敏感,比较敏感的有头孢噻肟 75%(15/20)、氨苄西林/舒巴坦75%(15/20)、阿莫西林/克拉维酸70%(14/20)。对萘啶酸普遍耐药,其次是氨苄西林 75%(15/20)、甲氧苄啶/磺胺甲噁唑50%(10/20)、头孢噻肟 15%(3/20)、四环素5%(1/20)。耐3种或3种以上抗生素的菌株达75%(15/20),其中广州地区的多重耐药(Multidrug resistance,MDR)菌株占35%(7/20),新疆占30%(6/ 20),北京占10%(2/20)。
2.3 PCR产物电泳结果(图 1,2) | 注: M: DNA m ark er D2000; 1 ~ 10: 10株肠炎沙门菌gyrA 基因PCR扩增目的片段; 11: 阴性对照。 图 1 部分菌株gyrA 基因PCR 产物电泳图 |
 | 注: M: DNA m arker D2000; 1~ 10: 10 株肠炎沙门菌parC 基因PCR扩增目的片段; 11: 阴性对照。 图 2 部分菌株parC基因PCR 产物电泳图 |
20株菌gy rA、parC基因 PCR扩增结果均为阳性,目的片段大小分别为620,413 bp。
2.4 耐药基因测序
与野生敏感株Ty2序列比对结果显示,20株肠炎沙门菌gyrA基因Asp8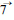 及Gly133密码子处发生了点突变,3株Asp8
及Gly133密码子处发生了点突变,3株Asp8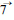 % Asn、2株Asp8
% Asn、2株Asp8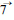 % Gly、15株Asp8
% Gly、15株Asp8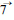 % Tyr,20株Gly133% Glu,其中Gly133是新的突变点。未发现parC基因密码子突变。
% Tyr,20株Gly133% Glu,其中Gly133是新的突变点。未发现parC基因密码子突变。
本研究结果显示,肠炎沙门菌对庆大霉素、头孢他定、亚胺培南的敏感率达100%,未检测到对环丙沙星耐药,这与某些报道一致〔4, 5〕。左氧氟沙星、氯霉素、氟哌酸的敏感率也较高,达95%(19/20),可为肠炎沙门菌感染的临床治疗提供一定参考。
近年来,肠炎沙门菌的耐药日益加重,对喹诺酮类等一线抗生素耐药的多重耐药(MDR)现象越来越多。陈建辉等〔6〕报道了一起由严重多重耐药的肠炎沙门菌引起的婴幼儿迁延性腹泻。本研究结果显示,20株肠炎沙门菌中75%(15//20) 的菌株对3种或3种以上抗生素耐药,其中广州地区的多重耐药(MDR)菌株占35%(7/20),新疆占30%(6/20),北京占 10%(2/20)。本研究中的菌株对萘啶酸均耐药,这可能与gyrA基因喹诺酮耐药决定区(QRDR)的突变相关。所有菌株的gy rA基因喹诺酮耐药决定区(QRDR)均存在Asp87、Gly133密码子的突变,Ser83和Asp87是喹诺酮类耐药株最常见的gy rA基因的突变密码子〔7, 8, 9, 10, 11〕,本研究未发现Ser83密码子突变,而普遍存在Asp87突变。gyrA基因编码DNA促旋酶的A亚基,包含喹诺酮耐药决定区,是一种Ⅱ型拓扑异构酶,在DNA超螺旋的形成中起重要作用,是喹诺酮类药物作用的首要靶位,parC基因编码DNA拓扑异构酶IV的一个亚基,与 gyrA基因是同源的,该基因编码喹诺酮类药物作用的次级靶位〔12〕。本研究未发现parC基因突变。G ly133密码子突变是新的突变,其对沙门菌耐药性的影响和作用尚不明确,有待进一步的研究。肠炎沙门菌临床分离株多重耐药情况严重及可能与抗生素的使用不当有关,腹泻患者就诊前不规范服用抗生素或者医院治疗时不合理的滥用抗生素导致了细菌对萘啶酸、氨苄西林、甲氧苄啶/磺胺甲噁唑、头孢噻肟、四环素的耐药,给沙门菌感染的控制和治疗带来不利影响。为防止多重耐药的蔓延和加重,应加强对细菌耐药性和耐药基因的监测,并规范临床及畜牧业抗生素的应用。
| 〔1〕 | 黄金林,焦新安,刘佩红,等.PCR快速检测沙门菌试剂盒的研制与应用.[J].中国公共卫生,2004,20(4):452-252. |
| 〔2〕 | Performance standards for antmiicrobial susceptibility testing:Nineteenth Informational Supplement.Clinical and Laboratory Standards Institute(CLSI)document M 100 S19[R].Wayne,PA:Clinical and Laboratory Standards Institute;2009,29(3):34-38. |
| 〔3〕 | Ling J,Chan E,Lam A,et al. Mutations in topoisomerase genes of fluoroqu inolone resistant Samlonellae in Hong Kong[J].Antmiicrobial Agents and Chemotherapy,2003,47(11):3567-3573. |
| 〔4〕 | Diasde Oliveira S,Siqueira Flores F,Dos Santos L,et al. Antmii crobial resistance in Salmonella enteritid is strains isolated from broiler carcasses,food,human and poultry related samples[J].International Journal of Food Microbiology,2005,97(3):297-305. |
| 〔5〕 | 周俊英,付有荣,郭清莲,等.生鲜肉中肠炎沙门菌分子生物学特征分析[J].中国公共卫生,2007,23(10):1227-1228. |
| 〔6〕 | 陈建辉,李海丹,谢一俊,等.严重耐药肠炎沙门菌引起婴幼儿迁延性腹泻1例报告[J].海峡预防医学杂志,2002,8(002): 78-78. |
| 〔7〕 | Izumiya H,MoriK,Kurazono T,et al. Characterization of isolates of Salm onella enterica serovar Typhmiurium displaying high level fluo roquinolone resistance in Japan[J].Journal of Clinical Microbiology,2005,43(10):5074-5079. |
| 〔8〕 | Hirose K,Hashmioto A,Tamura K,et al. DNAsequence analysis of DNAgyrase and DNA topoisomerase IV quinolone resistance determining regions of Samlonella enterica serovar and serovarParatyphi A[J].Antimicrobial Agents and Chemotherapy,2002,46(10): 3249-3252. |
| 〔9〕 | Eaves D,Liebana E,WoodwardM,et al. Detection of gyrAmutations in quino lone resistant Salm onella enterica by denaturing high performancel iquid chromatography[J].Journal of Clinical Microbiology,2002,40(11):4121-4125. |
| 〔10〕 | Levy D,Sharma B,Cebula T.Single nucleotide polymorphism mutation spectra and resistance to quinolones in Saml onella enterica serovar Enteritid is with a mutator phenotype[J].An timicrobial Agents and Chemotherapy,2004,48(7):2355-2363. |
| 〔11〕 | Baucheron S,ChaslusDancla E,Cloeckaert A,et al. High level resistance to fluoroqu inolones linked to mutations in gyrA,parC,and parEin Samlonella enterica serovar Schwarzeng rundisolates from humans in Taiwan[J].An timicrobial Agents and Chemotherapy, 2005,49(2):862-862. |
| 〔12〕 | Casin I,Breuil J,Darchis J,et al. Fluoroquinolone resistancelinked to GyrA,GyrB,and ParC mutations in Salmonella enterica Typhimu riumisolates in humans[J].Emerging Infectious Diseases, 2003,9(11):1455-1457. |
 2010, Vol. 26
2010, Vol. 26

 , 王勇, 王中强, 陈琛, 李婧, 张伶, 杜昕颖, 汪舟佳, 薛文仲, 黄留玉, 宋宏彬, 刘雪林
, 王勇, 王中强, 陈琛, 李婧, 张伶, 杜昕颖, 汪舟佳, 薛文仲, 黄留玉, 宋宏彬, 刘雪林