2. 郑州大学生物工程系
(1)菌株:大肠埃希菌DH5α,临床分离志贺菌耐多药株H24(郑州大学公共卫生学院流行病教研室收集保存); (2)载体:pMD18-T载体(宝生物大连有限公司); (3)工具酶及试剂:SacⅠ内切酶、BamHⅠ内切酶、胰RNA酶、DNA回收试剂盒、SYBR Premix Ex TaqTM荧光扩增体系(宝生物大连有限公司); 抗生素(中国药品生物制品检定所); 酵母提取物和蛋白胨(英国Oxiod公司)。营养肉汤水解酪蛋白培养基(MH)(北京奥博星公司)。
1.2 方法 1.2.1 志贺菌H24基因组DNA提取按文献〔4〕方法进行。
1.2.2 志贺菌临床耐多药株H24外排泵emrE基因片段扩增按文献〔5〕方法进行。
1.2.3 易错聚合酶链式反应(Error prone PCR, EP-PCR)EP-PCR使用的引物与扩增H24外排泵emrE基因使用的引物相同〔5〕, 以用H24为模板扩增的emrE基因为模板。采用MgCl2 25 mmol/L,MnCl2 5 mmol/L EP-PCR反应体系, EP-PCR产物用DNA快速回收试剂盒回收。
1.2.4 序列测定纯化后的易错emrE基因与pMD18-T载体于16 ℃连接过夜, 连接产物转化到E.coli DH5α感受态细胞中。利用蓝白斑法筛选重组克隆; 碱裂解法从菌液中提取质粒,用SacⅠ单酶切及SacⅠ和BamHⅠ双酶切鉴定emrE阳性质粒。验证均为阳性的质粒由上海生工生物工程技术服务有限公司测序。
1.2.5 微量稀释生长曲线法测定菌株耐药性采用MH液体培养基, 在无菌操作下将倍比稀释后不同浓度的抗生素溶液利福霉素、红霉素、硫酸新霉素、盐酸四环素、氯霉素、环丙沙星和盐酸强力霉素分别加到灭菌的96孔板中,每孔10 μl,只加培养基作为阴性对照。0.5麦氏比浊标准的菌悬液,经MH肉汤1:1000稀释后,向每孔中加100 μl,每个处理3个并行重复。置于37 ℃培养箱中震荡培养,4 h后在酶标仪450 nm处测定菌体浓度吸光度(A)值,以后每隔3 h测1次A值, 结果取平均值。为防止染色体突变造成的假阳性结果,所有重组菌株在测得耐药性变化后,再提取质粒重新转化新的大肠埃希菌DH5ɑ菌株。如果再次测得同样的耐药性,则确认耐药是由质粒携带的外源突变基因引起的。
1.2.6 荧光实时定量RT-PCR利用Primer 5.0设计引物,以大肠埃希菌内稳定表达的看家基因gapA基因的380 bp片段作为内参,emrE基因内部的190 bp序列为目的序列,引物由上海生工生物工程技术服务有限公司合成。目的基因引物为GGGTTTACACGGTTATGGC和GGGTTTACACGGTTATGGC,看家基因的引物为CTGGTCCGTCTAAAGACAACA和ACGAACGGTCAGGTCAACTA。分别以对照株pMD (DH5α)和pMD-emrE(DH5α)及emrE基因突变重组株cDNA为模板,同时扩增emrE基因片段及甘油醛3-磷酸脱氢酶gapA基因片段。荧光实时定量PCR采用Rotor-Gene 3000荧光实时定量PCR仪(澳大利亚Corbett Research公司); 采用SYBR Premix Ex TaqTM荧光扩增体系,进行40个循环并于60 ℃记录荧光强度,然后测定熔解曲线。每个样品重复做3管。通过Rotor-Gene 3000分析软件,得到目的基因和看家基因的循环域值(Ct值)和熔解曲线,采用相对定量的Comparative Delta-delta Ct方法,得到目的基因相对的拷贝数。
1.3 生物信息学分析肽链序列比对分析由DNASTAR Inc.的DNAStar Lasergene软件处理。
2 结果 2.1 EP-PCR扩增(图 1)经过条件摸索和优化,2次EP-PCR (epA, epB)扩增产物经1%琼脂糖凝胶电泳分析,均在约1100 bp处有一特异扩增条带,分子量大小与预期扩增值相符。将EP-PCR扩增产物与pMD-18T载体连接,并转入DH5ɑ菌株进行筛选,共得到31株重组株。提取质粒酶切电泳检测后,送样测序。
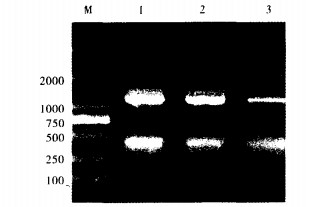
|
注:M:DL2000 Marker; 1, 2:epA-emrE; 3:epB-emrE。 图 1 EP-PCR扩增产物凝胶电泳分析 |
2.2 菌株的耐药性变化(图 2)
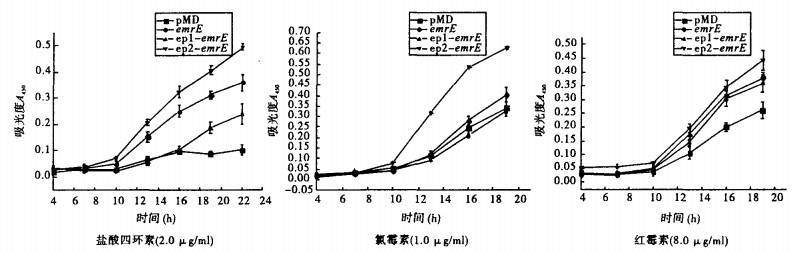
|
图 2 pMD-epl-emrE(DH5ɑ)和pMD-ep2-emrE(DH5ɑ)耐药性变化 |
选用不同EP-PCR变异株pMD-ep1-emrE(DH5ɑ)和pMD-ep2-emrE(DH5ɑ),通过微量稀释生长曲线法测定这2株进化菌株及出发基因对照株pMD-emrE(DH5ɑ)和空质粒对照株pMD (DH5α)对利福霉素、红霉素、硫酸新霉素、四环素、氯霉素、环丙沙星和强力霉素的耐药性。从生长曲线可以看出,pMD-ep2-emrE(DH5ɑ)菌株对四环素、红霉素和氯霉素的抗性比含原始基因株pMD-emrE(DH5ɑ)增高,其中氯霉素为新获得的耐药性, 表明经进化突变的ep2-emrE基因外排活性增强。
2.3 重组菌株中EP-PCR emrE基因表达为近一步确认突变基因的表达情况,对重组菌中emrE基因的表达进行实时荧光定量反转录分析。在溶解曲线中未检测到引物二聚体和其他非特异性荧光信号。荧光实时定量RT-PCR获得pMD (DH5a)、pMD-emrE(DH5ɑ)、pMD-ep1-emrE(DH5ɑ)、pMD-ep2-emrE(DH5ɑ)4个样本的平均Ct值和mRNA相对表达量。其emrE基因的相对表达量分别为1,20300,23600和49000倍。ep2-emrE基因不但得到表达,而且它的mRNA表达量最高。
2.4 ep1-emrE、ep2-emrE的序列分析和氨基酸序列同源性比对对原始emrE和突变基因ep1-emrE、ep2-emrE的DNA测序结果用DNASTAR 7.1.0软件对3个基因序列进行比对。结果显示, 在开放阅读框内ep1-emrE有5个点突变,ep2-emrE有9个点突变。经氨基酸同源性比较,ep1-emrE有5个氨基酸突变,分别是Glu-14→Gly, Met-21→Ile, Ile-54→Met, Ile-71→Asn, Trp-76→Arg; ep2-emrE有6个氨基酸突变, 分别是Thr-28→Ala,Cys-39→Ser,Tyr-40→终止子,Ser-72→Arg,Leu-93→Phe,Ile-101→Phe。ep2-emrE虽然Tyr-40突变成了UAA终止子,但从pMD-ep2-emrE(DH5ɑ)的耐药表现看,肽链合成并没有被完全中止,UAA终止子可能被无义抑制子(nonsense suppressor)所抑制〔6〕。
3 讨论本研究结果表明,在31个突变子中筛选出的突变子ep2-emrE,不但有增强的四环素和红霉素耐药能力,还有新产生的氯霉素耐药能力, 显示这种外排泵基因具有突变增强耐药性的潜力。根据本研究前期结果〔5〕分析,本研究结果尚不能表明这种外排泵基因在临床志贺菌对四环素、红霉素等的耐药性的发展中起着重要作用。原因为:(1)通过对emrE基因的系统发育分析表明,它有很强的进化规律和保守性,在不同种属中有很强的归属性,与个体耐药性无关; (2)本实验所用志贺菌H24是2002年在江西省铜鼓县分离得到的临床耐药株,其emrE基因与20世纪50年代在北京分离的宋氏志贺菌Ss046基因序列竟然完全一致〔5〕,说明50年来志贺菌红霉素和四环素耐药性的迅速发展与emrE基因突变没有关系。
实验结果表明,在细菌中有一个强大的耐药基因武器库,新的药物环境会进化筛选出新的改进型耐药基因。在志贺菌H24中,可能还有更强的红霉素、四环素耐药基因和耐药突变在起作用。因此,象emrE基因这样耐药能力较弱基因的突变在临床上未被筛选。在志贺菌中起作用的红霉素、四环素耐药基因还待进一步研究。
| [1] | Piddock LJ. Clinically relevant chromosomally encoded multidrug resistance efflux pumps in bacteria[J]. Clinical Microbiology Review, 2006, 19(2) : 382–402. DOI:10.1128/CMR.19.2.382-402.2006 |
| [2] | Nishino K, Yamaguchi A. Analysis of a complete library of putative drug transporter genes in Escherichia coli[J]. J Bacteriol, 2001, 183(20) : 5803–5812. DOI:10.1128/JB.183.20.5803-5812.2001 |
| [3] | Sharoni M, Steiner-Mordoch S, Schuldiner S. Exploring the binding domain of emrE, the smallest multidrug transporter[J]. J Biol Chem, 2005, 280(38) : 32849–32855. DOI:10.1074/jbc.M504910200 |
| [4] | 吴健, 张少平, 任存邦, 等. 志贺菌外排泵cmr基因克隆及耐药作用[J]. 中国公共卫生, 2007, 23(12) : 1494–1495. |
| [5] | 吴健, 张少平, 穆瑞瑞, 等. 志贺菌外排泵基因emrE的克隆表达及进化分析[J]. 中国抗生素杂志, 2007, 32(10) : 621–626. |
| [6] | Eggertsson G, Söll D. Transfer ribonucleic acid-mediated suppression of termination codons in Escherichia coli[J]. Microbiological Reviews, 1988, 52(3) : 354–374. |
 2009, Vol. 25
2009, Vol. 25

