2. 第四军医大学西京医院中医科;
3. Faculty ofLife Science, University of Manchester, UK, M139P;
4. 复旦大学公共卫生学院流行病学教研室
戊型肝炎(Hepatitis E, HE)是我国非常重要的传染病之一,属于消化道传播疾病,是导致亚热带和卫生条件相对较弱国家急性肝炎流行的重要病毒之一。在亚州、非洲和墨西哥都有暴发〔1〕。血清流行病学数据和RNA序列分析进一步显示,戊型肝炎病毒(Hepatitis E virus, HEV)的型别具有多样性与分布的全球性〔2〕。流行病学监测显示,HE的发病率和暴发次数在近年都有上升的趋势。作为易于变异的RNA病毒,明确HEV的进化规律对HE的防治具有重要的指导作用〔3〕。本次研究拟对HEV的2个主要读码框(ORF)进行阳性选择分析,探讨戊型肝炎病毒(HEV)是否也存在阳性选择位点,为该病的控制提供基础依据。
1 材料与方法 1.1 基因数据以31个HEV全序列为研究对象进行适应性进化研究。31个全序列共包含Ⅰ型9个,Ⅱ型1个,Ⅲ型11个,Ⅳ型10个。
1.2 阳性选择分析方法〔4, 5〕根据系统树和序列对位排列结果,以Yang et al创立的密码子置换模式M0、M3、M1a、M2a、M7和M8来计算每个位点上的ω(ω=dN/dS)〔6, 7〕。M0模型假定各位点都有同样的ω值; 与M0相对应的是M3模型,该模型是离散模型,假设基础是各个位点上的ω是不同的; Mla模型为近似中性模型,它将密码子分为2类:(1)位点pl所编码的氨基酸几乎没有变化(0 < ω < 1);(2)位点Pl为中性近化位点(ω=1);M2a模型与M1a相对应,为阳性选择模型,它是在M1a模型基础上又增添了第3类位点,此类位点(p2)所预测的ω可以 > 1;M7和M8模型均采用离散β分布来估计何位点的ω值,并通过参数P和Q来描述β分布,M8和M7的不同之处在于M8增添了一类ω > 1的位点,用于检测阳性选择。本研究采用了M0、M3、Mla、M2a以及M7、M8模型进行成对比较,用PAML 3.14b软件包中的codeml程序对蛋白编码序列检测阳性选择位点。用似然比检验(LRT)检测模型间差异是否有统计学意义,LRT=2δ(h),自由度为2个模型间自由参数之差。
1.3 序列对比和进化树分析应用MEGA 3.0软件进行序列比对,校对密码子,确保密码子翻译的准确性,邻接法构建NJ进化树,分析纳入序列的基因型别分布。应用PHYLIP 3.6软件包构建PHYLOP格式的进化树。构建PHYLIP格式的进化树步骤如下:(1)用CLUSTAL把FAS格式数据转化成PHYLIP格式; (2)用seqboot、Dnasist、Neigboor、consense分别对进化树进行构建和修饰。
2 结果 2.1 序列比对与校正经过序列比对、密码子校正后、空位删除后ORF 2保留了672个密码子; ORF 1共保留了1 694个密码子。
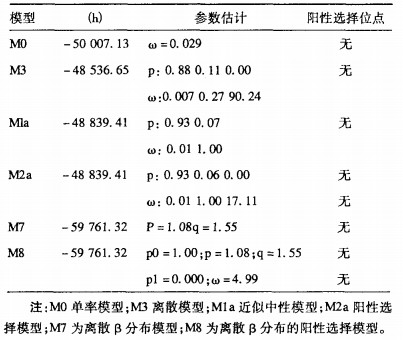
2.2 ORF 1不同模型ω值的最大似然参数估计(表 1)| 表 1 HEV ORF 1片段的阳性选择分析 |
M0模型的平均进化率估计提示ORF 1以净化选择为主,平均ω为0.029。M1a模型发现93%的位点属于保守位点,平均ω=0.018;7%的位点属于中性位点。M2a M8阳性选择模型分析结果提示ORF 1,片段没有阳性选择位点。经χ2检验发现,所有的假设检验均不成立,提示HEV ORF 1基因片段不存在阳性选择位点。
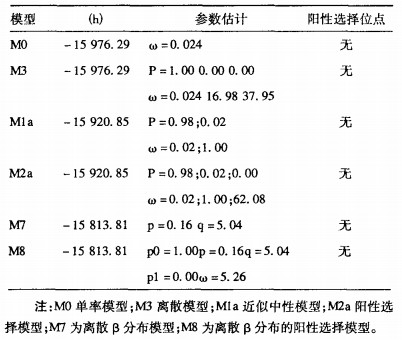
2.3 不同模型ω值的最大似然参数估计(表 2)| 表 2 HEV ORF 2片段的阳性选择分析 |
M0模型的平均进化率估计提示ORF 2以净化选择为主。中性进化模型(M1a)显示,98%的位点属于保守位点,2%的位点属于中性位点。M2a、M8阳性选择模型分析结果提示,ORF 2片段没有阳性选择位点。χ2检验发现,所有的假设检验均不成立,提示HEV ORF 2基因片段不存在阳性选择位点。
3 讨论阳性选择分析是综合分子遗传学、数理统计学和计算机模拟等技术对生物大分子进化进行分析〔8〕。氨基酸密码子的同义突变和非同义突变是分子进化分析的依据,非同义突变和同义突变的率比ω(ω=dN/dS)是反应选择压力的重要指标:ω=1表示中性突变,ω < 1表示净化突变,ω>1表示阳性选择〔9, 10〕。阳性选择算法有很多,其中以Yang et al基于贝叶斯理论建立的最大似然算法最为学术界所认可〔6, 7〕。该模型认为基因所有氨基酸位点的进化率不一致,并用最大似然算法对每个密码子位点进行ω值估计。进一步通过分子的三维结构分析表明,这些位点均分布在抗原提呈分子的抗原结合面〔11, 12〕。在分析精子膜蛋白的过程中也发现,阳性选择位点均分布于精子膜蛋白的入侵功能侧,体现了该模型的实际应用价值〔13〕。
ORF 1和ORF 2是HEV的2个最重要的读码框,占全基因的95%以上〔14〕,ORF 1编码一些非结构蛋白包括甲基转移酶、RNA解旋酶、RNA依赖性RNA聚合酶等,ORF 2编码衣壳蛋白,与衣壳抗原性有重要的关系。由此可见,研究这2个读码框对于深入了解HEV的分子进化规律具有重要意义。本次研究采用了Yang et al基于密码子替代模型的最大似然算法,分析了包括了目前所有的基因型31个全序列,但是无论从统计量LRT还是位点ω分析方面均末检测到阳性选择位点,说明这2个片段的进化可能不存在阳性选择位点。分析可能有以下原因:(1)戊型肝炎不是致死性疾病,而且宿主广泛(人、猪),尤其是4月龄猪HEV感染率高达100%,数量十分庞大。(2)病毒容易生存,具有很好的耐热性。(3)宿主带毒时间短,尚无很好的治疗药物,疫苗也仅仅是处于研究阶段,这些病毒抗原进化的主要影响因素尚末对HEV施加作用。因此认为,在HEV进化过程中阳性选择不是主要模式。
| [1] | Panda SK, Thakral D, Rehman S. Hepatitis E virus[J]. Rew Med Virol, 2007, 17(3) : 151–180. DOI:10.1002/(ISSN)1099-1654 |
| [2] | Acharya SK, Panda SK. Hepatitis E virus:epidemiology, diagnosis, pathology and prevention[J]. Trop Gastroenterol, 2006, 27(2) : 63–68. |
| [3] | Okamoto H. Genetic variability and evolution of hepatitis E virus[J]. Virus Res, 2007, 127(2) : 216–228. DOI:10.1016/j.virusres.2007.02.002 |
| [4] | 张文娟, 张原, 钟扬. 应用最大似然法检测丙肝病毒包膜蛋白编码基因的适应性进化[J]. 科学通报, 2006, 51(16) : 1894–1899. |
| [5] | 张原, 郑楠, 郝沛, 等. SARS冠状病毒S基因的最近共同祖先序列重建及Spike蛋白的适应性进化检测[J]. 科学通报, 2004, 49(11) : 113–114. |
| [6] | Anisimova M, Bielawski J P, Yang Z. Accuracy and power of the likelihood ratio test in detecting adaptive molecular evolution[J]. Mol Biol Evol, 2001, 18(8) : 1585–1592. DOI:10.1093/oxfordjournals.molbev.a003945 |
| [7] | Yang Z. Maximum likelihood analysis of adaptive evolution in HIV-1 gp120 env gene[J]. Pac Symp Biocomput, 2001 : 226–237. |
| [8] | Suzuki Y, Gojobori T. A method for detecting positive selection at single amino acid sites[J]. Mol Biol Evol, 1999, 16(10) : 1315–1328. DOI:10.1093/oxfordjournals.molbev.a026042 |
| [9] | Yang W, Bielawski J P, Yang Z. Widespread adaptive evolution in the human immunodeficiency virus type 1 genome[J]. J Mol Evol, 2003, 57(2) : 212–221. DOI:10.1007/s00239-003-2467-9 |
| [10] | Anisimova M, Bielawski JP, Yang Z. Accuracy and power of bayes prediction of amino acid sites under positive selection[J]. Mol Biol Evol, 2002, 19(6) : 950–958. DOI:10.1093/oxfordjournals.molbev.a004152 |
| [11] | Yang Z, Nielsen R, Goldman N, et al. Codon-substitution models for heterogeneous selection pressure at amino acid sites[J]. Genetics, 2000, 155(1) : 431–449. |
| [12] | Yang Z, Nielsen R. Codon-substitution models for detecting molecular adaptation at individual sites along specific lineages[J]. Mol Biol Evol, 2002, 19(6) : 908–917. DOI:10.1093/oxfordjournals.molbev.a004148 |
| [13] | Yang Z, Swanson WJ, Vacquier VD. Maximum-likelihood analysis of molecular adaptation in abalone sperm lysin reveals variable selective pressures among lineages and sites[J]. Mol Biol Evol, 2000, 17(10) : 1446–1455. DOI:10.1093/oxfordjournals.molbev.a026245 |
| [14] | Mushahwar IK. Hepatitis E virus: molecular virology, clinical features, diagnosis, transmission, epidemiology, and prevention[J]. J Med Virol, 2008, 80(4) : 646–658. DOI:10.1002/(ISSN)1096-9071 |
 2009, Vol. 25
2009, Vol. 25

 , 孙静2, 栗燕2, 杨林2
, 孙静2, 栗燕2, 杨林2