2. 郑州大学公共卫生学院;
3. 河南省分子医学重点学科开放实验室
霍乱弧菌毒力岛(Vibrio cholerae pathogenicity, VPI)可合成霍乱毒素共调菌毛, 它不仅是一个重要的定居因子, 而且可以作为溶源性噬菌体CTXΦ的受体, 介导CTXΦ进入霍乱弧菌, 且VPI也可以以丝状噬菌体的形式存在, 命名为VPIΦ, 在不同的霍乱菌群间传递〔1-3〕。已知VPIΦ有相同的插入位点〔4〕, 所以有必要研究VPI两侧不可移动的染色体基因组固有序列的同源性来推断O139群霍乱弧菌的来源。本文主要研究O139群霍乱弧菌国际标准株MO45的VPI的分子特征, 并与O1群El Tor型霍乱弧菌菌株N16961进行比较, 分析O139霍乱弧菌和El Tor霍乱弧菌的相互遗传关系。
1 材料与方法 1.1 材料O139群霍乱弧菌国际标准株MO45的DNA (中国疾病预防控制中心传染病研究); 大肠埃希菌TB1、克隆载体pNEB193(英国NEB公司)。地高辛核酸标记及检测试剂盒(德国Roche公司); 凝胶回收试剂盒(中国杭州维特洁生化技术有限公司); 凝胶图象分析仪(美国Syugene公司); 温度梯度基因扩增仪(德国Biometra公司)。
1.2 PCR扩增VC0815基因(1)引物:应用软件Primer Premier 5.0设计VPI上游基因VC0815引物(中国北京赛百盛公司合成), 上游引物引入Hind Ⅲ酶切位点, 下游引物引入BamHⅠ酶切位点:上游引物: 5'-CCCAAGCTTTAAGTCGTGGCTGCTAT-3'(Hind Ⅲ); 下游引物: 5'-TTCGGATCCCTTTCACGCTGTTTCACT-3'(BamH Ⅰ)。(2) PCR扩增体系: PCR反应体系25 μl, 其中10 × PCR缓冲液2.5 μl, 4 × dNTPs 2 μl (各200 μmol/L), MO45基因组DNA 2 μl, 引物各0.75 μl (20 μmol/L), 高保真DNA聚合酶0.25 μl (1.25 U)加三蒸水至终体积。95 ℃预变性5 min, 然后按以下PCR参数进行30个循环: 94 ℃变性1 min, 57 ℃退火1 min, 72 ℃延伸2 min, 最后1个循环72 ℃终末延伸10 min。
1.3 pNEBVC0815重组质粒的构建VC0815基因的PCR产物和pNEB193空质粒分别用限制性内切酶BamH Ⅰ、Hind Ⅲ双酶切, 酶切产物用凝胶回收试剂盒回收, 凝胶图像分析定量。VC0815基因的PCR产物与pNEB193空质粒的酶切回收产物以摩尔比8:1混合, T4DNA连接酶16 ℃过夜进行连接后, 进行蓝白筛选, 挑选白色菌落, 碱裂解法提取质粒, 酶切鉴定, 阳性重组质粒命名为pNEBVC0815。
1.4 VC0815基因探针的制备以回收、纯化的pNEBVC0815外源片段为模板, PCR掺入法制备VC0815基因探针。PCR扩增体系为50 μl, 其中10 × PCR缓冲液5.0 μl, 3 × dNTPs 4.0 μl (各200 μmol/L), 模板DNA 4.0 μl, 引物各1.5 μl (20 μmol/L), Taq DNA聚合酶1.0 μl (2.5 U), 此外标记组加入dTTP 3.5 μl (175 μmol/L), DIG-dUTP1.25 μl (25 μmol/L); 对照组加入dTTP4.0 μl, (200 μmol/L)加三蒸水至终体积。PCR扩增参数同前。
1.5 从MO45基因组中筛选含VC0815及其上游基因的片段从GenBank上发表的N16961全基因组序列中找到包含VC0815及其上游基因的片段约6.8 kb, 两端的酶切位点KpnⅠ和BamHⅠ。用KpnⅠ和BamHⅠ酶切MO45基因组DNA。取20 μl酶切产物于0.8 %的琼脂糖凝胶电泳后用VC0815基因探针按地高辛核酸标记及检测试剂盒说明进行Southern杂交。将杂交阳性附近条带切下, 回收并克隆到pNEB193中, 阳性重组质粒命名为pNEBVC081545。阳性克隆再经特异PCR鉴定, PCR体系、参数同前。
1.6 pNEBVC081545限制性酶切图谱分析已知El Tor型霍乱弧菌和O139群霍乱弧菌的VPI序列是相同的, 且VPI阳性的霍乱弧菌其插入位点包括VPI上游1.5 kb片段(其最上游基因是VC0815基因)是相同的〔5〕。El Tor型霍乱弧菌菌株N16961全基因组序列GenBank已经发表, 通过Primer premier 5.0软件分析N16961中这段序列的限制性酶切位点所在位置, 并综合pNEB193上已知的限制性酶切位点位置, 选用多种限制性内切酶(BamHⅠ、BglⅡ、PstⅠ、SalⅠ、XbaⅠ、Hind Ⅲ、KpnⅠ、EcoRⅠ、PvuⅠ等)酶切pNEBVC081545, 利用通过GeneTools软件计算出酶切片段的大小, 配合Southern分子杂交, 分析克隆片段中酶切位点的位置和数目, DNAMAN绘制酶切图谱, 并与N16961该段序列的限制性酶切图谱比较。
1.7 从pNEBVC081545中亚克隆VPI上游片段(包含VC0812、VC0813、VC0814基因)根据pNEBVC081545的限制性酶切图谱, 选用EcoRⅠ和SalⅠ消化该重组质粒, 找出所需亚克隆的片段(包含VC0812、VC0813、VC0814基因), 回收该片段重组至pNEB193中, 提质粒酶切鉴定, 方法同前, 重组质粒命名为pNEB45。
1.8 亚克隆片段测序分析将pNEB45的外源片段测序(大连TaKaRa公司)后与N16961中该段核酸序列进行同源性比较。
2 结果 2.1 VC0815基因定向克隆结果(图 1)
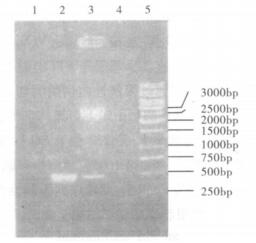
|
注: 1: pNEBVC0815的Hind Ⅲ单酶切; 2: MO45的VC0815基因PCR产物对照; 3: pNEBVC0815的BamHⅠ和Hind Ⅲ双酶切; 4: pNEB193空质粒对照; 5: 1 kb DNA ladder。 图 1 pNEBVC0815的酶切鉴定结果 |
小量碱裂解法提取质粒pNEBVC0815, 进行单酶切、双酶切鉴定。
2.2 VC0815基因探针标记的结果(图 2)
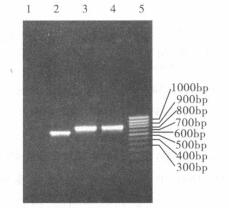
|
注: 1: PCR水对照; 2:未标记的VC0815基因PCR产物对照; 3, 4:标记的VC0815基因探针; 5: 100 bp DNA laddr。 图 2 VC0815基因探针标记结果 |
取2 μl标记及未标记地高辛的VC0815基因的PCR产物, 经1%的琼脂糖凝胶电泳。掺入地高辛的PCR产物条带稍落后于未标记地高辛的, 表明探针标记成功。
2.3 筛选MO45基因组中含VC0815及其上游基因片段(图 3)
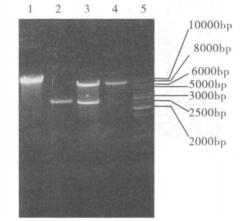
|
注: 1: pNEBVC081545的BamHⅠ单酶切; 2: pNEB193空质粒对照; 3: pNEBVC081545的BamHⅠ、KpnⅠ双酶切; 4:从MO45基因组中回收的6~8 kb的片段对照; 5: 1 kb DNA ladder。 图 3 pNEBVC081545的酶切鉴定结果 |
用KpnⅠ和BamHⅠ双酶切MO45基因组DNA后进行Southern杂交。结果显示, 用VC0815基因探针杂交KpnⅠ和BamHⅠ MO45基因组DNA正是6.8 kb左右的片段阳性。该片段与pNEB193重组质粒pNEBVC081545的酶切鉴定结果如图 3, 特异PCR鉴定证明已从MO45基因组中成功克隆了含有VC0815基因及其上游基因的片段。
2.4 pNEBVC081545限制性酶切图谱(图 4)
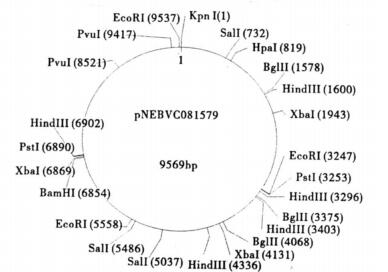
|
注:黑体字为pNEBVC081545中VC0815基因上游片段的限制性内切酶位点; 斜体字为pNEB193中限制性内切酶位点其余为VPI内部片段限制性内切酶位点。 图 4 pNEBVC081545的限制性酶切图谱 |
图 4可见, 第1个碱基即限制性酶切位点KpnⅠ位置到第3247位即EcoRⅠ限制性酶切位点位置为霍乱弧菌VC0812、VC0813、VC0814基因所在位置, 这3个基因是VPI上游VC0815基因的上游基因。这段序列通过限制性内切酶酶切分析, 绘制到pNEBVC081545的限制性酶切图谱上为黑体字部分。第1位限制性内切酶位点为KpnⅠ, 第732位为SalⅠ, 第819位为HpaⅠ, 第1578位为BglⅡ, 第1600位为Hind Ⅲ, 第1943位为XbaⅠ, 第3247位为EcoRⅠ, 此段序列限制性内切酶图谱与N16961同段序列限制性酶切图谱完全相同。
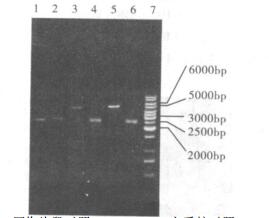
2.5 pNEBVC081545中VPI上游片段亚克隆结果(图 5)
|
注: 1: 2.5 kb回收片段对照; 2: pNEB193空质粒对照; 3, 5: pNEB45的EcoRⅠ单酶切; 4, 6: pNEB45的EcoRⅠ、SalⅠ双酶切; 7: 1 kb DNA ladder。 图 5 pNEB45的酶切鉴定结果 |
根据pNEBVC081545的限制性酶切图谱, 用EcoRⅠ、SalⅠ双酶切后回收2.5 kb片段即为VPI上游片段, 包含VC0812、VC0813、VC0814基因。将其重组至pNEB193质粒载体, 重组质粒命名为pNEB45, 鉴定结果表明已成功克隆了该段基因序列。
2.6 pNEB45外源片段的测序及序列比较将pNEB45的外源片段测序后与GenBank上N16961的该段核酸序列经同源性比较得出此O139群霍乱弧菌菌株MO45与El Tor型霍乱弧菌菌株N16961此片段的核酸序列完全相同。
3 讨论O139群霍乱弧菌是1992年以后新出现的霍乱病原菌。自出现以来有关其来源一直是研究的热点。目前有关O139群霍乱弧菌的来源主要有2种假设〔2, 3〕:来源于El Tor型霍乱弧菌产毒株或来源于O139群霍乱弧菌非产毒株。
限制酶切图谱是以限制性内切酶为标记的物理图谱, 即指DNA链的限制性酶切片段的排列顺序, 即酶切片段在DNA链上的定位。因限制性内切酶在DNA链上的切口是以特异序列为基础的, 核酸序列不同的DNA经酶切后就会产生不同长度的DNA片段, 由此而构成独特的酶切图谱。因此, DNA限制性酶切图谱是DNA分子结构的特征之一。本研究从O139群霍乱弧菌国际标准株MO45的基因组中筛选出含VPI上游基因VC0815及其上游基因的片段, 构建了该片段重组质粒的限制性酶切图谱, 并对其中部分片段进行了亚克隆、测序、分析。研究发现, MO45的VPI上游基因VC0815上游2.5 kb的片段与GenBank上发表的El Tor霍乱弧菌菌株N16961同段序列完全相同, 为O139群霍乱弧菌有可能是El Tor型霍乱弧菌发生抗原突变的观点〔5-8〕提供了证据。
| [1] | Craig L, Taylor RK, PiqueM E, et al. Type IV pilin structure and assembly:X-ray and EM analyses of Vibrio cholerae toxin coregulated pilus and Pseudomonas aeruginosa PAK pilin[J]. Mol Cell, 2003, 11(5) : 1139–1150. DOI:10.1016/S1097-2765(03)00170-9 |
| [2] | Miller J F. Bacteriophage and the evolution of epidemic cholera[J]. Infect Immun, 2003, 71(6) : 1981–1982. |
| [3] | Faruque SM, Albert ML, Mekalanos JJ. Epidemiology, genetics, and ecology of toxicgenic Vibrio cholerae[J]. Mol Biol Rev, 1998, 62 : 1301–1314. |
| [4] | Karaolis DK, Lan R, Kaper JB, et al. Comparison of Vibrio cholerae pathogenicity islands in sixth and seventh pandemic strains[J]. Infect Immun, 2001, 69(3) : 1947–1952. DOI:10.1128/IAI.69.3.1947-1952.2001 |
| [5] | Faruque SM, Sack DA, Sack RB, et al. Emergence and evolution of Vibrio cholerae O139[J]. Proc Natl Acad Sci USA, 2003, 100 : 1301–1309. |
| [6] | Kotetishvili M, Stine O C, Chen Y, et al. Multilocus sequence typing has better discriminatory ability for typing Vibrio cholerae than does pulsed field gel electrophoresis and provide a measure of phylogenetic relatedness[J]. J Clin Microbiol, 2003, 41(5) : 2191–2196. DOI:10.1128/JCM.41.5.2191-2196.2003 |
| [7] | 芮勇宇, 阚飙, 高守一, 等. 霍乱弧菌4种新类型tcpA基因发现及序列分析[J]. 中国公共卫生, 2005, 21(12) : 1418–1420. |
| [8] | 芮勇宇, 许锐恒, 阚飙, 等. 霍乱弧菌主要毒力和管家基因序列分析[J]. 中国公共卫生, 2006, 22(1) : 31–33. |
 2009, Vol. 25
2009, Vol. 25

 , 谢婧2, 郗园林2, 范清堂3
, 谢婧2, 郗园林2, 范清堂3