人类基因组中编码的蛋白据预测约有3 000多种可结合药物小分子, 被认为是可靶向成药的[1, 2]; 然而, 迄今为止, 这些蛋白质中仅有一小部分作为靶标用于人类相关疾病的治疗[3]。蛋白质除了自身功能外, 还通过翻译后修饰、高度组织化和反应灵敏的蛋白质网络来拓展功能并维持生命活动, 这些网络通过蛋白质节点之间的相互作用传递蛋白质功能, 即所谓的蛋白-蛋白相互作用(protein-protein interactions, PPIs)[4-7]。PPIs网络将细胞生物学的各个方面联系起来(如伴侣蛋白、骨架蛋白、传输细胞信号等), PPIs的功能失调与肿瘤、神经性病变和自身免疫性疾病等许多疾病密切相关, 是潜在的干预靶点[8], 但在成药性估计中却未被考虑在内。据估计, 人类细胞存在约有130 000多种PPIs, 如果可以设计出有效的策略来调节这种相互作用, 将为疾病治疗干预和药物发现创造巨大机会[9], 因为原则上每个具有病理学意义的PPI都是潜在的药物靶点[4-7]。尽管如此, 由于所涉及的蛋白质表面在拓扑结构上是平坦的, 且不提供小分子结合所必需的腔穴或裂隙, 故蛋白-蛋白界面难以靶向[10]。
天然产物是新化学实体药物的重要来源, 大约50%的FDA批准的小分子药物与天然产物有关[11]。传统的小分子药物通过直接靶向一种蛋白调节其功能来发挥治疗作用, 这些蛋白通常是被药物抑制或激活的酶、受体或转运蛋白等。然而, 传统的小分子药物很难靶向PPIs的界面, 对PPIs的调控几乎无能为力。早在上世纪90年代, Liu等[12]在研究环孢菌素A (cyclosporin A, CsA) 和FK506等大环天然产物的作用机制时发现, 这些化合物通过与蛋白结合改变其构象, 赋予其“获得性功能” (gain-of-function), 将两种彼此之间几乎没有亲和力的蛋白“粘连”在一起, 从而发挥新的作用, 这类化合物现在统称为“分子胶水”。分子胶水作为一种诱导邻近(proximity-inducing)、调节PPIs的小分子, 可以对各种生物过程进行精确的时间控制, 例如信号转导、转录以及蛋白质折叠、定位和降解等[13]。同时, 作为邻近化学诱导剂(chemical inducers of proximity, CIPs), 分子胶水以形成三元复合物的模式促进两种蛋白质的二聚化或共定位, 可以产生多种生物学和药理学功能[13]。
近年来, 随着沙利度胺等多种分子胶水降解剂(molecular glue degraders) 的出现, 分子胶水的范畴得到了很大的拓展。与CsA和FK506等“获得性功能”的分子胶水稳定剂不同, 分子胶水降解剂通过结合泛素化连接酶或LC3蛋白, 从而招募蛋白酶体或自噬体降解目标蛋白。不得不提的是, 分子胶水降解技术与近年来研究如火如荼的其他蛋白降解技术, 比如PROTAC、LYTAC和AUTAC等有所不同, 后者通常采用双功能分子降解剂, 即“两端靶向小分子+中间Linker”的模式, 而分子胶水不需要linker的介入。
与传统的小分子药物相比, 分子胶水具有选择性高、用量小和脱靶效应低等优点。同时, 分子胶水的出现打破了许多“不可成药”靶标的传说, 包括蛋白磷酸酶、蛋白激酶和蛋白-蛋白相互作用等, 极大拓展了可成药靶标的范畴。本文将聚焦天然产物及其衍生物来源的分子胶水, 根据其作用结果分类进行阐述, 主要包括稳定剂和降解剂。希望通过回顾已有研究, 加深对分子胶水发现和作用机制的认识来探寻新的分子胶水实体和新的作用模式。
1 分子胶水稳定剂本部分将从具有免疫抑制活性的天然产物CsA、FK506和雷帕霉素(rapamycin, RAPA) 的经典案例出发, 阐述其发现、作用机制和功能等; 同时, 这类天然产物的衍生物也在重点讨论之列, 如FK1012和rapadocin, 这些化合物要么诱导二聚化, 要么稳定调节蛋白复合物。由于篇幅有限, 本文不对其他天然产物如紫杉醇、forskolin及brefeldin A等进行讨论, 有兴趣的读者可参考已有文献及综述[14-16]。
1.1 Cyclosporin A免疫抑制剂CsA和FK506是最早发现的两个分子胶水[12]。尽管两者具有不同的受体蛋白, 分别为亲环蛋白(cyclophilin, CyP) 和FK506结合蛋白(FK506 binding protein 12, FKBP12), 但均通过相似的作用机制抑制细胞信号传导。CyP和FKBP是内源性亲免蛋白(immunophilins, 免疫抑制剂结合的蛋白质家族) 的两大家族, CsA、FK506和RAPA作为天然存在的3种亲免蛋白配体, 都因免疫抑制功能而被发现[17-19]。1991年, Liu等[12]发现并报道了钙调磷酸酶(cacineurin, CaN) 是二元复合物CyPA-CsA和FKBP-FK506的共同靶标, 而游离的CyP和FKBP不能结合CaN, 并首次引入分子胶水的概念。
CsA是子囊菌Tolypocladium inflatum产生的代谢物, 是一种由11个氨基酸组成的33元大环天然产物(图 1A), 1971年由瑞士Sandoz公司从收集的挪威土壤样本中鉴定出来, 随后发现其有免疫抑制活性[20]。1979年, 该化合物被用作器官移植免疫排斥的抑制剂, 于1983年被FDA批准上市[20], 奠定并推动了器官移植的发展。
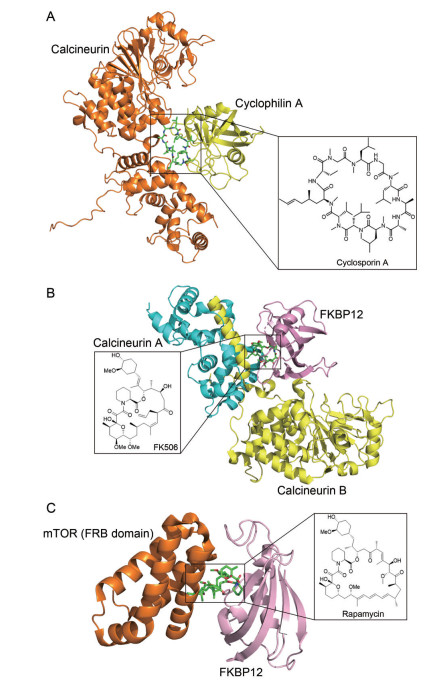
|
Figure 1 Crystal structures of natural molecular glues induced protein complex formation: cyclophilin A-cyclosporin A-calcineurin (A), FKBP12-FK506-calcineurin (B) and FKBP12-rapamycin- mTOR (FRB domain) (C) |
CsA的主要结合蛋白于1984年被鉴定并命名为亲环蛋白A (cyclophilin A, CyPA)[21], 是一种高度保守且广泛分布于原核及真核细胞的蛋白, 具有肽基脯氨酰顺反异构酶(peptidyl prolyl cis-trans isomerase, PPIase) 催化活性[22]。虽然CsA抑制了这种PPIase活性, 但亲环蛋白与免疫抑制之间没有显著关联, 真正发挥作用的是CyPA-CsA二元复合物的靶蛋白钙调磷酸酶CaN (图 1A)。CaN是一种独特的丝氨酸-苏氨酸蛋白磷酸酶, 受第二信使Ca2+和钙调蛋白的调节, 参与包括免疫反应在内的许多生物过程[23, 24]。CsA进入细胞后与CyPA结合形成CyPA-CsA二元复合物, 进一步与CaN结合使其失去磷酸酶活性, 导致活化的T细胞核因子(nuclear factor of activated T cells, NFAT) 等不能被去磷酸化和转位, 进而不能与核内相关基因启动子结合, 抑制了白介素2 (interleukin-2, IL-2) 等细胞因子的转录(图 2), 使T细胞无法从G0期向G1期过渡[25, 26], 从而阻断T细胞增殖。简言之, 三元复合物CyPA-CsA-CaN的形成有效抑制了CaN的磷酸酶活性, 生化实验和X射线晶体学证明了三元复合物的形成(图 1A)[27]。CyPA-CsA不但能结合在CaN的催化和调节亚基共同形成的复合表面上, 也与CaN催化活性位点上的Arg-122相互作用, 表明CyPA-CsA能直接参与CaN催化的调节。该晶体结构的解析为后续相关酶抑制剂的设计和探索提供了有力的支持。
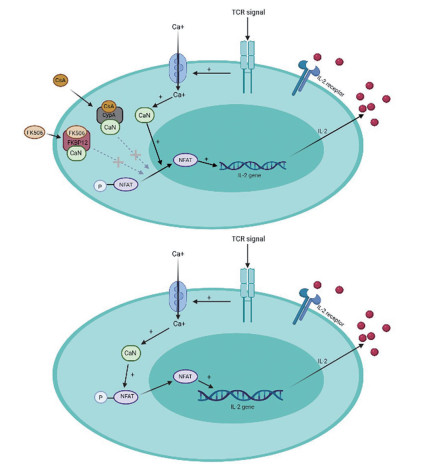
|
Figure 2 Signal pathways for immune regulation of cyclosporin A and FK506 |
FK506是1987年日本Fujisawa公司从Streptomyces tsukubaensis中分离出来的23元大环内酯化合物, 具有免疫抑制作用[19]。两年后, FK506的主要蛋白受体FKBP12被鉴定出来, 这是一种肽基-脯氨酰异构酶PPIase, 与CyPA相似但不完全相同[28]。FK506由一个FKBP结合域和一个传递免疫抑制活性的效应域组成(图 1B), 具有与CsA相似的活性, 甚至在器官移植患者中表现出比CsA更好的效果[29]。与CyPA-CsA一样, FKBP12-FK506二元复合物结合并变构抑制其靶标蛋白CaN, 从而诱导相同的免疫抑制作用[26, 30, 31]。Vertex和Agouron公司相继于1995年发表的X射线晶体结构也表明, FKBP12-FK506-CaN三元复合物的形成具有抑制CaN远端磷酸酶催化位点的作用[30, 31]。不同于CyPA-CsA直接参与CaN催化调节, 二元复合物FKBP12-FK506在没有直接与活性位点相互作用的情况下抑制CaN活性; 其在CaN上的物理存在会阻止大分子磷酸化底物(如NFAT转录因子) 进入磷酸酶活性位点[30]。三元复合物的三维结构为了解FKBP12-FK506对CaN的抑制作用提供了理论基础。CyPA-CsA-CaN与FKBP-FK506-CaN结构的对比十分有助于理解亲免蛋白-免疫抑制剂调节CaN活性的分子基础。
1.3 雷帕霉素(rapamycin, RAPA)RAPA是来自于Streptomyces hygroscopicus的抗真菌代谢物, 最初于1975年从复活节岛(Rapa Nui)[18]的土壤中分离出来, 不久后发现对哺乳动物免疫系统具有抑制活性[32]。在20世纪90年代初期, 人们首先发现RAPA与FK506具有相同的FKBP结合域, 能首先结合FKBP12蛋白。1994年, Sabatini等[33]和Brown等[34], 先后报道了二元复合物FKBP12-RAPA的靶标蛋白不是CaN, 而是mTOR (mechanistic target of rapamycin, mTOR), 阐明了RAPA是一个新型的分子胶水。mTOR是一类丝氨酸-苏氨酸蛋白激酶, 其变构抑制导致各种下游信号通路被阻断, 而这些信号通路控制着细胞的生长、凋亡和自噬等[26, 35]。
1996年, FKBP12-RAPA-mTOR (FRB domain, FKBP12-rapamycin binding domain) 三元复合物晶体结构的解析揭示了RAPA对FKBP12和mTOR的作用机制(图 1C)[36]。RAPA通过与两种蛋白的直接相互作用来形成并稳定三元复合物, 而两种蛋白之间的相互作用较少。三元复合物通过空间位阻效应阻碍mTOR下游底物对其接近。同样是抑制T细胞的激活, 不同于CsA和FK506, RAPA不抑制IL-2的释放, 而是通过mTOR抑制T细胞对IL-2信号的响应, 从而抑制T细胞从G1向S期过渡[26, 36]。
上述化学诱导蛋白复合物的新机制正成为探索生物过程和开发新治疗药物的有效策略; 通过稳定蛋白复合物等方式赋予增强、抑制或新的蛋白功能, 是一种潜在的、新的、有趣的治疗方式, 激发了从天然产物和合成分子中发现分子胶水的灵感[37]。
1.4 Sanglifehrin A受FK506和RAPA特殊作用机制的启发, Novartis公司于1997年对CyP的配体小分子进行筛选鉴定, 希望找到和CsA具有不同作用模式的新化合物[38]。筛选微生物Streptomyces sp. A92-308110浆提取物发现并鉴定出一类新的化合物, 称为sanglifehrins。Sanglifehrin A (SFA, 图 3) 是一种含混合聚酮结构的22元大环天然产物, 具有复杂而独特的分子结构, 对其受体CyPA具有皮摩尔级的亲和力。2017年, Pua等[39]报道了肌苷单磷酸脱氢酶2 (inosine monophosphate dehydrogenase 2, IMPDH2) 是CyPA-SFA二元复合物的体外特异性靶标。IMPDH2是细胞内鸟嘌呤核苷生物合成的限速酶, 并参与了由烟酰胺腺嘌呤二核苷酸(NAD) 介导的从IMP (inosine-5′-monophosphate) 到XMP (xanthosine-5′-monophosphoate) 的转化反应。然而, 形成这种三元复合物不会抑制IMPDH2的催化活性, 而是通过与IMPDH2的CBS (胱硫醚-β-合酶) 结构域相互作用来调节细胞生长。当然, 是否存在其他靶标蛋白来抑制免疫反应后续还需要进行严格的靶标鉴定和确证。
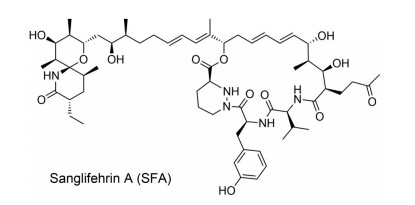
|
Figure 3 Structure of sanglifehrin A |
2020年, Shigdel等[40]报道了利用基因挖掘方法发现大环内酯分子胶水WDB002 (图 4)。与FK506和RAPA一样, WDB002首先结合FKBP12蛋白; 不同的是, FKBP12-WDB002二元复合物结合着丝粒蛋白CEP250。CEP250是中心体的组成成分, 中心体是哺乳动物细胞的主要微管组织中心。每个细胞内有一个中心体, 分裂前期发生复制, 在分裂间期分裂成两个中心体, 并通过丝状蛋白质连接体(proteinaceous linker) 紧密相连。CEP250是这种丝状结构的一个组成部分, 连接着两个中心体[41]。在有丝分裂开始时, NEK2磷酸化CEP250并触发其从中心体置换, 这导致接头的分解, 使中心体分离, 有丝分裂顺利进行[42, 43]。干扰接头分解会改变中心体分离的时间, 促进有丝分裂中染色体分离错误[44]。FKBP12-WDB002结合CEP250之后会影响中心体连接组织, 干扰其功能导致中心体分离减少。此外, WDB002介导的FKBP12募集到CEP250似乎干扰了NEK2通过CEP250磷酸化刺激中心体分离的能力。
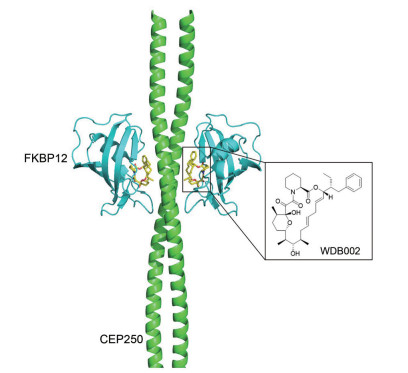
|
Figure 4 Crystal structure of the FKBP12-WDB002-CEP250 ternary complex |
FKBP12-WDB002-CEP250三元复合物的晶体结构为具有C2对称性的四聚体, 其中CEP250形成同源二聚体卷曲线圈, 并在每一侧结合一个FKBP12-WDB002复合物(图 4)。与RAPA和FK506类似, WDB002嵌入FKBP12目标蛋白界面, 与FKBP12和CEP250进行广泛的相互作用。两个FKBP12-WDB002二元复合物独立接触CEP250, 两者都与CEP250二聚体相互作用, 接触盘绕线圈的两条链。FKBP12-WDB002通过复合界面结合CEP250, FKBP12结合WDB002的恒定区并将可变区呈现给CEP250。尽管CEP250蛋白上的扁平结构没有任何突出的腔穴或裂隙, WDB002的可变区提供了互补的疏水热点, 因此两者可以很好的结合。这样的疏水热点正是PPIs的标志, 且通常是必不可少的特征[30, 36, 45]。
1.6 Asukamycin和manumycin具有明显生物活性和含有多个能与蛋白上的氨基酸形成共价结合的亲电天然产物, 很有可能是分子胶水新的来源。基于这个假设, Isobe等[46]重点考察了链霉菌衍生物manumycin聚酮类家族成员, 尤其是asukamycin和manumycin A, 具有抗生素和抗增殖特性; 同时, asukamycin含有多个亲电位点, 包括不饱和酰胺和环氧酮(图 5), 可以通过Michael加成与目标蛋白共价结合。在系统测定了asukamycin的体外抗癌活性后, 研究者通过基于ABPP (activity-based protein profiling) 的化学蛋白质组学平台, 发现manumycin聚酮类家族成员靶向乳腺癌细胞中E3连接酶UBR7的氨基酸残基C374, 并与新底物抑癌基因p53蛋白进行分子胶水相互作用, 导致p53转录激活和细胞死亡[46]。有趣的是, 和沙利度胺等分子胶水降解剂不同, asukamycin结合UBR7之后不是降解p53而是稳定p53, 形成UBR7-asukamycin-p53三元复合物并发挥抗癌活性。
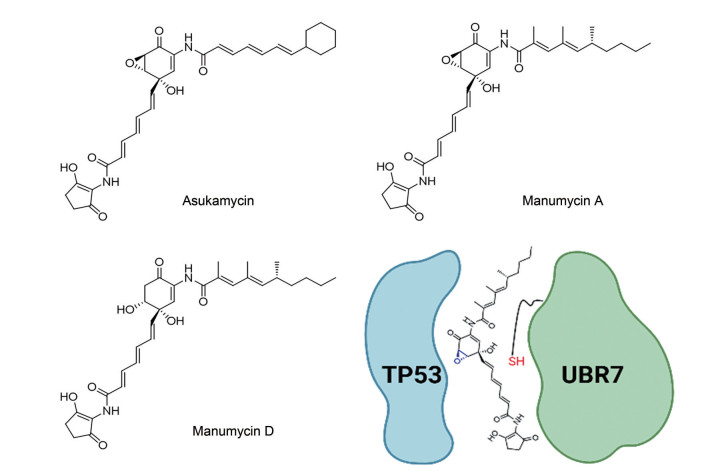
|
Figure 5 Structures of manumycin polyketides and schematic model of the mechanism of action (bottom right) |
对于asukamycin的类似物manumycin A也观察到了UBR7与p53之间的分子胶水型相互作用, 而缺失环氧基团的manumycin D仍保留与UBR7的结合, 但与p53的结合作用较弱, 且不能以相同程度共价修饰p53, 无法诱导UBR7与p53之间的PPIs。因此, manumycin聚酮化合物的不饱和酰胺有助于与UBR7中C374残基的共价作用, 而环氧基团负责与p53的相互作用[46]。
然而, 几个悬而未决的问题仍然存在。① Manumycin聚酮类化合物-UBR7复合物如何与p53相互作用以激活p53功能并不清楚; UBR7-asukamycin复合物就像一个分子伴侣, 可以提高p53的热稳定性和折叠性, 这可能会提高其与DNA结合并激活p53转录活性的能力。②三元复合物UBR7-asukamycin-p53具体的结合模式也不明确。这些都需要进一步的实验来阐明。
尽管存在待解决问题, 该研究不仅揭示了多共价小分子充当新型分子胶水的可能性, 也为其他带有多个反应位点的天然产物或者合成小分子是否可作为特定的分子胶水来赋予新的蛋白功能提供了参考。许多天然产物中含有类似manumycins化合物的不饱和侧链, 其中有的具有各种亲电性弹头; 此外, 含有一个或多个潜在蛋白反应性官能团的多环天然产物数量众多, 意味着这一概念可能会扩展到更大范围的天然产物空间。
1.7 FK506衍生物FK10121994年, 受FK506作用机制的启发, Pruschy等[47]报道了首个针对基因融合的“双功能”分子胶水FK1012 (图 6), 是FK506通过C21位烯丙基的烯烃复分解反应连接而成的二聚体。FK1012被证明能将受体蛋白聚集在一起, 启动细胞中相应的信号通路。比如, FK1012可以使T细胞受体MAF3E (一种包含ζ链的细胞内结构域和FKBP的嵌合受体) 二聚化, 从而启动依赖于CaN和NFAT的TCR信号传导。该结果阐明了分子胶水可以通过诱导PPIs在细胞内重建天然TCR信号。
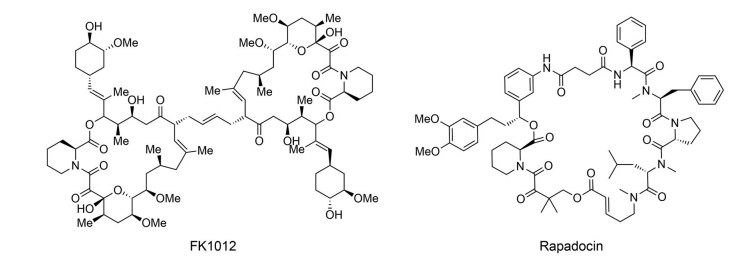
|
Figure 6 Structures of FK1012 and rapadocin |
此外, FK1012可以调节Src激酶的活化[48]。Src激酶家族(SFK) 是一类膜相关非受体酪氨酸激酶, 包括c-Src、Fyn、Lyn和Lck等, 可以调控许多生物学过程[49]。为了将Src激酶可逆地募集到膜上, Spencer等[48]将Fyn、Lyn和Lck的N-末端豆蔻酰化靶向肽除去, 并替换为FKBP12蛋白, 同时在细胞膜上表达FKBP12蛋白; FK1012可以通过连接两个FKBP12人为地诱导Src激酶的二聚化和活化, 对研究Src激酶的生物学功能提供了强有力的工具。
诸如此类的CIPs还有很多[50], 通过分子胶水诱导邻近蛋白关联, 可以远程控制基因表达和抑制、染色质甲基化和重构、细胞器和膜定位以及蛋白降解。深入理解诱导邻近在细胞内信号传导中的作用, 可以为设计新分子以控制用于实验和治疗目的的信号传导提供明确的途径。
1.8 RAPA衍生物rapadocinRAPA和FK506的分子结构可以简单划分为两部分: 一部分是类似结构的FKBP蛋白结合结构域(FKBD); 另一部分是不同结构的效应结构域(effector domain), 介导不同靶标(mTOR和CaN) 的结合。如果把RAPA和FK506的效应结构域互换, 那么其靶标蛋白就可以互换。受此启发, Guo等[51]通过寡肽组合文库替换RAPA效应结构域以构建靶向新蛋白的类RAPA大环分子库。通过化学合成并筛选出对FKBP12具有高亲和力的刚性片段FKBD10和FKBD11, 再在刚性片段上延伸出不同的四肽片段, 并采用关环复分解(RCM) 反应实现大环环化, 最终合成了具有45 000个化合物的分子库(命名为Rapafucins)。经过高通量筛选, 研究者发现了一个rapafucin能特异性抑制平衡型核苷转运蛋白1 (equilibrative nucleoside transporter 1, ENT1) 的转运功能, 并命名为rapadocin[51] (图 6)。
Rapadocin和RAPA/FK506在作用方式上既有相似也有差异之处。与RAPA和FK506一样, rapadocin以高亲和力与FKBP12结合, 然后靶向ENT1形成三元复合物。不同之处在于, rapadocin在没有FKBP的情况下对ENT1依然表现出明显的亲和力。FKBP12与mTOR之间的接触比FKBP12与CaN之间的接触面积少得多, 可能是由于RAPA比FK506的体积更大, 导致mTOR远离FKBP12。Rapadocin的结构甚至比RAPA还大, 因此不太可能允许靶标蛋白与FKBP有广泛的相互作用, 高亲和力很可能是通过rapadocin效应结构域的构象改变介导的, 从而促进了与ENT1的结合。Rapadocin是首个具有和FK506与RAPA类似作用机制的合成分子胶水。
ENT1的转运底物腺苷是一类重要的信号分子, 抑制ENT1导致的胞外腺苷增多被证明对缺血性再灌注损伤等无数疾病具有治疗益处。动物实验表明rapadocin对小鼠肾脏缺血再灌注损伤具有保护作用[51], 阐明了其在体内依然具有功效。从Rapafucins文库中鉴定出ENT1新型抑制剂表明了对RAPA分子胶水进行重塑和改造可以靶向新的蛋白。
2 分子胶水降解剂如前所述, 沙利度胺等分子胶水降解剂的出现极大拓展了分子胶水的范畴。然而, 沙利度胺系列降解剂是合成化合物, 不属于天然产物及其衍生物。鉴于目前已有关于沙利度胺等合成的分子胶水降解剂的详尽综述[52, 53], 故本文不再做介绍。天然产物来源的植物激素如生长素auxin和茉莉酸jasmonate是分子胶水降解剂的早期例子[16], 然而其降解作用没有在人源的细胞做过研究, 因此也不在本文的讨论范畴。本部分内容只简单回顾来源于天然产物的分子胶水降解剂醋酸棉酚(gossypol acetate, 图 7) 的发现与作用机制。
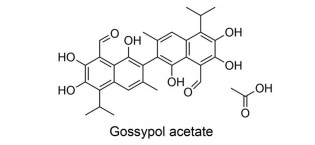
|
Figure 7 Structure of gossypol acetate |
自噬是除蛋白酶体降解之外的另一种蛋白降解途径[54]。2019年, Li等[55]报道了一种自噬小体绑定化合物(autophagosome-tethering compound, ATTEC) 可以触发自噬降解, 通过同时靶向自噬体蛋白LC3和亨廷顿致病突变蛋白(mHTT) 以促进自噬降解mHTT, 在治疗亨廷顿舞蹈症(HD) 方面显示出有效的治疗活性。醋酸棉酚是一种来源于棉花的棉籽及棉根皮中的多酚类物质, 用于橡胶抗氧剂和杀虫剂, 近年来也被证明具有抗癌及抗病毒作用。2021年, Li等[56]报道了天然产物醋酸棉酚是一种新型的ATTEC。VCP (valosin-containig protein) 是蛋白质稳态的关键调节剂, 通过蛋白酶体和自噬调节受损蛋白质的降解。VCP与HD等其他神经退行性疾病的发病机制密切相关, 因此特异性调节VCP活性的小分子有可能成为治疗HD等疾病患者的先导化合物。研究者通过基于VCP ATPase活性的高通量筛选方法, 发现了醋酸棉酚能有效地抑制VCP的酶活。醋酸棉酚直接作用在VCP蛋白的N和D1区域, 诱导VCP-LC3-mHTT三元复合物的形成, 从而利用自噬体降解mHTT。进一步的研究证明, 醋酸棉酚不但能降低mHTT水平, 减轻HD神经元的毒性, 还可以改善果蝇和小鼠模型中HD疾病的相关行为, 使醋酸棉酚成为开发新HD疗法非常有前途的先导化合物。
3 总结与展望分子胶水是一类可诱导PPIs形成并进而产生新的生物学功能或治疗效果的小分子。与结合活性位点或变构位点以调节其靶蛋白活性的常规小分子不同, 分子胶水通过诱导邻近效应来影响其靶蛋白的活性, 甚至改变其功能。分子胶水可以在没有药物结合腔穴的情况下靶向蛋白, 有极大的潜力扩展可成药的蛋白质靶点库和化学空间。
分子胶水的发现打破了很多蛋白不可成药的传说。在20世纪80年代, 细胞内信号传导、蛋白磷酸酶和蛋白激酶被认为是不可成药的靶点。CsA被发现通过抑制蛋白磷酸酶CaN, 从而阻断细胞内信号转导。CsA的上市打破了信号转导和蛋白磷酸酶不可成药的说法。之后, 随着mTOR抑制剂RAPA (西罗莫司) 的批准, 蛋白激酶也变成可靶的药物靶标。长期以来, 缺乏小分子结合腔穴的蛋白质也被认为是不可成药的。大环内酯WDB002通过二元复合物FKBP12-WDB002结合CEP250几乎没有特征的卷曲盘状表面, 进一步增加了靶标范围。分子胶水降解剂具有催化作用机制和靶向降解非酶蛋白的能力, 醋酸棉酚等ATTEC分子胶水的发现进一步拓展了可成药蛋白的范围, 从而治疗之前无法治疗的疾病。
早期的基于天然产物及其衍生物的分子胶水的发现都是偶然。20世纪90年代, 在相继发现cyclosporin、FK506、rapamycin和sanglifehrin之后, 这类分子胶水的发展停滞了一段时间。近年来, 研究者尝试用合理的设计和革新的生物技术去挖掘新的分子胶水, WDB002、asukamycin、manumycin、rapadocin和醋酸棉酚的发现标志着基于天然产物及其衍生物的分子胶水又迎来了新的发展时期。WDB002的发现表明了利用合理的基因挖掘方法能有效地从细菌中发现新的分子胶水; asukamycin和manumycin的发现表明了含有多个亲电位点的天然产物是分子胶水新的来源; rapadocin和醋酸棉酚的发现则证明了传统的组合化学和高通量筛选依然是发现新型分子胶水的有力手段。
尽管近年来基于天然产物及其衍生物的分子胶水的研究取得了一定进展, 但仍处于起步阶段, 要合理地设计并发展成治疗药物还有很长的路要走。学界内发展的一系列合理发现分子胶水的技术, 如高通量筛选、多共价化学蛋白质组学分析、CRISPR技术、系统数据挖掘和化学遗传学等, 促进了新分子胶水的识别和结合模式鉴定。但这些方法主要依赖于密集筛选和随后的机制验证, 缺乏合理的设计方法与策略, 限制了分子胶水发现的效率和适用性。系统性地深入理解PPIs对发现分子胶水至关重要, 通过晶体学和冷冻电子显微镜(cryo-EM) 对PPIs的结构进行分析, 同时开发新的计算工具来预测分子胶水诱导的PPI复合物的结合模型, 都将有助于虚拟筛选和基于结构的新型分子胶水的合理设计。此外, 人工智能(AI) 技术的整合将有助于提高数据挖掘和分子设计的效率, 例如机器学习(ML) 算法可能具有从大量高通量筛选、多组学和PPIs网络数据构建可靠模型, 并精确预测潜在的“蛋白-分子胶水-蛋白”组合的优势。此外, ML通过基于可用结构和构效关系(structure activity relationships, SAR) 信息构建模型, 也将有助于指导新型分子胶水的设计和优化。
我国有着丰富的天然产物资源, 合理利用和整合多种新兴技术, 从中发现和挖掘分子胶水, 也许是拓展天然产物的药用价值和作为新型工具分子用于基础研究的潜在方向。
作者贡献: 何佳撰写中英文摘要及正文; 宋坤玲画图; 郭祖奉和党永军提供综述的全文思路和结构; 所有作者共同参与了文章的撰写及修改。
利益冲突: 本文作者声明没有利益冲突。
| [1] |
Hopkins AL, Groom CR. The druggable genome[J]. Nat Rev Drug Discov, 2002, 1: 727-730. DOI:10.1038/nrd892 |
| [2] |
Russ AP, Lampel S. The druggable genome: an update[J]. Drug Discov Today, 2005, 10: 1607-1610. DOI:10.1016/S1359-6446(05)03666-4 |
| [3] |
Dixon SJ, Stockwell BR. Identifying druggable disease-modifying gene products[J]. Curr Opin Chem Biol, 2009, 13: 549-555. DOI:10.1016/j.cbpa.2009.08.003 |
| [4] |
Bonetta L. Interactome under construction[J]. Nature, 2010, 468: 851-854. DOI:10.1038/468851a |
| [5] |
Vidal M, Cusick ME, Barabasi AL. Interactome networks and human disease[J]. Cell, 2011, 144: 986-998. DOI:10.1016/j.cell.2011.02.016 |
| [6] |
Lu HC, Fornili A, Fraternali F. Protein-protein interaction networks studies and importance of 3D structure knowledge[J]. Expert Rev Proteom, 2013, 10: 511-520. DOI:10.1586/14789450.2013.856764 |
| [7] |
Cesa LC, Mapp AK, Gestwicki JE. Direct and propagated effects of small molecules on protein-protein interaction networks[J]. Front Bioeng Biotechnol, 2015, 3: 119. |
| [8] |
Stumpf MPH, Thorne T, de Silva E, et al. Estimating the size of the human interactome[J]. Proc Natl Acad Sci U S A, 2008, 105: 6959-6964. DOI:10.1073/pnas.0708078105 |
| [9] |
Venkatesan K, Rual JF, Vazquez A, et al. An empirical framework for binary interactome mapping[J]. Nat Methods, 2009, 6: 83-90. DOI:10.1038/nmeth.1280 |
| [10] |
Arkin MR, Wells JA. Small-molecule inhibitors of protein-protein interactions: progressing towards the dream[J]. Nat Rev Drug Discov, 2004, 3: 301-317. DOI:10.1038/nrd1343 |
| [11] |
Newman DJ, Cragg GM. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019[J]. J Nat Prod, 2020, 83: 770-803. DOI:10.1021/acs.jnatprod.9b01285 |
| [12] |
Liu J, Farmer JD, Lane WS, et al. Calcineurin is a common target of cyclophilin-cyclosporin A and FKBP-FK506 complexes[J]. Cell, 1991, 66: 807-815. DOI:10.1016/0092-8674(91)90124-H |
| [13] |
Stanton BZ, Chory EJ, Crabtree GR. Chemically induced proximity in biology and medicine[J]. Science, 2018, 359: eaao5902. DOI:10.1126/science.aao5902 |
| [14] |
Wurtele M, Jelich-Ottmann C, Wittinghofer A, et al. Structural view of a fungal toxin acting on a 14-3-3 regulatory complex[J]. EMBO J, 2003, 22: 987-994. DOI:10.1093/emboj/cdg104 |
| [15] |
Milroy LG, Grossmann TN, Hennig S, et al. Modulators of protein-protein interactions[J]. Chem Rev, 2014, 114: 4695-4748. DOI:10.1021/cr400698c |
| [16] |
Che Y, Gilbert AM, Shanmugasundaram V, et al. Inducing protein-protein interactions with molecular glues[J]. Bioorg Med Chem Lett, 2018, 28: 2585-2592. DOI:10.1016/j.bmcl.2018.04.046 |
| [17] |
Ruegger A, Kuhn M, Lichti H, et al. Cyclosporin-A, a peptide metabolite from Trichoderma-polysporum (link ex pers) Rifai, with a remarkable immunosuppressive activity[J]. Helv Chim Acta, 1976, 59: 1075-1092. DOI:10.1002/hlca.19760590412 |
| [18] |
Sehgal SN, Baker H, Vezina C. Rapamycin (AY-22, 989), a new antifungal antibiotic II. fermentation, isolation and characterization[J]. J Antibiot, 1975, 28: 727-732. DOI:10.7164/antibiotics.28.727 |
| [19] |
Tanaka H, Kuroda A, Marusawa H, et al. Structure of FK506: a novel immunosuppressant isolated from Streptomyces[J]. J Am Chem Soc, 1987, 109: 5031-5033. DOI:10.1021/ja00250a050 |
| [20] |
Stahelin HF. The history of cyclosporin A (sandimmune(R)) revisited: another point of view[J]. Experientia, 1996, 52: 5-13. DOI:10.1007/BF01922409 |
| [21] |
Handschumacher RE, Harding MW, Rice J, et al. Cyclophilin: a specific cytosolic binding-protein for cyclosporin A[J]. Science, 1984, 226: 544-547. DOI:10.1126/science.6238408 |
| [22] |
Nigro P, Pompilio G, Capogrossi MC. Cyclophilin A: a key player for human disease[J]. Cell Death Dis, 2013, 4: e888. DOI:10.1038/cddis.2013.410 |
| [23] |
Klee CB, Ren H, Wang X. Regulation of the calmodulin-stimulated protein phosphatase, calcineurin[J]. J Biol Chem, 1998, 273: 13367-13370. DOI:10.1074/jbc.273.22.13367 |
| [24] |
Schreiber SL. Immunophilin-sensitive protein phosphatase action in cell signaling pathways[J]. Cell, 1992, 70: 365-368. DOI:10.1016/0092-8674(92)90158-9 |
| [25] |
Schreiber SL. Chemistry and biology of the immunophilins and their immunosuppressive ligands[J]. Science, 1991, 251: 283-287. DOI:10.1126/science.1702904 |
| [26] |
Crabtree GR, Schreiber SL. SnapShot: Ca2+-calcineurin-NFAT signaling[J]. Cell, 2009, 138: 210, 210.e1. |
| [27] |
Huai Q, Kim HY, Liu YD, et al. Crystal structure of calcineurin-cyclophilin-cyclosporin shows common but distinct recognition of immunophilin-drug complexes[J]. Proc Natl Acad Sci U S A, 2002, 99: 12037-12042. DOI:10.1073/pnas.192206699 |
| [28] |
Siekierka JJ, Hung SHY, Poe M, et al. A cytosolic binding-protein for the immunosuppressant FK506 has peptidyl-prolyl isomerase activity but is distinct from cyclophilin[J]. Nature, 1989, 341: 755-757. DOI:10.1038/341755a0 |
| [29] |
Schreiber SL, Crabtree GR. The mechanism of action of cyclosporine-A and FK506[J]. Immunol Today, 1992, 13: 136-142. DOI:10.1016/0167-5699(92)90111-J |
| [30] |
Griffith JP, Kim JL, Kim EE, et al. X-ray structure of calcineurin inhibited by the immunophilin immunosuppressant FKBP12-FK506 complex[J]. Cell, 1995, 82: 507-522. DOI:10.1016/0092-8674(95)90439-5 |
| [31] |
Kissinger CR, Parge HE, Knighton DR, et al. Crystal-structures of human calcineurin and the human FKBP12-FK506-calcineurin complex[J]. Nature, 1995, 378: 641-644. DOI:10.1038/378641a0 |
| [32] |
Martel RR, Klicius J, Galet S. Inhibition of immune-response by rapamycin, a new antifungal antibiotic[J]. Can J Physiol Pharmacol, 1977, 55: 48-51. DOI:10.1139/y77-007 |
| [33] |
Sabatini DM, Erdjument-Bromage H, Lui M, et al. RAFT1: a mammalian protein that binds to FKBP12 in a rapamycin-dependent fashion and is homologous to yeast TORs[J]. Cell, 1994, 78: 35-43. DOI:10.1016/0092-8674(94)90570-3 |
| [34] |
Brown EJ, Albers MW, Shin TB, et al. A mammalian protein targeted by G1-arresting rapamycin-receptor complex[J]. Nature, 1994, 369: 756-758. DOI:10.1038/369756a0 |
| [35] |
Saxton RA, Sabatini DM. mTOR signaling in growth, metabolism, and disease[J]. Cell, 2017, 168: 960-976. DOI:10.1016/j.cell.2017.02.004 |
| [36] |
Choi JW, Chen J, Schreiber SL, et al. Structure of the FKBP12-rapamycin complex interacting with the binding domain of human FRAP[J]. Science, 1996, 273: 239-242. DOI:10.1126/science.273.5272.239 |
| [37] |
Schreiber SL. The rise of molecular glues[J]. Cell, 2021, 184: 3-9. DOI:10.1016/j.cell.2020.12.020 |
| [38] |
Sedrani R, Kallen J, Cabrejas LM, et al. Sanglifehrin-cyclophilin interaction: degradation work, synthetic macrocyclic analogues, X-ray crystal structure, and binding data[J]. J Am Chem Soc, 2003, 125: 3849-3859. DOI:10.1021/ja021327y |
| [39] |
Pua KH, Stiles DT, Sowa ME, et al. IMPDH2 is an intracellular target of the cyclophilin A and sanglifehrin A complex[J]. Cell Rep, 2017, 18: 432-442. DOI:10.1016/j.celrep.2016.12.030 |
| [40] |
Shigdel UK, Lee SJ, Sowa ME, et al. Genomic discovery of an evolutionarily programmed modality for small-molecule targeting of an intractable protein surface[J]. Proc Natl Acad Sci U S A, 2020, 117: 17195-17203. DOI:10.1073/pnas.2006560117 |
| [41] |
Fry AM, Bayliss R, Roig J. Mitotic regulation by NEK kinase networks[J]. Front Cell Dev Biol, 2017, 5: 102. DOI:10.3389/fcell.2017.00102 |
| [42] |
Hardy T, Lee M, Hames RS, et al. Multisite phosphorylation of C-Nap1 releases it from Cep135 to trigger centrosome disjunction[J]. J Cell Sci, 2014, 127: 2493-2506. |
| [43] |
Mardin BR, Schiebel E. Breaking the ties that bind: new advances in centrosome biology[J]. J Cell Biol, 2012, 197: 11-18. DOI:10.1083/jcb.201108006 |
| [44] |
Silkworth WT, Nardi IK, Paul R, et al. Timing of centrosome separation is important for accurate chromosome segregation[J]. Mol Biol Cell, 2012, 23: 401-411. DOI:10.1091/mbc.e11-02-0095 |
| [45] |
Wells JA, McClendon CL. Reaching for high-hanging fruit in drug discovery at protein-protein interfaces[J]. Nature, 2007, 450: 1001-1009. DOI:10.1038/nature06526 |
| [46] |
Isobe Y, Okumura M, McGregor LM, et al. Manumycin polyketides act as molecular glues between UBR7 and P53[J]. Nat Chem Biol, 2020, 16: 1189-1198. DOI:10.1038/s41589-020-0557-2 |
| [47] |
Pruschy MN, Spencer DM, Kapoor TM, et al. Mechanistic studies of a signaling pathway activated by the organic dimerizer FK1012[J]. Chem Biol, 1994, 1: 163-172. DOI:10.1016/1074-5521(94)90006-X |
| [48] |
Spencer DM, Graef I, Austin DJ, et al. A general strategy for producing conditional alleles of Src-like tyrosine kinaSES[J]. Proc Natl Acad Sci U S A, 1995, 92: 9805-9809. DOI:10.1073/pnas.92.21.9805 |
| [49] |
Kim LC, Song L, Haura EB. Src kinases as therapeutic targets for cancer[J]. Nat Rev Clin Oncol, 2009, 6: 587-595. DOI:10.1038/nrclinonc.2009.129 |
| [50] |
Gerry CJ, Schreiber SL. Unifying principles of bifunctional, proximity-inducing small molecules[J]. Nat Chem Biol, 2020, 16: 369-378. DOI:10.1038/s41589-020-0469-1 |
| [51] |
Guo Z, Hong SY, Wang J, et al. Rapamycin-inspired macrocycles with new target specificity[J]. Nat Chem, 2019, 11: 254-263. DOI:10.1038/s41557-018-0187-4 |
| [52] |
Dong G, Ding Y, He S, et al. Molecular glues for targeted protein degradation: from serendipity to rational discovery[J]. J Med Chem, 2021, 64: 10606-10620. DOI:10.1021/acs.jmedchem.1c00895 |
| [53] |
Kozicka Z, Thoma NH. Haven't got a glue: protein surface variation for the design of molecular glue degraders[J]. Cell Chem Biol, 2021, 28: 1032-1047. DOI:10.1016/j.chembiol.2021.04.009 |
| [54] |
Mizushima N, Levine B, Cuervo AM, et al. Autophagy fights disease through cellular self-digestion[J]. Nature, 2008, 451: 1069-1075. DOI:10.1038/nature06639 |
| [55] |
Li Z, Wang C, Wang Z, et al. Allele-selective lowering of mutant HTT protein by HTT-LC3 linker compounds[J]. Nature, 2019, 575: 203-209. DOI:10.1038/s41586-019-1722-1 |
| [56] |
Li XJ, Zhang YY, Fu YH, et al. Gossypol, a novel modulator of VCP, induces autophagic degradation of mutant huntingtin by promoting the formation of VCP/p97-LC3-mHTT complex[J]. Acta Pharmacol Sin, 2021, 42: 1556-1566. DOI:10.1038/s41401-020-00605-0 |
 2022, Vol. 57
2022, Vol. 57


