单细胞组学技术作为近年发展迅速的生命科学前沿技术, 是指在对样品中单个细胞进行分离之后, 采用基因组测序、转录组测序、蛋白质组检测和代谢组检测等技术进行研究, 从而获取单个细胞的基因组、转录组和蛋白质组等组学信息[1, 2]。在信息采集的深度与广度方面, 单细胞组学技术能够在以往针对组织或器官整体的研究层次上进一步深入到“细胞”水平, 并对各种层次的组学信息进行解析, 在全面揭示细胞间异质性方面具有独特优势。目前以单细胞转录组测序为代表的单细胞组学技术已经广泛应用于肿瘤[3, 4]、免疫[5, 6]、神经系统[7]以及发育[8]等领域。而单细胞组学技术在药物研究尤其是药理领域的应用也逐渐成为人们关注的焦点, 并且相关研究已取得关键性进展与突破[9-11]。
中药是中医治疗、保健和预防疾病的主要载体, 临床主要用药形式包括复方、单味药等, 具有多成分、多靶点、多途径协同作用的药理特点。目前在中药药理研究中主要面临有效成分不清晰、作用靶标谱不完整、作用机制不明确等局限, 成为限制中药现代化和国际化的主要瓶颈, 也是亟待突破的关键问题[12]。细胞是药物分子直接的作用对象, 也是药物具体效能的客观反映。笔者认为, 通过以细胞作为研究切入点与突破口, 将单细胞组学技术应用于中药药理的“成分—靶标—机制”研究之中, 构建“单细胞药理学(single cell pharmacology, SCP)”的研究体系, 将为攻克上述复杂难题提供可行途径与有效策略, 从而创造中药现代化研究的崭新模式。
为此, 本文综述以单细胞转录组测序为代表的单细胞组学技术体系及其在现代药物药理中的研究进展, 以此为基础探究单细胞药理学在中药领域的研究思路。通过应用单细胞组学技术, 促进对中药单药或复方中有效成分的筛选, 以及对中药成分的作用机制与效应规律的探究, 并推进针对作用靶点的新药创制或者拓展经典药物的适应范围, 从而加速中医药现代化与国际化的进程。
1 单细胞组学技术体系概述 1.1 单细胞组学技术发展现状以中心法则为主线, 单细胞组学技术主要包括基于高通量测序平台开展的单细胞基因组、转录组测序, 以及基于质谱等平台的单细胞蛋白质组、代谢组检测技术[13, 14]。其中, 单细胞基因组测序技术通过对分离的单细胞基因组进行多重链置换扩增(multiple displacement amplification, MDA)、多次退火环状循环扩增技术(multiple annealing and looping-based amplification cycles, MALBAC) 等全基因组扩增, 并使用高通量测序对其变异位点进行检测, 从而解析同一个体不同细胞之间在基因组序列上的差异[15, 16], 其应用主要集中在生殖遗传[17]、肿瘤异质性与亚克隆进化[3, 18]、液体活检[19, 20]等研究领域。单细胞蛋白质组学以及单细胞代谢组学检测技术则是分别对单个细胞的蛋白质以及内源性代谢物进行定性和定量研究[21-23]。由于蛋白质和代谢物均不具备扩增特性, 并且单个细胞中的所含物质含量较低、种类繁多、丰度分布差异较大, 对于检测方法的灵敏度与特异性提出了极高要求, 导致目前发展相对滞后[24]。尽管目前已有基于串联质谱标签(tandem mass tag, TMT) 标记和液质联用技术(liquid chromatography and tandem mass spectrometry, LC-MS/MS) 的SCoPE-MS[25]/SCoPE-MS2[26]等单细胞蛋白质组检测技术, 以及基于基质辅助激光解吸(matrix-assisted laser desorption/ionization, MALDI) 成像质谱的SpaceM[27]等单细胞代谢组检测技术涌现, 其具有较高的细胞检测通量与蛋白质/代谢物定量准确性, 然而鲜有应用方面的报道。上述技术囿于其应用范围、检测通量或者技术瓶颈, 目前仍难以成为现代药物乃至中药药理领域中普适性、可行性的研究方法。
自2009年Tang等[28]首次报道对小鼠单个卵裂球进行转录组测序以来, 单细胞转录组测序(single cell RNA sequencing, scRNA-Seq) 历经十余年的发展与革新, 技术流程的成熟稳健与应用范围的灵活多样使其在单细胞组学技术体系之中独树一帜。该技术通过对样品中单个细胞进行分离裂解, 提取以mRNA为主的RNA并逆转录合成cDNA, 随后进行扩增建库以及高通量测序, 从而获取样品中各个细胞的基因表达信息[29]。scRNA-Seq目前已广泛用于表征细胞亚群功能状态、揭示基因表达模式异质性、构建细胞发育轨迹、研究细胞-细胞通讯互作等[30, 31]。上述方法使得研究人员能够深入研究细胞层次的生命活动过程, 揭示疾病发生发展规律, 同时也为探索药物的作用靶点以及分子机制提供崭新的方法学支撑。
基于单细胞组学技术体系的发展现状, 笔者认为应通过以点带面, 协同推进的策略, 将以scRNA-Seq为代表的单细胞组学技术应用于单细胞药理学研究之中, 促进对中药单药或复方中有效成分的高效筛选, 以及对药物成分的作用机制与效应规律等方面的深入探究。
1.2 单细胞转录组测序技术scRNA-Seq的检测原理与组织转录组测序(bulk cell RNA sequencing, Bulk RNA-Seq) 相似, 不同之处在于其研究对象从群体组织转移到单个细胞。如图 1所示, 其关键技术流程主要包括对单细胞进行分离捕获、对微量RNA分子信号进行扩增, 以及后续对scRNA-Seq数据进行数据分析。
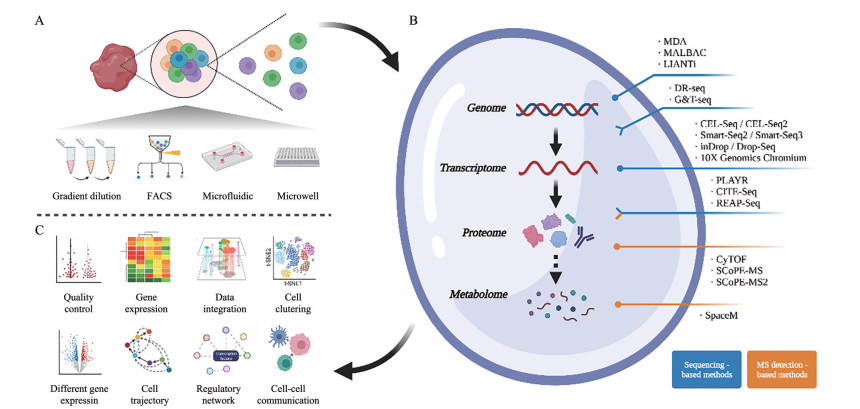
|
Figure 1 Single cell omics technology and single-cell RNA-sequencing analysis workflow. (A) Single cell dissociation; (B) Single cell omics technology; (C) Bioinformatics analysis of scRNA-Seq. FACS: Fluorescence activated cell sorting; MDA: Multiple displacement amplification; LIANTI: Linear amplification via transposon insertion; G & T-seq: Genome and transcriptome sequencing; CEL-Seq: Cell expression by linear amplification and sequencing; SMART-Seq: Switching mechanism at 5' end of the RNA transcript sequencing; PLAYR: Proximity ligation assay for RNA; CITE-Seq: Cellular indexing of transcriptomes and epitopes by sequencing; REAP-Seq: RNA expression and protein sequencing assay; CyTOF: Cytometry time of flight; SCoPE-MS: Single cell ProtEomics by Mass spectrometry; SpaceM: MALDI-based single-cell metabolomics |
单细胞分离技术根据其原理不同可分为梯度稀释、荧光激活细胞分选(fluorescence activated cell sorting, FACS)、微流控分离(microfluidics)、微孔板分离等。梯度稀释法[32]操作简易、成本低廉, 然而分离准确度较低, 主要用于人工培养样品相关研究。FACS[33]基于流式细胞仪对细胞悬液中激发特定荧光信号的靶细胞进行收集, 准确高效, 然而分选过程可能会对细胞活性与形态造成影响, 适用于对细胞活性和形态要求较低的相关研究。微流控分离技术[34, 35]采用微流体芯片装置, 将细胞封闭于微滴中进行分离, 系统集成, 选样灵活, 分选效率与稳定性较高, 对细胞造成的损伤较低, 然而对细胞大小有所限制。而微孔板分离技术[36]主要将细胞分散在单个微孔从而实现细胞分离, 能够克服细胞大小的影响, 而其细胞检测通量与微流控分离技术等相比较低。上述方法的建立, 促进了细胞活性以及检测通量的提升, 也为后续优化与整合转录组扩增技术奠定基础。
随着近10年的发展与迭代, 各种高效、灵敏的scRNA-Seq扩增方法不断出现, 其中主要包括基于体外转录(cell expression by linear amplification and sequencing) 的CEL-Seq/CEL-Seq2技术、基于模板转换(switching mechanism At 5' end of the RNA transcript, SMART) 的Smart-Seq2/Smart-Seq3技术、结合微流控系统的inDrop/Drop-Seq技术以及10X Genomics的Chromium平台。其中, CEL-Seq/CEL-Seq2[37, 38]技术对转录组进行体外线性扩增, 能够降低扩增偏倚, 然而其具有较高的3'端偏好性, 以及对低丰度转录本的低灵敏度。Smart-Seq2[39, 40]/Smart-Seq3[41]技术基于逆转录与模板转换捕获并扩增基因的全长cDNA, 除了能够捕获较高数量及覆盖度的转录本, 还能够用于检测单细胞水平的单核苷酸变异位点、可变剪接等相关研究。inDrop[42]和Drop-Seq[43]技术结合微流控分离和细胞条形码标记技术, 通过微珠捕获同一细胞来源的RNA并于液滴中进行扩增反应, 在提升产物浓度、扩大细胞检测通量的同时也降低了建库成本, 而10X Genomics的Chromium平台[44]在前者基础上进一步采用油滴对反应体系进行封装, 使得细胞检测数量以及平台拓展性显著提高, 并且采用独特分子识别标识(unique molecular identifiers, UMIs) 标记单个mRNA片段, 在后续分析中能够有效降低PCR扩增所带来的偏倚, 提升转录本定量的准确性。
由于检测方法的多样、信号类型的复杂以及批次效应的影响, scRNA-Seq所产生的测序数据中包含大量细胞的基因表达信息, 呈现“高维”、“稀疏”的数据分布特征, 有赖于后续生物信息学的方法进行存储、分析、处理以及可视化[30, 45, 46]。在整个分析流程中, 需要依次进行数据质控、基因表达定量与标准化、批次整合、降维聚类以及差异基因表达等多个步骤, 各个步骤需要根据检测方式和数据分布特征等对分析方法进行评估与选择, 以保障分析结果能够准确表征细胞状态或功能, 并揭示其与组学信息的联系[47, 48]。此外, 随着转录调控分析[49]、拟时序分析[50]以及细胞通讯分析[51]等新颖分析方法的建立, 极大丰富研究人员对单细胞的基因共表达调控网络、细胞状态转变与基因表达动态变化, 以及细胞间基于配体-受体对的互作通讯的理解。
综上, 细胞检测通量与效率的增加、信息捕获完整和准确程度的提升以及分析方法类型与应用的丰富, 共同促进scRNA-Seq技术的快速发展, 研究人员能够根据自身的研究目的以及技术特点, 采用相应的scRNA-Seq分析方案进行课题研究[52, 53]。随着各种自动化、高通量、低成本的商业化平台如10X Genomics Chromium/Connect、BD Rhapsody的普及与应用, 将极大地推进基于scRNA-Seq的研究工作的开展, 也为scRNA-Seq在现代药物以及中药药物研究的应用提供坚实的研究平台。
1.3 单细胞多组学联合技术单细胞组学技术的应用使得研究人员能够逐一对单个细胞的基因组、转录组、蛋白质组和代谢组信息进行研究, 对于细胞多样性产生变革性的见解。然而由于疾病机制的高复杂性以及各组学层次间的低关联性, 使得在单一层次模式近似的细胞亚群, 在其他层次中仍然可能具有差异[54]。因此, 在单细胞中整合多个组学信息能够弥补单一组学视角的局限, 更加全面表征细胞的状态与功能, 充分揭示细胞之间异质性以及不同组学信息之间的动态联系, 上述研究策略也与中医药中的“整体观”和“系统观”不谋而合。
目前单细胞多组学联合技术主要以单细胞基因组-转录组联合测序和单细胞转录组-蛋白质联合检测技术为主。单细胞基因组-转录组联合测序能够从单个细胞中获得基因组和转录组的联合信息, 研究人员相继开发了对同一单细胞裂解物中的DNA和RNA进行同步和分别扩增与测序的DR-seq[55]技术和G & T-seq[56]技术, 有助于检测细胞间表达的致病遗传变异, 从而揭示遗传信息对于基因表达与细胞状态的调控机制。而单细胞转录组-蛋白质联合检测方面的代表性例子为PLAYR (proximity ligation assay for RNA) 技术、CITE-Seq (cellular indexing of transcriptomes and epitopes by sequencing) 以及REAP-Seq (RNA expression and protein sequencing assay)。PLAYR[57]分别采用同位素标记的探针序列与抗体分别标记RNA与蛋白质, 并通过质谱流式细胞技术(mass cytometry) 检测数千个细胞的转录组与蛋白质组的信息。而CITE-Seq[58]和REAP-Seq[59]两种方法基于寡聚核苷酸链偶联的抗体标记, 从而整合胞外蛋白质信号捕获与转录组测序步骤, 但细胞检测通量以及蛋白鉴定数量相对较少, 大约为80~100种。
随着细胞检测通量的提高、组学信息的丰富以及检测成本的降低, 单细胞多组学技术具备极大的发展潜力, 也被《Nature Methods》评为“2019年年度技术”[54]。对于单细胞多个组学信息的整合分析, 能够促进对于细胞的复杂调控过程尤其是对于中药药物作用靶点、治疗机制和治疗反应等药理机制的理解[60]。
2 单细胞组学技术在现代药物研究的应用现状目前, 以scRNA-Seq为主的单细胞组学技术已广泛应用于化疗药物、小分子抑制剂、抗体等。如图 2所示, 相关研究通过以药物治疗前后、治疗方法与剂量或时间因素差异、治疗响应程度高低等对样品划分组别, 使用scRNA-Seq等技术分析不同组别的细胞功能状态与基因表达模式, 进而揭示药物作用机制、高效筛选药物、评估药物疗效以及阐明耐药机制等。故此, 本章节通过回顾单细胞组学技术在现代药物研究领域的应用进展, 以期为该技术在中药药理研究的应用提供思路借鉴和方法参考。
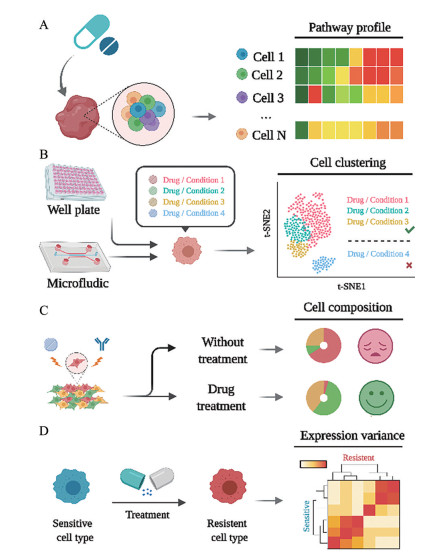
|
Figure 2 The application of single cell omics technology in modern drugs research. (A) Promoting mechanistic research. (B) Facilitating drug screening. (C) Drug efficacy evaluation. (D) Revealing drug resistant mechanism |
scRNA-Seq能够大规模、高分辨率地捕获基因表达信息, 可以全面、准确地研究健康、恶性以及药物处理后组织中的细胞类型和分子状态, 为药物作用靶点和效应通路的发现提供更多信息, 从而推动理解药物在疾病中的作用机制[10, 61]。
近年来几项研究采用scRNA-Seq用于揭示不同肿瘤类型的肿瘤微环境(tumor micro environment, TME) 的细胞构成和分子表达模式, 通过对比药物治疗前后TME中对肿瘤浸润淋巴细胞和基质细胞细胞类型与状态, 从而在复杂微环境系统中揭示药物的作用机制[62, 63]。Zheng等[64]则在单细胞水平对肝癌肿瘤微环境中的5 000个T淋巴细胞转录组及TCR进行综合分析, 揭示了肿瘤相对于其他部位的T细胞在功能、分布和发展状态方面的异质性。研究发现Layilin基因在效应CD8+ T细胞以及抑制性T细胞(Treg) 中的特异性表达, 并通过体外实验证明该基因能够抑制CD8+ T细胞的杀伤作用, 可能作为免疫疗法的潜在作用靶点, 从而促进新型肝癌免疫疗法的建立。
对于其他疾病领域, scRNA-Seq技术也有助于解析疾病背景下各种类型细胞的基因表达谱、丰度以及相互作用, 从而为检测药物作用靶标和作用途径, 进而指导疾病的靶向治疗提供可能。药物诱发的超敏反应综合征/伴有嗜酸性粒细胞增多和全身症状的药物反应(drug-induced hypersensitivity syndrome/drug reaction with eosinophilia and systemic symptoms, DiHS/DRESS) 是一种潜在致命的多器官炎症性疾病, 与疱疹病毒再激活和随后发生的自身免疫性疾病相关[65]。Kim等[66]对难治嗜酸性粒细胞增多和全身症状患者的皮肤和血液样本进行单细胞转录组测序, 研究鉴定JAK-STAT信号通路为潜在治疗靶标, 中央记忆CD4+ T细胞富含人类疱疹病毒DNA。通过托法替尼的干预在体外抑制病因引起的T细胞增殖, 进一步验证JAK-STAT途径和疱疹病毒在介导药物不良反应中的作用。
2.2 提高药物筛选效率药物筛选是药物研发流程中至关重要的环节, 其中筛选通量的提高直接缩短药物的研发周期, 进而降低其研发成本。基于高通量测序的基因表达分析能够反映处理样品对于药物作用的响应程度并作为评估药物疗效的筛选依据之一, 目前已经广泛地应用于新药研究之中[67]。而scRNA-Seq则能将评估药物响应程度的分辨率从组织水平提升到细胞类型水平上, 既能评估同种细胞对于不同药物作用条件如种类、时间、剂量等的响应程度异同; 也能反映不同类型细胞对相同药物处理的响应程度的差异。
Ye等[68]开发了一种高通量药物筛选平台DRUG-seq, 基于不同化合物作用后的基因表达谱对细胞进行tSNE聚类, 其中同一亚簇中不同化合物的药理作用机制相似。该研究采用433种化合物处理骨肉瘤U2OS细胞, 其中88个被鉴定为导致具有50个以上基因显著改变的有效化合物, 涉及信号转导、细胞周期和翻译等作用途径。通过该技术能够对作用机制相似的化合物进行高通量筛选, 并快速确定未知化合物的可能作用途径与作用靶标。Shin等[69]采用瞬时转染短条形码寡核苷酸(short barcoding oligos, SBOs) 用于标记多种实验条件下的细胞样品, 并结合Drop-Seq单细胞测序技术对药物作用的基因表达特征进行分析。该研究探究5种药物处理时间和45种药物处理慢性髓系白血病K562细胞的单细胞测序结果, 揭示了不同处理时间下基因表达轨迹, 同时检测了不同药物作用的靶标特异性基因, 为临床药物选择提供参考依据。Srivatsan等[70]开发出sci-Plex, 结合细胞核散列(nuclear hashing) 标记与单细胞转录组测序技术, 在单细胞分辨率下对数千个药物干扰的全局转录反应进行评估, 并且将单个细胞的建库成本降低到每个细胞低于0.01美元。研究对使用188种化合物处理的3个癌细胞系, 应用sci-Plex在单次实验中对约5 000个独立样本中的约65万个单细胞转录组进行解析, 高效鉴定了不同类型细胞对特定化合物反应的差异, 以及对同一类化合物家族反应的共性。
上述研究表明, 通过应用单细胞组学技术进行药物的高通量筛选, 能够在提高药物类型及作用条件筛选效率的同时降低筛选成本, 从而为新药的快速研制提供强大助力。
2.3 评估药物治疗效果单细胞组学技术在评价药物治疗疗效方面, 能够通过对比同一研究对象中治疗前后、不同组别对象的细胞丰度、基因表达模式变化, 从而评估药物治疗响应以及疾病进展情况。
在化疗效果研究方面, Park等[71]通过scRNA-Seq研究三种结直肠癌系的10 000多个细胞对于5-氟尿嘧啶(5FU) 诱导DNA损伤的转录组反应, 反应类型包括细胞凋亡、细胞分裂以及应激反应, 其中不同反应类型细胞具有特定的DNA损伤反应相关基因表达模式, 伴随其相应细胞命运的走向。部分研究结果也通过流式细胞术在蛋白水平进行验证, 并在其他DNA损伤药物喜树碱和依托泊苷处理的细胞观察到类似现象。上述研究为通过单细胞测序技术评估化疗药物的有效性, 以及后续研究不同细胞亚群的分子特征提供新颖见解。
在免疫疗法相关研究方面, 免疫检查点抑制剂例如抗CTLA-4和抗PD-1/PD-L1已经极大地变革多种肿瘤的治疗方式, 然而目前临床上只对部分特定肿瘤类型的患者提供有效、持久的肿瘤抑制反应, 研究人员对于影响肿瘤对免疫疗法响应率的影响因素和作用机制尚不清楚。为此, Goswami等[72]对5名接受和7名未接受PD-1抑制剂治疗的胶质母细胞瘤(glioblastoma, GBM) 患者进行scRNA-Seq, 研究发现神经胶质瘤中CD73 (hi) 巨噬细胞亚群在抗PD-1治疗后依然存在。体外实验表明CD73的缺失可以提高GBM小鼠模型对于抗PD-1和抗CTLA-4联合治疗的响应, 表明CD73是一种特异性免疫治疗靶点, 能够改善胶质母细胞瘤对于免疫检查点抑制剂的响应程度。上述研究表明scRNA-Seq正逐渐成为评估免疫检查点抑制剂对肿瘤细胞和TME影响以及鉴定新的潜在药物靶标分子的重要手段, 为后续其他类型疗法的响应程度评估提供思路借鉴。
2.4 揭示耐药分子机制在以抗癌药物为代表的耐药机制研究方面, 单细胞组学技术作为剖析肿瘤异质性的有力工具, 能够鉴定导致肿瘤细胞耐药的肿瘤亚克隆群, 检测出与耐药表型相关的基因变异与基因表达模式并作为候选的靶标分子或通路, 从而深入理解肿瘤细胞从对药物敏感到耐受两种状态间转变的分子机制[73, 74]。
单细胞转录组研究方面, Maynard等[75]使用Smart-Seq2技术, 对30名非小细胞肺癌患者分别处于靶向治疗前期、治疗间期以及疾病进展期的49个肿瘤样本进行单细胞转录组测序, 共获得23 261个单细胞的基因表达谱。其中靶向治疗后残留(residual disease, RD) 的肿瘤细胞上调表达肺泡修复与再生信号如WNT/β-catenin信号通路, 而治疗中进展(progressive disease, PD) 的肿瘤细胞则上调犬尿氨酸、血纤维蛋白溶酶原和细胞缝隙连接相关通路。研究结果表明通过早期联合用药同时抑制WNT/β-catenin信号通路以及EGFR或ALK则能提高肿瘤细胞对药物响应的程度。该研究揭示了非小细胞肺癌在不同治疗阶段的肿瘤细胞的可塑性特征, 并且为后续临床联合用药的方案制定提供了新颖见解。单细胞蛋白质组研究方面, Su等[76]采用微流控的单细胞条形码芯片(single cell barcode chip, SCBC) 技术对BRAF V600E突变型黑色素瘤细胞的单细胞蛋白质组与代谢组进行检测, 基于FLOW-MAP算法对数据集的降维结果表明, 肿瘤细胞可能通过两种相互独立的途径由药物反应状态转变为药物耐受状态, 模块分析与实验验证表明特定肿瘤细胞所采取的轨迹由药物治疗之前的转录因子MITF水平决定, 同时还发现两条耐药路径与不同的信号转导和细胞代谢密切相关。通过小分子药物抑制糖酵解丙酮酸激酶PKM2或转录因子NFκB可以分别抑制两条耐药途径, 并且三药组合疗效优于双药组合, 后者优于单药疗法。该研究结果将有助于增进对黑色素瘤耐药性转化的理解, 为后续开发更有效的肿瘤联合疗法提供了新的视角。
应用单细胞组学技术对耐药机制进行研究, 有助于采取可行的治疗策略来抑制或逆转耐药状态的转变, 并为筛选联合用药方案的候选药物分子提供依据, 从而为后续临床针对耐药患者治疗方案的制定与优化奠定基础。
3 单细胞组学技术在中药现代化研究中的应用以及思路探究基于scRNA-Seq等单细胞组学技术在现代药物研究的应用基础与项目经验, 将为单细胞组学技术在中药药理的研究应用提供思路借鉴与启发, 有助于揭示中药的药效物质基础及其作用机制, 并由此促进单细胞药理学这一新兴领域的产生。如图 3所示, 单细胞药理学的研究内容主要包括中药活性成分筛选、作用靶点研究、作用机制阐明以及药代动力学分析等。
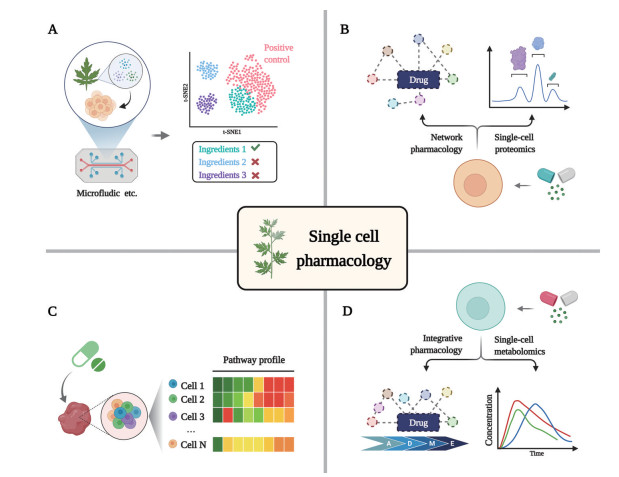
|
Figure 3 The application of single cell pharmacology in traditional Chinese medicine research. (A) Screening of active ingredients. (B) Identification of drug targets. (C) Elucidating mechanism of action. (D) Revealing pharmacokinetics |
相对于化学成分单一的化学药物, 中药的单味药物或复方方剂中具有组成复杂、数量众多的化学成分, 并且各种药物成分中组合与作用的形式在不同病症中具有差异, 采用现代科学的分析技术对中药中的活性成分进行筛选是中药现代化的重要手段与研究策略[77, 78]。前期研究采用基于活性成分分离鉴定、代谢组学和分子识别等技术对青蒿素[79]、川芎嗪[80]、淫羊藿苷[81]等中药活性药效基础进行大量探索, 然而存在筛选效率较低、活性评价指标单一, 难以体现中药系统性与整体性等局限。在单细胞药理学研究中, 通过单细胞组学技术高通量评估细胞对药物作用的响应程度, 同时有助于阐明药物作用的物质基础和效应机制, 在对中药活性成分筛选方面具有广阔的应用前景。
瞬时受体电位(transient receptor potential, TRP) 离子通道是细胞膜上重要的阳离子通道蛋白超家族, 而寻找新型的TRP通道调节剂成为药物筛选领域的热点之一。Ai等[82]开发出针对TRP通道调节剂筛选的单细胞微流控装置, 该装置通过优化微孔尺寸捕获单个细胞, 能够对近200个中药成分对5种TRP通道亚型的调控活性进行高通量筛选, 相比传统的钙荧光FlexStation 3筛选方法能将筛选结果的假阳性/假阴性率从76.2%降低到4.8%。该研究从中药九里香中发现4个香豆素衍生物活性成分, 进一步通过体内模型证实香豆素衍生物活性成分B304能够逆转TRPA1介导的炎症性疼痛。尽管并未对药物处理后的单细胞开展转录组等组学分析, 该研究仍表明了单细胞微流控装置在筛选中药、天然药物的化合物方面具有的卓越性能。后续研究中能够基于类似的微流控系统平台, 将中药药物成分筛选步骤与单细胞组学检测技术如DRUG-seq、sci-Plex等进行整合, 根据细胞在药物成分及其组合作用下的基因表达与通路变化模式的异同, 在单次研究中对具体发挥作用的药物成分及其组合进行高通量筛选。此外, 通过在该系统中拓展药物浓度加样单元, 则能够在此基础上进一步研究各种药物成分的量效关系, 从而提升整个筛选流程的集成化、自动化与标准化。
3.2 中药作用靶点鉴定与中药的药物成分组成复杂、组方配伍形式多样的特点相对应, 中药的多种活性成分通过协同作用于多种靶标蛋白, 引起下游多条通路途径的激活或抑制从而发挥治疗作用, 明确药物作用靶点将进一步促进中药药理机制的阐明。针对具有多成分、多靶点和多途径特点的中药药物研究, 目前研究人员主要通过组织蛋白质组学以及网络药理学等策略进行研究。
对于蛋白质组学, 在前期研究中研究人员已经对中药单体成分如青蒿素[83]、藤黄酸[84]等, 单味中药如黄芩[85]、三七[86]等, 以及中药复方如黄黛片[87]、茵陈五苓散[88]等, 采用活性蛋白质组(activity-based protein profiling, ABPP) 等技术对给药处理前后的细胞系或动物模型的药物作用靶点进行鉴定。而在单细胞蛋白质组学技术方面, 尽管由于其技术发展仍处于早期阶段, 目前在中药领域乃至现代药物研究都鲜有相关研究报道。然而鉴于两者在检测原理与分析策略上的相似性, 后续研究将通过应用单细胞蛋白质组学技术, 在药物干预的背景下进行单细胞蛋白质表达谱分析, 全面、准确地反映药物在不同类型细胞的作用靶点。
网络药理学的研究策略是基于数据库数据信息, 构建成分-靶点-疾病等互作网络, 从而预测药物的药效物质、作用靶点以及效应通路, 目前已广泛应用于单味中药和复方方剂的作用靶点研究[89-91]。如高凤凤等[92]利用网络药理学和分子对接技术从整体层面阐释黄精治疗动脉粥样硬化的作用机制, 拓扑分析结果鉴定到血清白蛋白、丝裂原活化蛋白激酶3等5个关键靶点, 并且分子对接结果表明黄精成分与潜在关键靶点具有较好的结合活性。而黄友等[93]采用类似的研究策略, 筛选到82个附子理中丸治疗溃疡性结肠炎的潜在靶点, 主要涉及有机物反应、细胞凋亡的调节、细胞程序性死亡的调控等生物过程。网络药理学分析结果的准确程度高度依赖于中药成分与靶点网络数据库的完整性以及网络计算模型与指标设定的合理性, 同样也离不开组学技术的支持与验证[94]。随着单细胞组学数据的引入, 网络药理学在后续应用中将整合“细胞”维度的信息, 完善“成分-细胞-组学-靶点-疾病”互作网络的构建, 为今后中药单药和方剂的作用靶点研究提供新颖的线索。
3.3 中药作用机制阐明由于随着二代测序与生物信息学的发展与应用, 目前已有不少研究采用Bulk RNA-Seq用于中药成分或复方的药理机制研究之中, 通过检测药物作用于细胞或动物模型后而引起的总体水平的基因表达谱变化, 从而对药物作用的潜在靶点与功能通路进行揭示[95]。以往的转录组学研究对象主要聚焦于组织水平, 不同类型细胞对于药物作用的响应异质性在此过程被掩盖。而scRNA-Seq技术的应用, 则能够充分反映药物作用于不同类型细胞而引起的转录差异, 从而深入揭示药物调控的细胞类型及其作用的分子机制。
糖耐量受损(impaired glucose tolerance, IGT) 表现为由正常血糖向糖尿病过渡的异常代谢状态, 先前研究表明中药经典方剂左归丸对于IGT具有疗效, 为了进一步研究左归丸治疗IGT的作用分子机制, Liang等[96]采用显微操作技术分离2细胞期小鼠胚胎的单个受精卵, 分为对照组(细胞培养基)、模型组(高糖培养基)以及加药组(在高糖培养基中添加含0.01% v/v左归丸的大鼠血清的培养基) 进行体外培养以及scRNA-Seq。该研究揭示了高糖对于胚胎细胞中糖代谢相关的基因调控变化, 其中左归丸能够通过上调氧化呼吸链中的基因和氧化磷酸化从而促进糖代谢, 对由葡萄糖高负荷引起的胚胎细胞死亡具有保护作用, 为后续研究中药方剂在疾病模型上的潜在分子机制提供了启示。
通过将scRNA-Seq等单细胞组学技术应用于单细胞药理相关研究之中, 获取中药药物作用于不同类型细胞所引起的基因表达或通路变化信息, 将有助于揭示药物的直接作用对象——细胞在“药物-细胞-靶点-途径-表型”中的枢纽作用, 从而全面阐释中药发挥效用的分子机制。
3.4 中药药代动力学研究中药方剂多以口服制剂形式应用, 各种药物成分历经体内一系列吸收、分布、代谢与排泄(ADME) 过程后抵达靶标器官或组织处发挥作用, 其中各种成分在不同代谢过程的效应形式、生理活性、生物利用度等尚待明确。因此, 中药成分的药理机制研究需要密切结合其代谢动力过程, 才能更加全面地揭示中药药物的效应机制以及切合中药的实际应用情况[97-99]。然而, 传统的药代动力学研究往往存在如下问题: ①以偏概全, 通过一种或数种成分来代表复杂多样的复方系统; ②孤立静止, 关注药物原型成分而忽略药物成分之间的相互作用以及在ADME过程中的动态变化; ③片面单一, 缺乏生物机体对于药物处理的代谢反应等信息, 因而难以系统准确地表征中药整体性和动态性的药代动力学过程及其与药理机制的内在联系。
针对上述局限, 研究人员分别提出了基于组织代谢组学以及整合ADME模型的网络药理学等策略进行中药药代动力学研究。在组织代谢组学方面, Lan等[100]首次提出多药药物动力学Poly-PK (poly-pharmacokinetics) 研究策略, 基于代谢组学技术, 对药物自身化学成分、生物体的内源性代谢物以及药物的次级代谢成分进行定性和定量检测, 并进行差异性和关联性分析。Xie等[101]对服用黄芪汤后人体内的代谢反应谱进行表征, 在人血浆中鉴定了56种黄芪汤化合物成分以及292代谢衍生物并构建其浓度-时间曲线。而Liu等[102]则对服用四神丸的正常和结肠炎大鼠的血浆中的26种活性成分及其生物利用度进行研究, 上述研究表明Poly-PK研究思路的有效性和可行性。在此研究策略的基础上, 单细胞药理学能够通过单细胞代谢组学技术, 在药物处理的背景下进行不同作用部位或过程的单细胞药物代谢图谱进行分析, 从而全面动态地反映药物的活性成分和生物利用度, 系统揭示不同细胞中药物代谢变化以及动力学过程的异质性。
在整合ADME模型的网络药理学方面, 许海玉等[103, 104]首次提出“整合药理学”的概念, 通过将中药方剂成分的体外ADME模块与网络药理学进行整合分析, 从而构建“化学指纹-代谢指纹-分子生物网络-药理效应”多维研究模式, 目前已有对青黛片[105]、清火柔肝方[106]等进行整合分析的相关报道。鉴于整合药理学具有交叉融合、兼收并蓄的特点, 其与单细胞药理学也具有较好的学科结合相性。通过在此分析框架上整合单细胞的转录组、蛋白质组等组学信息, 则能建立单细胞药效学—单细胞药动学的研究模型, 进一步揭示药物成分的药代动力学过程与细胞药理效应之间的内在关联, 从而构建完整的中药—细胞—疾病的分子生物网络。
4 讨论与展望在过去10年中, 单细胞组学技术能够以高通量、无偏倚的方式精确表征单个细胞中基因组、转录组、蛋白质组以及代谢组等成分信息及其动态关系, 已经广泛应用于生物学、医学等研究领域并取得重大进展, 该技术在中药药理研究领域也具备巨大的应用潜力。因此, 本文介绍了以scRNA-Seq为代表的单细胞组学技术体系的发展现状以及技术流程, 并综述了该技术在药理机制、药物筛选以及药效评估等方面的研究进展。通过借鉴其研究策略与思路, 从而探究其在中药药理的有效成分筛选、作用靶点鉴定以及药物机制探索等研究模式, 系统阐述了单细胞药理学在中药领域的研究价值与应用前景。
研究领域方面, 中药相对于现代药物具有以下3种特点: 一是在药物来源上, 中药来源以本草植物为主, 以植物总体或取其根、茎、叶等药用部位等经采收、炮制和存储过程后作为药材, 不同处理条件会导致其所含成分及含量具有较大差异[107]。二是在作用成分上, 中药蕴含成千上万种化学成分, 历经体内ADME过程后抵达靶标器官或组织处发挥作用, 并且历经宿主和肠道微生物的生物转换而形成活性形式, 其活性成分的代谢过程及其相互作用机制尚待明确[108]。三是在应用方式上, 中药强调以“君臣佐使, 七情和合”的复方方剂进行治疗, 具有“药有个性之特长, 方有合群之妙用”的应用特点, 其各种药物成分之间的协同作用机制复杂、涉及的药物靶点与通路众多[109, 110]。因此, 笔者认为, 在通过单细胞组学技术开展单细胞组药理学研究的同时, 也需要高度结合中药自身的研究背景与学科特点, 针对中药研究领域的痛点、难点提出解决方案, 才能切实提高中药的研究水平, 进而促进中药的现代化与国际化。
技术体系方面, 单细胞组学技术目前仍然存在较大的局限与挑战。在单细胞转录组测序方面, 尽管在单细胞组学技术中相对成熟, 其技术流程较为复杂、检测成本相对较高, 并且在细胞检测通量和检测基因表达的完整程度方面仍有局限, 对前期样品处理以及后期数据质控提出较高要求[111, 112]。单细胞蛋白质组和单细胞代谢组学发展与应用相对滞后, 目前仍然没有成熟的商业化解决方案, 其技术瓶颈一方面是需要高效、稳定获取单细胞样品, 在样品制备过程尽可能减少样品损失、缩短样品制备时间以及降低外界因素而引入的误差。随着微流控芯片以及纳米材料等装置的引入, 后续细胞样品处理过程将能进一步提升高细胞检测通量、稳健性以及自动化程度[113, 114]。而另一方面在于对蛋白组和代谢组学数据进行准确检测和分析, 需要进一步扩大对蛋白质和代谢产物的检测范围、提升检测信号的灵敏度、以及不同检测流程之间的兼容和拓展性。而随着上述局限的克服, 单细胞蛋白质组学和单细胞代谢组学等技术将为表征细胞状态与功能, 药物靶点鉴定以及活性成分筛选等发挥重要作用, 在单细胞药理学的研究之中具有巨大的应用潜力。
发展方向方面, 目前单细胞组学技术的研究与应用仍然处于起步阶段, 特别在于中药研究领域仍然是相对前沿与新颖的技术。为了更好地将单细胞组学技术应用于中药药理研究, 仍然需要研究人员在纵向和横向两个维度进行探索与攻坚。纵向方面, 需要促进单细胞组学技术在单细胞药理学的应用深度。对其多组分、多靶点、多途径的特点逐一剖析, 并在此过程借助生物信息学和网络药理学等多种方法、引入单细胞多组学的研究策略、整合各种新兴技术与平台等, 从而深入揭示药用成分对于各类细胞相应作用靶点的作用机制。而横向方面, 需要拓宽单细胞药理学与各种疾病如肿瘤、微生物感染等交叉融合的应用广度。研究药物治疗疾病的作用机制, 并鉴定相关分子标志物, 用于预测患者的治疗反应、预后状态等, 从而助力具有中国特色的精准医学发展。
单细胞组学技术的迅速发展逐渐成为驱动现代药物研发流程革新的强大助力, 为高效筛选中药活性成分、鉴定作用靶点、阐明药物作用机制提供更加全面、精准的研究策略和工具。随着单细胞组学技术的应用与普及, 其技术门槛与成本限制也随之降低, 为广大科研工作者探索中药研究领域给予有力的支持。而在应用单细胞组学技术挖掘中药资源宝库的过程之中, 产生的技术需求也为推动单细胞组学的进一步发展提供强大的动力。综上, 中药药理研究与单细胞组学技术的有机结合促进了单细胞药理学这一学科的兴起, 终将实现两者的共同发展与飞跃, 在促进中药现代化与国际化进程中具有广阔的应用前景。
作者贡献: 陈嘉鋆负责本论文主要内容的撰写与可视化; 郭秋岩负责本论文部分内容的撰写与修改, 王继刚研究员和徐承超研究员负责本论文的框架构思、项目管理和文字审阅。
利益冲突: 文章内容不涉及相关利益冲突, 该研究未涉及任何厂家及相关雇主或其他经济组织直接或间接的经济或利益的赞助。本文为原创作品, 内容真实, 内容不涉及泄密, 无一稿两投, 无内容抄袭与剽窃, 作者无署名争议, 全体作者都同意最终的稿件文本。
| [1] |
Eberwine J, Sul JY, Bartfai T, et al. The promise of single-cell sequencing[J]. Nat Methods, 2014, 11: 25-27. DOI:10.1038/nmeth.2769 |
| [2] |
Schier AF. Single-cell biology: beyond the sum of its parts[J]. Nat Methods, 2020, 17: 17-20. DOI:10.1038/s41592-019-0693-3 |
| [3] |
Baslan T, Hicks J. Unravelling biology and shifting paradigms in cancer with single-cell sequencing[J]. Nat Rev Cancer, 2017, 17: 557-569. DOI:10.1038/nrc.2017.58 |
| [4] |
Nam AS, Chaligne R, Landau DA. Integrating genetic and non-genetic determinants of cancer evolution by single-cell multi-omics[J]. Nat Rev Genet, 2021, 22: 3-18. |
| [5] |
Papalexi E, Satija R. Single-cell RNA sequencing to explore immune cell heterogeneity[J]. Nat Rev Immunol, 2018, 18: 35-45. DOI:10.1038/nri.2017.76 |
| [6] |
Neu KE, Tang Q, Wilson PC, et al. Single-cell genomics: approaches and utility in immunology[J]. Trends Immunol, 2017, 38: 140-149. DOI:10.1016/j.it.2016.12.001 |
| [7] |
Ofengeim D, Giagtzoglou N, Huh D, et al. Single-cell RNA sequencing: unraveling the brain one cell at a time[J]. Trends Mol Med, 2017, 23: 563-576. DOI:10.1016/j.molmed.2017.04.006 |
| [8] |
Potter SS. Single-cell RNA sequencing for the study of development, physiology and disease[J]. Nat Rev Nephrol, 2018, 14: 479-492. DOI:10.1038/s41581-018-0021-7 |
| [9] |
Yofe I, Dahan R, Amit I. Single-cell genomic approaches for developing the next generation of immunotherapies[J]. Nat Med, 2020, 26: 171-177. DOI:10.1038/s41591-019-0736-4 |
| [10] |
Valdes-Mora F, Handler K, Law A, et al. Single-cell transcriptomics in cancer immunobiology: the future of precision oncology[J]. Front Immunol, 2018, 9: 2582. DOI:10.3389/fimmu.2018.02582 |
| [11] |
Gohil S, Iorgulescu J, Braun D, et al. Applying high-dimensional single-cell technologies to the analysis of cancer immunotherapy[J]. Nat Rev Clin Oncol, 2021, 18: 244-256. DOI:10.1038/s41571-020-00449-x |
| [12] |
Yang X, Kui L, Tang M, et al. High-throughput transcriptome profiling in drug and biomarker discovery[J]. Front Genet, 2020, 11: 19. DOI:10.3389/fgene.2020.00019 |
| [13] |
de Souza N. Single-cell genetics[J]. Nat Methods, 2013, 10: 820. DOI:10.1038/nmeth.2626 |
| [14] |
Method of the Year 2012[J]. Nat Methods, 2013, 10: 1.
|
| [15] |
Gawad C, Koh W, Quake S. Single-cell genome sequencing: current state of the science[J]. Nat Rev Genet, 2016, 17: 175-188. DOI:10.1038/nrg.2015.16 |
| [16] |
Huang L, Ma F, Chapman A, et al. Single-cell whole-genome amplification and sequencing: methodology and applications[J]. Annu Rev Genomics Hum Genet, 2015, 16: 79-102. DOI:10.1146/annurev-genom-090413-025352 |
| [17] |
Deleye L, De C, Deforce D, et al. Genome-wide copy number alteration detection in preimplantation genetic diagnosis[J]. Methods Mol Biol, 2018, 1712: 27-42. |
| [18] |
Vogelstein B, Papadopoulos N, Velculescu V, et al. Cancer genome landscapes[J]. Science, 2013, 339: 1546-1558. DOI:10.1126/science.1235122 |
| [19] |
Rossi E, Zamarchi R. Single-cell analysis of circulating tumor cells: how far have we come in the -omics era?[J]. Front Genet, 2019, 10: 958. DOI:10.3389/fgene.2019.00958 |
| [20] |
Wang Z, Shen XH, Shi QH. Advances in single-cell whole genome sequencing technology and its application in biomedicine[J]. Hereditas (Beijing) (遗传), 2021, 43: 108-117. |
| [21] |
Labib M, Kelley SO. Single-cell analysis targeting the proteome[J]. Nat Rev Chem, 2020, 4: 143-158. DOI:10.1038/s41570-020-0162-7 |
| [22] |
Marx V. A dream of single-cell proteomics[J]. Nat Methods, 2019, 16: 809-812. DOI:10.1038/s41592-019-0540-6 |
| [23] |
Zenobi R. Single-cell metabolomics: analytical and biological perspectives[J]. Science, 2013, 342: 1243259. DOI:10.1126/science.1243259 |
| [24] |
Kelly R. Single-cell proteomics: progress and prospects[J]. Mol Cell Proteomics, 2020, 19: 1739-1748. DOI:10.1074/mcp.R120.002234 |
| [25] |
Budnik B, Levy E, Harmange G, et al. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation[J]. Genome Biol, 2018, 19: 161. DOI:10.1186/s13059-018-1547-5 |
| [26] |
Specht H, Emmott E, Petelski A, et al. Single-cell proteomic and transcriptomic analysis of macrophage heterogeneity using SCoPE2[J]. Genome Biol, 2021, 22: 50. DOI:10.1186/s13059-021-02267-5 |
| [27] |
Rappez L, Stadler M, Triana S, et al. SpaceM reveals metabolic states of single cells[J]. Nat Methods, 2021, 18: 799-805. DOI:10.1038/s41592-021-01198-0 |
| [28] |
Tang F, Barbacioru C, Wang Y, et al. mRNA-Seq whole-trans-criptome analysis of a single cell[J]. Nat Methods, 2009, 6: 377-382. DOI:10.1038/nmeth.1315 |
| [29] |
Slovin S, Carissimo A, Panariello F, et al. Single-cell RNA sequencing analysis: a step-by-step overview[J]. Methods Mol Biol, 2021, 2284: 343-365. |
| [30] |
Hwang B, Lee J, Bang D. Single-cell RNA sequencing technologies and bioinformatics pipelines[J]. Exp Mol Med, 2018, 50: 1-14. |
| [31] |
Luecken M, Theis F. Current best practices in single-cell RNA-seq analysis: a tutorial[J]. Mol Syst Biol, 2019, 15: e8746. |
| [32] |
Bull N, Bartlett P. The adult mouse hippocampal progenitor is neurogenic but not a stem cell[J]. J Neurosci, 2005, 25: 10815-10821. DOI:10.1523/JNEUROSCI.3249-05.2005 |
| [33] |
Stepanauskas R, Sieracki M. Matching phylogeny and metabolism in the uncultured marine bacteria, one cell at a time[J]. Proc Natl Acad Sci U S A, 2007, 104: 9052-9057. DOI:10.1073/pnas.0700496104 |
| [34] |
Shields CW, Reyes CD, López GP. Microfluidic cell sorting: a review of the advances in the separation of cells from debulking to rare cell isolation[J]. Lab Chip, 2015, 15: 1230-1249. DOI:10.1039/C4LC01246A |
| [35] |
Liu Y, Chen X, Zhang Y, et al. Advancing single-cell proteomics and metabolomics with microfluidic technologies[J]. Analyst, 2019, 144: 846-858. DOI:10.1039/C8AN01503A |
| [36] |
Han X, Wang R, Zhou Y, et al. Mapping the mouse cell atlas by microwell-Seq[J]. Cell, 2018, 173: 1307. DOI:10.1016/j.cell.2018.05.012 |
| [37] |
Yanai I, Hashimshony T. CEL-Seq2-single-cell RNA sequencing by multiplexed linear amplification[J]. Methods Mol Biol, 2019, 1979: 45-56. |
| [38] |
Hashimshony T, Senderovich N, Avital G, et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq[J]. Genome Biol, 2016, 17: 77. DOI:10.1186/s13059-016-0938-8 |
| [39] |
Picelli S, Bjorklund A, Faridani O, et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells[J]. Nat Methods, 2013, 10: 1096-1098. DOI:10.1038/nmeth.2639 |
| [40] |
Picelli S, Faridani O, Bjorklund A, et al. Full-length RNA-seq from single cells using smart-seq2[J]. Nat Protoc, 2014, 9: 171-181. DOI:10.1038/nprot.2014.006 |
| [41] |
Hagemann-Jensen M, Ziegenhain C, Chen P, et al. Single-cell RNA counting at allele and isoform resolution using Smart-seq3[J]. Nat Biotechnol, 2020, 38: 708-714. DOI:10.1038/s41587-020-0497-0 |
| [42] |
Klein AM, Mazutis L, Akartuna I, et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells[J]. Cell, 2015, 161: 1187-1201. DOI:10.1016/j.cell.2015.04.044 |
| [43] |
Macosko EZ, Basu A, Satija R, et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets[J]. Cell, 2015, 161: 1202-1214. DOI:10.1016/j.cell.2015.05.002 |
| [44] |
Zheng G, Terry J, Belgrader P, et al. Massively parallel digital transcriptional profiling of single cells[J]. Nat Commun, 2017, 8: 14049. DOI:10.1038/ncomms14049 |
| [45] |
Andrews T, Hemberg M. Identifying cell populations with scRNASeq[J]. Mol Aspects Med, 2018, 59: 114-122. DOI:10.1016/j.mam.2017.07.002 |
| [46] |
Chen G, Ning B, Shi T. Single-cell RNA-seq technologies and related computational data analysis[J]. Front Genet, 2019, 10: 317. DOI:10.3389/fgene.2019.00317 |
| [47] |
Ning L, Liu G, Li G, et al. Current challenges in the bioinformatics of single cell genomics[J]. Front Oncol, 2014, 4: 7. |
| [48] |
Lahnemann D, Koster J, Szczurek E, et al. Eleven grand challenges in single-cell data science[J]. Genome Biol, 2020, 21: 31. DOI:10.1186/s13059-020-1926-6 |
| [49] |
Aibar S, Gonzalez-Blas C, Moerman T, et al. SCENIC: single-cell regulatory network inference and clustering[J]. Nat Methods, 2017, 14: 1083-1086. DOI:10.1038/nmeth.4463 |
| [50] |
Saelens W, Cannoodt R, Todorov H, et al. A comparison of single-cell trajectory inference methods[J]. Nat Biotechnol, 2019, 37: 547-554. DOI:10.1038/s41587-019-0071-9 |
| [51] |
Armingol E, Officer A, Harismendy O, et al. Deciphering cell-cell interactions and communication from gene expression[J]. Nat Rev Genet, 2021, 22: 71-88. |
| [52] |
Liu F, Zhang Y, Zhang L, et al. Systematic comparative analysis of single-nucleotide variant detection methods from single-cell RNA sequencing data[J]. Genome Biol, 2019, 20: 242. DOI:10.1186/s13059-019-1863-4 |
| [53] |
Ziegenhain C, Vieth B, Parekh S, et al. Comparative analysis of single-cell RNA sequencing methods[J]. Mol Cell, 2017, 65: 631-643.e4. DOI:10.1016/j.molcel.2017.01.023 |
| [54] |
Method of the Year 2019: single-cell multimodal omics[J]. Nat Methods, 2020, 17: 1
|
| [55] |
Dey S, Kester L, Spanjaard B, et al. Integrated genome and transcriptome sequencing of the same cell[J]. Nat Biotechnol, 2015, 33: 285-289. DOI:10.1038/nbt.3129 |
| [56] |
Macaulay I, Haerty W, Kumar P, et al. G & T-seq: parallel sequencing of single-cell genomes and transcriptomes[J]. Nat Methods, 2015, 12: 519-522. DOI:10.1038/nmeth.3370 |
| [57] |
Frei A, Bava F, Zunder E, et al. Highly multiplexed simultaneous detection of RNAs and proteins in single cells[J]. Nat Methods, 2016, 13: 269-275. DOI:10.1038/nmeth.3742 |
| [58] |
Stoeckius M, Hafemeister C, Stephenson W, et al. Simultaneous epitope and transcriptome measurement in single cells[J]. Nat Methods, 2017, 14: 865-868. DOI:10.1038/nmeth.4380 |
| [59] |
Peterson V, Zhang K, Kumar N, et al. Multiplexed quantification of proteins and transcripts in single cells[J]. Nat Biotechnol, 2017, 35: 936-939. DOI:10.1038/nbt.3973 |
| [60] |
Stein-O'Brien G, Ainslie MC, Fertig EJ. Forecasting cellular states: from descriptive to predictive biology via single-cell multiomics[J]. Curr Opin Syst Biol, 2021, 26: 24-32. DOI:10.1016/j.coisb.2021.03.008 |
| [61] |
Giladi A, Amit I. Single-cell genomics: a stepping stone for future immunology discoveries[J]. Cell, 2018, 172: 14-21. DOI:10.1016/j.cell.2017.11.011 |
| [62] |
Ren X, Zhang L, Zhang Y, et al. Insights gained from single-cell analysis of immune cells in the tumor microenvironment[J]. Annu Rev Immunol, 2021, 39: 583-609. DOI:10.1146/annurev-immunol-110519-071134 |
| [63] |
Guo X, Zhang Y, Zheng L, et al. Global characterization of T cells in non-small-cell lung cancer by single-cell sequencing[J]. Nat Med, 2018, 24: 978-985. DOI:10.1038/s41591-018-0045-3 |
| [64] |
Zheng C, Zheng L, Yoo J, et al. Landscape of infiltrating T cells in liver cancer revealed by single-cell sequencing[J]. Cell, 2017, 169: 1342-1356.e16. DOI:10.1016/j.cell.2017.05.035 |
| [65] |
Kang S, Kim J, Kim M, et al. Drug-induced hypersensitivity syndrome/drug reaction with eosinophilia and systemic symptoms syndrome induced by cilostazol and carbamazepine[J]. J Dermatol, 2012, 39: 723-724. DOI:10.1111/j.1346-8138.2011.01500.x |
| [66] |
Kim D, Kobayashi T, Voisin B, et al. Targeted therapy guided by single-cell transcriptomic analysis in drug-induced hypersensitivity syndrome: a case report[J]. Nat Med, 2020, 26: 236-243. DOI:10.1038/s41591-019-0733-7 |
| [67] |
Menard T, Barros A, Ganter C. Clinical quality considerations when using next-generation sequencing (NGS) in clinical drug development[J]. Ther Innov Regul Sci, 2021, 55: 1066-1074. DOI:10.1007/s43441-021-00308-6 |
| [68] |
Ye C, Ho D, Neri M, et al. DRUG-seq for miniaturized high-throughput transcriptome profiling in drug discovery[J]. Nat Commun, 2018, 9: 4307. DOI:10.1038/s41467-018-06500-x |
| [69] |
Shin D, Lee W, Lee J, et al. Multiplexed single-cell RNA-seq via transient barcoding for simultaneous expression profiling of various drug perturbations[J]. Sci Adv, 2019, 5: eaav2249. DOI:10.1126/sciadv.aav2249 |
| [70] |
Srivatsan SR, McFaline-Figueroa J, Ramani V, et al. Massively multiplex chemical transcriptomics at single-cell resolution[J]. Science, 2020, 367: 45-51. DOI:10.1126/science.aax6234 |
| [71] |
Park S, Namkoong S, Friesen L, et al. Single-cell transcriptome analysis of colon cancer cell response to 5-fluorouracil-induced DNA damage[J]. Cell Rep, 2020, 32: 108077. DOI:10.1016/j.celrep.2020.108077 |
| [72] |
Goswami S, Walle T, Cornish A, et al. Immune profiling of human tumors identifies CD73 as a combinatorial target in glioblastoma[J]. Nat Med, 2020, 26: 39-46. DOI:10.1038/s41591-019-0694-x |
| [73] |
Schmidt F, Efferth T. Tumor heterogeneity, single-cell sequencing, and drug resistance[J]. Pharmaceuticals (Basel), 2016, 9: 33. DOI:10.3390/ph9020033 |
| [74] |
Sun G, Li Z, Rong D, et al. Single-cell RNA sequencing in cancer: applications, advances, and emerging challenges[J]. Mol Ther Oncolytics, 2021, 21: 183-206. DOI:10.1016/j.omto.2021.04.001 |
| [75] |
Maynard A, McCoach C, Rotow J, et al. Therapy-induced evolution of human lung cancer revealed by single-cell RNA sequencing[J]. Cell, 2020, 182: 1232-1251.e22. DOI:10.1016/j.cell.2020.07.017 |
| [76] |
Su Y, Ko M, Cheng H, et al. Multi-omic single-cell snapshots reveal multiple independent trajectories to drug tolerance in a melanoma cell line[J]. Nat Commun, 2020, 11: 2345. DOI:10.1038/s41467-020-15956-9 |
| [77] |
Lin A, Chan G, Hu Y, et al. Internationalization of traditional Chinese medicine: current international market, internationalization challenges and prospective suggestions[J]. Chin Med, 2018, 13: 9. DOI:10.1186/s13020-018-0167-z |
| [78] |
Yu Y, Yao C, Guo D. Insight into chemical basis of traditional Chinese medicine based on the state-of-the-art techniques of liquid chromatography-mass spectrometry[J]. Acta Pharm Sin B, 2021, 11: 1469-1492. DOI:10.1016/j.apsb.2021.02.017 |
| [79] |
Tu YY, Ni MY, Zhong YR, et al. Studies on the chemical constituents of Artemisia annua LI[J]. Sci Technol Rev (科技导报), 2015, 33: 124-126. |
| [80] |
Kong L, Yu Z, Bao Y, et al. Screening and analysis of an antineoplastic compound in Rhizoma Chuanxiong by means of in vitro metabolism and HPLC-MS[J]. Anal Bioanal Chem, 2006, 386: 264-274. DOI:10.1007/s00216-006-0621-0 |
| [81] |
Li F, Lu X, Liu H, et al. A pharmaco-metabonomic study on the therapeutic basis and metabolic effects of Epimedium brevicornum Maxim. on hydrocortisone-induced rat using UPLC-MS[J]. Biomed Chromatogr, 2007, 21: 397-405. DOI:10.1002/bmc.770 |
| [82] |
Ai X, Wu Y, Lu W, et al. A precise microfluidic assay in single-cell profile for screening of transient receptor potential channel modulators[J]. Adv Sci (Weinh), 2020, 7: 2000111. DOI:10.1002/advs.202000111 |
| [83] |
Wang J, Zhang CJ, Chia W, et al. Haem-activated promiscuous targeting of artemisinin in Plasmodium falciparum[J]. Nat Commun, 2015, 6: 10111. DOI:10.1038/ncomms10111 |
| [84] |
Li D, Song X, Yue Q, et al. Proteomic and bioinformatic analyses of possible target-related proteins of gambogic acid in human breast carcinoma MDA-MB-231 cells[J]. Chin J Nat Med, 2015, 13: 41-51. |
| [85] |
Ong E, Len S, Lee A, et al. Proteomic analysis of mouse liver for the evaluation of effects of Scutellariae Radix by liquid chromatography with tandem mass spectrometry[J]. Rapid Commun Mass Spectrom, 2004, 18: 2522-2530. DOI:10.1002/rcm.1654 |
| [86] |
Yao Y, Wu W, Guan S, et al. Proteomic analysis of differential protein expression in rat platelets treated with notoginsengnosides[J]. Phytomedicine, 2008, 15: 800-807. DOI:10.1016/j.phymed.2008.06.013 |
| [87] |
Wang L, Zhou G, Liu P, et al. Dissection of mechanisms of Chinese medicinal formula Realgar-Indigo naturalis as an effective treatment for promyelocytic leukemia[J]. Proc Natl Acad Sci U S A, 2008, 105: 4826-4831. DOI:10.1073/pnas.0712365105 |
| [88] |
Li R, Zhao L, Wu N, et al. Proteomic analysis allows for identifying targets of Yinchenwuling Powder in hyperlipidemic rats[J]. J Ethnopharmacol, 2016, 185: 60-67. DOI:10.1016/j.jep.2016.03.029 |
| [89] |
Hopkins A. Network pharmacology: the next paradigm in drug discovery[J]. Nat Chem Biol, 2008, 4: 682-690. DOI:10.1038/nchembio.118 |
| [90] |
Hopkins A. Network pharmacology[J]. Nat Biotechnol, 2007, 25: 1110-1111. DOI:10.1038/nbt1007-1110 |
| [91] |
Wang L, Li Z, Shao Q, et al. Dissecting active ingredients of Chinese medicine by content-weighted ingredient-target network[J]. Mol Biosyst, 2014, 10: 1905-1911. DOI:10.1039/C3MB70581A |
| [92] |
Gao FF, Pei YL, Ren Y, et al. Possible mechanisms by which Polygonati rhizoma opposes atherosclerosis based on network pharmacology and molecular docking analyses[J]. Acta Pharm Sin (药学学报), 2020, 55: 2642-2650. |
| [93] |
Huang Y, Yang SS, Lin X, et al. The mechanism of action of Fuzi-Lizhong pill in treatment of ulcerative colitis based on network pharmacology-molecular docking[J]. Acta Pharm Sin (药学学报), 2020, 55: 1812-1822. |
| [94] |
Li XJ, Xu HY. Network pharmacology and traditional Chinese medicine research[J]. Acta Pharm Sin (药学学报), 2018, 53: 1385-1386. |
| [95] |
Liu Y, Liu X, Zhang N, et al. Berberine diminishes cancer cell PD-L1 expression and facilitates antitumor immunity via inhibiting the deubiquitination activity of CSN5[J]. Acta Pharm Sin B, 2020, 10: 2299-2312. DOI:10.1016/j.apsb.2020.06.014 |
| [96] |
Liang Q, Qu Z, Liang Y, et al. Zuo Gui Wan alters expression of energy metabolism genes and prevents cell death in high-glucose loaded mouse embryos[J]. Evid Based Complement Alternat Med, 2018, 2018: 2409471. |
| [97] |
Hao HP, Zheng CN, Wang GJ. Thoughts and experimental exploration on pharmacokinetic study of herbal medicines with multiple-components and targets[J]. Acta Pharm Sin (药学学报), 2009, 44: 270-275. |
| [98] |
Liu CX. Difficulty and hot points on pharmacokinetics studies of traditional Chinese medicine[J]. Acta Pharm Sin (药学学报), 2005, 40: 395-401. |
| [99] |
Lv L, Sun HJ, Han GZ. Research progresses of pharmacokinetics of traditional Chinese medicine[J]. Acta Pharm Sin (药学学报), 2013, 48: 824-833. |
| [100] |
Lan K, Xie G, Jia W. Towards polypharmacokinetics: pharmacokinetics of multicomponent drugs and herbal medicines using a metabolomics approach[J]. Evid Based Complement Alternat Med, 2013, 2013: 819147. |
| [101] |
Xie G, Wang S, Zhang H, et al. Poly-pharmacokinetic study of a multicomponent herbal medicine in healthy Chinese volunteers[J]. Clin Pharmacol Ther, 2018, 103: 692-702. DOI:10.1002/cpt.784 |
| [102] |
Liu L, Wang S, Xu QX, et al. Poly-pharmacokinetic strategy represented the synergy effects of bioactive compounds in a traditional Chinese medicine formula, Si Shen Wan and its separated recipes to normal and colitis rats[J]. J Sep Sci, 2021, 44: 2065-2077. DOI:10.1002/jssc.202001258 |
| [103] |
Xu HY, Yang HJ. Integrative pharmacology: new mode of modern research on traditional Chinese medicine[J]. China J Chin Mater Med (中国中药杂志), 2014, 39: 357-362. |
| [104] |
Xu H, Zhang Y, Wang P, et al. A comprehensive review of integrative pharmacology-based investigation: a paradigm shift in traditional Chinese medicine[J]. Acta Pharm Sin B, 2021, 11: 1379-1399. DOI:10.1016/j.apsb.2021.03.024 |
| [105] |
Zhou C, Liu L, Zhuang J, et al. A systems biology-based approach to uncovering molecular mechanisms underlying effects of traditional Chinese medicine Qingdai in chronic myelogenous leukemia, involving integration of network pharmacology and molecular docking technology[J]. Med Sci Monit, 2018, 24: 4305-4316. DOI:10.12659/MSM.908104 |
| [106] |
Jing C, Sun Z, Xie X, et al. Network pharmacology-based identification of the key mechanism of Qinghuo Rougan Formula acting on uveitis[J]. Biomed Pharmacother, 2019, 120: 109381. DOI:10.1016/j.biopha.2019.109381 |
| [107] |
Chen SL, Sun YZ, Xu J, et al. Strategies of the study on herb genome program[J]. Acta Pharm Sin (药学学报), 2010, 45: 807-812. |
| [108] |
Buriani A, Garcia-Bermejo M, Bosisio E, et al. Omic techniques in systems biology approaches to traditional Chinese medicine research: present and future[J]. J Ethnopharmacol, 2012, 140: 535-544. DOI:10.1016/j.jep.2012.01.055 |
| [109] |
Saqi M, Pellet J, Roznovat I, et al. Systems medicine: the future of medical genomics, healthcare, and wellness[J]. Methods Mol Biol, 2016, 1386: 43-60. |
| [110] |
Ma T, Tan C, Zhang H, et al. Bridging the gap between traditional Chinese medicine and systems biology: the connection of Cold Syndrome and NEI network[J]. Mol Biosyst, 2010, 6: 613-619. DOI:10.1039/b914024g |
| [111] |
Chen W, Zhao Y, Chen X, et al. A multicenter study benchmarking single-cell RNA sequencing technologies using reference samples[J]. Nat Biotechnol, 2020, 39: 1103-1114. |
| [112] |
Butler A, Hoffman P, Smibert P, et al. Integrating single-cell transcriptomic data across different conditions, technologies, and species[J]. Nat Biotechnol, 2018, 36: 411-420. DOI:10.1038/nbt.4096 |
| [113] |
Lin C, Lee D, Chang H, et al. Single-cell enzyme-free dissociation of neurospheres using a microfluidic chip[J]. Anal Chem, 2013, 85: 11920-11928. DOI:10.1021/ac402724b |
| [114] |
Verboket P, Borovinskaya O, Meyer N, et al. A new microfluidics-based droplet dispenser for ICPMS[J]. Anal Chem, 2014, 86: 6012-6018. DOI:10.1021/ac501149a |
 2021, Vol. 56
2021, Vol. 56


