2. 汉中职业技术学院药学院, 陕西 汉中 723000;
3. 北京大学深圳医院, 深圳北京大学-香港科技大学医学中心, 广东 深圳 518036
2. Department of Pharmacy, Hanzhong Vocation and Technology College, Hanzhong 723000, China;
3. Peking University Shenzhen Hospital, Shenzhen Peking University-The Hong Kong University of Science and Technology Medical Center, Shenzhen 518036, China
胃癌是全球死亡率最高的恶性肿瘤之一, 多数患者确诊时已发展为进展期或晚期。靶向治疗、免疫治疗及多药联合化疗成为胃癌治疗的主要手段。人表皮生长因子受体2 (human epidermal growth factor receptor 2, HER-2) 是由erbB2基因编码的致癌基因, 其过表达与胃癌的发生及发展相关, 且HER-2过表达与肿瘤耐药及不良预后关系密切[1, 2]。赫赛汀为靶向HER-2的免疫球蛋白G1型单克隆抗体药物, 通过抑制HER-2信号转导及抗体依赖细胞介导的细胞毒作用发挥功能。尽管赫赛汀能延长胃癌进展期及提高患者生存期, 然而肿瘤获得性耐药制约其在临床的有效治疗。因此, 探究胃癌赫赛汀耐药机制, 发现耐药靶标, 对提高化疗有效性具有重要意义。
Runt相关转录因子3 (Runt related transcription factor 3, RUNX3) 属于Runt家族转录因子, 具有保守的Runt-DNA结构域, 在消化道上皮细胞及间叶细胞中广泛表达。研究显示, RUNX3在上皮细胞增殖-凋亡平衡及信号转导过程中发挥重要作用, 其缺失或异常表达与肿瘤发生、发展相关[3-5]。RUNX3具有广泛的生物学功能, 作为抑癌基因, 其功能失活与基因表观修饰改变、蛋白质错误定位有关[5, 6]; 作为致癌基因, RUNX3可激活Myc表达, 引起癌细胞快速增殖并转移[7]。
近年来, RUNX3突变与肿瘤耐药备受关注, 如RUNX3过表达可导致凋亡抑制因子2 (apoptosis inhibitor 2, IAP2) 上调, 诱导卵巢癌细胞对卡铂耐药[8]; RUNX3缺失可诱发多药耐药蛋白(multidrug resistance proteins, MRP) 高表达, 导致肝癌细胞对5-氟尿嘧啶及顺铂耐药[9]以及RUNX3缺失导致胰腺癌细胞对吉西他滨耐药[10]。前期研究发现[11], 对赫赛汀耐药的胃癌细胞NCI N87R中, RUNX3与DNA的结合活性显著增加, 提示RUNX3可能与胃癌赫赛汀耐药有关, 然而相关机制尚未阐明。本实验以NCI N87R为研究对象, 基于CRISPR/Cas9构建RUNX3敲除细胞株(△RUNX3/NCI N87R), 基于非标记定量蛋白质组学研究△RUNX3/NCI N87R细胞蛋白质表达谱的变化, 结合生物信息学分析△RUNX3/NCI N87R细胞信号通路的改变及RUNX3调控胃癌赫赛汀耐药的潜在机制。
材料与方法主要仪器 LTQ Orbitrap Velos Pro轨道阱质谱仪及Easy-nLC 2000 nano液相色谱仪、细胞培养箱(美国Thermo Fisher公司); 2695 HPLC液相色谱系统(美国Waters公司); SP8荧光显微镜(德国Leica公司); 化学发光成像系统(Tanon公司); 电泳仪(美国Bio-Rad公司); 多功能超声仪(美国Sonics公司); 5810R真空浓缩仪(德国Eppendorf公司)。
细胞株及试剂 人源NCI N87细胞由国家蛋白质组研究中心提供; NCI N87R及△RUNX3/NCI N87R细胞由实验室自建。赫赛汀(440 mg, S20060026, 罗氏制药); 胎牛血清(fetal bovine serum, FBS, 美国Gibco公司); DMEM培养基、胰酶(美国Mediatech公司); 1%青霉素-链霉素、抗兔/鼠二抗(碧云天生物科技); 二硫苏糖醇(dithiothreitol, DTT)、吲哚-3-乙酸(indole-3-acetic acid, IAA)、尿素、碳酸氢钠, 甲醇、乙醇、乙腈、甲酸、蛋白酶抑制剂(美国Thermo Fisher公司); Bradford蛋白浓度试剂盒(北京康为生物有限公司); 一抗RUNX3 (9647)、自噬相关蛋白(autophagy-related protein, ATG) 13 (13468)、ATG7 (8558) 及丝氨酸/苏氨酸蛋白激酶Chk2 (serine/threonine-protein kinase Chk2, CHEK2, 6334) (美国Cell Signaling Technology公司); 凋亡调节分子Bcl-2 (apoptosis regulator Bcl-2, BCL2, 7382)、BECN1 (48341)、丝氨酸/苏氨酸蛋白激酶AKT (271149)、磷酸化AKT (p-AKT, 52940)、mitogen-activated protein kinase 3 (ERK1, 271291)、磷酸化ERK1 (p-ERK1, 7383) 及β-actin (376421) (美国Santa Cruz公司); Lipofectamine 2000 (美国Invitrogen公司); BsmBI (美国NEB公司); T4连接酶(日本TAKARA公司); CCK-8 kit (日本同仁化学)。
细胞培养 NCI N87、NCI N87R及△RUNX3/NCI N87R细胞按常规方法培养于DMEM培养液(10% FBS和1%青霉素-链霉素)。NCI N87R在培养过程中按照文献方法[12, 13]加入赫赛汀, 药物终质量浓度为80 μg·mL-1。
向导RNA (sgRNA) 寡核苷酸链合成及载体构建 依据http://www.e-crisp.org/E-CRISP设计sgRNA RUNX3, 选取得分最高的两个核苷酸序列。核苷酸1: 正向5'-CACCGGCGCGCCCTCCGGGCCCCA-3', 反向5'-AAACTGGGGCCCGGAGGGCGCGCC-3'; 核苷酸2: 正向5'-CACCGGCAAGATGGGCGAGAACAG-3', 反向5'-AAACCTGTTCTCGCCCATCTTGCC-3'。核苷酸序列由华大公司合成, 通过T4连接酶将其连入两个BbsI位点的U6-sgRNA-SFFV-spCas9-puro质粒中。
细胞转染及目标细胞株筛选 将2 μg Cas9、1 μg pMD 2.G和1.5 μg psPAX2混合质粒及15 μL Lipofectamine 2000混合, 加入293T细胞, 孵育8 h去除包含慢病毒质粒的培养基, 加入新鲜培养基, 培养48 h, 10 000 ×g离心10 min, 收集包含病毒颗粒的上清液, 备用。将NCI N87R细胞按每毫升1×105个接种于六孔板, 培养至70%~80%更换培养液, 加入慢病毒感染, 48 h后加入0.5 μg·mL-1嘌呤霉素培养细胞72 h, 流式分选单细胞, 96孔板培养, 免疫印迹(Western blot) 验证。
细胞活力检测 对数期生长的NCI N87、NCI N87R和△RUNX3/NCI N87R细胞分别接种于96孔板中, 细胞数约为5 000个/孔, 按文献方法[14-16]检测细胞活力。
免疫荧光实验 对数期生长的NCI N87、NCI N87R和△RUNX3/NCI N87R细胞分别用PBS洗3次, 4%多聚甲醛固定10 min, 0.2% Triton X-100透化10 min; QuickBlockTM封闭2 h。一抗孵育过夜(4 ℃) (稀释倍数1∶300), 二抗孵育1 h (稀释倍数1∶2 000); DAPI染核2~3 min, PBS洗3次, 随机选取视野拍照。
Western blot检测蛋白质的表达 收集对数期生长的NCI N87R和△RUNX3/NCI N87R细胞, 提取蛋白, 依次进行变性、聚丙烯酰胺凝胶电泳、转膜、封闭、一抗孵育过夜(4 ℃)、洗膜、孵育二抗(常温)、洗膜、显影。一抗ATG13、ATG7、CHEK2、BECN1、BCL2、AKT、ERK1、p-AKT、p-ERK1及β-actin分别稀释1 000倍, 二抗稀释3 000倍; 使用Image J进行灰度值计算, GraphPad Prism 8.0进行定量分析。
色谱及质谱色谱条件 Easy-nLC 2000纳升高效液相色谱系统(3 µm×75 µm×15 cm), 搭载反相C18分离柱(2 cm×100 μm×3 μm) 和C18分析柱(15 cm×75 μm×3 μm) 均购自美国Thermo Fisher公司。液相分离条件: 流动相A为水(0.1%甲酸, v/v), 流动相B为乙腈(0.1%甲酸, v/v), 梯度洗脱: 0~5 min, 0~5% B; 6~90 min, 6%~35% B; 91~110 min, 36%~98% B; 111~120 min, 2% B; 流速200 nL·min-1。
质谱及检测参数 LTQ rbitrap Velos Pro型质谱仪, 纳升级电喷雾(nano-ESI) 离子源, 电压为2.5 kV, 离子传输毛细管温度300 ℃, Orbitrap一级扫描质荷比(m/z) 300~2 000, 一级扫描的分辨率为30 000, 选取丰度最高的Top 20离子进行二级(MS/MS) 分析, MS/MS扫描分辨率为7 500, 二级碎裂模式为碰撞诱导解离(CID), 归一化碰撞能(NCE) 为35%, 活化Q值为0.25; 活化时间为30 ms; 排除同位素干扰峰, 动态排除时间为40 s, 重复次数为1, 重复持续时间为30 s, 排除列表为50, 排除离子时间为180 s。
细胞蛋白质提取 收集细胞, 预冷PBS洗2次, 加入8 mol·L-1尿素(1%蛋白酶抑制剂), 充分震荡, 冰上裂解10 min, 超声2 min, 12 000 ×g离心15 min (4 ℃), 取上清, Bradford法测定蛋白浓度。
还原烷基化及FASP酶切 还原烷基化及FASP酶切参照文献[17]。100 μg样品加入20 mmol·L-1 DTT, 56 ℃反应30 min, 加入50 mmol·L-1 IAA, 避光30 min, 加入20 mmol·L-1 DTT, 避光15 min, 12 000 r·min-1离心5 min, 取上清, 加入10 kD超滤管, 12 000 ×g离心40 min, 加入50 mmol·L-1 NH4HCO3 (300 μL), 12 000 ×g离心3次, 每次40 min。加入50 mmol·L-1 NH4HCO3 (50 μL) 和胰蛋白酶混合液(5 μg), 37 ℃孵育4 h, 再加50 mmol·L-1 NH4HCO3和胰蛋白酶5 μg, 酶切至过夜, 离心, 收集肽段。
肽段预分离 肽段分离参照文献[18]。采用Waters 2695 HPLC液相系统分离肽段, 流动相A (水), 流动相B (乙腈), 洗脱梯度为: 0~34 min, 0~98% B; 35~40 min, 98% B, 流速0.5 mL·min-1, 收集洗脱液(每分钟1管), 合并洗脱液: ① 1、6、11、16、21; ② 2、7、12、17、22; ③ 3、8、13、18、23; ④ 4、9、14、19、24; ⑤ 5、10、15、20、25; ⑥ 26~30; ⑦ 31~35; ⑧ 36~40, 将收集的混合组分减压浓缩, 质谱检测。
肽段检测、定量及差异表达蛋白筛选 肽段用0.1%甲酸复溶, 12 000 ×g离心10 min, 取上清, 经Easy-nLC 2000 nano分离进入LTQ Orbitrap Velos Pro质谱仪, 采用Proteome Discover 1.4软件搜库, 数据库为NCBI人源蛋白质序列库。检索参数为: 母离子质量公差20 ppm, 子离子0.5 Da。蛋白质修饰为: 半胱氨酸脲甲基化为固定修饰, 蛋氨酸N-乙酰化及氧化为动态修饰, 肽段水平错误发现率(false discovery rate, FDR) 小于1%。
基于数据依赖分析(data dependence analysis, DDA) 采集数据, 按总面积进行归一化定量, 方法为每个蛋白峰面积除以样本中全部蛋白峰面积之和, 为便于计算, 将所有蛋白的相对峰面积扩大105倍。肽段筛选标准为: 每个蛋白质被检测到的特异性肽段数≥ 1, Mascot ≥ 20。采用差异倍数及t检验进行差异蛋白质筛选, 若某蛋白的定量值在两组样本中的均值之比≥ 2或≤ 0.5, 且P < 0.05, 则认为该蛋白在两样本中的表达具有显著差异。
统计学分析 数据用均数±标准误(mean ± SEM) 表示, n表示样本量。使用SPSS 26.0进行统计学分析, 两组间均数比较用t检验, 以α = 0.05为检验水平, P < 0.05表示差异具有统计学意义, P < 0.01表示差异具有显著统计学意义, P < 0.001表示差异具有极显著统计学意义。定量作图采用GraphPad Prism 8.0软件, 蛋白-蛋白网络图采用Cytoscape (version 3.7.2) 软件, 生物信息学作图采用RStudio (version 3.6.2)。
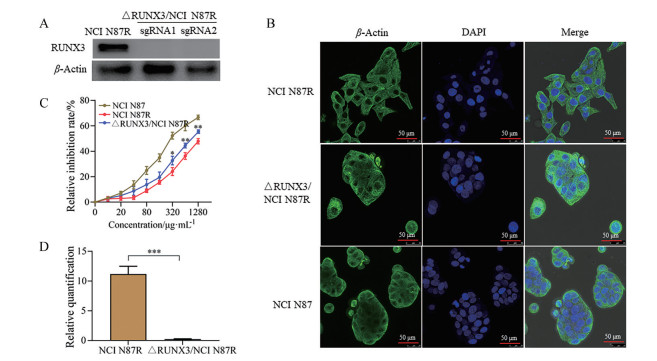
结果 1 △RUNX3/NCI N87R细胞株的构建基于CRISPR/Cas9构建RUNX3敲除细胞株, 为防止基因编辑可能出现的脱靶效应, 设计能够靶向切割RUNX3的两条sgRNA序列。免疫印迹表明, 两条sgRNA均能发挥作用, 引导Cas9切除目标基因, 表明△RUNX3/NCI N87R细胞株构建成功(图 1A)。免疫荧光显示, NCI N87R细胞由“不规则形”变为“卵圆形”, 生长状态由“弥散型”变为“抱团型”; 且△RUNX3/NCI N87R形态更接近于母本细胞(图 1B), 提示敲除RUNX3导致NCI N87R细胞形态重塑。为探究赫赛汀对△RUNX3/NCI N87R细胞的抑制作用, 采用CCK-8分别检测△RUNX3/NCI N87R、NCI N87R及NCI N87细胞在不同浓度赫赛汀作用下的细胞相对抑制率。结果显示, 赫赛汀对3种细胞的增殖抑制具有浓度依赖关系, 且抑制强度分别为: NCI N87 > △RUNX3/NCI N87R > NCI N87R。当药物的质量浓度达到320 μg·mL-1时, △RUNX3/NCI N87R与NCI N87R的相对抑制率差异具有统计学意义(P < 0.05); 当质量浓度大于320 μg·mL-1时, 相对抑制率差异具有显著统计学意义(P < 0.01)。在实验条件下, 赫赛汀对NCI N87、NCI N87R及△RUNX3/NCI N87R的半数抑制浓度(IC50) 分别为134.2、651.8和328.5 μg·mL-1 (图 1C), 提示敲除RUNX3增强了赫赛汀对耐药细胞的敏感性。蛋白质数据显示, RUNX3在耐药细胞中被敲除, 证实△RUNX3/NCI N87R细胞株构建成功(图 1D)。
|
Figure 1 Establishment of △RUNX3/NCI N87R cell line. A: RUNX3 expression detected in NCI N87R and △RUNX3/NCI N87R cell lines by Western blot; B: The morphological comparison among NCI N87, NCI N87R, and △RUNX3/NCI N87R cells after immunofluorescence, respectively; C: Relative inhibition rate of NCI N87, NCI N87R, and △RUNX3/NCI N87R cells was detected by CCK-8 assays, respectively; D: RUNX3 in NCI N87R and △RUNX3/NCI N87R cell lines were quantified by mass spectrometry. n = 3, mean ± SEM. *P < 0.05, **P < 0.01, ***P < 0.001 vs NCI N87R |
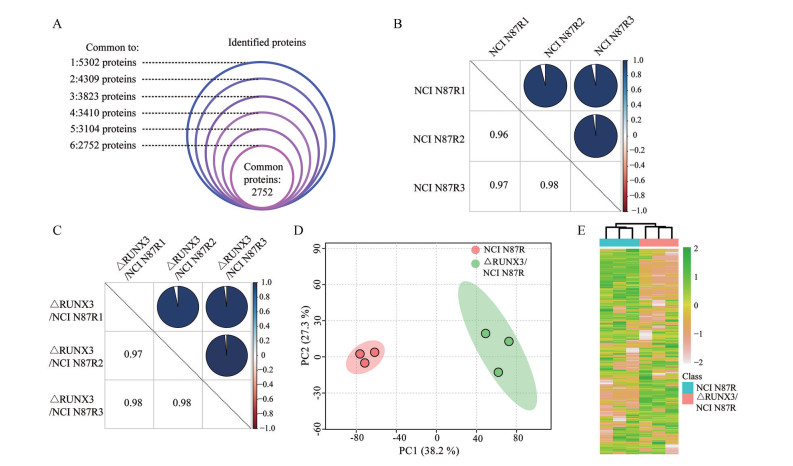
为明确RUNX3敲除对耐药细胞表达谱的影响, 基于定量蛋白质组学分别检测了NCI N87R和△RUNX3/NCI N87R细胞的蛋白质组, 采用生物信息学进行差异蛋白质筛选及通路分析。为保证肽段鉴定的可靠性及蛋白定量的准确性, 将肽段水平的卡值设定为“严格匹配”, 而蛋白水平的卡值设定为FDR < 1%。两组样本中共检测到蛋白5 302个, 且这些蛋白至少在1个样本中被鉴定。以此类推, 4 309个蛋白至少在2个样本中被检测, 3 823个蛋白至少在3个样本被检测, 2 752个蛋白在所有样本中被检测。根据经验, 将3个及以上样本中被检测到的蛋白(3 823个) 纳入生物信息学及统计学分析(图 2A)。基于Spearman方法对各组数据进行相关性分析。结果显示, 样本组内相关系数均大于0.95 (图 2B、C), 表明实验重复性良好。主成分分析(principal composition analysis, PCA) 显示, NCI N87R和△RUNX3/NCI N87R数据分别沿第1和第2主成分(PC1和PC2) 方向分开, PC1与PC2之和能够解释组间65.5%的变异, 且组内样本聚集良好, 而组间分离明显(图 2D)。聚类分析证实, NCI N87R和△RUNX3/NCI N87R细胞的表达谱差异明显(图 2E), 提示敲除RUNX3对NCI N87R细胞的蛋白质谱产生影响。
|
Figure 2 Proteomics profiling of NCI N87R and △RUNX3/NCI N87R cell lines. A: Proteins detected in different samples; B, C: Spearman's correlation coefficients analysis of NCI N87R and △RUNX3/NCI N87R cell samples. The lower-left half shows pairwise Spearman correlation coefficients of three biological repeats, the upper-right half shows pairwise pie plots; D: Principal component scores of NCI N87R and △RUNX3/NCI N87R cells, respectively; E: Clustering analysis of proteome of NCI N87R and △RUNX3/NCI N87R cells |
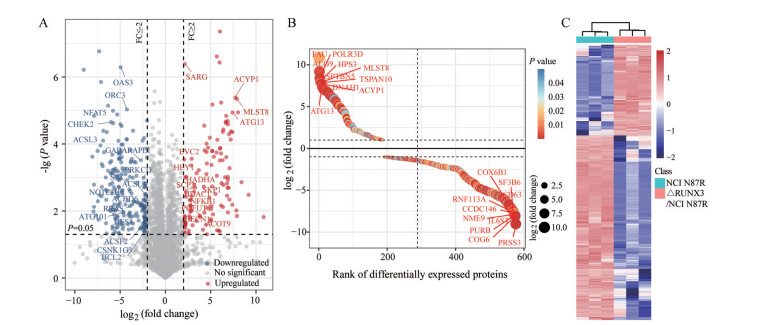
首先, 对样本中所有变量过滤(将缺失值超过50%的蛋白以及角蛋白剔除)。随后, 基于K-Nearest Neighbors (KNN, K = 10) 方法对剩余缺失值填补, 并对每个变量按总面积归一化。差异表达蛋白的筛选原则为: 若某蛋白在3次生物学重复数据中的均值在两组中的比值≥ 2或≤ 0.5 (FC ≥ 2), 且P < 0.05, 则认为该蛋白在两组中的表达具有差异。火山图中上调蛋白被标注为红色, 下调蛋白被标注为蓝色, 除此以外的蛋白认为在两组中的表达无显著变化, 标注为灰色(图 3A)。结果显示, 共有577种基因在△RUNX3/NCI N87R中的表达发生了显著改变, 其中上调191种, 下调386种, 未显著变化3 246种。进一步对差异表达基因按倍数变化排序, 发现上调倍数最大的分子分别为MLST8、DNAH1及ATG13等; 而下调倍数最大的分子分别为PRSS3、COG6及CD63等(图 3B)。聚类分析显示, 与NCI N87R相比, △RUNX3/NCI N87R细胞中多数分子显著下调(图 3C), 提示敲除RUNX3对NCI N87R细胞具有潜在的抑制作用。
|
Figure 3 Identification and analysis of differentially expressed proteins. A: Differentially expressed proteins shown by volcano plot; B: Rank of differentially expressed proteins; C: Heat map of differentially expressed proteins |
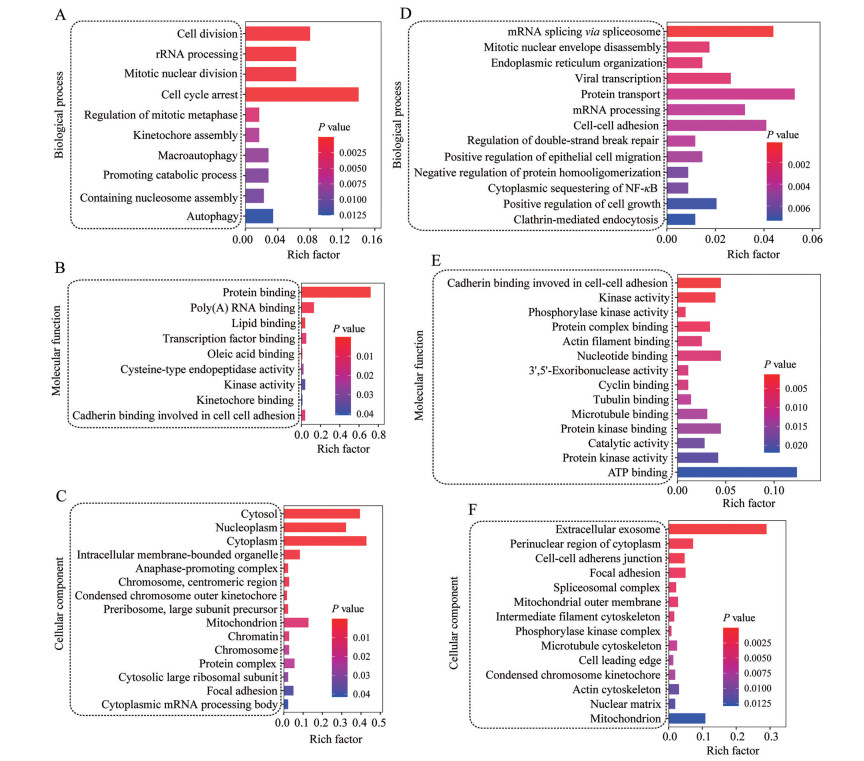
通过DAVID Bioinformatics Resources 6.8数据库对差异表达蛋白从生物学过程(biological process, BP)、细胞成分(cellular component, CC) 和分子功能(molecular function, MF) 进行基因本体分析(图 4A~F)。富集参数设置如下: 物种为Homo sapiens, 富集最大层级为4, 矫正后的阈值为0.05, 显示数目为TOP20, 基因背景为人类基因组。结果显示, 在生物学过程方面, 上调分子主要与调控细胞分裂、rRNA处理、细胞周期调控及自噬有关; 而下调分子主要与mRNA剪接、有丝分裂核膜解体、调控内质网功能及细胞黏附有关。分子功能方面, 上调分子在蛋白、脂质及RNA连接方面发挥作用; 而下调分子在细胞-细胞黏附、激酶活性及蛋白复合物链接方面具有作用。就细胞成分而言, 上调分子主要定位在胞浆、核质、细胞质等区域; 而下调分子主要定位于胞外外泌体、细胞核外围、线粒体外膜及细胞连接区域。
|
Figure 4 Gene ontology enrichment analysis of differential proteins. Analysis of upregulated (A-C) and downregulated (D-F) proteins involved in cell component, molecular function, and biological process |
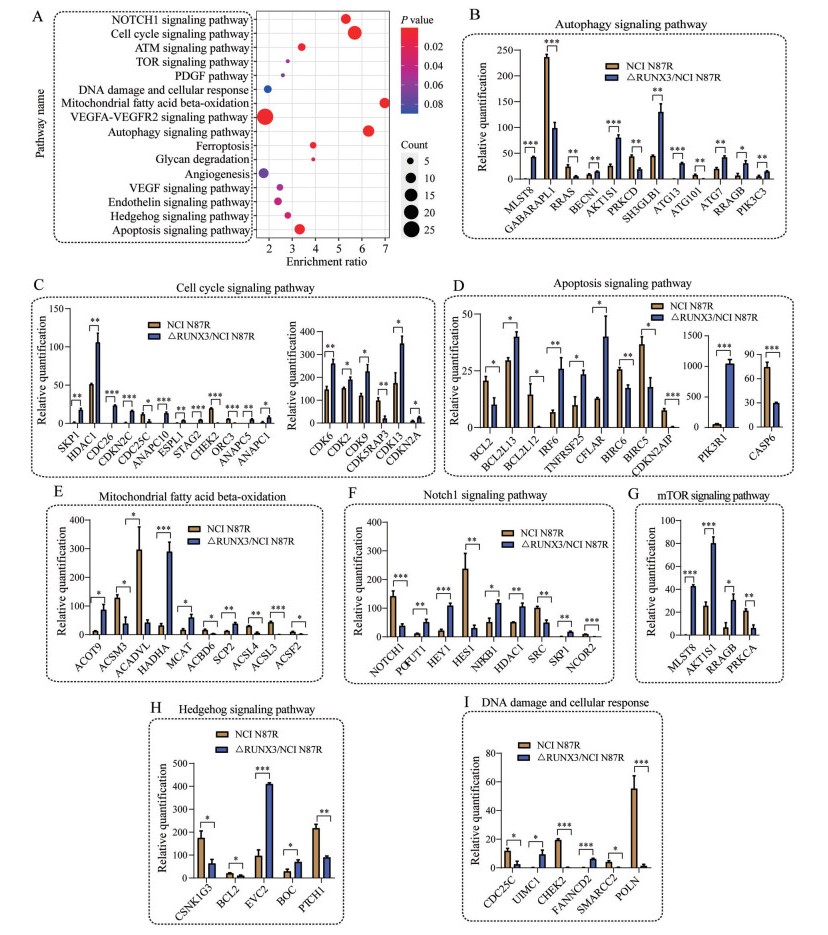
基于GeneAnalytics数据库对差异表达蛋白进行通路富集分析。根据显著性水平(P) 及富集率(enrichment ratio, ER), 采用气泡图对变化最显著的前18条通路进行可视化展示(图 5A)。结果显示, 细胞周期(P = 3.28×10-4、ER = 5.62)、NOTCH1 (P = 3.24×10-3、ER = 5.24)、自噬(P = 2.75×10-4、ER = 6.32)、线粒体脂肪酸β氧化(P = 2.17×10-3、ER = 6.93) 及凋亡通路(P = 6.11×10-3、ER = 3.27) 在富集的所有通路中变化最显著; ATM (P = 7.51×10-3、ER = 3.37)、VEGFA-VEGFR2 (P = 3.59×10-3、ER = 1.75) 及铁死亡(P = 8.78×10-3、ER = 3.87) 富集率相对较低; 而血管生成(P = 0.073×10-3、ER = 1.72)、DNA损失及细胞反应(P = 0.089、ER = 1.91) 通路的显著性水平及富集率较低。从通路相关蛋白定量结果看, 在自噬通路中, 除了RRAS、GABARAPL1、PRKCD及ATG101表达下调外, 其他分子包括ATG13、ATG7、BECN1、AKT1S1、MLST8、RRAGB、SH3GLB1及PIK3C3在△RUNX3/NCI N87R细胞中均显著上调(P < 0.001, P < 0.01) (图 5B), 表明敲除RUNX3促进NCI N87R细胞自噬。在细胞周期通路中, 上调分子有SKP1、HDAC1、CDC26、CDKN2C、ESPL1、STAG2、ANAPC1/5/10、CDK6、CDK2、CDK9、CDK13及CDKN2A (P < 0.001, P < 0.01); 而下调分子有CDC25C、CHEK2、ORC3及CDK5RAP3 (图 5C)。在凋亡信号中, 抗凋亡分子BCL2、BCL2L12及BIRC5/6表达下调, 促凋亡分子BCL2L13表达上调(图 5D), 提示敲除RUNX3导致NCI N87R细胞的促凋亡能力增强、而抗凋亡能力降低; 线粒体脂肪酸β氧化信号中, ACOT9、HADHA、MCAT及SCP2表达上调, 而ACSM3、ACADVL、ACBD6、ACSL4/3及ACSF2表达下调(图 5E), 表明△RUNX3/NCI N87R细胞线粒体功能异常。此外, NOTCH1、mTOR、Hedgehog通路及DNA损伤分子在△RUNX3/NCI N87R细胞中也显著改变(图 5F~I), 且部分分子同时参与多条通路的信号调控, 如MLST8、AKT1S1和RRAG8既是自噬调控分子, 又是mTOR通路核心分子; 而CHEK2在细胞周期及DNA损伤信号中具有重要作用, 也是DNA修复激活、DNA双链断裂引起凋亡所需激酶。综上所述, RUNX3对胃癌赫赛汀耐药具有重要作用, 敲除RUNX3导致耐药细胞多条通路异常, 提示这些通路异常可能是改变其耐药的重要机制, 也可能是逆转或降低耐药的潜在治疗靶标。
|
Figure 5 Enrichment analysis of signaling pathways of differentially expressed proteins. A: Bubble plot visualization of signaling pathways; B-I: Quantitative analysis of proteins in activated signaling pathways. *P < 0.05, **P < 0.01, ***P < 0.001 |
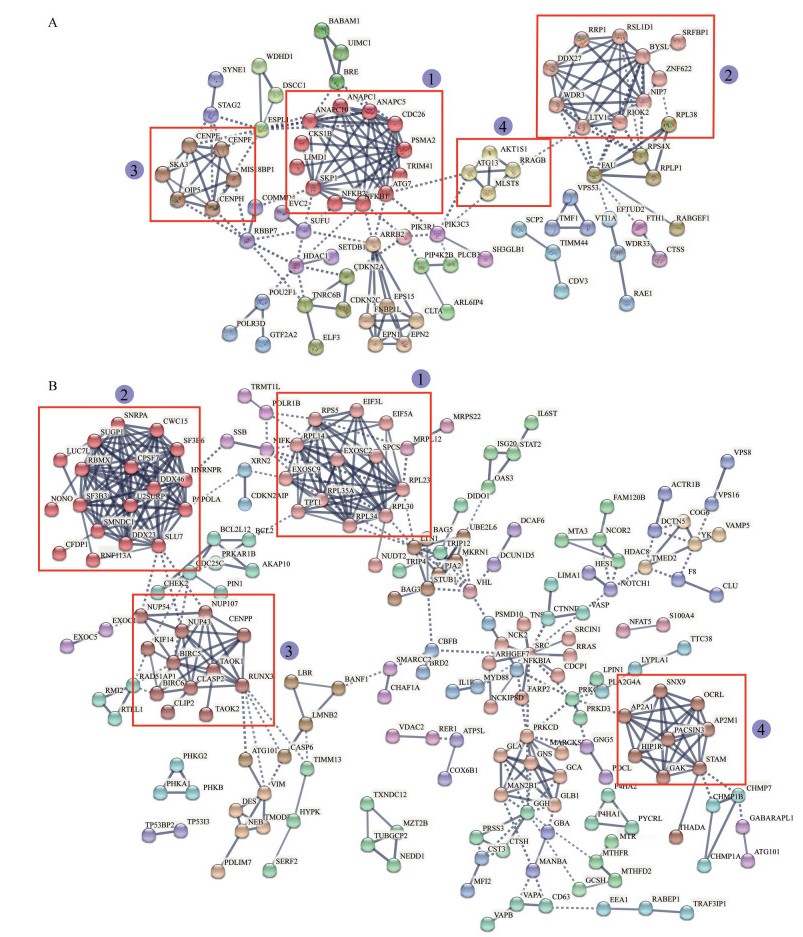
基于STRING (version 11.0) 数据库对差异表达蛋白进行蛋白-蛋白网络分析, 网络构建参数设置为: 类型为全局STRING网络、最低互作得分为0.7、聚类形式为马尔科夫算法(inflation参数≥ 3)。由网络可知, 功能相似的分子在网络中具有密切的互作关系, 且各分子依据不同的生物学功能组成独立的子网络, 实线相连的分子间具有较高的互作得分和相似的生物学功能, 虚线相连的分子归属于不同的子网络, 节点蛋白为各子网络间的连接枢纽分子。结果显示, △RUNX3/NCI N87R细胞中上调分子构成的网络主要包含4个子网络(图 6A), 其中子网络1拥有最多的分子, 分析其功能, 它们在细胞周期调控(CDK26、CKS1B、SKP1)、细胞凋亡(NFKB1、NKKB2、PSMA2)、自噬(ATG7)、蛋白质泛素化修饰及分裂(ANAPC1/5/10、TRIM41)、细胞骨架重构及组装(LIMD1) 中发挥重要作用; 而子网络2相关分子(RRP1、BYSL、DDX27、WDR3、LTV1、RIOK2、NIP7) 在rRNA加工处理、DNA损伤响应、细胞凋亡及衰老(RSL1D1、SRFBP1) 中具有重要作用; 子网络3分子在细胞有丝分裂、细胞周期、细胞信号交流、细胞免疫、蛋白转运(CENPE、CENPF、CENPH、SKA3、OIP5、MIS18BP1) 中至关重要。值得注意的是, 子网络4所有分子(ATG13、AKT1S1、RRAGB、MLST8) 在细胞大自噬及线粒体自噬过程中具有重要作用, 且AKT1S1、MLST8及RRAGB是mTOR通路中关键信号分子, 它们在细胞能量感知、外界刺激响应及细胞形态调控中起重要作用。相应地, 表达下调分子的互作网络主要包含4个子网络(图 6B), 其中子网络1含多个RPL家族成员(RPL14/5/35A/34/30/23), 作为核糖体的重要组成部分, 它们在蛋白质的合成、基因表达、肽链延伸、rRNA加工及蛋白泛素化调控过程中发挥重要作用; 作为线粒体核糖体蛋白, MRPL12和MRPS22与线粒体RNA聚合酶结合并激活其转录, 在线粒体翻译起始、延伸及终止过程具有重要作用; 子网络2包括多个与mRNA剪接相关的分子(SNRPA、CWC15、SUGP1、RBMX、NONO、SUL7、DDX23), 它们在RNA加工、DNA修复及重组方面发挥重要作用; 子网络3拥有的分子具有多功能特点, 在蛋白磷酸化、DNA修复响应、信号通路活化(TAOK1/2、RUNX3)、细胞增殖、凋亡及分化(BIRC5/6、CLASP2、RUNX3) 中至关重要; 而子网络4分子在细胞分裂、内吞、蛋白质转运和脂质代谢过程中具有重要功能。值得注意的是, RUNX3不仅是构成子网络3的成员分子, 而且作为枢纽分子与CASP6、ATG101、VIM及TIMM13相互作用。众所周知, CASP6与细胞凋亡、上皮细胞分化相关; ATG101作为自噬体形成的必需因子, 对ATG13起稳定作用, 保护其免受蛋白体酶降解; VIM参与caspase调控的细胞骨架蛋白的切割; 而TIMM13作为线粒体膜间伴侣分子、对线粒体蛋白的转运起重要作用。综上所述, 差异表达分子网络从不同角度诠释了RUNX3调控分子互作的内在关系, 为了解RUNX3调控胃癌赫赛汀耐药提供了广阔的洞察视角。
|
Figure 6 Protein-protein interaction network plot of up- (A) and down- (B) regulated proteins in △RUNX3 /NCI N87R cells |
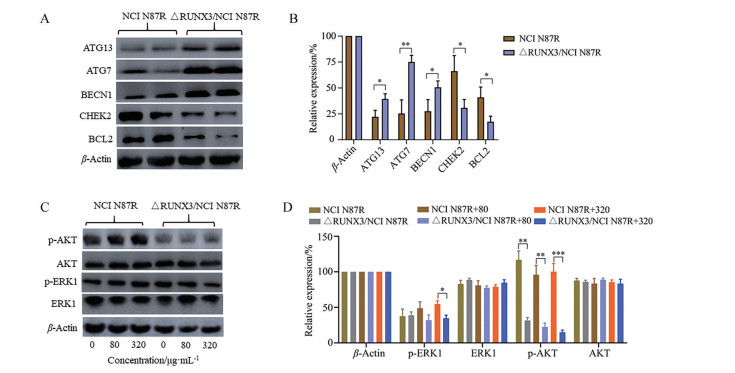
采用免疫印迹分别检测了NCI N87R和△RUNX3/NCI N87R细胞中ATG13、ATG7、BECN1、CHEK2、BCL2及HER-2下游分子p-AKT和p-ERK1的水平。结果显示, 与NCI N87R相比, ATG13、ATG7及BECN1在△RUNX3/NCI N87R中表达增加, 而CHEK2和BCL2的表达降低(图 7A、B), 提示敲除RUNX3改变NCI N87R细胞周期, 促进其自噬。此外, 敲除RUNX3导致耐药细胞p-AKT表达降低(P < 0.01), 且在实验条件下, 赫赛汀对p-AKT的抑制作用具有依赖性。与NCI N87R相比, p-ERK1在△RUNX3/NCI N87R中的表达无显著变化(P = 0.8), 当赫赛汀浓度达到320 μg·mL-1时, 其抑制作用具有统计学意义(P = 0.03) (图 7C、D), 提示敲除RUNX3增强赫赛汀对耐药细胞敏感性可能与抑制p-AKT的表达有关。
|
Figure 7 Expression of some proteins detected by Western blot. A, C: The expression of ATG13, ATG7, BECN1, CHEK2, BCL2, ERK1, p-ERK1, AKT, and p-AKT in NCI N87R and △RUNX3/NCI N87R cells by Western blot; B, D: Quantitative analysis of ATG13, ATG7, BECN1, CHEK2, BCL2, ERK1, p-ERK1, AKT, and p-AKT in NCI N87R and △RUNX3/NCI N87R cells. n = 3, mean ± SEM. *P < 0.05, **P < 0.01, **P < 0.001 |
自噬是真核细胞自我降解的生物学行为, 也是独立于caspase依赖的细胞程序性死亡方式, 在细胞代谢应激、维护基因组完整性及内稳态中具有重要作用[19-22]。癌细胞凋亡耐受是导致其化疗失败的重要因素, 也是诱发肿瘤耐药的重要机制[23]。肿瘤细胞可通过增强自噬降低化疗药物的作用, 然而, 由于过度自噬导致的自噬性死亡也是耐药细胞的重要死亡途径[24]。因此, 自噬在肿瘤细胞耐药过程中具有双重作用, 这为逆转或降低肿瘤耐药提供了一种新的治疗途径。
作为自噬体形成枢纽分子, ATG13直接与ATG1和ATG17连接, 通过介导ATG1与ATG17-ATG29-ATG31复合物作用调控大自噬和线粒体自噬过程[25]。ATG7为类泛素修饰剂激活酶, 通过激活ATG12与ATG5以及ATG8家族蛋白与磷脂酰乙醇胺结合, 参与调控caspase-8诱导的自噬性死亡[26]; ATG7还可通过调控线粒体自噬, 影响线粒体ROS水平, 参与线粒体启动的内源性凋亡[27]。而ATG101对维持ATG13的稳定性起重要作用, 有效保护ATG13不被蛋白酶体降解而失活[28]。作为自噬启动分子, ULK1的磷酸化水平受ATG13的调节, 当ATG13表达改变, ULK1的磷酸化状态随之变化, 诱发自噬启动[29]。蛋白质组数据表明, △RUNX3/NCI N87R细胞中自噬因子ATG13、ATG7及ATG101的表达显著改变, 其中ATG13和ATG7上调, 而ATG101下调, 提示敲除RUNX3促进NCI N87R细胞自噬。此外, 作为细胞增殖、凋亡及自噬的核心分子, mTOR通路受到多条信号的调节, 也是连接细胞自噬多条通路的枢纽中心, 通过调控ATG13与ULK1的磷酸化水平及ATG13-ULK1-RB1CC1复合体来调节自噬。结果显示, mTOR通路分子AKT1S1、MLST8及RRAGB均显著上调, 而PPKCA显著下调, 提示mTOR信号在RUNX3介导的细胞自噬中发挥作用。
作为自噬的特异性基因, BECN1可结合PI3K形成复合物(PI3KC3-C1和PI3KC3-C2), 以调节其他ATG基因在自噬中的定位, 进而调节自噬的活性, 且PI3KC3-C1参与自噬体的启动过程, 而PI3KC3-C2参与自噬体的成熟和内吞作用[30]。也有研究显示, BECN1还可通过影响自噬活性, 在肿瘤发生、发展及耐药过程中发挥作用, 且通过与BCL2结合同时参与自噬和凋亡两种程序性死亡过程, 成为连接细胞程序性死亡的交点[31]。结果显示, BECN1及BCL2在△RUNX3/NCI N87R中表达显著改变, 前者上调, 后者下调, 提示RUNX3引起的耐药细胞自噬性凋亡可能与BECN1与BCL2之间的失衡有关。
另一方面, 细胞凋亡分子在△RUNX3/NCI N87R中的表达显著改变, 如与内源性凋亡相关的分子, 包括BCL2、BCL2L12、BIRC5和BIRC6表达显著下调, 而BCL2L13显著上调, 前者具有抗凋亡作用, 后者具有促凋亡作用; 与内质网应激及外源性凋亡相关分子(PIK3R1) 显著上调; 通过DNA损伤调控细胞凋亡及老化的分子(CDKN2AIP) 显著下调; 通过影响半胱氨酸内肽酶活性诱导凋亡的分子既有上调(CFLAR), 也有下调(CASP6); 此外, TNFRSF25表达显著上调, 其直接与适配器TRADD作用, 介导NF-κB激活并诱导细胞凋亡, 这些分子与多种凋亡途径有关, 提示敲除RUNX3诱导NCI N87R细胞凋亡可能涉及多种凋亡机制, 这可能是敲除RUNX3导致耐药细胞对赫赛汀增敏的重要机制。
众所周知, 线粒体是细胞生命活动的重要场所, 不仅对维持细胞稳态至关重要, 而且是细胞能量交换所在地。应激状态时, 线粒体脂肪酸β氧化是细胞代谢所需能量的主要来源, 而相关酶的表达或活性的改变是脂肪酸β氧化异常的主要原因, 也是引起线粒体功能障碍的重要因素。蛋白质组数据显示, 与线粒体脂肪酸合成、转运及代谢相关的多个分子在△RUNX3/NCI N87中变化, 其中ACOT9在调节线粒体酰基辅酶A、游离脂肪酸和辅酶A的平衡中具有重要作用; HADHA是线粒体三功能酶, 能够催化单心磷脂酰化转化为心磷脂, 后者为线粒体膜磷脂的主要成分, 且在细胞凋亡中起关键作用; MCAT与线粒体脂肪酸合成有关; 而SCP2在支链脂肪酸的过氧化物酶体氧化中起作用, 这些分子在△RUNX3/NCI N87R中显著上调。相应地, ACSM3、ACADVL、ACBD6、ACSL4/3及ACSF2在△RUNX3/NCI N87R中显著下调, 它们在线粒体酰基辅酶A的合成、转运及脂肪酸代谢中发挥重要作用。以上分子表达改变可能是导致线粒体功能异常的重要原因, 进而引起线粒体代谢紊乱, 促进线粒体自噬, 并诱导细胞凋亡。
此外, 与细胞周期、DNA损伤响应相关分子在△RUNX3/NCI N87R细胞中显著改变, 这些分子在细胞周期调控、细胞代谢和细胞监控过程中具有重要作用。如CHEK2是DNA修复异常、DNA双链断裂时诱导细胞凋亡的关键激酶, 也是DNA损伤检控核心分子, 在响应DNA损伤凋亡信号中至关重要; ORC3是启动DNA复制所必需的复合体; CDC25C在细胞DNA损伤响应、p53信号转导引起细胞周期阻滞中发挥作用; 而CDK5RAP3在细胞凋亡核膜破裂阶段起重要作用。这些分子在△RUNX3/NCI 87R中表达下调, 提示敲除RUNX3导致耐药细胞周期异常、DNA修复及监控失调。
NOTCH为高度保守的跨膜蛋白, 与细胞发育、分化及凋亡有关。文献报道[32], IL-6/STAT3/Jagged-1/NOTCH环路激活是胃癌赫赛汀耐药的重要机制, 且NOTCH活化可上调IL-6表达, 后者诱导肿瘤细胞表型转换。不同于文献报道, 本研究数据表明, NOTCH1在△RUNX3/NCI N87R细胞表达下调, 推测敲除RUNX3可能对NOTCH1信号起抑制作用, 而且NOTCH的靶基因HES1与HEY1表达趋势相反, 其机制待进一步探究。
作者贡献: 刘文虎负责实验设计指导、实验操作、文章撰写及修改; 常晋霞负责文献调研、参与文章撰写及数据分析; 袁江北负责数据采集; 王仕宝负责文献调研。
利益冲突: 所有作者均声明无利益冲突。
| [1] |
Mitani S, Kawakami H. Emerging targeted therapies for HER2 positive gastric cancer that can overcome trastuzumab resistance[J]. Cancers, 2020, 12: 400. DOI:10.3390/cancers12020400 |
| [2] |
Sampera A, Sánchez-Martín FJ, Arpí O, et al. HER-family ligands promote acquired resistance to trastuzumab in gastric cancer[J]. Mol Cancer Ther, 2019, 18: 2135-2145. DOI:10.1158/1535-7163.MCT-19-0455 |
| [3] |
Liu Z, Zhang X, Xu X, et al. RUNX3 inhibits survivin expression and induces cell apoptosis in gastric cancer[J]. Eur J Cell Biol, 2014, 93: 118-126. DOI:10.1016/j.ejcb.2014.02.002 |
| [4] |
Tong D, Jiang Y, Li M, et al. RUNX3 inhibits cell proliferation and induces apoptosis by TGF-beta-dependent and -independent mechanisms in human colon carcinoma cells[J]. Pathobiology, 2009, 76: 163-169. DOI:10.1159/000218332 |
| [5] |
Whittle MC, Hingorani SR. RUNX3 and cell fate decisions in pancreas cancer[J]. Adv Exp Med Biol, 2017, 962: 333-352. |
| [6] |
Han Y, Liang D. The role of the tumor suppressor RUNX3 in giant cell tumor of the bone[J]. Int J Oncol, 2012, 40: 673-678. |
| [7] |
Selvarajan V, Osato M, Nah GSS, et al. RUNX3 is oncogenic in natural killer/T-cell lymphoma and is transcriptionally regulated by MYC[J]. Leukemia, 2017, 31: 2219-2227. DOI:10.1038/leu.2017.40 |
| [8] |
Barghout SH, Zepeda N, Vincent K, et al. RUNX3 contributes to carboplatin resistance in epithelial ovarian cancer cells[J]. Gynecol Oncol, 2015, 138: 647-655. DOI:10.1016/j.ygyno.2015.07.009 |
| [9] |
Kataoka J, Shiraha H, Horiguchi S, et al. Loss of Runt-related transcription factor 3 induces resistance to 5-fluorouracil and cisplatin in hepatocellular carcinoma[J]. Oncol Rep, 2016, 35: 2576-2582. DOI:10.3892/or.2016.4681 |
| [10] |
Horiguchi S, Shiraha H, Nagahara T, et al. Loss of RUNT-related transcription factor 3 induces gemcitabine resistance in pancreatic cancer[J]. Mol Oncol, 2013, 7: 840-849. DOI:10.1016/j.molonc.2013.04.004 |
| [11] |
Chang JX, Wang Y, Zhang F, et al. Proteomic study of transcription factors in trastuzumab-resistant gastric cancer based on liquid chromatography-mass spectrometry technique[J]. Chin J Anal Chem (分析化学), 2019, 47: 1035-1044. |
| [12] |
Liu W, Yuan J, Liu Z, et al. Label-free quantitative proteomics combined with biological validation reveals activation of Wnt/β-catenin pathway contributing to trastuzumab resistance in gastric cancer[J]. Int J Mol Sci, 2018, 19: 1981. DOI:10.3390/ijms19071981 |
| [13] |
Chang J, Wang Q, Bhetuwal A, et al. Metabolic pathways underlying GATA6 regulating trastuzumab resistance in gastric cancer cells based on untargeted metabolomics[J]. Int J Med Sci, 2020, 17: 3146-3164. DOI:10.7150/ijms.50563 |
| [14] |
Liu WH, Yuan JB, Chang JX. Label-free quantitative proteomics for investigation of signaling pathways of GATA6 regulating trastuzumab resistance in gastric cancer cells[J]. Chin J Anal Chem (分析化学), 2020, 48: 187-196. DOI:10.1016/S1872-2040(19)61215-6 |
| [15] |
Liu WH, Wang Y, Li SM, et al. Label-free quantitative proteomic analysis of acquired Herceptin resistance in gastric cancer cells[J]. Acta Pharm Sin (药学学报), 2018, 53: 553-560. |
| [16] |
Liu WH, Yuan JB, Yang L, et al. Mechanisms of curcumin to reverse Herceptin resistance in gastric cancer cells[J]. Acta Pharm Sin (药学学报), 2018, 53: 1818-1824. |
| [17] |
Wi-Niewski JR, Zougman A, Nagaraj N, et al. Universal sample preparation method for proteome analysis[J]. Nat Methods, 2009, 6: 359-362. DOI:10.1038/nmeth.1322 |
| [18] |
Fu Q, Yuan J, Wang L, et al. Proteomic analysis of murine macrophages mitochondria and lysosomes reveal cathepsin D as a potential broad-spectrum antimicrobial protein[J]. J Proteomics, 2020, 223: 103821. DOI:10.1016/j.jprot.2020.103821 |
| [19] |
Yu L, Chen Y, Tooze SA. Autophagy pathway: cellular and molecular mechanisms[J]. Autophagy, 2018, 14: 207-215. DOI:10.1080/15548627.2017.1378838 |
| [20] |
Denton D, Kumar S. Autophagy-dependent cell death[J]. Cell Death Differ, 2019, 26: 605-616. DOI:10.1038/s41418-018-0252-y |
| [21] |
Yang WZ, Tu J, Liu N, et al. Advances in signal regulation and detection strategies for autophagy in fungal cells[J]. Acta Pharm Sin (药学学报), 2020, 55: 1431-1438. |
| [22] |
Tan CY, Tian HZ, Kuang H, et al. Medications regulate autophagy for treatment of Alzheimer's disease[J]. Acta Pharm Sin (药学学报), 2019, 54: 984-990. |
| [23] |
Ng CP, Bonavida B. A new challenge for successful immunotherapy by tumors that are resistant to apoptosis: two complementary signals to overcome cross-resistance[J]. Adv Cancer Res, 2002, 85: 145-174. |
| [24] |
Chen P, Huang H, Wang Y, et al. Curcumin overcome primary gefitinib resistance in non-small-cell lung cancer cells through inducing autophagy-related cell death[J]. J Exp Clin Cancer Res, 2019, 38: 254. DOI:10.1186/s13046-019-1234-8 |
| [25] |
Li W, Zhang L. Regulation of ATG and autophagy initiation[J]. Adv Exp Med Biol, 2019, 1206: 41-65. |
| [26] |
Yu L, Alva A, Su H, et al. Regulation of an ATG7-beclin 1 program of autophagic cell death by caspase-8[J]. Science, 2004, 304: 1500-1502. DOI:10.1126/science.1096645 |
| [27] |
Yu L, Wan F, Dutta S, et al. Autophagic programmed cell death by selective catalase degradation[J]. Proc Natl Acad Sci U S A, 2006, 103: 4952-4957. DOI:10.1073/pnas.0511288103 |
| [28] |
Hosokawa N, Sasaki T, Iemura S, et al. Atg101, a novel mammalian autophagy protein interacting with Atg13[J]. Autophagy, 2009, 5: 973-979. DOI:10.4161/auto.5.7.9296 |
| [29] |
Li Z, Tian X, Ji X, et al. ULK1-ATG13 and their mitotic phospho-regulation by CDK1 connect autophagy to cell cycle[J]. PLoS Biol, 2020, 18: e3000288. DOI:10.1371/journal.pbio.3000288 |
| [30] |
Wang L, Ma H, Huang P, et al. Down-regulation of beclin1 promotes direct cardiac reprogramming[J]. Sci Transl Med, 2020, 12: 7856. DOI:10.1126/scitranslmed.aay7856 |
| [31] |
Lian J, Karnak D, Xu L. The Bcl-2-beclin 1 interaction in (-)-gossypol-induced autophagy versus apoptosis in prostate cancer cells[J]. Autophagy, 2010, 6: 1201-1203. DOI:10.4161/auto.6.8.13549 |
| [32] |
Yang Z, Guo L, Liu D, et al. Acquisition of resistance to trastuzumab in gastric cancer cells is associated with activation of IL-6/STAT3/Jagged-1/Notch positive feedback loop[J]. Oncotarget, 2015, 6: 5072-5087. DOI:10.18632/oncotarget.3241 |
 2021, Vol. 56
2021, Vol. 56


