综观上市药物, 其结构类型多隶属于化学合成小分子以及生物大分子(如抗体药物、核酸等)。但小分子药物研发周期较长, 体内代谢复杂, 常会造成不可忽视的毒性及安全性等问题。此外, 更重要的一点是有限的小分子化合物库难以与一些具有挑战性的潜力靶点相结合。这增加了发现苗头化合物的难度, 使得新靶点的早期研究受阻。相比于小分子药物, 生物大分子药物的组成都是由人体内已存在的氨基酸或者核苷酸组成, 除会导致一些免疫反应外, 其他由药物本身导致的不良反应发生概率较低。但大分子药物也存在一些不足, 主要是其制造复杂, 均一性和稳定性较差, 同时因药物递送问题, 给药途径多为静脉给药, 使其开发和应用受到很大限制。而肽类药物介于大分子和小分子药物之间, 兼具两者优点, 一方面组成类似大分子药物, 为人体内常见的氨基酸, 同时因相对分子质量较小, 可采用合成方法制备, 具有优良的均一性和稳定性。同时肽类药物还可靶向一些特殊靶点体系, 特别是蛋白-蛋白相互作用界面, 因而关注度逐渐提升, 激发了越来越多的科学家开展肽类药物研发[1-3], 并成为了近年来药物研究的新兴方向。
肽类化合物可分为链状线性肽及约束环肽。其中环肽作为构象受限的肽类, 结构上较链状线性肽更为刚性, 具有一些独特的优良性质: 其一, 肽类药物分子质量介于化学小分子和抗体大分子之间, 一般大小为500~2 000 Da, 通常因构象变化多, 具有非常不利的熵效应, 而环肽结构极大约束了潜在的构象, 减少熵效应的自由能损失, 通常具有更佳的结合亲和力和靶标选择性。同时因环肽分子具有较大尺寸结构, 从而与大分子靶点接触面大, 对于小分子无法有效靶向的结合位点, 也能发生较多的相互作用[4]。其二, 环肽分子具有更优的代谢稳定性和膜通透性。不同于线性肽, 环肽凭借引入多种修饰手段可增加肽类药物的代谢稳定性, 包括血浆的蛋白酶水解稳定性、肝脏代谢稳定性。另外因为环肽分子刚性较强, 更易透过细胞膜, 进入细胞内发挥效用[1], 因而表现出更高的药理活性。这些成药性方面的性质特点也为后续的治疗方面的应用带来一定优势, 增加了患者的依从性。
鉴于如上所述的诸多有利特性, 环肽在药物研发方面已取得较多成果[5-7], 经由FDA批准上市的环肽类药物就有9种, 包括阿尼芬净(anidulafungin)、兰瑞肽(lanreotide)、替拉万星(telavancin)、罗米地辛(romidepsin)、培吉沙肽(peginesatide已退市)、利那洛肽(linaclotide)、帕瑞肽(pasireotide)、达巴万星(dalbavancin)、奥利万星(oritavancin)。开展临床研究的环肽药物也已占有一定比例, 日益获得了关注。目前已上市的环肽药物中绝大多数是从微生物、人体激素或其衍生物中分离出来的天然产物[8]。
自1985年由Smith[9]首次报道在丝状噬菌体pⅢ蛋白基因中插入外源基因可在噬菌体表面以融合蛋白的形式表达后, 开创了一种组合生物学技术—噬菌体展示技术。噬菌体展示技术利用基因编码手段构建组合文库, 带来了技术上的飞跃, 被广泛应用于抗体、骨架蛋白、多肽等分子的展示[10], 为药物发现提供了有力的工具平台。因此, 除筛选天然来源的环肽作为药物研发起点外, 采用噬菌体展示技术来开展环肽研究是另一个重要的研究策略。例如专注于环肽药物的Bicycle Therapeutics公司就利用该技术开展研究, 目前已取得了较好的进展, 公开的6个在研项目涉及多个靶点(https://www.bicycletherapeutics.com/programs/)。采用噬菌体展示技术所产生的多样性肽库可达1012量级, 这使得该技术可针对小分子难以靶向的靶点开展多肽抑制剂的研发。随着生物展示技术的逐渐成熟与发展, 编码多样性文库也变得更为便利。因此, 快速建立生物展示环肽库的方法作为药物发现平台已逐步获得了广大药物科学家的认可。目前基于基因编码的环肽库除了借助噬菌体展示技术之外, 也有其他选择, 如mRNA展示[11, 12]、SICLOPPS[13]等。但与mRNA展示和SICLOPPS方法相比, 噬菌体展示的方法具有成本低、操作简单等优势, 是快速实现有效筛选的最常用的展示方法[14]。
本篇综述主要分为3个部分, 第一部分将简要综述近年来已上市及在研的环肽药物, 着重介绍通过噬菌体展示技术发现的环肽药物。第二部分系统性阐述噬菌体展示环肽的构建方法和环肽库筛选的特点, 为构建新颖多样性环肽库提供参考, 以期引发同行研究者思考更优的环化方法或更多样的环结构。第三部分则着重介绍各种噬菌体展示环肽库的类型特点以及研究个例, 使同行研究者更为直观地了解各种环肽库的特点, 从而根据不同的研究目的, 找到适合的建库及筛选策略。
1 环肽药物的研发现状环肽药物分子的大小通常介于小分子药物与生物大分子药物之间, 凭借独特优良的性质如毒性小、代谢稳定、膜通透性高等特点, 在新药研发中具有非常重要的应用前景。目前处于临床研究约有40个环肽药物, 并且近年来临床前及临床在研的环肽类药物数目持续增加, 平均每年都有一个新的环肽药物进入临床的发展态势。目前已上市的环肽类药物多为抗菌肽或激素类似物, 除了知名的催产素(oxytocin)、万古霉素(vancomycin)、环孢菌素(cyclosporine), 还有近年来获批上市的环肽药物anidulafungin、lanreotide、linaclotide等。它们均来源于天然产物及其衍生物, 适应症集中于感染性疾病或肿瘤。
目前临床在研的环肽类药物也已占有一定比例, 成为上市药物的潜在力量。本文以研发环肽药物的代表性公司为基础, 将其进展较快的环肽类药物列表所示(表 1), 直观呈现目前环肽药物的研究平台、结构类型及治疗应用方向, 为科研同行提供环肽药物的研究方向与灵感。据表 1所示, 目前环肽研究较多采用了理性设计的药物发现平台, 其中环肽形式不仅有双环肽, 还有订书肽等。
| Table 1 Part of cyclopeptide drugs in clinical research. a PEM (protein epitope mimetic) |
鉴于篇幅, 本文将重点介绍表 1中由噬菌体展示技术开发的临床在研的环肽药物(图 1)。由Apellis开发的APL-2是经由体外进化筛选噬菌体展示组合肽库衍生而来的二硫键形式的环肽, 含13个氨基酸, 是一类C3补体抑制剂。APL-2是对多肽结构进行了聚乙二醇链(PEG) 修饰, 增加其半衰期, 有效提高了其在血浆中的稳定性。补体C3分子在血浆中的含量高, 在多种疾病中表现为过度激活, APL-2能够实现特异性结合C3及C3b, 而C3处于C5的上游。C5蛋白的抑制是治疗阵发性睡眠性血红蛋白尿症(paroxysmal nocturnal hemoglobinuria, PNH) 的关键因素, 因此通过抑制C3也有望实现治疗PNH的功能。2020年1月, Apellis公布了APL-2在治疗PNH成人患者的3期临床数据(NCT03531255、NCT03500549;https://investors.apellis.com/news-releases/news-release-details/apellis-reports-positive-top-line-results-phase-3-head-head), 包括提高血红蛋白水平, 避免输血依赖的评估方面等都具有不错的效果, 相比于依库珠单抗(eculizumab), APL-2在提高血红蛋白水平上具有明显改善。APL-2除了获批治疗PNH及地图样萎缩症[15] (geographic atrophy, GA) 患者的快速通道之外, 还在积极开展干性老年性黄斑变性(age-related macular degeneration, AMD)、C3肾炎等疾病的临床研究。鉴于其在临床上具有良好的应用前景, 有望为Apellis公司创造巨大的经济效益。
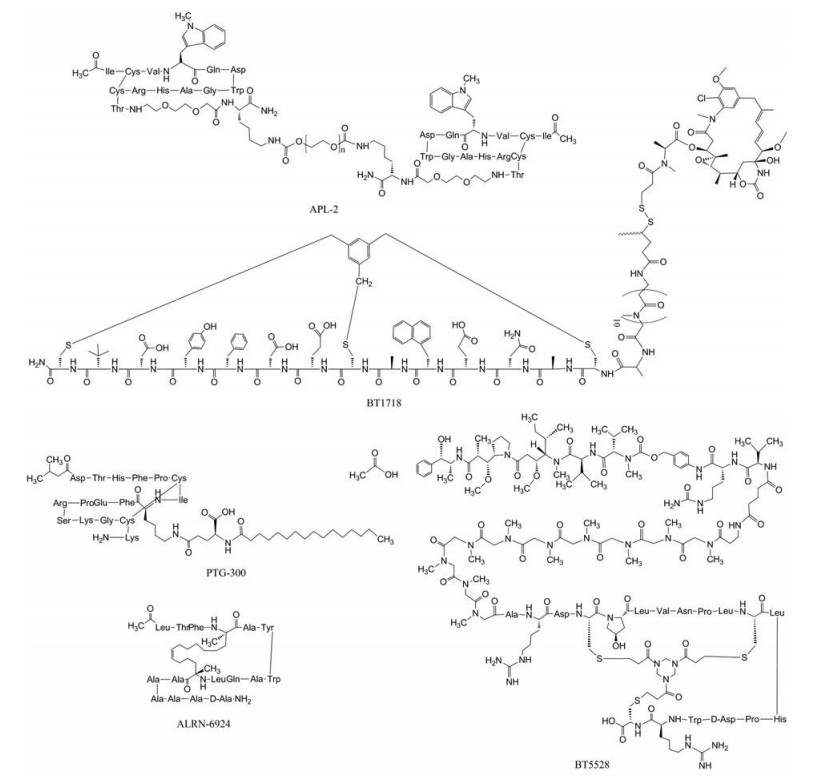
|
Figure 1 The structure of cyclic peptide drug developed by phage display technology in clinical research |
Bicycle Therapeutics公司是研发双环约束肽药物的翘楚。公司基于Greg Winter及Christian Heinis教授开发的噬菌体展示技术, 构建及筛选通过化学试剂环化的噬菌体展示环肽库, 该类试剂具有在温和条件下与游离巯基发生反应的特征。这些环肽多为9~15个氨基酸的短肽, 具有较好的组织渗透性, 可实现肿瘤组织的穿透及有效驻留, 同时具有较高的靶向选择性。目前有6个公开的在研项目, 主要集中于癌症治疗方面, 其开发的代表性药物是靶向膜型基质金属蛋白酶-1 (MT1-MMP) 的环肽-毒物DM1偶联药物BT-1718 (https://www.bicycletherapeutics.com/publications/), 目前已处于临床2期(NCT03486730)。MT1-MMP在人体正常组织中处于较低水平, 而在实体肿瘤组织中是过表达的, 并且在肿瘤侵袭及转移中扮演着重要角色。尽管该靶点对于传统小分子及抗体大分子而言开发配体较为困难, 但BT1718可实现有效靶向MT1-MMP, 在实体瘤如三阴性乳腺癌(triple negative breast cancer, TNBC)、非小细胞肺腺癌(non-small cell lung carcinoma, NSCLC) 上具有较好的治疗效果, 目前的临床数据显示其具有良好的耐受性。其他用于肿瘤治疗的候选药物如靶向EphA2的BT5528及靶向血浆激肽释放酶KLKB1的THR-149均已经进入临床研究, 除此以外, 该公司还积极推进多个临床前环肽候选药物。
PTG300是Protagonist Therapeutics公司采用噬菌体展示技术开发的基于二硫键成环的皮下注射铁调素模拟物(https://investors.protagonist-inc.com/news-releases/news-release-details/protagonist-therapeutics-announces-initial-phase-2-results), 主要针对真性红细胞增多症(polycythemia vera, PV) 和遗传性血色素沉着症(hereditary hemochromatosis, HH)。于2020年5月7日已公布在真性红细胞增多症患者上开展的2期临床试验的初步结果, 临床数据显示其具有良好的耐受性, 安全性高, 没有出现严重的不良反应事件(NCT04057040)。2020年6月17日, 该药物也获得FDA批准作为孤儿药用于治疗真性红细胞增多症。
不同于上述几种环肽药物, 由Aileron Therapeutics公司开发的ALRN-6924 (https://www.aileronrx.com/clinical-development/), 是通过噬菌体展示技术研发的鼠双微染色体2基因(mouse double minute 2, MDM2) 和鼠双微体基因X (mouse double minute X, MDMX) 的双重抑制剂装订肽。该公司凭借噬菌体展示订书肽技术, 利用其专有的化学交联手段, 通过与具有刚性结构的碳氢化合物形成碳碳键从而稳固多肽的α-螺旋结构。由此形成的订书肽具有良好的细胞膜穿透性质, 能靶向细胞内蛋白-蛋白相互作用靶标。代表性订书肽ALRN-6924与ATSP-7041[16]是类似物, 具有稳定良好的细胞渗透性, 可同时靶向MDM2/MDMX, 破坏其对抑癌基因p53蛋白的抑制作用, 激活p53蛋白使其行使正常功能包括对细胞周期的调控、DNA修复、细胞凋亡等, 从而发挥抗肿瘤效应。截止于2019年8月, 已完成了Ⅰ/Ⅱa期临床试验(NCT04022876、NCT03725436), 评估其在过表达野生型p53的实体瘤及血液系统恶性肿瘤患者中的多项数据, 包括药效学、药代动力学、耐受性及抗肿瘤作用等, 显示了进一步开展研究的价值。ALRN-6924还可与其他药物如单抗连用, 其联合托泊替康(topotecan) 在小细胞肺癌模型中具有增强的抗肿瘤活性。它的成功进展为订书肽在医药领域的发展打开了想象大门。
2 噬菌体展示技术应用于环肽库构建及筛选 2.1 噬菌体展示的发展历程1983年Renato[17]就提出通过噬菌体表面展示外源多肽的概念。随后, 1985年Smith首次将编码多肽序列的外源DNA片段插入丝状噬菌体f1的基因Ⅲ中, 产生融合蛋白, 从而在噬菌体表面展示外源多肽, 利用与靶标分子的特异性亲和力筛选噬菌体结合物, 随着病毒感染宿主细菌的繁殖扩增, 模拟自然进化的淘筛过程, 快速发现与靶标特异性结合富集的多肽序列, 开创了噬菌体展示技术[9]。在1990年Winter教授[18]通过构建噬菌体展示抗体库, 在试管内实现了对抗体药物的发现研究。他们通过不断进行“突变引入”及“自然淘筛”的过程, 进一步优化筛选以期获得高特异性的抗体, 最终采用该技术成功开发了第一个全人源的抗体药物即阿达木单抗(adalimumab), 将噬菌体展示技术真正应用到医药研发领域。此外, 他还打开了利用化学修饰手段构建噬菌体展示特定环肽库的大门[19, 20], 启发Heinis Christian等其他学者在环肽领域进一步深入探索。
2.2 环肽库的构建及筛选的常规流程噬菌体展示环肽库的构建通常是将肽段和蛋白展示于m13丝状噬菌体[21]的pⅲ或者pvⅲ蛋白上, 考虑到展示效价的差别, 一般选择将外源cdna序列插入pⅲ基因中, 获得较低效价展示, 适用于针对特定靶点筛选较高亲和力的环肽配体[22]。此外, 也可通过使用噬菌粒载体与辅助噬菌体(如m13k07等) 一同构建完整的噬菌体颗粒[23], 进而表达噬菌体展示环肽, 实现单价展示, 这也是筛选高亲和力配体的常用的展示系统。
以筛选高亲和配体为例, 选择以pⅢ蛋白作为展示融合蛋白的噬菌粒展示系统, 借助于简并密码子(NNK)n编码随机氨基酸序列[24-26], 插入到pⅢ衣壳蛋白的结构域D1和D2基因中, 在辅助噬菌体协助下构建完整噬菌体环肽库。由于靶点的性质及结构千差万别, 构建多样性丰富的环肽库显得极为重要。除了如上编码序列带来的多样性之外, 环化结构的差异也是多样性的来源之一。综合已发表的噬菌体展示环肽库文献, 环肽库大多表现为单环或双环形式, 少量报道形成多环肽库。环肽库的成键形式主要以二硫键为主, 其他方式则以借助化学试剂或在肽链上引入非天然氨基酸, 进而通过与肽链上的半胱氨酸反应成环(图 2)。

|
Figure 2 The format of the phage cyclic peptide libraries |
噬菌体环肽库淘选可分为体外和体内淘选[27, 28]两种方式, 以发现先导物为目的的噬菌体环肽库筛选大多采用体外淘选。淘选的流程大致如下: 先将靶标蛋白固定, 再添加噬菌体展示环肽库, 共同孵育一段时间, 通过多次清洗洗去非特异性的噬菌体环肽, 再用较低pH (如pH 2.2) 的酸性洗脱液破坏靶标蛋白与噬菌体展示环肽间的结合从而收集洗脱下来的特异性结合的噬菌体展示环肽, 再次感染大肠杆菌后繁殖扩增, 经过多轮这样的淘筛形式后, 可以有效富集特异性结合的噬菌体环肽。然后借助常规一代测序如Sanger测序或者二代测序Roche's 454、Illumina测序技术等[29]及特定的分析程序[30, 31]来鉴定序列。此过程中, 根据靶标蛋白固定方式的不同, 又可分为固相淘筛和液相淘选[32, 33]。其中固相淘筛一般固定靶标蛋白于免疫吸附材料如酶标板上。该方法虽然操作简单方便, 但是固定于免疫吸附材料上, 会对一些蛋白的天然构象产生影响, 甚至可能破坏其结合表位, 并且蛋白包被固定通常不够稳定。液相淘选通过将蛋白固定于带有亲和涂层如链霉亲和素或是Ni2+的磁珠上, 整个淘筛体系处于液相, 可以增加靶标蛋白与噬菌体文库的接触几率, 提高筛选效率。为减少直接结合于磁珠的噪声序列的干扰, 可采取不同磁珠交替使用的筛选方式[32]以及差减淘筛的方式[33]。
除了上述最常用的两大类体外淘选方式外, 还有细胞淘选[34]。主要是针对那些性质不稳定、难以提纯, 且要求获取空间结构的抗原靶点, 例如癌细胞的表面受体, 可考虑将细胞直接作为抗原筛选, 但该方法也存在一些缺陷, 如通过该方法很容易筛选到细胞表面的其他非特异性的噬菌体, 增加了筛选的复杂度。
总之, 每种筛选策略都有其独特的优势和一些缺陷, 只有根据不同的筛选目的和靶标蛋白的特性来选择合适的筛选方式才能达到事半功倍的效果。
3 噬菌体展示环肽库的特征类型及应用案例 3.1 噬菌体展示单环肽库噬菌体展示单环肽库应用经历了从偶然事件到理性设计的发展过程。在构建的随机展示肽库或是抗体库中往往会筛选到序列中含两个半胱氨酸的序列。鉴于半胱氨酸易氧化形成二硫键的特点, 且测试活性时发现二硫键成环限制构象, 其活性往往大于线性肽, 这促使科学家开始尝试主动设计含定点半胱氨酸的肽库。Merck制药的O'Neil[35]于1992年首次报道了构建噬菌体展示环肽库, 肽库形式为CX6C库(其中X指可变氨基酸, C指半胱氨酸, 下标数字为可变氨基酸数目), 肽段在噬菌体表面表达后, 半胱氨酸之间会自发氧化成环, 从而形成环肽库。McLafferty等[36]也成功设计了含18个氨基酸的较大的噬菌体展示环肽库, 库容大小为8.55×106。自此越来越多的文献报道了以两端半胱氨酸成环的单环肽库。
采用这些基于成对的半胱氨酸侧链巯基氧化形成二硫键的单环肽库进行筛选也获得了不错的应用成果。为了能够更好地在动物模型上研究尿激酶型纤溶酶原激活物(urokinase plasminogen activator, uPA) 靶点的癌症机制, Andersen等[37, 38]针对鼠源uPA筛选了CX8C型的二硫桥键噬菌体展示单环肽库, 经过四轮的迭代筛选, 最终分离得到了较高亲和力的序列CPAYSRYLDC, 即mupain-1 (图 3A), 其Ki为0.55 ± 0.08 μmol·L-1, 并且相对于人源uPA, mupain-1对于鼠源uPA具有较高的特异性, 同样针对人源uPA筛选得到的单环肽序列upain-1 (CSWRGLENHRMC), 其对鼠源uPA的亲和力则很低。通过丙氨酸突变扫描的方法, 分别探究了靶点蛋白中与结合相关的重要残基, 以及mupain-1中的个别氨基酸的重要性(Pro2、Tyr4、Ser5、Arg6及Tyr7) 和环化结构的重要性。通过针对提高亲和力进行修饰改造得到的两变体可将亲和力提升2倍及10倍, 有望使mupain-1成为新的鼠源uPA高亲和力抑制剂, 在肿瘤侵袭转移模型中成为有用的探针分子。
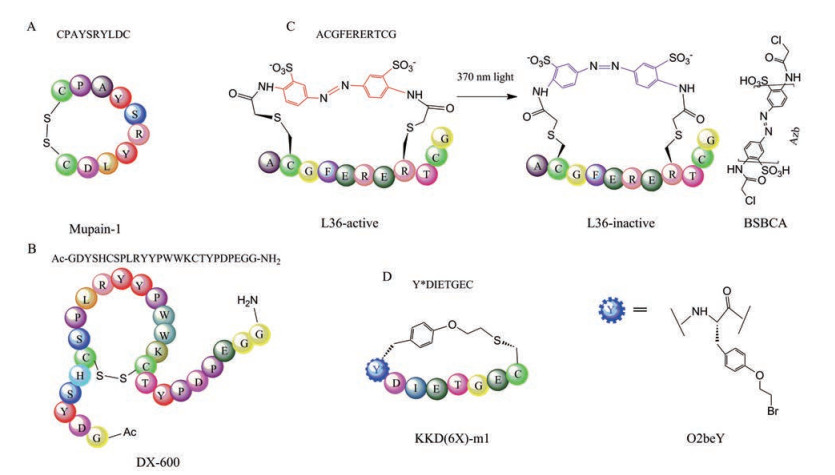
|
Figure 3 Schematic drawings of cases of phage-displayed monocyclic peptide. A: Mupain-1, obtained from the screening which is against murine uPA; B: DX600 targeting ACE2; C: Schematic diagram of the function of the "light-inhibited ligand" L36 obtained by phage display screening and key chemical modification reagents; D: Monocyclic peptide KKD(6X)-m1 with a non-natural amino acid screened against the keap1 |
针对血管紧张素转化酶2 (recombinant angiotensin I converting enzyme 2, ACE2) 蛋白的已有研究表明其与调节心脏功能相关。它虽然是羧肽酶ACE的同源物, 但其在血压调节中的作用仍不清楚。为研究ACE2在血压调节中的机制, Huang等[39]通过MANP即M13mp18型噬菌体载体的一种衍生物, 构建了6个通过一对二硫键构成的噬菌体展示环肽库并用于筛选ACE2抑制剂。此外, 同时他们还针对FLAG-ACE2淘筛了2个商业可获得的噬菌体线性肽库Ph.D.-7和Ph.D.-12, 但是线性肽库中并未筛得活性肽。相反在上述构建的6个噬菌体展示约束肽库的筛选中, 根据噬菌体ELISA阳性菌株的情况, 最终分析得到了10个共有基序, 根据这些基序及淘筛时出现频率, 共获得23个活性肽序列。其中, 序列Ac-GDYSHCSPLRYYPWW KCTYPDPEGG-NH2即DX600是活性最好的抑制剂(图 3B), 其Ki值为2.8 nmol·L-1, 既可以很好地抑制ACE2对合成底物M-2195的酶活性, 同样也可以很好地抑制ACE2对其天然底物十肽血管紧张素I (AngI) 的酶解活性, 此外DX600也具有较高的选择性, 对ACE并无抑制作用, 已成为目前最广泛应用的ACE2抑制剂。而同样有较强抑制作用的DX512 (Ac-GDRLHCKPQRQSPWMKCQHLDPEGG-NH2) 已完成了在自发性高血压大鼠中的研究, 具有明确的剂量依赖性的降压效应, 在3 mg·kg-1剂量水平下降压作用持续约6 min, 可从155 ± 10 mm Hg的平均动脉压降压至70.5 ± 4.6 mm Hg。这些通过筛选构象限制的噬菌体约束肽库所获得的高效的ACE2抑制剂, 有助于进一步揭示ACE2在血压调节中的作用机制。
尽管二硫键构建噬菌体单环肽库已有许多成功应用, 但是仍存在一定限制。主要由于二硫键结构尺寸小且具有柔韧性, 只是施加较弱的构象约束, 并且二硫键不稳定, 在细胞内环境还原条件下结构容易受到破坏。为了满足各种研究需求及筛选目的, 也开发了其他方式构建噬菌体单环肽库。Ratmir Derda小组通过携带光响应性偶氮苯(Azb) 化学结构的试剂来修饰在噬菌体上展示的线性肽序列, 从而构建了光响应型遗传编码文库[40]。使用商业的M13噬菌体单环文库ph.D-C7C, 利用固定化的三(2-羰基乙基) 磷盐酸盐iTCEP, 在不破坏pⅢ蛋白内部二硫键条件下, 选择性还原外源插入序列上的二硫键, 借助连接剂3, 3'-双(磺基)-4, 4'-双(氯乙酰胺基)-偶氮苯(BSBCA, 图 3C) 与游离巯基发生亲核取代反应实现对线性肽库的环化。针对模型底物链霉亲和素进行3轮亲和淘筛, 由于50%的环化产率, 80个克隆中经鉴定32个未成功掺入BSBCA, 最终通过ELISA鉴定获得了3个通过BSBCA连接剂修饰后显示结合的“光抑制性配体”, 即L36 (ACGFERERTCG, Kddark = 452 ± 63 μmol·L-1)、L42 (ACLSQRDGNCG, Kddark = 368 ± 103 μmol·L-1)、L57 (ACSVKLHTHCG, Kddark = 506 ± 89 μmol·L-1)。相比于从原始商业库筛的对照序列P81 (ACTSHQPRVCG, Kddark = 229 ± 21 μmol·L-1, Kdlight = 239 ± 41 μmol·L-1) 经BSBCA修饰后, 其光照前后Kd值未显著变化, 而“光抑制性配体”L36、L42和L57均在370 nm的光照射后, 发生顺式-反式的构型异构化, Kd值显著增大, 结合变弱。通过含有光响应型官能团的化学试剂修饰构建噬菌体展示单环肽库, 有望针对多种蛋白质靶标开展光响应型配体的筛选, 从而作为工具分子应用于光药理学和光化学遗传学研究。
Rudi Fasan研究小组通过在噬菌体展示体系中引入非天然氨基酸构建了稳定不被还原的硫醚键成环的噬菌体展示组合单环文库, 即MOrPH-PhD[41]。采用噬菌粒(pSEX81) 载体与辅助噬菌体的组合构建外源序列插入的完整M13型噬菌体展示, 利用引入非天然氨基酸O-(2-溴乙基)-酪氨酸(O2beY, 图 3D) 与序列中固定的半胱氨酸自由巯基发生亲核取代反应生成稳定硫醚键, 形成单环。根据这种成环技术成功构建多种组合文库, 并针对3种靶标链霉亲和素、Kelch样ECH相关蛋白1 (kelch like ECH-associated protein 1, Keap1)、Sonic Hedgehog (Shh) 均筛得高亲和力结合肽。尤其是针对keap1靶点, 平行筛选了O2beY-(Xaa)6-Cys形式的完全随机(NNK) 六聚体序列的MOrPH库以及包含Glu(X)nGlu基序的半理性随机组合肽库, 筛得了来自完全随机库的KD值为43 ± 14 nmol·L-1的KKD(6X)-m1 (Y*DIETGEC, 其中Y* = O2beY)、40 ± 13 nmol·L-1的KKD(6X)-m2 (Y*DAETGEC) 以及基于Glu/Glu半理性随机库的KKD-m1 (Y*DSETGEC), KD值为110 ± 8 nmol·L-1; 同样针对Shh筛选了O2beY-(Xaa)6-Cys形式的随机噬菌体单环肽库及基于围绕hedgehog相互作用蛋白(hedgehog interacting protein, HHIP) 中L2环的半理性设计噬菌体展示单环肽库, 其中, 来自于半理性设计噬菌体展示组合文库的最优序列Shh-m1 (GSSPY*EAMDMCTDTG), 其KD为0.55 ± 0.07 μmol·L-1。有趣的是, 不同于二硫键成环方式, 硫醚键成键方向即O2beY/Cys还是Cys/O2beY排列对于调节环肽结构对其结合或抑制能力具有一定的影响。
3.2 噬菌体展示订书肽库订书肽不同于上述这些约束环肽, 其通过引入刚性烃钉链基团, 稳定多肽的α-螺旋结构赋予约束肽更高的特异性、结合亲和力、良好的细胞穿透性以及延长的血浆半衰期。由于这些良好的特性, 特别是细胞穿透性, 订书肽在药物开发方面潜力巨大。Anananuchatkul等[42]采用pⅢ基因上无半胱氨酸的噬菌粒载体fdg3p0ss21成功构建16个氨基酸序列的噬菌体展示订书肽GAX1ECX2X3KEX4X5CKX6AG, 其中6个是随机肽序列以保证文库多样性可达6.4×107, 选择刚性联苯衍生物即4, 4-双(溴甲基) 联苯(BP) 与序列中固定的半胱氨酸发生取代反应形成环状结构, 稳定α-螺旋。针对肿瘤治疗与诊断相关的重要靶点半乳糖凝集素3 (galectin-3, Gal-3) 进行四轮筛选, 每轮投入量超过1.0×1010, 通过链霉亲和素磁珠捕获结合的噬菌体。为了筛选高亲和力配体, 筛选过程中加入竞争结合剂即过量的乳糖进行竞争洗脱, 同时提高筛选压力, 在第三、第四轮时, 将靶蛋白数目从1.0 μg降低至0.5 μg, 最终得到了有效的富集, 经测序及噬菌体ELISA共得到了5个荧光信号较强的序列。其中, 荧光标签修饰的p4-flu-BP (H-GAQECVYKEQSCKSAGGGGK-(flu)-NH2) 经荧光滴定测定(图 4), 发现对靶蛋白具有较高的结合力, Kd为0.45 ± 0.18 μmol·L-1, 此外, 经特异性实验验证其可特异性结合到Gal-3蛋白上。而不含烃钉试剂固定的p4-flu则没有明显的荧光变化, 可见, 刚性烃钉成环对于筛选高结合力配体具有重要作用。

|
Figure 4 Schematic drawing of a case of phage-displayed staple peptide |
不同于单环肽, 双环肽的构象更受限。针对一些复杂难靶的蛋白有更大的结合潜力和选择特异性。在1997年时, Klaus Mosbach小组[43]就已通过氧化形成二硫键手段成功构建M13噬菌体展示双环九肽库, 并筛选出α-胰凝乳蛋白酶的双环肽抑制剂。构建投入多达1012噬菌体颗粒用于筛选, 且采用了3种不同pH条件(pH 6.5, pH 7.0, pH 7.5), 在洗脱过程中还加入阳性抑制剂Bowman-Birk抑制剂进行竞争洗脱, 经过6轮迭代淘筛后, 最终每个pH下都淘筛获得了一些结合肽序。pH条件对于筛选的序列也有一定影响, 如pH 7.5时, 淘筛测序的序列在羧基末端均含有基序Arg-Cys-Arg-Cys, 且pH 6.5时, 大部分序列中均含有相似基序Leu-Trp, 对于所有在pH 6.5及7.0条件下, 淘筛测序的肽序列其第二或第三位置均为丙氨酸。鉴于α-胰凝乳蛋白酶的最适pH为7.8, 最终选择对pH 7.5条件下淘筛获得的肽序列进一步研究, 其中肽序列Cys-Cys-Phe-Ser-Trp-Arg-Cys-Arg-Cys抑制活性显著(图 5A), 在10 μmol·L-1浓度下即可实现对α-胰凝乳蛋白酶的完全抑制, 而非氧化形式则需提高50倍浓度才能实现几乎完全抑制的效果。此外, 该序列具有较高的特异性, 对牛胰蛋白酶和弹性蛋白酶(猪) 仅有微弱抑制作用, 而对苯甲基磺酰氟化物(PMSF) 修饰的α-糜蛋白酶的亲和力低于对天然α-糜蛋白酶的亲和力。
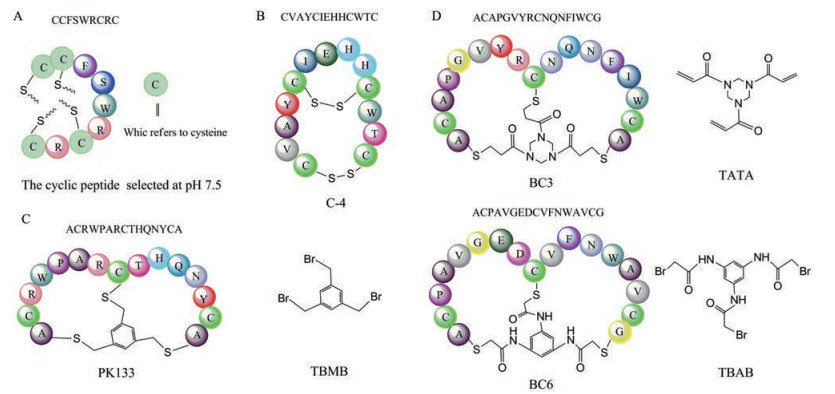
|
Figure 5 Schematic drawing of cases of phage-displayed bicyclic peptide. A: The dominant sequence screened for α-chymotrypsin at pH 7.5; B: C-4 sequence targeting PSA; C: The dominant sequence constructed by TBMB targeting the mouse PK; D: Schematic drawing of the dominant sequences from the TATA library and the TBAB library against β-catenin |
2000年, Wu等[44]针对前列腺特异性抗原(prostate specific antigen, PSA), 利用fUSE5噬菌体构建并筛选了4种不同的噬菌体展示约束肽库CX8C、CX10C、CX3CX3CX3C和CX3CX4CX2C。为避免直接包被可能会破坏PSA结构从而影响筛选结果, 他们通过单抗5E4将PSA固定在微量滴定孔中, 最终经过三轮迭代筛选, 共获得了14条结合肽序列, 并发现序列中含有CVAYC基序的肽与PSA具有较强结合。其中, 以融合蛋白GST-C-4序列CVAYCIEHHCWTC (图 5B) 结合能力最强, 可以显著增强PSA对发色底物s-2586的酶活性, KD值为2.9 μmol·L-1, 且具有高度选择特异性, 对糜蛋白酶、组织蛋白酶G、激肽释放酶、胰蛋白酶和hK2均无结合, 甚至对于PSA-α1-抗胰凝乳蛋白酶复合物PSA-ACT及PSA-α1蛋白酶抑制剂PSA-API同样没有结合。此外, 研究过程中还发现相比于低聚物, 多聚物形式的GST肽具有较强的结合力, 鉴于噬菌体fUSE5可同时表达3~5个拷贝的肽插入物, 说明该筛选肽通过高价结合增强了结合力, 但是, 并不表示单体肽毫无结合能力。总之, 通过噬菌体展示手段筛选到的具有4个半胱氨酸的肽序列GST-C-4是PSA高特异性的结合肽, 其环肽形式对于结合能力至关重要。
除去上述较为常见的通过二硫键手段形成双环肽之外, 还可以借助各种化学连接剂来构建多样性的噬菌体展示双环肽库。Christian Heinis小组及合作的bicycle公司一直致力于研究通过噬菌体展示技术构建各种多样性的环肽库, 从而针对各种靶点筛选有效的结合物。他们在通过化学试剂构建双环肽库上具有丰富的经验, 发表了一系列的文献及相应专利, 开发了许多高效的双环肽。所发现的环肽有一些已经进入临床研究(详见上述临床药物讨论部分)。针对人源血浆激肽释放酶(human plasma kallikrein, hPK) 筛选噬菌体展示双环肽库, 分离得到双环PK抑制剂PK15[45] (序列为: ACSDRFRNCPADEALCG), 其Ki值为2.9 ± 0.9 nmol·L-1, 同时具有高度特异性, 不会抑制与hPK高度同源的人源丝氨酸蛋白hfXIa及其他丝氨酸蛋白酶, 但由于高度特异性也导致其很难在动物模型中展开研究。通过构建AC-(Xaa)3-C-(Xaa)3-CA型的噬菌体展示线性肽, 随后用含有3个反应基团, 同时与半胱氨酸反应生成硫醚键的1, 3, 5-三(溴甲基) 苯(TBMB, 图 5C) 进行化学环化, 生成噬菌体展示双环肽库[46]。在仅经过2~3轮的迭代筛选, 即可获得一些共有序列, 虽然从这些3×3库筛选到的肽序列可以显著抑制大鼠源PK, 其Ki值在低纳摩尔范围内。但是由于对高度同源的人源fXIa也具有较强的抑制效果, 不利于进一步研究, 于是又在TBMB的环化作用下, 生成了5×5系列的噬菌体展示组合双环肽库AC-(Xaa)5-C-(Xaa)5-CA。成功筛得一批仅对人源PK及大鼠源PK等直系同源蛋白酶具有较高抑制活性的双环肽, 而对人源fXIa的活性仅为微摩尔量级。其中, PK133 (序列为ACRWPARCTHQNYCA) 抑制效果显著, 对人源PK的Ki仅为2.9 ± 1.2 nmol·L-1, 与PK15效果相当, 同时可以6.6 ± 2.3 nmol·L-1的Ki值抑制大鼠源PK, 此外, 在 > 100 μmol·L-1的高浓度下对人源fXIa仅有轻微抑制。
除了上述常用的TBMB反应模块外, 他们还开发了基于TBMB衍生的小分子反应模块, 优先考虑了易于与硫醇基团反应的丙烯酰胺和溴乙酰胺官能团, 成功构建了3个化学连接剂, 即1, 3, 5-三(丙烯酰基)-1, 3, 5-三嗪烷(TATA)、N, N′, N′′-苯-1, 3, 5-三基-三(2-溴乙酰胺) (TBAB) 和N, N′, N′′-苯-1, 3, 5-三基-三(丙-2-烯酰胺) (TAAB), 其中TATA与TBAB (图 5D) 成功应用于构建结构高度多样化的噬菌体展示双环肽库[47]。在靶向β-catenin抑制其作为转录激活因子的能力从而干扰Wnt信号通路研究中, 使用TBMB、TATA及TBAB成功构建了3种ACX6CX6CG形式的噬菌体展示双环肽库[48], 利用磁珠交替筛选的方法(1、3轮使用链霉亲和素磁珠, 而第2轮使用中性生物素涂层的磁珠) 针对已生物素化的β-catenin靶蛋白进行筛选, 并利用软件分析比对三类分离出的肽序列, 对应3种肽库, 分别获得12、5及4种不同的共有序列。其中共发现6个具有微摩尔Kd值的双环肽序列, 序列BC1 (ACAQKLDGCSY ISWSCG, Kd = 4.57 ± 0.57 μmol·L-1) 和BC2 (ACSGW WPKCQGYIPGCG, Kd = 3.82 ± 0.17 μmol·L-1) 均来自TBMB环化肽库, BC3 (ACAPGVYRCNQNFIWCG, Kd = 3.89± 0.32 μmol·L-1) 来自TATA环化肽库, 而BC4 (ACGNAFQDCLRDYSVCG, Kd = 25 μmol·L-1)、BC5 (ACFQVHEDCTWDYSLCG, Kd = 20 μmol·L-1) 和BC6 (ACPAVGEDCVFNWAVCG, Kd = 6.34 ± 0.72 μmol·L-1) 均来自TBAB环化库。在与axin的α-螺旋、β-catenin与TCF的抑制剂ICAT竞争结合β-catenin实验中, 发现BC1、BC4、BC5和BC6的结合被ICAT完全阻断, 这些双环肽可能与ICAT的扩展结合区域具有相似的结合表位。鉴于ICAT是β-catenin的天然抑制剂, 这些筛选获得的环肽在不干扰β-catenin其他活性功能下, 有望实现抑制β-catenin介导的转录。
3.4 噬菌体展示的多环肽库的未来发展通过二硫键成环的环肽库中, 一旦展示肽序列中出现2对以上半胱氨酸时, 就能形成富含二硫键的多环肽。由于二硫键的形成无方向性, 此类多环肽库很难预测其确定的环化方式, 吴川六等团队[49]致力于采用非天然氨基酸penicillamine (Pen) 对序列中的半胱氨酸做定位替换(图 6A), 在完成富含3对二硫键的多环肽库筛选后, 再对高亲和力序列进行二硫键替换设计, 从而得到结构明确且不易被还原的多环肽。这个研究方向对于筛选得到活性多环肽的后续开发有一定的意义。
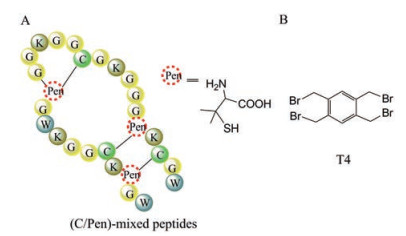
|
Figure 6 Examples of potential polycyclic peptides and useful reagents in the future. A: Schematic drawing of the construction of polycyclic peptides using non-natural amino acid penicillamine (Pen); B: T4 structure containing four cysteine reactive groups |
此外, 通过小分子化学反应模块成环方式中, 除了上述的单桥试剂构建的双环肽库, 还可以考虑同时与含有4个固定半胱氨酸的随机肽序列形成多元环, Timmerman等[50]研究发现了可通过T4 (图 6B) 即1, 2, 4, 5-四(溴甲基) 苯与含有4个半胱氨酸的线性肽进行环化从而生成三环肽, 若反应条件能兼顾不影响噬菌体感染复制能力, 则可以将这样的化学试剂成功应用于噬菌体展示线性肽的环化, 这将极大地丰富库的形式和筛选文库容量。
4 结论与展望在多肽化学合成领域, 已有丰富多样的成环方式和成键形式[51, 52], 如何在兼顾不影响噬菌体感染复制能力的情况下, 巧妙地将这些化学试剂应用于到噬菌体展示线性肽的环化, 丰富成环方式将极大地增强噬菌体展示环肽库在药物早期发现中的作用。期望在不久的将来有基于噬菌体展示环肽药物获批上市, 进一步推动该领域的发展。
作者贡献: 曹佳文负责综述的文献资料收集和主要内容撰写, 绘制图表; 曹丹燕负责综述的选题和内容框架, 撰写部分内容; 熊兵研究员对本文提出了重要的指导和修改意见。
利益冲突: 本文不涉及个人与组织利益冲突。
| [1] |
Dougherty PG, Sahni A, Pei D. Understanding cell penetration of cyclic peptides[J]. Chem Rev, 2019, 119: 10241-10287. DOI:10.1021/acs.chemrev.9b00008 |
| [2] |
Driggers EM, Hale SP, Lee J, et al. The exploration of macrocycles for drug discovery--an underexploited structural class[J]. Nat Rev Drug Discov, 2008, 7: 608-624. DOI:10.1038/nrd2590 |
| [3] |
Gang D, Kim D, Park HS. Cyclic peptides: promising scaffolds for biopharmaceuticals[J]. Genes, 2018, 9: 557. DOI:10.3390/genes9110557 |
| [4] |
Villar EA, Beglov D, Chennamadhavuni S, et al. How proteins bind macrocycles[J]. Nat Chem Biol, 2014, 10: 723-731. DOI:10.1038/nchembio.1584 |
| [5] |
Deyle K, Kong XD, Heinis C. Phage selection of cyclic peptides for application in research and drug development[J]. Acc Chem Res, 2017, 50: 1866-1874. DOI:10.1021/acs.accounts.7b00184 |
| [6] |
Zorzi A, Deyle K, Heinis C. Cyclic peptide therapeutics: past, present and future[J]. Curr Opin Chem Biol, 2017, 38: 24-29. DOI:10.1016/j.cbpa.2017.02.006 |
| [7] |
Jing X, Jin K. A gold mine for drug discovery: strategies to develop cyclic peptides into therapies[J]. Med Res Rev, 2019, 40: 753-810. |
| [8] |
Giordanetto F, Kihlberg J. Macrocyclic drugs and clinical candidates: what can medicinal chemists learn from their properties?[J]. J Med Chem, 2014, 57: 278-295. DOI:10.1021/jm400887j |
| [9] |
Smith G. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface[J]. Science, 1985, 228: 1315-1317. DOI:10.1126/science.4001944 |
| [10] |
Nixon AE, Sexton DJ, Ladner RC. Drugs derived from phage display: from candidate identification to clinical practice[J]. MAbs, 2014, 6: 73-85. DOI:10.4161/mabs.27240 |
| [11] |
Huang Y, Wiedmann MM, Suga H. RNA display methods for the discovery of bioactive macrocycles[J]. Chem Rev, 2019, 119: 10360-10391. DOI:10.1021/acs.chemrev.8b00430 |
| [12] |
Nemoto N, Miyamoto-Sato E, Husimi Y, et al. In vitro virus: bonding of mRNA bearing puromycin at the 3'-terminal end to the C-terminal end of its encoded protein on the ribosome in vitro[J]. FEBS Lett, 1997, 414: 405-408. DOI:10.1016/S0014-5793(97)01026-0 |
| [13] |
Tavassoli A. SICLOPPS cyclic peptide libraries in drug discovery[J]. Curr Opin Chem Biol, 2017, 38: 30-35. DOI:10.1016/j.cbpa.2017.02.016 |
| [14] |
Omidfar K, Daneshpour M. Advances in phage display technology for drug discovery[J]. Expert Opin Drug Dis, 2015, 10: 651-669. DOI:10.1517/17460441.2015.1037738 |
| [15] |
Tian M, Li S, Wang JJ, et al. Multimodal imaging quantitative analysis of geographic atrophy in aged-related macular degeneration[J]. Chin J Ocul Fundus Dis (中华眼底病杂志), 2017, 33: 580-583. |
| [16] |
Chang YS, Graves B, Guerlavais V, et al. Stapled α-helical peptide drug development: a potent dual inhibitor of MDM2 and MDMX for p53-dependent cancer therapy[J]. Proc Natl Acad Sci U S A, 2013, 110: E3445-3454. DOI:10.1073/pnas.1303002110 |
| [17] |
Renato D. Viruses with recombinant surface proteins: EP, 19830900590[P]. 1983-01-07.
|
| [18] |
McCafferty J, Griffiths AD, Winter G, et al. Phage antibodies: filamentous phage displaying antibody variable domains[J]. Nature, 1990, 348: 552-554. DOI:10.1038/348552a0 |
| [19] |
Heinis C, Rutherford T, Freund S, et al. Phage-encoded combinatorial chemical libraries based on bicyclic peptides[J]. Nat Chem Biol, 2009, 5: 502-507. DOI:10.1038/nchembio.184 |
| [20] |
Luzi S, Kondo Y, Bernard E, et al. Subunit disassembly and inhibition of TNFα by a semi-synthetic bicyclic peptide[J]. PEDS, 2015, 28: 45-52. |
| [21] |
Wu R. Recombinant DNA Methodology[M]. San Diego: Academic Press, 1989: 225-233.
|
| [22] |
Hamzeh-Mivehroud M, Alizadeh AA, Morris MB, et al. Phage display as a technology delivering on the promise of peptide drug discovery[J]. Drug Discov Today, 2013, 18: 1144-1157. DOI:10.1016/j.drudis.2013.09.001 |
| [23] |
Qi H, Lu H, Qiu HJ, et al. Phagemid vectors for phage display: properties, characteristics and construction[J]. J Mol Biol, 2012, 417: 129-143. DOI:10.1016/j.jmb.2012.01.038 |
| [24] |
Barbas CF Ⅲ, Burton DR, Scott JK, et al. Phage Display: A Laboratory Manual[M]. New York: Cold Spring Harbor Laboratory Press, 2001: 4.2-4.5.
|
| [25] |
Sidhu SS. Phage Display in Biotechnology and Drug Discovery[M]. Boca Raton: CRC Press, 2015: 111-142.
|
| [26] |
Smith GP, Petrenko VA. Phage display[J]. Chem Rev, 1997, 97: 391-410. DOI:10.1021/cr960065d |
| [27] |
Deramchia K, Jacobin-Valat MJ, Vallet A, et al. In vivo phage display to identify new human antibody fragments homing to atherosclerotic endothelial and subendothelial tissues[J]. Am J Pathol, 2012, 180: 2576-2589. DOI:10.1016/j.ajpath.2012.02.013 |
| [28] |
Sánchez-Martín D, Martínez-Torrecuadrada J, Teesalu T, et al. Proteasome activator complex PA28 identified as an accessible target in prostate cancer by in vivo selection of human antibodies[J]. Proc Natl Acad Sci U S A, 2013, 110: 13791-13796. DOI:10.1073/pnas.1300013110 |
| [29] |
Dias-Neto E, Nunes D, Giordano R, et al. Next-generation phage display: integrating and comparing available molecular tools to enable cost-effective high-throughput analysis[J]. PLoS One, 2009, 4: e8338. DOI:10.1371/journal.pone.0008338 |
| [30] |
Rentero Rebollo I, Sabisz M, Baeriswyl V, et al. Identification of target-binding peptide motifs by high-throughput sequencing of phage-selected peptides[J]. Nucleic Acids Res, 2014, 42: e169. DOI:10.1093/nar/gku940 |
| [31] |
Villequey C, Kong XD, Heinis C. Bypassing bacterial infection in phage display by sequencing DNA released from phage particles[J]. PEDS, 2017, 30: 761-768. |
| [32] |
Rentero Rebollo I, Heinis C. Phage selection of bicyclic peptides[J]. Methods, 2013, 60: 46-54. DOI:10.1016/j.ymeth.2012.12.008 |
| [33] |
Nixon AE. Therapeutic Peptides[M]. Totowa New Jersey: Humana Press, 2014: 67-79.
|
| [34] |
Zhang X, Zhan JB. Screening method and application of phage display peptide library[J]. Fujian Med J (福建医药杂志), 2004, 26: 153-156. |
| [35] |
O'Neil KT, Hoess RH, Jackson S, et al. Identification of novel peptide antagonists for GPⅡb/Ⅲa from a conformationally constrained phage peptide library[J]. Proteins, 1992, 14: 509-515. DOI:10.1002/prot.340140411 |
| [36] |
McLafferty MA, Kent RB, Ladner RC, et al. M13 bacteriophage displaying disulfide-constrained microproteins[J]. Gene, 1993, 128: 29-36. DOI:10.1016/0378-1119(93)90149-W |
| [37] |
Xu P, Andreasen PA, Huang M. Structural principles in the development of cyclic peptidic enzyme inhibitors[J]. Int J Biol Sci, 2017, 13: 1222-1233. DOI:10.7150/ijbs.21597 |
| [38] |
Andersen LM, Wind T, Hansen HD, et al. A cyclic peptidylic inhibitor of murine urokinase-type plasminogen activator: changing species specificity by substitution of a single residue[J]. Biochem J, 2008, 412: 447-457. DOI:10.1042/BJ20071646 |
| [39] |
Huang LL, Sexton DJ, Skogerson K, et al. Novel peptide inhibitors of angiotensin-converting enzyme 2[J]. J Biol Chem, 2003, 278: 15532-15540. DOI:10.1074/jbc.M212934200 |
| [40] |
Jafari MR, Deng L, Kitov PI, et al. Discovery of light-responsive ligands through screening of a light-responsive genetically encoded library[J]. ACS Chem Biol, 2014, 9: 443-450. DOI:10.1021/cb4006722 |
| [41] |
Owens AE, Iannuzzelli JA, Gu Y, et al. MOrPH-PhD: an integrated phage display platform for the discovery of functional genetically encoded peptide macrocycles[J]. ACS Cent Sci, 2020, 6: 368-381. DOI:10.1021/acscentsci.9b00927 |
| [42] |
Anananuchatkul T, Chang IV, Miki T, et al. Construction of a stapled α-helix peptide library displayed on phage for the screening of galectin-3-binding peptide ligands[J]. ACS Omega, 2020, 5: 5666-5674. DOI:10.1021/acsomega.9b03461 |
| [43] |
Krook M, Lindbladh C, Eriksen JA, et al. Selection of a cyclic nonapeptide inhibitor to α-chymotrypsin using a phage display peptide library[J]. Mol Divers, 1997, 3: 149-159. DOI:10.1023/A:1009697515328 |
| [44] |
Wu P, Leinonen J, Koivunen E, et al. Identification of novel prostate-specific antigen-binding peptides modulating its enzyme activity[J]. Eur J Biochem, 2010, 267: 6212-6220. |
| [45] |
Heinis C, Rutherford T, Freund S, et al. Phage-encoded combinatorial chemical libraries based on bicyclic peptides[J]. Nat Chem Biol, 2009, 5: 502-507. DOI:10.1038/nchembio.184 |
| [46] |
Baeriswyl V, Rapley H, Pollaro L, et al. Bicyclic peptides with optimized ring size inhibit human plasma kallikrein and its orthologues while sparing paralogous proteases[J]. ChemMedChem, 2012, 7: 1173-1176. DOI:10.1002/cmdc.201200071 |
| [47] |
Chen S, Morales-Sanfrutos J, Angelini A, et al. Structurally diverse cyclisation linkers impose different backbone conformations in bicyclic peptides[J]. ChemBioChem, 2012, 13: 1032-1038. DOI:10.1002/cbic.201200049 |
| [48] |
Bertoldo D, Khan MMG, Dessen P, et al. Phage selection of peptide macrocycles against β-catenin to interfere with Wnt signaling[J]. ChemMedChem, 2016, 11: 834-839. DOI:10.1002/cmdc.201500557 |
| [49] |
Zheng Y, Meng X, Wu Y, et al. De novo design of constrained and sequence-independent peptide scaffolds with topologically-formidable disulfide connectivities[J]. Chem Sci, 2018, 9: 569-575. DOI:10.1039/C7SC03956E |
| [50] |
Timmerman P, Beld J, Puijk WC, et al. Rapid and quantitative cyclization of multiple peptide loops onto synthetic scaffolds for structural mimicry of protein surfaces[J]. ChemBioChem, 2005, 6: 821-824. DOI:10.1002/cbic.200400374 |
| [51] |
Rhodes CA, Pei D. Bicyclic peptides as next-generation therapeutics[J]. Chemistry, 2017, 23: 12690-12703. DOI:10.1002/chem.201702117 |
| [52] |
Jing X, Jin K. A gold mine for drug discovery: strategies to develop cyclic peptides into therapies[J]. Med Res Rev, 2020, 40: 753-810. DOI:10.1002/med.21639 |
 2021, Vol. 56
2021, Vol. 56


