2. 枣庄学院信息科学与工程学院, 山东 枣庄 277160;
3. 山东大学齐鲁医学院药学院, 山东 济南 250012;
4. 山东中医药大学药学院, 山东 济南 250355
2. College of Information Science and Engineering, Zaozhuang University, Zaozhuang 277160, China;
3. School of Pharmaceutical Sciences, CheeLoo College of Medicine, Shandong University, Jinan 250012, China;
4. College of Pharmaceutical Science, Shandong University of Traditional Chinese Medicine, Jinan 250355, China
急性肺损伤(acute lung injury, ALI)是一种严重的呼吸系统疾病, 可导致急性呼吸衰竭或死亡, 其特征是急性呼吸窘迫(acute respiratory distress syndrome, ARDS)、难治性低氧血症和非心源性肺水肿[1]。急性肺损伤的发病机制非常复杂, 已经发现的可能因素有: 炎症反应(炎症瀑布)作用[2]、氧化应激系统失衡[3]、肺泡毛细血管受损和上皮细胞损伤[4]等。ALI是许多疾病共同的病理反应, 新型冠状病毒肺炎(COVID-19)的发病机制与ALI的发生发展息息相关[5], 而且, 采用中医药对其干预治疗有较好的效果。从COVID-19的中医药治疗效果看, 从轻症、重症/危重症到康复期, 中医药全过程起效, 彰显其独特的优势和作用。
中医认为, “热、毒、痰、瘀”贯穿急性肺损伤的始终, “热、毒”为其的发病基础, “痰、瘀”为致病因素[6]。所以, 治疗上可辨证应用“清热、解毒、祛痰、化瘀”等方法, 用药主要涉及清热解毒、活血化瘀、益气扶正等中药及方剂。二黄汤是传统清热解毒方剂, 由黄芩、黄连、甘草组成, 记载于清·《医宗金鉴》中, “治上焦火旺, 头面大肿, 目赤肿痛, 心胸咽喉口舌耳鼻热盛及生疮毒者”[7]。而且, 文献报道显示, 黄芩中的黄芩苷[8]、黄芩素[9]、黄连中小檗碱[10]、甘草中的甘草酸[11]、甘草次酸[12]等成分均表现出治疗ALI的效果, 涉及到抗炎、抗氧化、抑制细胞凋亡和调节免疫等机制。
但是, 以上研究多以单体成分进行单个靶点的实验研究, 在一定程度上阐释了治疗ALI的作用机制, 但还不全面、不系统, 缺少多成分、多靶点、多通路整体观念。本文采用中药复方网络药理学方法[13, 14], 以入血成分为研究对象, 全面预测二黄汤治疗ALI的多靶点、多通路、多途径的机制, 为ALI的治疗和研究提供思路, 为二黄汤进一步开发利用奠定基础。本研究思路流程图见图 1。
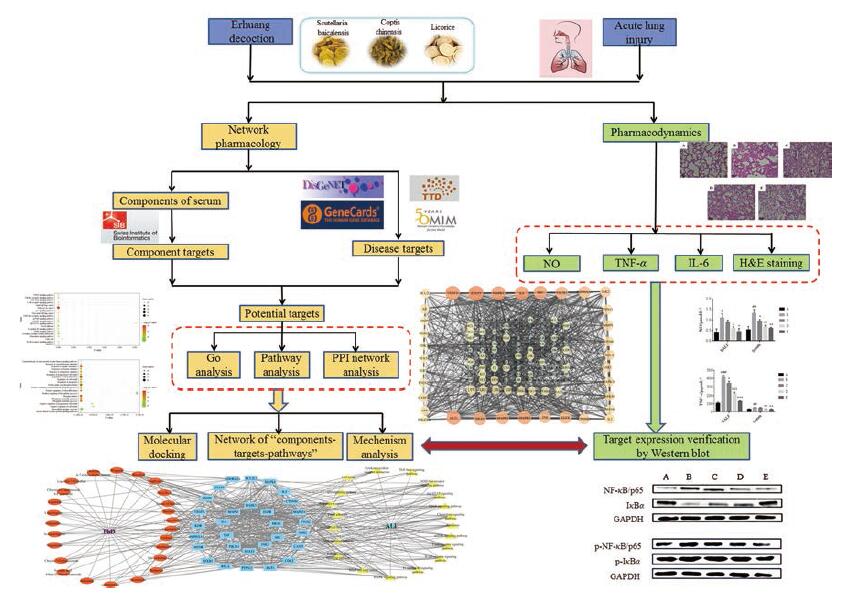
|
Figure 1 Flow chart of research |
药品与试剂黄芩、黄连、甘草(批号: 200201、200201、200301, 济南众和中药饮片有限公司), 经鉴定为黄芩Scutellaria baicalensis Georgi、黄连Coptis chinensis Franch.、甘草Glycyrrhiza uralensis Fisch.加工成的饮片, 且符合2015年版《中国药典》相关规定; 脂多糖(lipopolysacchatide, LPS)(大肠杆菌055:B5)(批号: 0000081275, 美国Sigma-Aldrich公司)。一氧化氮(nitric oxide, NO)微板检测试剂盒(批号: 20190508, 南京建成生物工程研究所); 小鼠肿瘤坏死因子α(tumor necrosis factorα, TNF-α)、小鼠白介素6 (interleukin 6, IL6) ELISA试剂盒(批号: A28200722、A2060064370, 上海联科生物科技有限公司)。NF-κB/p65、IκBα的抗体(批号: 4764S、4812S, Cell Signaling Technology公司)。甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase, GAPDH)(批号: GR208234-1, Protein-Tech Group公司)。二级抗体(批号: 7074p2, Cell Signaling Technology公司), 稀释使用(1∶3 000)。取黄芩、黄连、甘草饮片各等分, 按照参考文献[15]中的方法提取浓缩, 制备二黄汤(EhD)灌胃液(质量浓度1 g·mL-1)。
动物 BALB/c小鼠(8~10周), 雄性, 体重18~22 g, 购自山东大学实验动物中心, 合格证号: SCXK(鲁) 20190003。动物实验伦理要求已获得山东大学实验动物中心伦理委员会的批准(批准号: 2016020)。
主要仪器 高速低温离心机Sigma3-18k (德国Sigma公司); 超纯水仪Milli-Q (美国Millipore公司); 酶标仪Spectra Max M3 Plus (美国MD公司); 显微镜DM2500 (德国Leica公司); 凝胶成像系统Alpha Imager EP (美国Protein Simple公司); 切片机SYD-S3020 (沈阳誉德电子仪器有限公司)。
潜在活性成分收集 根据前期二黄汤血清药物化学实验结果, 共鉴定出入血成分23个原型成分和2个代谢产物[16]。以此25个入血成分为研究对象, 通过Chem Bio Draw Ultra12.0软件, 绘制结构化学结构。见表 1。
| 表 1 Information sheet of chemical compounds. * indicates metabolites |
靶点的预测与筛选 将25个活性成分变换格式导入Swiss Target Prediction数据库[17](http://www.swisstargetprediction.ch/)进行靶点预测, 得到入血成分作用靶点。然后, 将靶点信息输入Uni Prot数据库(https://www.uniprot.org/)规范靶点名称。在DisGeNET (http://www.disgenet.org/)、TTD (http://db.idrblab.net/ttd/)、Gene Cards (https://www.genecards.org/)、OMIM (http://omim.org/)数据库中检索关键词“acute lung injury”, 汇总整理疾病靶点, 并与入血成分作用靶点整合取交集, 获得EhD治疗ALI的潜在靶点。
蛋白互作(PPI)网络的构建 ALI的发病分子机制较复杂, 与疾病相关的靶蛋白往往存在直接的物理相互作用或间接的功能关联, 从而影响疾病的发生发展。为了更好地分析靶点蛋白间的相互作用, 将潜在作用靶点导入String数据库, 物种选择“Human”, 选择0.7的高置信度, 以确保数据的可靠性。保存结果并导入Cytoscape 3.6.1软件构建PPI网络, 对其网络进行分析, 并将节点(node)大小和颜色设置用于反映度值(degree)的大小, 边(edge)的粗细设置用于反映结合分数的大小。
KEGG通路分析与GO分类富集分析 将潜在作用靶点导入DAVID 6.8数据库(https://david.ncifcrf.gov/), 物种选择“Human”, 进行GO分析和KEGG通路分析(P < 0.05), 分析潜在靶点聚类于ALI相关的生物学过程或信号通路。
“成分-靶点-通路”网络构建 将EhD中鉴定出的入血成分、治疗ALI的潜在靶点、作用通路分析结果导入Cytoscape 3.6.1软件, 构建“成分-靶点-通路”网络图。
分子对接虚拟计算 采用Chem Bio Draw Ultra12.0软件, 将25个入血成分和阳性对照药地塞米松(DXMS)[18]转化为“.mol2”文件, 并进行能量最小化处理。从RCSB PDB蛋白质结构数据库得到筛选关键靶蛋白三维结构, 并去除非结合位点水分子, 保存为pdb格式。登录http://www.swissdock.ch/docking网站[19, 20], 分别导入相应格式成分和靶蛋白, 点击“Start Docking”开始分子对接实验, 预测对接结合能[EstimatedΔG(kcal·mol-1)]。根据结合能分值排序, 验证靶蛋白与小分子结合能力。
小鼠ALI模型建立与药效学实验 取BALB/c小鼠30只, 随机分组, 每组6只, 适应性饲养1周后, 设立空白组、模型组、EhD低剂量组(1 g·kg-1)、EhD中剂量组(2.5 g·kg-1)、EhD高剂量组(5 g·kg-1)。每日给药1次, 连续7天, 空白组和模型组给予相同体积生理盐水; 除空白组外, 其他各组于第7天给药前1 h气管注射LPS磷酸盐缓冲液(5 mg·kg-1), 造成小鼠急性肺损伤模型。
造模后24 h后, 麻醉, 眼眶采集全血, 3 500 r·min-1离心10 min, 上清液测定NO、TNF-α和IL-1β。处死实验动物, 同时行右肺结扎, 立即用0.8 mL预冷生理盐水以三进三出的主支气管灌注方式灌洗左肺, 记录收集灌洗液(bronchoalveolar lavage fluid, BALF)的总体积, 并分离无细胞上清液, 采用ELISA法测定肺灌洗液中IL6、TNF-α和NO含量。肺组织用4%甲醛磷酸盐缓冲液处理过夜, 脱水石蜡包埋, 切片4μm, H & E染色, 光学显微下观察肺组织形态学变化。
Western blot法靶点表达验证 肺组织加入放射免疫沉淀分析缓冲液, 匀浆处理, 提取蛋白, 采用双辛酸分析试剂盒测定总蛋白含量。在8%~12%十二烷基硫酸钠–聚丙烯酰胺凝胶上分离等量蛋白质并转移到聚偏氟乙烯膜上, 进行封闭等操作, 采用ECL底物化学发光显色, 显影, 定影, 扫描。将膜在4℃下与一抗孵育过夜, 然后在室温下与辣根过氧化物酶(HRP)结合的抗兔或抗鼠抗体孵育1 h。用增强化学发光检测器测量光密度。利用Alpha View-SA软件对各波段的相对光密度进行定量分析。
统计学分析 数据均以“均数±标准差”表示, 利用SPSS 20.0软件进行统计分析。
结果 1 靶点的预测与筛选EhD入血成分导入Swiss Target Prediction数据库预测整合得到637个靶点。检索DisGeNET、TTD、Gene Cards、OMIM数据库, 得到与ALI有关的疾病靶基因1 057个。Venn软件作图取交集, 得到148个二黄汤治疗ALI的潜在靶点。
2 蛋白互作(PPI)网络的构建潜在靶点的蛋白质相互作用PPI网路中共有142个节点(靶点蛋白)、1 057条边(蛋白相互作用), 见图 2。节点大小和颜色表示该节点度值的大小, 节点越大、橙色越深, 对应的度值越大, 表明在所预测的疾病相关靶点中, 能与该靶点产生有效互相作用的靶点越多。边的粗细表示结合分数, 边越粗结合分数值越大, 表明相关靶点相互作用置信度越高。其中, 信号传导转录激活因子3 (STAT3, 58)、血管内皮生长因子A(VEGFA, 54)、丝氨酸/苏氨酸蛋白激酶1 (AKT1, 51)、丝裂原活化蛋白激酶1 (MAPK1, 53)、白介素6 (IL6, 52)度值较大, 可能是关键靶点。另外, 肿瘤坏死因子(TNF, 41)、核转录因子-κB/p65 (RELA, 31)、前列腺素内过氧化物合酶2 (prostaglandin g/h synthase 2, PTGS2, 24)、凋亡抑制基因(B cell lymphoma/lewkmia-2-like protein 1, BCL2L1, 25)、一氧化氮合酶2 (nitric oxide synthase 2, NOS2, 12), 也可能是有效靶点。
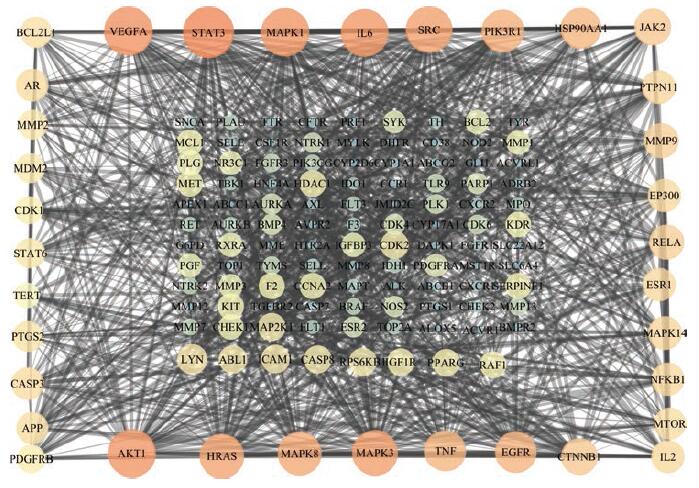
|
Figure 2 Protein-protein interaction (PPI) network of potential targets for Erhuang decoction (EhD) treatment of acute lung injury (ALI) |
通过DAVID 6.8数据平台对二黄汤治疗ALI的潜在靶点进行KEGG通路富集分析(P < 0.05)绘制气泡图, 见图 3和表 2。KEGG分析主要涉及疾病通路、炎症通路、氧化和凋亡通路相关通路等。疾病通路主要包括癌症通路(pathways in cancer)、非小细胞肺癌(non-small cell lung cancer)、小细胞肺癌(small cell lung cancer)等通路; 与炎症相关通路主要包括VEGF信号通路(VEGF signaling pathway)、MAPK信号通路(MAPK signaling pathway)、TLRs信号通路(TLRs signaling pathway)、趋化因子信号通路(chemokine signaling pathway)、细胞因子-细胞因子-受体相互作用(cytokine-cytokine receptor interaction)等通路; 免疫应激有关的通路有B细胞受体信号通路(B cell receptor signaling pathway)、T细胞受体信号通路(T cell receptor signaling pathway); 凋亡相关的通路有肿瘤蛋白53(p53)信号通路(tumor protein 53 signaling pathway)、转化生长因子β(TGF-β)信号通路(transforming growth factor-β signaling pathway)、受体酪氨酸激酶-信号转导和转录激活子(Jak-STAT)信号通路(Janus kinase/signal transducer and activator of transcription signaling pathway); 其中, 雷帕霉素靶蛋白(mTOR)信号通路(mammalian target of rapamycin signaling pathway)还有调节物质代谢的作用。
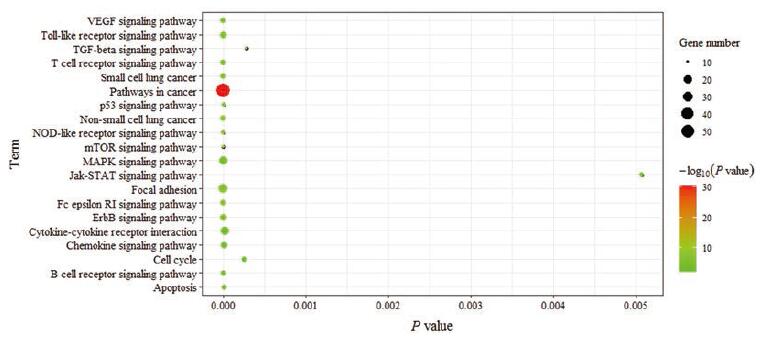
|
Figure 3 Pathways enrichment analysis diagram |
| 表 2 Enrich KEGG pathways analysis of potential targets for EhD for treatment of ALI |
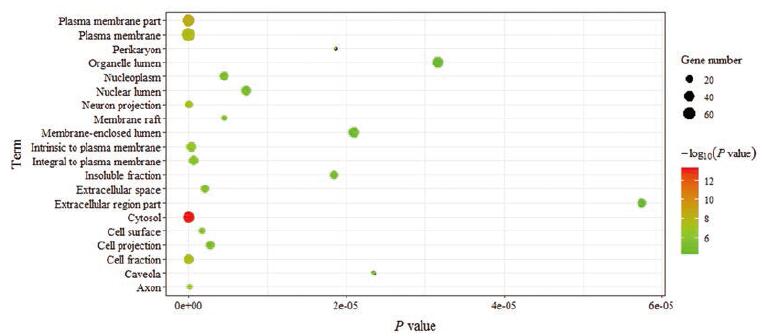
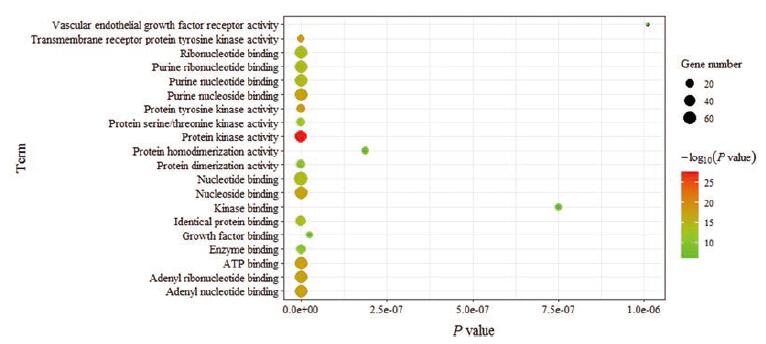
GO分析结果按P值排序选取前20绘制气泡图。图 4显示, 生物过程中涉及跨膜受体蛋白酪氨酸激酶信号、细胞内信号级联、酶联受体蛋白信号途径、细胞增殖与凋亡信号通路以及磷酸化、大分子代谢过程等代谢通路调控(P < 0.05);图 5显示, 细胞组分涉及细胞膜、细胞核和胞外间隙等多个方面(P < 0.05);图 6显示, 分子功能集中于血管内皮生长因子受体活性、跨膜受体蛋白酪氨酸激酶活性、蛋白质二聚活性、核苷酸活动、腺苷酸活动及ATP活动等多个过程(P < 0.05)。
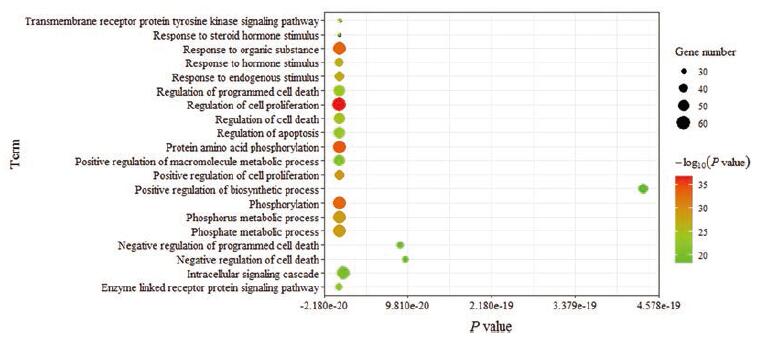
|
Figure 4 Biological process enrichment analysis diagram |
|
Figure 5 Cellular component enrichment analysis diagram |
|
Figure 6 Molecular function enrichment analysis diagram |
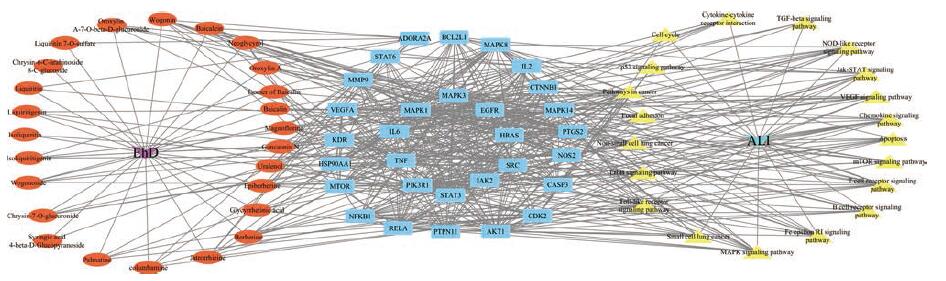
由于预测出的潜在靶点较多, 在采用Cytoscape3.6.1软件构建网络时, 筛选PPI蛋白互作度值排名靠前的30个靶点参与“成分-靶点-通路”网络构建, 见图 7。该网络有77个节点(包括1个中药复方、25个入血成分、30个靶点、20条通路和1种疾病)和575条边。度值越大, 代表与该节点相连的其他节点越多, 证明该节点在网络中的贡献率较大。乌拉尔醇(uralenol, 11)、木兰碱(magnoflorine, 11)、黄芩素(baicalein, 9)、汉黄芩素(wogonin, 9)、小檗碱(berberine, 8)、药根碱(jatrorrhizine, 7)、甘草次酸(glycyrrhetinic acid, 6)、巴马汀(palmatine, 5)、异甘草素(isoliquiritigenin, 5)、黄芩苷(baicalin, 5)等成分度值较大, 均与多个靶点连接。度值较大的靶点包括丝氨酸/苏氨酸蛋白激酶1(AKT1, 39)、丝裂原活化蛋白激酶3 (MAPK3, 40)、丝裂原活化蛋白激酶1 (MAPK1, 37)、信号传导转录激活因子3 (STAT3, 32)、血管内皮生长因子A (VEGFA, 26)、白介素6 (IL6, 29)、核转录因子p65亚基(NF-κB/p65)(RELA, 28)、前列腺素内过氧化物合酶2 (PTGS2, 28)、凋亡调节因子(BCL2L1, 22)、诱导型一氧化氮合酶2 (NOS2, 19)等。对应度值较大的通路除癌症通路(22)外, 还涉及了MAPK信号通路(12)、Toll样受体信号通路(11)、VEGF信号通路(11)、NOD样受体(NLRs)信号通路(NOD-like receptor signaling pathway, 11)、趋化因子信号通路(10)和Jak-STAT信号通路(10)。
|
Figure 7 Network of drug ingredients-targets-pathways |
将PPI网络中排名靠前的靶点蛋白, 分别与DXMS和入血成分进行分子对接验证, 结果见表 3。根据ALI的疾病特点, 靶点蛋白选取时, 分别选取了影响炎症、细胞凋亡和氧化应激3个方面的靶蛋白, 参与分子对接。分子对接结合能越小, 表明其结合性越好。表中96%入血成分都能与所选的靶点蛋白较好地结合。25个入血成分与MAPK3、STAT3、IL6、RELA (NF-κB/p65)、BCL2L1和NOS2靶蛋白的平均结合性能优于对照药DXMS, 与VEGFA靶蛋白结合能力稍低于DXMS。以上结果表明, 二黄汤入血成分能较好地与ALI相关靶点对接, 潜在生物活性高。
| 表 3 Molecular docking result of absorbed ingredients of EhD in blood. "-" indicates that the molecular docking is not successful |
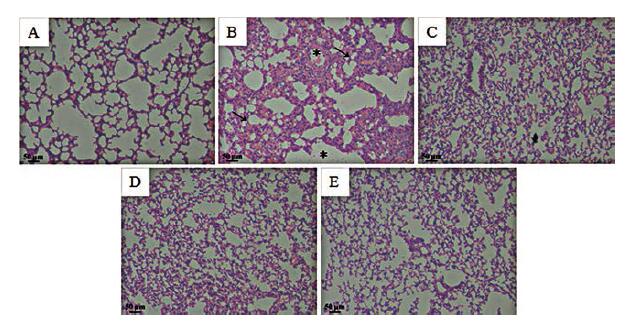
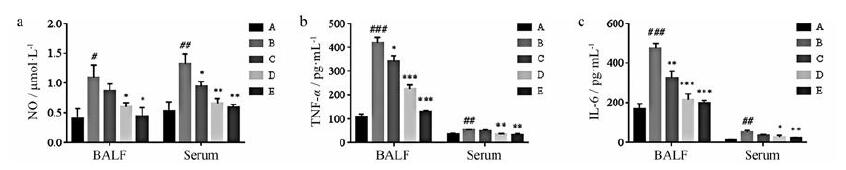
脂多糖(LPS) 5 mg·kg-1, 肺部滴入给药的方式, 建立LPS诱导的小鼠急性肺损伤模型。与空白组比较, 模型组的H & E染色病理切片中肺泡结构改变, 肺泡壁增厚, 炎症因子明显增多(图 8); 模型组血清和肺组织灌洗液中IL6、TNF-α和NO的含量显著升高(图 9), 造模成功。
|
Figure 8 Effects of EhD on lung histological changes of lipopolysacchatide (LPS)-induced ALI mice (H & E staining magnification 100×). A: Control; B: LPS; C: LPS + 1.0 g·kg-1 EhD; D: LPS + 2.5 g·kg-1 EhD; E: LPS + 5.0 g·kg-1 EhD. Asterisks denote fibrillar to granular eosinophilic protein strands. Solid arrows denote thickened alveolar wall and interstitial neutrophils infiltrating in the intercellular spaces |
|
Figure 9 Effects of EhD on the secretion of pro-inflammatory cytokines in bronchoalveolar lavage fluid (BALF) and serum. A: Control; B: LPS; C: LPS + 1.0 g·kg-1 EhD; D: LPS + 2.5 g·kg-1 EhD; E: LPS + 5.0 g·kg-1 EhD. Quantitative densitometric analyses of NO (a), TNF-α (b), IL6 (c). n = 6, mean ± SEM. #P < 0.05, ##P < 0.01, ###P < 0.001 vs control group; *P < 0.05, **P < 0.01, ***P < 0.001 vs LPS group |
与模型组比较, EhD干预给药治疗可明显改善肺组织形态, 降低炎症细胞浸润数目(图 8); 同时, 与模型组比较, 给药组的动物血清和肺组织灌洗液中IL6、TNF-α和NO含量显著降低, 而且, 低、中、高剂量组干预效果呈现明显的量-效相关(图 9)。
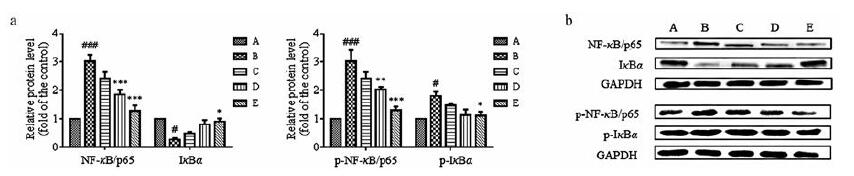
6.3 Western blot法验证EhD调控NF-κB/p65和IκBα蛋白表达ALI的重要临床表现是肺部炎症。在炎症进程中, NF-κB的活化与表达发挥了关键作用[21]。根据网络预测结果, RELA基因在构建的成分-靶点-通路网络中, 度值较大, 是核转录因子p65调控基因, 控制着NF-κB/p65蛋白的表达。所以, 本实验以NF-κB/p65和IκBα蛋白表达, 对网络药理学预测靶点进行验证(图 10)。结果显示, 与模型组相比, 二黄汤干预能显著降低NF-κB/p65、p-NF-κB/p65蛋白的表达, 提高IκBα蛋白的表达, 降低p-IκBα蛋白表达。而且, 与给药剂量呈现一定的量-效关系。
|
Figure 10 Effects of EhD on LPS-induced protein expressions of NF-κB/p65 and IκBα in the ALI mice. a: Quantitative densitometric analyses of NF-κB/p65, IκBα, p-NF-κB/p65, and p-IκBα; b: Western blot analyses of NF-κB/p65, IκBα, p-NF-κB/p65, and p-IκBα; normalized against glyceraldehyde-3-phosphate dehydrogenase (GAPDH). A: Control; B: LPS; C: LPS + 1.0 g·kg-1 EhD; D: LPS + 2.5 g·kg-1 EhD; E: LPS + 5.0 g·kg-1 EhD. n= 3, mean ± SEM. #P < 0.05, ###P < 0.001 vs control group; *P < 0.05, **P < 0.01, ***P < 0.001 vs LPS group |
ALI是包括新冠病毒在内的很多疾病的共同病理过程, 是一种临床常见的危重症, 死亡率较高。本文通过网络药理学对二黄汤治疗急性肺损伤的作用机制进行了预测分析, 发现相关靶点和通路涉及到了炎症反应、氧化应激和细胞凋亡等环节。
PPI网络显示, 靶点之间存在着多种关联, 连接度值越高, 靶点的潜在治疗作用越大。STAT3、VEGFA、AKT1、MAPK1、MAPK3和IL6排名靠前, 可能是关键靶点。STAT3是STAT家族重要成员, 可调控细胞增生、存活、凋亡和炎症等过程。甘草酸可以通过降低STAT3活性, 减少角叉菜胶引起的ALI的炎症反应[22]。MAPK家族包括调节激酶(ERK)、p38丝裂原活化蛋白激酶(p38MAKP)、c-Jun氨基末端激酶(JNK)和ERK5亚族, ERK1和ERK2 (也称为MAPK3和/MAPK1)信号转导通路调控细胞生长和分化, p38MAPK和JNKMAPK信号转导通路在炎症与细胞凋亡等应激反应中发挥重要作用。研究发现, LPS诱导大鼠发生ARDS时, 细胞质中p38MAKP和细胞外ERK蛋白明显上调, 同时p-p38MAPK和p-p ERK的蛋白表达水平明显增加[23]。黄芩素可增加p38、ERK1和ERK2的蛋白表达水平, 降低斑马鱼幼鱼的急性肝炎肝毒性损伤[24]。汉黄芩素通过阻断p38MAPK和JNK的磷酸化, 下调诱导型一氧化氮合成酶(inducible nitric oxide synthas, i NOS)和环氧化酶-2 (cyclooxygenase-2, COX-2)的表达, 抑制肺水肿和蛋白质渗漏, 对LPS诱导的ALI具有潜在的保护作用[25]。IL6基因可调控ALI等多种炎症疾病, 其编码的IL6是调节宿主免疫应答、炎症、血细胞生成和癌发生的多效细胞因子。黄芩苷通过刺激调节性T细胞(regulatory cell, Treg)和抑制IL6、白介素-23 (inter-leukin-23)的释放, 进而导致辅助性T细胞17 (T helper cell 17, Th17)细胞减少, 从而影响Th17与Treg反应的免疫平衡[26], 起到调控ALI炎症反应的作用。
VEGF在ALI中发挥双重作用, 它既参与早期急性肺水肿的形成, 又能促进恢复期肺泡上皮细胞的存活[27]。VEGFA主要在肺泡上皮细胞和支气管上皮细胞表达, ALI/ARDS急性期的肺泡上皮细胞和血管内皮细胞受损, 呼吸道上皮细胞中的VEGFA向血液中释放, 与血管内皮细胞的VEGF受体结合, 增加血管通透性。研究表明, 黄芩素可抑制分化抑制因子-1蛋白(inhibitor of differentiation-1 protein)、上皮间质转化(epithelialmesenchymal transition)相关的N-钙黏附蛋白(N-cadherin)、波形蛋白(vimentin)以及血管生成相关蛋白(VEGFA)的表达[28], 从而抑制人非小细胞肺癌原位移植瘤的生长。肺上皮细胞和肺毛细血管内皮细胞的凋亡是ALI发生的一个潜在的重要机制[29]。研究证实, 小檗碱可以调控BCL2L1等凋亡抑制基因的表达, 进而抑制B细胞淋巴瘤/白血病-2 (B-cell lymphoma/leukemia 2, Bcl-2)蛋白表达, 增加B细胞淋巴瘤/白血病-2-相关X蛋白(BCL-2-associated X, Bax)表达, 从而抑制细胞增殖, 促进细胞凋亡[30]。LPS诱导的ALI后, 炎性因子的大量产生会导致NOS2的过度表达, 在内皮细胞、血液和肺组织液中产生大量NO[31]。黄芩中的黄酮类成分对LPS诱导的ALI有明显的保护作用, 其保护作用可能与抑制COX-2和i NOS的表达有关[18]。
KEGG富集通路分析显示, VEGF信号通路[29]、MAPK1信号通路、MAPK3信号通路[24]、NLRs信号通路[32]、Toll样受体信号通路[33]、mTOR信号通路[34]、TGF-β信号通路[35]、T细胞受体信号通路、B细胞受体信号通路[36]和Jak-STAT信号通路[37]等是ALI的疾病调控相关的重要通路, 且有文献报道, 说明本研究预测富集的通路可信度较高。甘草次酸可降低肺组织TGF-β1、母亲DPP同源物2 (mothers against decapentaplegic homolog 2, Smad2)和母亲DPP同源物3 (mothers against decapentaplegic homolog 3, Smad3)的表达, 抑制TGF-β1/Smads信号通路, 干预ALI的治疗过程。黄芩素可下调T细胞Jak-STAT信号通路STAT3和STAT4的mRNA表达, 促进T细胞增殖, 从而发挥免疫抗炎作用。
通过动物实验证实, EhD具有减轻小鼠肺部损伤和减少炎症细胞因子释放的作用。同时, Western blot结果显示, EhD可以降低NF-κB/p65蛋白的表达, 提高IκBα蛋白的表达, 从而调控NF-κB通路, 发挥抗炎作用。NF-κB作为转录因子蛋白家族, 包括5个亚单位: Rel、p65 (RELA)、Rel B、p50和p52。RELA基因在调控细胞的基因转录过程中起着至关重要的作用, 活化的NF-κB/p65对机体的炎症反应和免疫反应等方面的作用十分关键, 同时与内毒素型ALI的发生发展联系密切。近年来的相关研究发现, 许多中药能够调控NF-κB的活化过程, 进而抑制炎症因子产生, 对多种疾病起到一定的治疗作用。文献报道, 二黄汤中黄芩素[38]、小檗碱[39]和甘草酸[40]等主要活性成分均可以通过抑制NF-κB的表达, 达到改善肺部ALI炎症的效果。
综上所述, 本研究以EhD中入血成分为研究对象, 通过网络药理学和分子对接方法, 对EhD治疗ALI的潜在作用靶点及作用机制进行了预测分析, 并通过动物实验和分子生物学方法, 验证EhD的药效和调控机制。本文为治疗发病机制复杂的ALI的研究提供了新的思路, 也为EhD的协同作用机制的深入研究奠定了基础。
作者贡献:本文设计思路由张永清、孙秀梅和赵忠熙教授提供。杨斌和付先军教授具体参与靶点筛选、通路富集、网络构建及分子对接工作。王京龙、郑丹丹和张立华老师负责ALI药效实验及Western blot蛋白表达实验。
利益冲突:本文不存在利益冲突。
| [1] |
Sweeney RM, Mc Auley DF. Acute respiratory distress syndrome[J]. Lancet, 2016, 388: 2416-2430. DOI:10.1016/S0140-6736(16)00578-X |
| [2] |
Ferrara JL. Cytokine dysregulation as a mechanism of graft versus host disease[J]. Curr Opin Immunol, 1993, 5: 794-799. DOI:10.1016/0952-7915(93)90139-J |
| [3] |
Jing H, Yao J, Liu X, et al. Fish-oil emulsion(omega-3 polyun-saturated fatty acids)attenuates acute lung injury induced by intestinal ischemia-reperfusion through adenosine 5'-monophos-phate-activated protein kinase-sirtuin1 pathway[J]. J Surg Res, 2014, 187: 252-261. DOI:10.1016/j.jss.2013.10.009 |
| [4] |
Choudhury SR, Babes L, Rahn JJ, et al. Dipeptidase-1 is an adhesion receptor for neutrophil recruitment in lungs and liver[J]. Cell, 2019, 178: 1205-1221. DOI:10.1016/j.cell.2019.07.017 |
| [5] |
Huang C, Wang Y, Li X, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China[J]. Lancet, 2020, 395: 497-506. DOI:10.1016/S0140-6736(20)30183-5 |
| [6] |
Lu Y, Zhang PP, Wang DQ, et al. Discussion on pathogenesis and pathogenesis of acute lung injury[J]. J Emerg Tradit Chin Med(中国中医急症), 2020, 29: 280-282. |
| [7] |
Wu Q. Yi Zong Jin Jian(医宗金鉴)[M]. 1st ed. China Medicine Science and Technology Press, 2011: 345.
|
| [8] |
Li HY, Yuan ZY, Chai YS, et al. Study on inflammatory mechanism between lung and brain by the action of baicalin on TLRsNOD2 signaling[J]. World Sci Tech Mod Chin Mater Med(世界科学技术-中医药现代化), 2012, 14: 1195-1200. |
| [9] |
Chen HJ, Zhang YL, Zhang WX, et al. Inhibition of myeloid differentiation factor 2 by baicalein protects against acute lung injury[J]. Phytomedicine, 2019, 63: 152997. DOI:10.1016/j.phymed.2019.152997 |
| [10] |
Li HM, Wang YY, Wang HD, et al. Berberine protects against lipopolysaccharide-induced intestinal injury in mice via alpha 2 adrenoceptor-independent mechanisms[J]. Acta Pharmacol Sin, 2011, 32: 1364-1372. DOI:10.1038/aps.2011.102 |
| [11] |
Kong DL, Wang ZF, Tian J, et al. Glycyrrhizin inactivates toll-like receptor(TLR)signaling pathway to reduce lipopolysaccharideinduced acute lung injury by inhibiting TLR2[J]. J Cell Physiol, 2019, 234: 4597-4607. DOI:10.1002/jcp.27242 |
| [12] |
Liu ZJ, Zhong J, Zhang M, et al. The alexipharmic mechanisms of five licorice ingredients involved in CYP450 and Nrf2 pathways in paraquat-induced mice acute lung injury[J]. Oxid Med Cell Longev, 2019, 2019: 7283104. |
| [13] |
Wu H, Wang JQ, Yang YW, et al. Preliminary exploration of the mechanism of Qingfei Paidu decoction against novel coronavirus pneumonia based on network pharmacology and molecular docking technology[J]. Acta Pharm Sin(药学学报), 2020, 55: 374-383. |
| [14] |
Liu AL, Du GH. Network pharmacology: new guidelines for drug discovery[J]. Acta Pharm Sin(药学学报), 2010, 45: 1472-1477. |
| [15] |
Wang JL, Zheng DD, Wang YZ, et al. Pharmacokinetics study of Erhuang decoction extracts in rats by HPLC-MS/MS[J]. JChromatogr B, 2017, 1059: 35-42. DOI:10.1016/j.jchromb.2017.05.019 |
| [16] |
Wang JL, Zheng DD, Xv N, et al. Attribution and identification of absorbed components by HPLC-DAD-ESI-MS after oral administration of Erhuang decoction[J]. J Anal Sci Technol, 2020, 11: 38. DOI:10.1186/s40543-020-00236-4 |
| [17] |
David G, Aurelien G, Matthias W, et al. Swiss Target Prediction: a web server for target prediction of bioactive small molecules[J]. Nucleic Acids Res, 2014, 42: W32-W38. DOI:10.1093/nar/gku293 |
| [18] |
Chen JJ, Huang CC, Chang HY, et al. Scutellaria baicalensis ameliorates acute lung injury by suppressing inflammation in vitro and in vivo[J]. Am J Chin Med, 2017, 45: 137-157. DOI:10.1142/S0192415X17500100 |
| [19] |
Bitencourt-Ferreira G, Azevedo WFD. Docking with Swiss Dock[J]. Methods Mol Biol, 2019, 2053: 189-202. |
| [20] |
Tang SJ, Rao L, Chen J, et al. The docking prediction of baicalin and BMP receptor using Swiss Dock[J]. Chin J Biol Pharm(中国生化药物杂志), 2017, 37: 34-36. |
| [21] |
Ding ZH, Zhong RX, Xia TY, et al. Advances in research into the mechanisms of Chinese Materia Medica against acute lung injury[J]. Biomed Pharmacother, 2020, 122: 109706. DOI:10.1016/j.biopha.2019.109706 |
| [22] |
Menegazzi M, Paola RD, Mazzon E, et al. Glycyrrhizin attenuates the development of carrageenan-induced lung injury in mice[J]. Pharmacol Res, 2008, 58: 22-31. DOI:10.1016/j.phrs.2008.05.012 |
| [23] |
Zhang LP, Li JG. Glabridin reduces lipopolysaccharide-induced lung injury in rats by inhibiting p38 mitogen activated protein kinase/extracellular regulated protein kinases signaling pathway[J]. Natl Med J Chin(中华医学杂志), 2016, 96: 3893-3897. |
| [24] |
Zhang J, Deng YY, Cheng B, et al. Protective effects and molecular mechanisms of baicalein on thioacetamide-induced toxicity in zebrafish larvae[J]. Chemosphere, 2020, 256: 127038. DOI:10.1016/j.chemosphere.2020.127038 |
| [25] |
Wei CY, Sun HL, Yang ML, et al. Protective effect of wogonin on endotoxin-induced acute lung injury via reduction of p38MAPK and JNK phosphorylation[J]. Environ Toxicol, 2017, 32: 397-403. DOI:10.1002/tox.22243 |
| [26] |
Liu T, Dai WJ, Li C, et al. Baicalin alleviates silica-induced lung inflammation and fibrosis by inhibiting the Th17 response in C57BL/6 mice[J]. J Nat Prod, 2015, 78: 3049-3057. DOI:10.1021/acs.jnatprod.5b00868 |
| [27] |
Takamitsu S, Yasuhiko K, Toru N, et al. Inhibition of epidermal growth factor receptor and vascular endothelial growth factor receptor phosphorylation on tumor-associated endothelial cells leads to treatment of orthotopic human colon cancer in nude mice[J]. Neoplasia, 2007, 9: 1066-1077. DOI:10.1593/neo.07667 |
| [28] |
Zhao ZX, Liu BJ, Sun J, et al. Baicalein inhibits orthotopic human non-small cell lung cancer xenografts via Src/Id1 pathway[J]. Evid Based Complement Alternat Med, 2019, 2019: 9806062. |
| [29] |
Guo Q, Huang F, Wang J, et al. Expressions of apoptosis gene Bcl-2 and Bad in the lung tissue of mice with acute lung injury[J]. Chin J Emerg Med, 2010, 19: 250-252. |
| [30] |
Li J, Liu F, Jiang SL, et al. Berberine hydrochloride inhibits cell proliferation and promotes apoptosis of non-small cell lung cancer via the suppression of the MMP2 and Bcl-2/Bax signaling pathways[J]. Oncol Lett, 2018, 15: 7409-7414. |
| [31] |
Agorreta J, Garayoa M, Montuenga LM, et al. Effects of acute hypoxia and lipopolysaccharide on nitric oxide synthase-2 expression in acute lung injury[J]. Am J Respir Crit Care Med, 2003, 168: 287-296. DOI:10.1164/rccm.200209-1027OC |
| [32] |
Liu QM, Lv HM, Wen ZM, et al. Isoliquiritigenin activates nuclear factor erythroid-2 related factor 2 to suppress the NOD-like receptor protein 3 inflammasome and inhibits the NF-κB pathway in macrophages and in acute lung injury[J]. Front Immunol, 2017, 8: 1518. DOI:10.3389/fimmu.2017.01518 |
| [33] |
Zheng L, Zhu Q, Xu C, et al. Glycyrrhizin mitigates radiationinduced acute lung injury by inhibiting the HMGB1/TLR4 signalling pathway[J]. J Cell Mol Med, 2020, 24: 214-226. DOI:10.1111/jcmm.14703 |
| [34] |
Qu LH, Chen C, He W, et al. Glycyrrhizic acid ameliorates LPS-induced acute lung injuryby regulating autophagy through the PI3K/AKT/mTOR pathway[J]. Am J Transl Res, 2019, 11: 2042-2055. |
| [35] |
Chen JM, Zhang WJ, Zhang LR, et al. Glycyrrhetinic acid alleviates radiation-induced lung injury in mice[J]. J Radiat Res, 2017, 58: 41-47. DOI:10.1093/jrr/rrw091 |
| [36] |
Han QH, Han N, Liu YZ, et al. Analysis of key genes and pathways involved in acute lung injury in a mouse model[J]. Genet Mol Res, 2014, 13: 4591-4598. DOI:10.4238/2014.June.18.1 |
| [37] |
Xu J, Liu JL, Yue GL, et al. Therapeutic effect of the natural compounds baicalein and baicalin on autoimmune diseases[J]. Mol Med Rep, 2018, 18: 1149-1154. |
| [38] |
Tsai CL, Lin YC, Wang HM, et al. Baicalein, an active component of Scutellaria baicalensis, protects against lipopolysaccharide-induced acute lung injury in rats[J]. J Ethnopharmacol, 2014, 153: 197-206. DOI:10.1016/j.jep.2014.02.010 |
| [39] |
Li ZH, Zheng J, Zhang N, et al. Berberine improves airway inflammation and inhibits NF-κB signaling pathway in an ovalbumin-induced rat model of asthma[J]. J Asthma, 2016, 53: 999-1005. DOI:10.1080/02770903.2016.1180530 |
| [40] |
Yao L, Sun TL. Glycyrrhizin administration ameliorates Streptococcus aureus-induced acute lung injury[J]. Int Immunopharmacol, 2019, 70: 504-511. DOI:10.1016/j.intimp.2019.02.046 |
 2021, Vol. 56
2021, Vol. 56


