2. 中国医学科学院、北京协和医学院药物研究所, 新药作用机制研究与药效评价北京市重点实验室, 北京 100050
2. Beijing Key Laboratory of New Drug Mechanisms and Pharmacological Evaluation Study, Institute of Materia Medica, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100050, China
正常机体免疫系统具有免疫监视功能, 当体内发生肿瘤时, 免疫系统能够识别并通过免疫机制特异地清除这些“非己”细胞, 抵御肿瘤的发生发展。而大多数肿瘤具有应对免疫系统的进化选择能力, 该进化过程发展的三个特征阶段包括:消除、平衡和逃避, 即所谓的“免疫编辑”[1, 2]。肿瘤细胞通过调整自身的内在状态和肿瘤微环境等多种机制, 使免疫介导的排斥反应不能被诱导产生, 从而导致机体对肿瘤的免疫应答反应处于低能状态, 进而使肿瘤逃避免疫监视而发生、发展[3]。21世纪初, 肿瘤免疫逃逸被认为是肿瘤的十大特征之一[4]。针对肿瘤的免疫逃逸机制研究开发了免疫检查点抑制剂、肿瘤疫苗等多种抗肿瘤免疫药物[5]。肿瘤免疫治疗因其疗效显著被《Science》评为2013年度十大科技突破之首。2018年, 细胞毒T淋巴细胞相关抗原4 (cytotoxic T lymphocyte-associated antigen-4, CTLA-4)和程序性死亡受体1 (programmed death protein 1, PD-1)的发现者詹姆斯•艾利森和本庶佑获得诺贝尔生理学或医学奖。肿瘤免疫相关领域的研究为肿瘤的治疗提供了新的治疗手段, 为转移性晚期肿瘤患者带来新的希望。因此, 肿瘤免疫治疗引起了国内外学者越来越多的关注[6]。
1924年, Otto Warburg[7]发现癌细胞在有氧条件下将葡萄糖代谢为乳酸, 即Warburg效应。科学家们开始意识到肿瘤细胞具有独特的代谢方式, 肿瘤细胞的代谢异常受突变基因的调控[8, 9], 各代谢通路之间的影响和联系错综复杂。随着各种分析技术和方法的发展, 肿瘤代谢的研究也更加深入, 各种糖类、氨基酸、脂肪酸等代谢物的变化都为肿瘤适应外部环境提供助力, 肿瘤代谢也成为研究肿瘤发生发展以及治疗的新突破点[10, 11]。而肿瘤代谢异常和肿瘤免疫逃逸之间的联系也被发现, 基于肿瘤代谢调控的免疫治疗成为研究热点。本文对近年来基于代谢调控的肿瘤免疫逃逸和肿瘤免疫治疗相关研究进展以及肿瘤代谢研究的前沿分析技术和方法进行综述。
1 肿瘤代谢与免疫 1.1 肿瘤免疫逃逸与免疫治疗肿瘤细胞可通过重编程或者微环境中的“优势因素”以多途径、多层面的方式抑制或逃避机体免疫系统的杀伤[3]。目前, 对于上述逃逸机制的研究主要集中在针对微环境中的肿瘤表面抗原[12], 肿瘤相关成纤维细胞(cancer-associated fibroblasts, CAFs)[13-15], 免疫细胞如T细胞[16-18], 自然杀伤细胞(natural killer cell, NK)[19], 巨噬细胞[20, 21], 抗原呈递细胞[22]表面蛋白标志及其分泌的细胞因子[23]、转录因子[24, 25]介导的信号通路等方面。Wang等[24]提出并证实纤维介素蛋白1 (fibrinogen-like protein 1, FGL1)-淋巴细胞活化基因3 (lymphocyte activation gene-3, LAG-3)通路抑制抗原介导的T细胞反应, 该项研究表明肿瘤细胞大量产生的FGL1是LAG-3发挥免疫抑制功能的重要配体, 癌症患者血浆中高水平的FGL1与不良预后相关, 阻断FGL1和LAG-3蛋白的相互作用可刺激T细胞的活化和增殖来提高机体对肿瘤的清除, 该发现可作为肿瘤免疫治疗的新靶点。此外, 抑制NK细胞介导的免疫应答的T细胞免疫球蛋白和ITIM结构域蛋白[26]、向巨噬细胞传递“别吃我”信号的CD47[27]以及提高肿瘤免疫原性的干扰素基因刺激蛋白都是近些年发现的潜在靶点[28]。
迄今为止, CTLA-4抗体、PD-1抗体、程序性死亡配体1 (programmed death ligand 1, PD-L1)抗体、嵌合抗原受体T细胞是肿瘤免疫治疗中数量增长最快、最具前景的药物类别, 纷纷在国内外获批上市, 并在血液系统恶性肿瘤、恶性黑色素瘤、肺癌和肾癌等临床治疗中表现出令人振奋的结果[29, 30]。《新英格兰医学杂志》公布了PD-1抗体Nivolumab和CTLA-4抗体Ipilimumab联合治疗晚期肾细胞癌(renal cell carcinoma, RCC)的Ⅲ期临床试验结果, 联合疗法组客观缓解率高达42%, 而标准靶向药物舒尼替尼治疗组只有27%, 患者总生存率和生活质量得以显著提高[31]。PD-1抗体Keytruda联合紫杉醇和铂类化疗是转移性鳞状非小细胞肺癌化疗免疫治疗的重大进步[32]。近期, 用于晚期不可切除肝细胞癌的PD-L1抗体Atezolizumab和血管内皮生长因子抑制剂Bevacizumab联合治疗获得国家药品监督管理局优先评审资格。肿瘤免疫治疗在医药领域已经取得了突破性的进展, 对肿瘤学和人类健康具有重大影响。
尽管如此, 肿瘤免疫治疗领域仍然存在许多问题, 如获益的患者有限; 出现获得性耐药[33, 34]; 免疫系统可能过度激活而发生“细胞因子风暴”等严重炎症反应[35]。因此, 肿瘤免疫逃逸的途径和影响因素必然是多方面的, 除免疫细胞功能状态之外, 局部肿瘤微环境也相当重要。Baitsch等[36]发现转移性黑色素瘤患者外周血中的效应性T细胞功能衰竭并增加共抑制受体的表达, 但是经体外短期培养, 肿瘤浸润性T细胞杀伤肿瘤细胞的功能可以恢复[37], 由此可见肿瘤局部微环境对机体的免疫行为和功能状态有极大的影响[38]。因此, 进一步深入进行肿瘤免疫药物相关的基础和临床研究, 同时全面理解肿瘤细胞在肿瘤微环境中如何逃逸免疫监视与杀伤的复杂机制, 寻找能够在治疗早期预测疗效的生物标志物, 对于改善肿瘤免疫治疗效果是极其重要的。
1.2 肿瘤代谢与免疫逃逸肿瘤组织主要由肿瘤细胞、基质细胞以及正常组织细胞等多种类型细胞构成, 其结构及生物化学微环境复杂。肿瘤微环境是宿主与肿瘤细胞相互作用的场所[39], 与肿瘤的发生、发展及转移密切相关。肿瘤细胞为了满足其快速增殖的需求, 合成代谢和分解代谢均发生了适应性变化, 即“代谢重编程”[40]。肿瘤代谢重编程涉及的代谢通路多且复杂, 从最开始的糖酵解到糖代谢、氨基酸代谢、脂代谢等[41](图 1)。肿瘤细胞受到基因调控和微环境的影响, 代谢重塑的同时, 代谢微环境也相应被改变。例如, 肿瘤细胞进行有氧糖酵解, 消耗大量葡萄糖并转化为乳酸, 使得微环境中乳酸堆积, 同时糖酵解的中间产物葡萄糖-6-磷酸、甘油醛-3-磷酸可以为核苷酸、脂质等物质合成提供原料[42]; 肿瘤细胞摄取的多种氨基酸增加; 肿瘤血管系统异常, 上述这些代谢变化使得肿瘤微环境呈现缺氧、低pH、营养物质缺乏和代谢物蓄积的特征[43], 促进了肿瘤微环境的免疫抑制能力和免疫逃逸的发生。
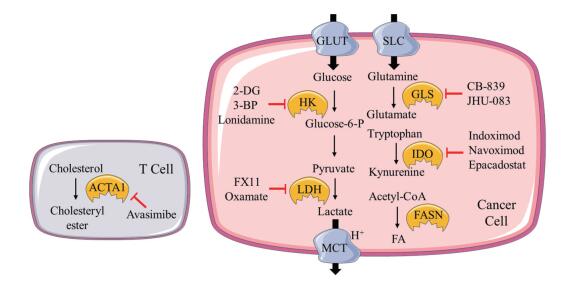
|
Figure 1 Some important metabolic pathways related to immune escape and relevant inhibitor in cancer cell and T cell. HK: Hexokinase; LDH: Lactate dehydrogenase; MCT: Monocarboxylte transporters; GLUT: Glucose transporter; SLC: Solute carrier; GLS: Glutaminase; IDO: Indoleamine-2, 3-dioxygenase; FASN: Fatty acid synthase; ACAT1: Acetyl-CoA cholesterol acyltransferase1; 2-DG: 2-Deoxy-d-glucose; 3-BP: 3-Bromopyruvic acid |
糖酵解是肿瘤细胞能量代谢的主要途径, 而糖酵解对于免疫细胞的增殖和功能活动也至关重要[44]。Chang等[45]发现初始T细胞依靠氧化磷酸化来维持能量需求, 而活化的T细胞通过有氧糖酵解消耗大量葡萄糖来发挥抗肿瘤效应。较高糖酵解活性的肿瘤细胞具有很强的逃避免疫监视的能力, 即使肿瘤的免疫原性强, 在施加葡萄糖限制后, T细胞的哺乳动物雷帕霉素靶蛋白(mammalian target of rapamycin, mTOR)活性和糖酵解活性减弱, 肿瘤施加的代谢限制导致T细胞表现低反应性。肿瘤细胞糖酵解的异常受到糖酵解相关酶和基因调控, 如抑癌基因p53、己糖激酶(hexokinase, HK)、果糖二磷酸醛缩酶A、乳酸脱氢酶(lactate dehydrogenase, LDH)等[46, 47]。
除了葡萄糖的代谢竞争外, 糖酵解的代谢产物乳酸也通过多种途径促进了肿瘤免疫逃逸。乳酸可以直接抑制NK细胞的毒杀伤活性, 同时通过增加骨髓源性抑制细胞(myeloid-derived suppressor cells, MDSCs)的数量间接抑制NK细胞功能[48]。此外, 乳酸可阻断单核细胞的迁移和向树突状细胞(dendritic cells, DCs)的分化[49], 抑制DCs和细胞毒性T细胞释放细胞因子[50], 并促进肿瘤相关巨噬细胞M2极化[51]。Zhang等[52]首次发现并证实乳酸可以直接和线粒体抗病毒信号转导蛋白结合并使其失活, 从而抑制细胞RIG-Ⅰ样受体介导的Ⅰ型干扰素产生, 靶向LDH-A可促进Ⅰ型干扰素产生。不仅如此, 肿瘤细胞中乳酸通过单羧酸转运体与H+共转运[53], 其对细胞毒性T细胞的抑制作用依赖质子的存在[54]。乳酸还可作为信号分子通过自分泌或旁分泌激活癌细胞或免疫细胞上的G蛋白偶联受体81, 参与单羧酸转运体的表达调控, 促进血管生成, 驱动免疫逃逸, G蛋白偶联受体81也被作为潜在靶点在探索中[55, 56]。
1.2.2 氨基酸代谢为了满足自身生长需要, 肿瘤细胞需要持续的氨基酸供应, 并且多种氨基酸代谢水平发生改变, 肿瘤细胞和免疫细胞之间的代谢竞争会导致营养物质的缺乏, 因此肿瘤细胞的氨基酸代谢也是研究的热点。
精氨酸(arginine, Arg)代谢是调节免疫细胞反应活性的重要途径之一, 其影响免疫逃逸的机制中涉及两个关键酶:一氧化氮合酶(nitric oxide synthase, NOS)和精氨酸酶(arginase, ARG)。NOS将Arg氧化成瓜氨酸和NO, ARG将Arg水解成鸟氨酸和尿素。肿瘤细胞中NOS或ARG过表达使肿瘤微环境中Arg耗竭, 抑制T细胞增殖和功能[57]。在黑色素瘤等多种肿瘤细胞中诱导型NOS产生的NO可以激活环氧化酶2, 诱导MDSCs中ARG1表达, 进一步导致肿瘤环境中Arg的耗竭[58-60]。补充L-Arg可以提高T细胞的存活率和抗肿瘤活性[61], 因此靶向Arg代谢可作为提高抗肿瘤免疫力的有效策略。
谷氨酰胺(glutamine, Gln)是人体中含量最丰富的氨基酸, 是细胞增殖最重要的营养物质, 仅次于葡萄糖[62], 许多肿瘤细胞表现出Gln依赖。谷氨酰胺转运蛋白和谷氨酰胺酶(glutaminase, GLS)的上调使细胞外Gln被肿瘤摄取并代谢[63, 64]。而在Gln缺乏的微环境中, 肾癌细胞可以通过EGFR/ERK/c-Jun途径来诱导PD-L1表达, 从而抑制了T细胞分泌干扰素γ, 使肿瘤细胞逃逸免疫系统的杀伤[65]。Fu等[66]发现在RCC中, Gln耗竭激活缺氧诱导因子通路诱导巨噬细胞分泌白介素23活化调节性T细胞(regulatory cells, Treg), 促进白介素10和转化生长因子β的表达, 抑制T细胞毒杀伤活性从而介导免疫逃逸。因此, 白介素23有望成为RCC免疫治疗的新靶点。
色氨酸(tryptophan, Trp)代谢对免疫抑制的影响也得到了极大的关注。吲哚胺-2, 3-双加氧酶(indoleamine-2, 3-dioxygenase, IDO)将Trp转化为犬尿氨酸(kynurenine, Kyn), 在抗原呈递细胞和肿瘤细胞中高表达。Trp是保证各种细胞功能的必需氨基酸, 其耗竭会引起包括效应T细胞在内的细胞死亡, 同时由此产生大量的Kyn, 促进Treg分化, 抑制免疫系统功能[67]。Kyn还可以通过芳烃受体影响CD8+ T细胞的活性[68], 而Trp耗竭可通过激活GCN2通路或抑制mTOR通路影响免疫反应[69]。因此, IDO表达上调被认为是抗PD-1等免疫治疗的耐药机制之一, IDO活性常以Kyn/Trp表示, 比值越高, 免疫治疗效果越差[70]。此外, Kyn下游代谢产物以及代谢酶也可能在肿瘤免疫逃逸中发挥作用。Pour等[71]发现3-羟基犬尿氨酸和犬尿喹啉酸等Kyn代谢产物具有免疫调节作用, 增强Kyn代谢关键酶犬尿氨酸3-单加氧酶(kynurenine-3-monooxygenase, KMO)或犬尿氨酸酶活性有可能恢复皮肤恶性黑色素瘤患者的抗肿瘤免疫。
亮氨酸(leucine, Leu)作为人体必需氨基酸在蛋白质合成中发挥重要作用, 同时也是激活mTOR通路的信号分子, mTOR通路可以调控蛋白合成、细胞生长、增殖、分化等功能。Leu在肿瘤细胞内因转运蛋白SLC7a5表达增加而发生积累, 激活mTORC1, 促进肿瘤的发展和生长[72]。而Leu对免疫细胞增殖、分化也至关重要。剥夺Leu的T细胞无法维持mTORC1活性, 而活化的T细胞中mTORC1活性依赖于T细胞抗原受体诱导的SLC7a5表达增强[73], SLC7a5的底物包括Leu、蛋氨酸等多种中性氨基酸, 是控制T细胞mTORC1活性的重要因素[74]。支链氨基酸转氨酶1在乳腺癌、胶质瘤等多种癌症以及活化的T细胞中上调[75], 短暂使用此酶的抑制剂可以诱导CD8+ T细胞代谢重编程, 增强PD-1抗体活性[76]。癌细胞和免疫细胞对氨基酸的代谢重编程往往具有相似过程, 因而两者的代谢竞争与肿瘤免疫逃逸机制相关, 深入研究两者的代谢关系有助于靶向代谢调控免疫应答。
1.2.3 脂代谢除了消耗性的营养物质外, 肿瘤细胞通过脂肪酸的从头合成途径产生大量脂肪酸, 富含脂肪酸的微环境影响效应T细胞功能和巨噬细胞M1型极化, 同时有利于产生Tregs和M2样巨噬细胞[77], 造成肿瘤部位的免疫抑制。脂肪合成酶(fatty acid synthase, FASN)是脂肪酸从头合成的关键代谢酶, 在卵巢癌、胃癌等多种肿瘤中上调[78]。Jiang等[79]发现卵巢癌中FASN过表达导致肿瘤浸润DCs中脂质积累, 造成T细胞功能障碍, 进而诱导抗肿瘤免疫应答受损, 因此抑制脂肪酸合成能增强抗肿瘤免疫。但肿瘤中脂质合成异常的机制尚不清楚, Gouw等[80]发现在MYC诱导的癌症中, MYC与胆固醇调节元件结合蛋白1协同促进脂肪酸合成途径中多种酶的表达, 如乙酰辅酶A羧化酶、FASN等, 共同调节肿瘤的脂肪生成。脂肪酸氧化在调节T细胞应答中也具有重要作用。组织驻留记忆T细胞(tissue- resident memory T, Trm)是一类肿瘤浸润淋巴细胞, Lin等[81]发现胃腺癌中Trm细胞不是利用葡萄糖而是依靠脂肪酸氧化供能, 癌细胞和Trm细胞对脂质的代谢竞争, 导致Trm死亡。而阻断PD-L1可以调节Trm细胞代谢, 促进其脂质摄取, 增强抗肿瘤免疫反应。
1.3 基于肿瘤代谢调控的免疫治疗随着肿瘤代谢的深入研究, 靶向肿瘤代谢中的关键转运体或代谢酶成为开发抗肿瘤代谢药物的新方法[82]。如前文所述, 多种肿瘤代谢途径的改变可引起肿瘤免疫抑制和免疫逃逸。因此, 基于代谢调控的肿瘤免疫治疗策略, 可以提高免疫治疗的有效性, 让更多患者受益, 为改善肿瘤免疫治疗开辟新方向。
IDO是最早用于免疫治疗的代谢酶靶点之一, 有IDO1和IDO2两种亚型, 两者都能催化Trp代谢为Kyn, 但IDO1的酶活性是IDO2的20~30倍[67, 83]。IDO抑制剂常与化疗药物或其他免疫治疗联合使用, 一项Ⅱ期临床试验数据显示, IDO2抑制剂Indoximod与Keytruda的联合疗法在晚期黑色素瘤治疗中客观缓解率为55.7%, 完全缓解率为18.6%, 明显高于Keytruda单独疗法[84]。Navoximod是双重抑制剂, 同时抑制IDO1和TDO, Ⅰ期临床试验表明Navoximod和Atezolizumab联合用药对多种晚期癌症患者显示出可接受的安全性、耐受性和药代动力学特征, 但是没有明显证据表明联合疗法优于PD-L1抗体单药治疗[85]。另一种IDO1抑制剂Epacadostat与Keytruda联合用于黑色素瘤治疗的Ⅲ期临床试验中, 与Keytruda单药治疗相比, 该联合用药方案并未改善无进展生存期或总生存期, IDO1抑制剂增强肿瘤抗PD-1/PD-L1治疗的有效性仍不能确定[86], 仅抑制IDO1也许不能明显改善免疫治疗的效果, 因此Kyn途径中失调的其他酶如KMO等逐渐受到更多关注[87]。Ray等[88]发现单独靶向KMO或联合PD-L1抗体可以增强细胞毒性, 恢复抗多发性骨髓瘤免疫。
基于Gln代谢调控的免疫治疗也是研究热点。Gln类似物6-重氮-5-氧代-L-正亮氨酸(6-diazo-5-oxo-L-norlucine, DON)通过抑制Gln代谢中多种酶活性而发挥作用, 但因其毒性大而限制了临床应用。Leone等[89]设计了DON的前体药物JHU-083, 通过阻断Gln代谢抑制肿瘤细胞氧化代谢和糖酵解, 降低肿瘤微环境免疫抑制能力, 直接影响T细胞的活化和功能, 使其呈现出长寿命、高度活化的表型。进一步研究发现JHU-083可以增强过继T细胞疗法和PD-1抗体的疗效, 但还未进入临床阶段。此外, 靶向Gln代谢还可以通过促进MDSCs凋亡, 抑制肿瘤细胞IDO表达使Kyn下调等方式抑制肿瘤转移和增强抗肿瘤免疫[90]。针对GLS或谷氨酰胺转运蛋白的抑制剂也显示出抗肿瘤活性, PD-1/PD-L1治疗中加入GLS抑制剂CB-839可以增强小鼠结肠癌肿瘤消退, 提高小鼠生存率[91]。CB-839与PD-1/PD-L1抗体联合治疗正在Ⅰ期和Ⅱ期临床试验研究当中[92, 93]。
基于糖酵解代谢的免疫治疗药物主要针对HK、LDH、葡萄糖转运蛋白等靶点设计开发。糖酵解的第一步是由HK催化葡萄糖磷酸化, 2-脱氧葡萄糖、3-溴丙酮酸和氯尼达明是常见的HK抑制剂, 正在体内外研究中, 但多组临床试验因有效性或毒性而暂停[94]。肿瘤细胞中上调的LDH-A使乳酸生成增加, 在黑色素瘤和非小细胞肺癌中, 患者血清高水平LDH与免疫治疗不良预后具有相关性[95], 因此靶向LDH也许可以克服免疫治疗耐药性。Oxamate、FX11等多种LDH抑制剂在临床前研究中表现出抗肿瘤活性, 但尚未在临床应用[96]。
通过调节胆固醇代谢可以增强小鼠CD8+ T细胞的抗肿瘤免疫反应[97]。药物Avasimibe抑制胆固醇代谢通路中关键的酰基辅酶A-胆固醇酰基转移酶1, 阻止T细胞内游离胆固醇转化为胆固醇酯, CD8+ T细胞的质膜胆固醇水平升高, 导致T细胞受体聚集和信号传递增强, 更有效地形成免疫突触。Avasimibe与PD-1抗体的联合疗法在控制肿瘤进展方面比单一疗法更有效, 两者可通过不同的途径发挥作用, 在癌症免疫治疗中具有相加的作用。这类代谢抑制剂与癌症疫苗的联用也有类似效果, 在Kras突变的肺癌动物模型中, Avasimibe和Kras疫苗联合使用可以显著降低Treg数量, 促进CD8+ T细胞的肿瘤浸润, 增强疫苗的疗效[98]。
2 肿瘤代谢分析方法 2.1 代谢组学代谢组学是一种新兴“组学”技术, 涉及生物系统中代谢物和代谢通路的综合表征。代谢物代表基因组的下游输出和来自环境的上游输入, 因此通过研究生物体系受机体基因影响或外部环境刺激所产生的代谢物及其代谢通路的变化规律, 有助于探索基因与环境相互作用的关系, 寻找与疾病发生发展、药物作用机制等相关的新型标志物[99]。随着分析技术的发展, 代谢组学在肿瘤学的应用日益增多, 包括癌症的诊断、分期分型、治疗和预后等。目前代谢组学研究中使用较多的分析检测技术包括核磁共振技术(NMR)、气相色谱-质谱联用技术(GC-MS)和液相色谱-质谱联用技术(LC-MS)。
NMR是一种非破坏性和非侵入性技术, 可以提供复杂混合物中有机分子完整结构分析, 因此可检测肿瘤和健康组织之间的代谢差异, 寻找不同进展程度的癌症中可能的生物标记物。因几乎所有代谢物中都含有质子, 1H NMR的应用最为广泛[100, 101]。Zheng等[102]利用基于1H NMR的代谢组学方法分析前列腺癌患者的肿瘤组织、血清和尿液样本, 发现前列腺癌进展过程中, 组织样本的代谢表型变化更明显, 能量代谢、氨基酸代谢、胆碱代谢等重要代谢紊乱, Gln、酪氨酸等多种代谢物可能作为前列腺癌进展的潜在生物标志物。Lin等[103]采用1H NMR结合模式识别, 比较健康人和结直肠癌患者的粪便代谢物, 发现乙酸的诊断性能最高。NMR具有无损伤性、定性能力强等优点, 缺点则是灵敏度低、仪器价格高。
质谱技术因其灵敏度高、特异性好、检测动态范围宽等优势, 已成为代谢组学研究中最常用的分析工具, 而色谱质谱联用技术能够实现复杂生物样本中多组分快速、同时、准确定性或定量分析, 更是在肿瘤代谢组学研究中有着不可或缺的地位[104]。Kibi等[105]采用基于GC-MS和LC-MS的代谢组学方法, 检测结直肠癌组织样本和相邻正常组织样本代谢物水平, 发现了多种代谢物变化, 如促进肿瘤免疫逃逸的高水平Kyn和奎宁酸、辅助结直肠癌细胞增殖的高水平抗氧化代谢物等。Jing等[106]用LC-MS技术对3种亚型RCC样本进行代谢组学分析, 建立OPLS-DA模型用于亚型鉴别, 独立样本队列中至少92%的样本被准确预测, 同时发现不同亚型RCC的氨基酸和脂肪酸代谢存在显著差异, 准确鉴别以及代谢特征分析有助于精准治疗。除了基础研究, 代谢组学也可指导临床免疫治疗。Li等[107]对3组免疫治疗临床试验患者的血清进行代谢组学分析, 发现了在部分黑色素瘤和RCC患者中, 阻断PD1可导致Trp向Kyn的转化增加, 并且Kyn/Trp的时间变化与患者总体生存期密切相关。因此, 检测血清Kyn/Trp变化用于患者分层, 对于肿瘤免疫治疗具有指导意义。
2.2 空间分辨代谢组学质谱成像(mass spectrometry imaging, MSI)是一种新型分子成像技术, 它不仅可同时获得生物体表面如细胞、组织等复杂基质中多肽、蛋白质、脂类、药物及其他小分子代谢物等多种内源性和外源性化合物的含量和空间分布信息, 还能够提供不同生理及病理过程中目标分子的动态变化以及分子结构信息[108, 109]。由于MSI灵敏度高、专属性好、无需特异性标记、所需时间短、耗费低、检测对象广, 可以对一些目标(已知)和非目标性(未知)的分子同时进行成像分析等特点和优势, 有望成为分子病理和分子诊断最为理想的检测工具, 因此在医学、生命科学和药学等领域具有重大应用前景而倍受国际上的关注[110]。
揭示生物组织中代谢物的空间分布特征和变化规律, 有助于理解代谢活动的时空变化与组织结构和生物功能的关联和分子机制。基于MSI的空间分辨代谢组学方法, 可获得整体动物或组织器官微区中上千代谢物空间分布信息, 并可直观呈现生理病理条件下的代谢途径的改变。然而, 代谢物的种类繁多、性质复杂以及生物组织的异质性使得有效地发现、挖掘真实的代谢组和代谢活动具有挑战性。MSI分析中的样品处理、数据采集、数据处理、信息挖掘及其生命科学领域应用等方面的不断进步, 促进了MSI空间分辨代谢组学的发展和完善。基于MSI的空间分辨代谢组学一般流程如图 2所示。
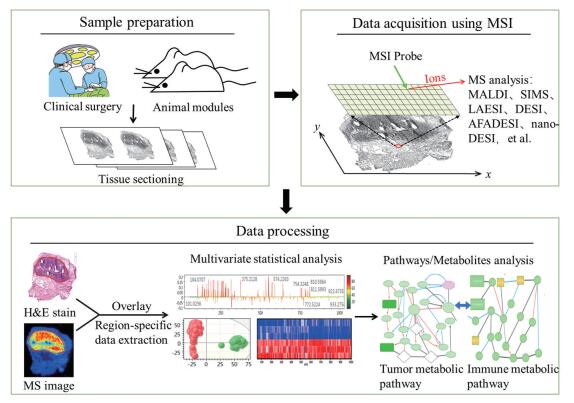
|
Figure 2 General procedure for spatial-resolved metabolomics using mass spectrometry imaging (MSI). MALDI: Matrix-assisted laser desorption/ionization; SIMS: Secondary ion mass spectrometry; LAESI: Laser ablation electrospray ionization; DESI: Desorption electrospray ionization; AFADESI: Air flow-assisted desorption electrospray ionization; MS: Mass spectrometry; H & E: Hematoxylin-eosin |
肿瘤的代谢异质性不仅体现在不同个体或癌症之间, 同种组织来源肿瘤的不同亚型, 甚至是同一肿瘤组织不同微区之间代谢特征也有差异, 这为肿瘤代谢的表征和研究带来了挑战。MSI空间分辨代谢组学能原位表征肿瘤组织中代谢物的变化, 为理解原位分子事件提供了新方法。Wang等[111]采用MALDI-FTICR-MSI, 利用两种互补基质, 检测到了前列腺癌组织中多达1 091个代谢物, 其中250种只在癌区被检测到, 217种只在非癌区被检测到, 并发现能量电荷增加和中性酰基甘油表达下调等明显代谢异常。Sun等[112]开发1, 1-二萘基-2, 2-二胺为MALDI-MSI新基质, 成功地描述了小鼠肺癌组织活组织区、坏死区和结缔组织区的大量代谢物的空间特征, 展示了肺癌组织的代谢异质性。本课题组建立了一种空间分辨代谢组学方法, 利用空气动力辅助解吸电喷雾离子化质谱成像技术筛选出一批在食管癌中异常表达的代谢物, 其中涉及了脯氨酸生物合成、多胺合成及代谢、精氨酸和脯氨酸代谢、脂肪酸合成、核苷代谢、组氨酸代谢等在内的多条代谢通路, 通过上下游分析及免疫组化验证, 发现并验证了GLS (图 3)、吡咯-5-羧酸还原酶2、尿苷磷酸化酶1等6个在食管癌中异常表达的代谢酶, 为理解肿瘤代谢重编程提供了新思路[113]。近期, Pareek等[114]利用代谢组学和气体簇离子束二次离子质谱成像技术揭示了HeLa细胞中嘌呤小体的存在和嘌呤从头合成途径, 嘌呤小体可能是高嘌呤需求的侵袭性癌症的代谢弱点, 该研究也说明了此成像技术在单细胞生物化学研究中有广阔应用前景。
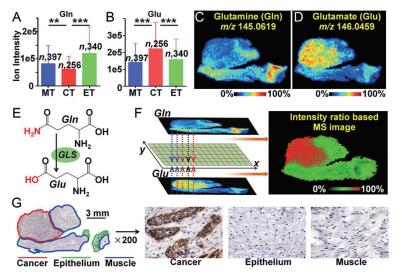
|
Figure 3 In situ visualization of crucial metabolites and metabolic enzyme in the glutamine metabolism pathway. A, B: Glutamine (Gln) and glutamate (Glu) levels in cancer and paired epithelium and muscle tissues from 256 ESCC patients (means ± SD). **P < 0.01; ***P < 0.001. C, D: MS images of Gln and Glu in ESCC tissue section. E: GLS-mediated metabolic process of converting Gln to Glu. F: The newly constructed MS image based on the ion-intensity ratio of Glu to Gln. G: Expression of GLS in different regions of the ESCC tissue section. CT: Cancer tissue; ET: Epithelial tissue; MT: Muscular tissue [113]. Reproduced from Ref.[113] with the permission of the National Academy of Sciences |
代谢流分析(metabolic flow analysis, MFA)是一种研究细胞内代谢反应速率和细胞与细胞外环境或细胞之间代谢物交换的技术, 通常利用稳定同位素标记某代谢物, 结合NMR或MS分析技术, 示踪其在体内的代谢过程, 得到代谢物在代谢通路中的动态变化[115]。以[1, 2-13C]葡萄糖为例(图 4), 当它进入糖酵解途径时, 产生M+2的糖酵解代谢物, 而当它进入磷酸戊糖途径时, 可以产生M+1的糖酵解代谢物, 比较同位素异数体含量, 可以得到两种途径对葡萄糖的消耗通量。而利用[U-13C]Gln可以测定Gln对TCA中间体的贡献[116]。其他稳定同位素2H、18O和15N等也可用于代谢流分析, 例如15N可用于研究氨基酸代谢等含有氮原子的代谢中间体的代谢途径[117]。MFA为阐明肿瘤代谢重编程和相关机制提供技术支持, 有利于研究营养依赖和发现治疗新靶点。Locasale等[118]利用NMR和LC-MS/MS检测肿瘤细胞中13C标记的葡萄糖向其他代谢物转化的时间过程, 发现大量葡萄糖通过磷酸甘油酸脱氢酶(phosphoglycerate dehydrogenase, PHGDH)转化为3-磷酸甘油酸后进入丝氨酸和甘氨酸合成途径, 降低PHGDH表达可以抑制肿瘤细胞增殖, PHGDH可能成为代谢治疗新靶点。Achreja等[119]基于13C-MFA成功预测了CAFs中代谢物的包装通量、癌细胞外泌体内化速率的动态变化以及代谢物随时间的释放通量, 该研究表明MFA有助于深入了解肿瘤细胞和其他细胞之间的代谢相互作用。Jin等[120]将MFA、代谢组学和转录组学整合分析, 发现了受体酪氨酸激酶激活引起的代谢偏好, 揭示了癌症基因型和代谢依赖的分子联系, 指导代谢抑制剂的个性化治疗。上述分析方法各有其优缺点(表 1[121, 122]), 多种分析方法的结合可以从静态、动态以及空间分布等方面更全面深入地理解肿瘤代谢重编程。
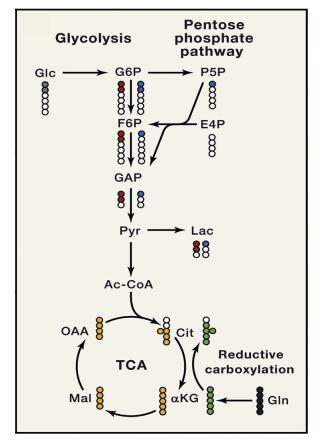
|
Figure 4 Atom mapping for [1, 2-13C]glucose tracing pentose phosphate pathway flux and for [U-13C]-glutamine tracing TCA flux. White balls are 12C atoms. Shaded balls are 13C atoms. Glycolysis, red; pentose phosphate pathway, blue; classical TCA turning, yellow; reductive carboxylation, green[116]. Reproduced from Ref. [116] with the permission of Elsevier |
| Table 1 The characteristics of three analytical method |
肿瘤代谢通过多种途径和信号通路影响或调节肿瘤微环境, 促进肿瘤免疫逃逸的发生。理解肿瘤代谢重编程及其机制以及肿瘤组织中不同类型细胞之间复杂的相互调控的代谢网络, 有助于发现代谢检查点和抗肿瘤的新药靶标。肿瘤细胞和免疫细胞通过代谢调控的相互作用是靶向代谢、增强抗肿瘤免疫功能的理论基础。基于代谢调控的肿瘤免疫治疗是解决当前免疫治疗存在问题的重要策略之一, 各种靶向代谢抑制剂和PD-1/PD-L1抗体等抗肿瘤免疫药物的联合疗法正处在临床前或临床试验阶段中, 为肿瘤免疫治疗带来了曙光。继续深入了解肿瘤免疫逃逸的代谢机制、发现代谢调控的新药靶标和抑制剂, 有望更好地实现代谢调控和免疫治疗的联合用药。
针对肿瘤组织代谢微环境的分析技术尚需进一步发展, 以满足肿瘤组织原位代谢表征需求。当前代谢组学研究大部分采用的是具有高效分离能力和强大分析能力的色谱质谱联用技术(LC-MS、GC-MS、CE-MS等), 它能够在单次分析中获得数千种内源性代谢物的信息, 在目前生物样品的代谢物分析中占有重要地位。然而色谱质谱联用分析方法一般是从血浆、血清、尿液等体液样本中获得生物体系整体水平的代谢表型, 其结果反映的是代谢物经过体循环后各组织器官混合、平均后的代谢水平, 甚至包含了病理状态所带来的营养失调, 与药物效应和疾病相关的代谢特异性或差异性会被一定程度地平均化、复杂化。组织器官作为大多数疾病的病灶部位(如实体肿瘤等)蕴含着最原始的代谢变化信息, 全面而准确地表征组织中代谢水平的变化, 能够为疾病的诊断、病理机制研究、药物治疗靶点发现提供重要的分子基础。尽管色谱质谱联用技术也能分析组织样本, 但是该技术方法需组织匀浆、代谢物提取、纯化和富集等前处理过程, 对于异质性较强的生物组织而言, 破坏了代谢物在组织中的空间分布信息, 无法实现代谢物的原位分析。
当前针对肿瘤免疫的代谢组学研究, 限于技术手段的局限性, 大多仅在体外细胞水平上进行, 无法真实反映肿瘤在生物组织中代谢微环境的客观状况。在组织层面的研究一般采用流式细胞技术分选出肿瘤细胞、免疫细胞等, 再研究其代谢物及其代谢通路水平的变化。对于肿瘤这种异质性和结构化非常强的组织, 分离后再分析的技术或方法会破坏细胞间的空间分布和相互作用信息; 另一方面, 免疫细胞是否处于肿瘤微环境中, 无论是在功能、代谢水平、蛋白水平、还是基因水平都存在着很大的差异。
综上所述, 如何从代谢水平深入理解肿瘤细胞的代谢重编程与免疫逃逸、药物的代谢调控、免疫细胞功能抑制与重激活以及三者之间的相互联系和相互影响是当前肿瘤药物免疫治疗研究需要解决的关键科学问题。如何真正反映并表征肿瘤细胞、基质细胞、免疫细胞与药物等在组织微区中的代谢状况及其相互作用和规律, 是当前肿瘤药物免疫治疗研究需要解决的关键技术难题。基于MSI的空间分辨代谢组学方法, 可从代谢组整体水平和组织原位层面开展深入研究, 可深入客观表征三者间的代谢调控和网络关联等关键信息, 有望发现和全面深入掌握肿瘤细胞的代谢重编程与免疫逃逸、药物的代谢调控、免疫细胞功能抑制与重激活的关键代谢通路。从而有望发现新型肿瘤免疫治疗的代谢检查点和抗肿瘤新药靶标。
作者贡献:王相宜查阅文献、撰写文章、修改文章; 张锦查阅文献、撰写文章、指导文章修改; 李燕指导文章修改; 贺玖明指导撰写思路和文章修改。
利益冲突:无利益冲突。
| [1] |
Van Der Burg SH, Arens R, Ossendorp F, et al. Vaccines for established cancer:overcoming the challenges posed by immune evasion[J]. Nat Rev Cancer, 2016, 16: 219-233. DOI:10.1038/nrc.2016.16 |
| [2] |
Swann JB, Smyth MJ. Immune surveillance of tumors[J]. J Clin Invest, 2007, 117: 1137-1146. DOI:10.1172/JCI31405 |
| [3] |
Zhang X, Cao XT. Recent advances in the mechanism of tumor immune escape[J]. Chin J Cancer Biother (中国肿瘤生物治疗杂志), 2001, 8: 72-74. DOI:10.1038/74955 |
| [4] |
Hanahan D, Robert A. Hallmarks of cancer:the next generation[J]. Cell, 2011, 144: 646-674. DOI:10.1016/j.cell.2011.02.013 |
| [5] |
Sanmamed MF, Chen L. A paradigm shift in cancer immunotherapy:from enhancement to normalization[J]. Cell, 2018, 175: 313-326. DOI:10.1016/j.cell.2018.09.035 |
| [6] |
Oiseth S, Aziz M. Cancer immunotherapy:a brief review of the history, possibilities, and challenges ahead[J]. J Cancer Metastasis Treat, 2017, 3: 250-261. DOI:10.20517/2394-4722.2017.41 |
| [7] |
Warburg O, Wind F, Negelein E. The metabolism of tumors in the body[J]. J Gen Physiol, 1927, 8: 519-530. DOI:10.1085/jgp.8.6.519 |
| [8] |
Furuta E, Okuda H, Kobayashi A, et al. Metabolic genes in cancer:their roles in tumor progression and clinical implications[J]. Biochim Biophys Acta, 2010, 1805: 141-152. DOI:10.1016/j.bbcan.2010.01.005 |
| [9] |
Linehan WM, Srinivasan R, Schmidt LS. The genetic basis of kidney cancer:a metabolic disease[J]. Nat Rev Urol, 2010, 7: 277-285. DOI:10.1038/nrurol.2010.47 |
| [10] |
Lehuédé C, Dupuy F, Rabinovitch R, et al. Metabolic plasticity as a determinant of tumor growth and metastasis[J]. Cancer Res, 2016, 76: 5201-5208. DOI:10.1158/0008-5472.CAN-16-0266 |
| [11] |
Liu JY, Ren LW, Li S, et al. Research progress of tumor immune and tumor metabolic drug targets[J]. Acta Pharm Sin (药学学报), 2019, 54: 1718-1727. |
| [12] |
Kaufman KL, Mactier S, Armstrong NJ, et al. Surface antigen profiles of leukocytes and melanoma cells in lymph node metastases are associated with survival in AJCC stage Ⅲ melanoma patients[J]. Clin Exp Metastasis, 2014, 31: 407-421. DOI:10.1007/s10585-014-9636-7 |
| [13] |
Fang T, Lv H, Lv G, et al. Tumor-derived exosomal miR-1247-3p induces cancer-associated fibroblast activation to foster lung metastasis of liver cancer[J]. Nat Commun, 2018, 9: 191. DOI:10.1038/s41467-017-02583-0 |
| [14] |
Covarrubias AJ, Aksoylar HI, Yu J, et al. Akt-mTORC1 signaling regulates Acly to integrate metabolic input to control of macrophage activation[J]. eLife, 2016, 5:e11612. DOI:10.7554/eLife.11612 |
| [15] |
Lakins MA, Ghorani E, Munir H, et al. Cancer-associated fibroblasts induce antigen-specific deletion of CD8+ T Cells to protect tumour cells[J]. Nat Commun, 2018, 9: 948. DOI:10.1038/s41467-018-03347-0 |
| [16] |
Xie F, Liang R, Li D, et al. Regulatory T cells and their clinical applications in antitumor immunotherapy[J]. Engineering, 2019, 5: 132-139. DOI:10.1016/j.eng.2018.12.002 |
| [17] |
Miller BC, Sen DR, Al Abosy R, et al. Subsets of exhausted CD8+ T cells differentially mediate tumor control and respond to checkpoint blockade[J]. Nat Immunol, 2019, 20: 326-336. DOI:10.1038/s41590-019-0312-6 |
| [18] |
Park SL, Buzzai A, Rautela J, et al. Tissue-resident memory CD8+ T cells promote melanoma-immune equilibrium in skin[J]. Nature, 2019, 565: 366-371. DOI:10.1038/s41586-018-0812-9 |
| [19] |
Sung AP, Tang JJJ, Guglielmo MJ, et al. An improved method to quantify human NK cell-mediated antibody-dependent cell-mediated cytotoxicity (ADCC) per IgG FcR-positive NK cell without purification of NK cells[J]. J Immunol Methods, 2018, 452: 63-72. DOI:10.1016/j.jim.2017.11.002 |
| [20] |
Colegio OR, Chu N, Szabo AL, et al. Functional polarization of tumour-associated macrophages by tumour-derived lactic acid[J]. Nature, 2014, 513: 559-563. DOI:10.1038/nature13490 |
| [21] |
Casazza A, Laoui D, Wenes M, et al. Impeding macrophage entry into hypoxic tumor areas by Sema3A/Nrp1 signaling blockade inhibits angiogenesis and restores antitumor immunity[J]. Cancer Cell, 2013, 24: 695-709. DOI:10.1016/j.ccr.2013.11.007 |
| [22] |
Shiao SL, Ganesan AP, Rugo HS, et al. Immune microenvironments in solid tumors:new targets for therapy[J]. Genes Dev, 2011, 25: 2559-2572. DOI:10.1101/gad.169029.111 |
| [23] |
Konjević GM, Vuletić AM, Mirjačić Martinović KM, et al. The role of cytokines in the regulation of NK cells in the tumor environment[J]. Cytokine, 2019, 117: 30-40. DOI:10.1016/j.cyto.2019.02.001 |
| [24] |
Wang J, Sanmamed MF, Datar I, et al. Fibrinogen-like protein 1 is a major immune inhibitory ligand of LAG-3[J]. Cell, 2019, 176: 334-347. DOI:10.1016/j.cell.2018.11.010 |
| [25] |
Gautam S, Fioravanti J, Zhu W, et al. The transcription factor c-Myb regulates CD8+ T cell stemness and antitumor immunity[J]. Nat Immunol, 2019, 20: 337-349. DOI:10.1038/s41590-018-0311-z |
| [26] |
Zhang Q, Bi J, Zheng X, et al. Blockade of the checkpoint receptor TIGIT prevents NK cell exhaustion and elicits potent anti-tumor immunity[J]. Nat Immunol, 2018, 19: 723-732. DOI:10.1038/s41590-018-0132-0 |
| [27] |
Advani R, Flinn I, Popplewell L, et al. CD47 blockade by Hu5F9-G4 and rituximab in non-Hodgkin's lymphoma[J]. N Engl J Med, 2018, 379: 1711-1721. DOI:10.1056/NEJMoa1807315 |
| [28] |
Ahn J, Xia T, Rabasa Capote A, et al. Extrinsic phagocyte-dependent STING signaling dictates the immunogenicity of dying cells[J]. Cancer Cell, 2018, 33: 862-873. DOI:10.1016/j.ccell.2018.03.027 |
| [29] |
Galon J, Bruni D. Approaches to treat immune hot, altered and cold tumours with combination immunotherapies[J]. Nat Rev Drug Discov, 2019, 18: 197-218. DOI:10.1038/s41573-018-0007-y |
| [30] |
Guo ZH, Cao XT. The status and prospect of tumor immune cell therapy[J]. Chin J Cancer Biother (中国肿瘤生物治疗杂志), 2016, 23: 6-17. DOI:10.3872/j.issn.1007-385X.2016.02.001 |
| [31] |
Motzer RJ, Tannir NM, Mcdermott DF, et al. Nivolumab plus ipilimumab versus sunitinib in advanced renal-cell carcinoma[J]. N Engl J Med, 2018, 378: 1277-1290. DOI:10.1056/NEJMoa1712126 |
| [32] |
Pacheco JM. KEYNOTE-407:changing the way we treat stage Ⅳ squamous non-small cell lung cancer[J]. Transl Lung Cancer Res, 2020, 9: 148-153. DOI:10.21037/tlcr.2020.01.12 |
| [33] |
Qian HL. The immune checkpoint inhibitors:opportunities and challenges of tumor immunotherapy[J]. Oncol Prog (癌症进展), 2017, 15: 1363-1364. DOI:10.1002/cncy.21951 |
| [34] |
Zaretsky JM, Garcia Diaz A, Shin DS, et al. Mutations associated with acquired resistance to PD-1 blockade in melanoma[J]. N Engl J Med, 2016, 375: 819-829. DOI:10.1056/NEJMoa1604958 |
| [35] |
Riddell SR. Adrenaline fuels a cytokine storm during immunotherapy[J]. Nature, 2018, 564: 194-196. DOI:10.1038/d41586-018-07581-w |
| [36] |
Baitsch L, Baumgaertner P, Devêvre E, et al. Exhaustion of tumor-specific CD8+ T cells in metastases from melanoma patients[J]. J Clin Invest, 2011, 121: 2350-2360. DOI:10.1172/JCI46102 |
| [37] |
Frey AB, Monu N. Suppression of proximal T cell receptor signaling and lytic function in CD8+ tumor-infiltrating T cells[J]. Cancer Res, 2007, 67: 11447-11454. DOI:10.1158/0008-5472.CAN-07-1441 |
| [38] |
Koukourakis MI, Giatromanolaki A, Harris AL, et al. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas:a metabolic survival role for tumor-associated stroma[J]. Cancer Res, 2006, 66: 632-637. DOI:10.1158/0008-5472.CAN-05-3260 |
| [39] |
Taddei ML, Giannoni E, Comito G, et al. Microenvironment and tumor cell plasticity:an easy way out[J]. Cancer Lett, 2013, 341: 80-96. DOI:10.1016/j.canlet.2013.01.042 |
| [40] |
Yoshida G. Metabolic reprogramming:the emerging concept and associated therapeutic strategies[J]. J Exp Clin Cancer Res, 2015, 34: 111. DOI:10.1186/s13046-015-0221-y |
| [41] |
Li L, Kou JJ, Du WJ. Metabolic reprogramming in cancer and its potential clinical implications[J]. Chin J Front Med Sci (中国医学前沿杂志电子版), 2019, 11: 28-37. |
| [42] |
Deberardinis RJ, Lum JJ, Hatzivassiliou G, et al. The biology of cancer:metabolic reprogramming fuels cell growth and proliferation[J]. Cell Metab, 2008, 7: 11-20. DOI:10.1016/j.cmet.2007.10.002 |
| [43] |
Sun Y. Tumor microenvironment and cancer therapy resistance[J]. Cancer Lett, 2015, 380: 205-215. DOI:10.1158/1078-0432.CCR-11-0768 |
| [44] |
Renner K, Singer K, Koehl GE, et al. Metabolic hallmarks of tumor and immune cells in the tumor microenvironment[J]. Front Immunol, 2017, 8: 248. DOI:10.1007/s10120-016-0642-0 |
| [45] |
Chang CH, Qiu J, O'sullivan D, et al. Metabolic competition in the tumor microenvironment is a driver of cancer progression[J]. Cell, 2015, 162: 1229-1241. DOI:10.1016/j.cell.2015.08.016 |
| [46] |
Zhou Y, Niu W, Luo Y, et al. p53/Lactate dehydrogenase A axis negatively regulates aerobic glycolysis and tumor progression in breast cancer expressing wild-type p53[J]. Cancer Sci, 2019, 110: 939-949. DOI:10.1111/cas.13928 |
| [47] |
Cascone T, Mckenzie JA, Mbofung RM, et al. Increased tumor glycolysis characterizes immune resistance to adoptive T cell therapy[J]. Cell Metab, 2018, 27: 977-987. DOI:10.1016/j.cmet.2018.02.024 |
| [48] |
Husain Z, Huang Y, Seth P, et al. Tumor-derived lactate modifies antitumor immune response:effect on myeloid-derived suppressor cells and NK cells[J]. J Immunol, 2013, 191: 1486-1495. DOI:10.4049/jimmunol.1202702 |
| [49] |
Villalba M, Rathore MG, Lopez Royuela N, et al. From tumor cell metabolism to tumor immune escape[J]. Int J Biochem Cell Biol, 2013, 45: 106-113. DOI:10.1016/j.biocel.2012.04.024 |
| [50] |
Hirschhaeuser F, Sattler UG, Mueller-Klieser W. Lactate:a metabolic key player in cancer[J]. Cancer Res, 2011, 71: 6921-6925. DOI:10.1158/0008-5472.CAN-11-1457 |
| [51] |
Colegio OR. Lactic acid polarizes macrophages to a tumor-promoting state[J]. Oncoimmunology, 2016, 5:e1014774. DOI:10.1080/2162402X.2015.1014774 |
| [52] |
Zhang WN, Wang GH, Xu ZG, et al. Lactate is a natural suppressor of RLR signaling by targeting MAVS[J]. Cell, 2019, 178: 176-189. DOI:10.1016/j.cell.2019.05.003 |
| [53] |
Payen VL, Mina E, Van Hee VF, et al. Monocarboxylate transporters in cancer[J]. Mol Metab, 2019, 33: 48-66. DOI:10.1016/j.bbamcr.2016.03.013 |
| [54] |
Mendler AN, Hu B, Prinz PU, et al. Tumor lactic acidosis suppresses CTL function by inhibition of p38 and JNK/c-Jun activation[J]. Int J Cancer, 2012, 131: 633-640. DOI:10.1002/ijc.26410 |
| [55] |
Brown TP, Ganapathy V. Lactate/GPR81 signaling and proton motive force in cancer:role in angiogenesis, immune escape, nutrition, and Warburg phenomenon[J]. Pharmacol Ther, 2020, 206: 107451. DOI:10.1016/j.pharmthera.2019.107451 |
| [56] |
Lund K, Trauelsen M, Pedersen SF, et al. Why Warburg works:lactate controls immune evasion through GPR81[J]. Cell Metab, 2020, 31: 666-668. DOI:10.1016/j.cmet.2020.03.001 |
| [57] |
Kim SH, Roszik J, Grimm EA, et al. Impact of L-arginine metabolism on immune response and anticancer immunotherapy[J]. Front Oncol, 2018, 8: 67. DOI:10.3389/fonc.2018.00067 |
| [58] |
Timosenko E, Hadjinicolaou AV, Cerundolo V. Modulation of cancer-specific immune responses by amino acid degrading enzymes[J]. Immunotherapy, 2016, 9: 83-97. |
| [59] |
Malek E, De Lima M, Letterio JJ, et al. Myeloid-derived suppressor cells:the green light for myeloma immune escape[J]. Blood Rev, 2016, 30: 341-348. DOI:10.1016/j.blre.2016.04.002 |
| [60] |
Vanichapol T, Chutipongtanate S, Anurathapan U, et al. Immune escape mechanisms and future prospects for immunotherapy in neuroblastoma[J]. Biomed Res Int, 2018, 2018: 1812535. DOI:10.1155/2018/1812535 |
| [61] |
Geiger R, Rieckmann JC, Wolf T, et al. L-Arginine modulates T cell metabolism and enhances survival and anti-tumor activity[J]. Cell, 2016, 167: 829-842. DOI:10.1016/j.cell.2016.09.031 |
| [62] |
Sikalidis AK. Amino acids and immune response:a role for cysteine, glutamine, phenylalanine, tryptophan and arginine in T-cell function and cancer?[J]. Pathol Oncol Res, 2015, 21: 9-17. DOI:10.1007/s12253-014-9860-0 |
| [63] |
Edwards DN, Ngwa VM, Wang S, et al. The receptor tyrosine kinase EphA2 promotes glutamine metabolism in tumors by activating the transcriptional coactivators YAP and TAZ[J]. Sci Signal, 2017, 10:eaan4667. |
| [64] |
Zhang T, Liu J, Ding Y. Research advances of glutamine metabolism-related targets in tumor treatment[J]. Acta Pharm Sin (药学学报), 2020, 55: 813-820. |
| [65] |
Ma G, Liang Y, Chen Y, et al. Glutamine deprivation induces PD-L1 expression via activation of EGFR/ERK/c-Jun signaling in renal cancer[J]. Mol Cancer Res, 2020, 18: 324-339. DOI:10.1158/1541-7786.MCR-19-0517 |
| [66] |
Fu Q, Xu L, Wang Y, et al. Tumor-associated macrophage-derived interleukin-23 interlinks kidney cancer glutamine addiction with immune evasion[J]. Eur Urol, 2019, 75: 752-763. DOI:10.1016/j.eururo.2018.09.030 |
| [67] |
Brochez L, Chevolet I, Kruse V. The rationale of indoleamine 2, 3-dioxygenase inhibition for cancer therapy[J]. Eur J Cancer, 2017, 76: 167-182. DOI:10.1016/j.ejca.2017.01.011 |
| [68] |
Greene LI, Bruno TC, Christenson JL, et al. A role for tryptophan-2, 3-dioxygenase in CD8 T-cell suppression and evidence of tryptophan catabolism in breast cancer patient plasma[J]. Mol Cancer Res, 2019, 17: 131-139. DOI:10.1158/1541-7786.MCR-18-0362 |
| [69] |
Wu H, Gong J, Liu Y. Indoleamine 2, 3-dioxygenase regulation of immune response[J]. Mol Med Rep, 2018, 17: 4867-4873. DOI:10.3892/mmr.2018.8537 |
| [70] |
Botticelli A, Cerbelli B, Lionetto L, et al. Can IDO activity predict primary resistance to anti-PD-1 treatment in NSCLC?[J]. J Transl Med, 2018, 16: 219. DOI:10.1186/s12967-018-1595-3 |
| [71] |
Pour S, Morikawa H, Kiani NA, et al. Immunometabolic network interactions of the kynurenine pathway in cutaneous malignant melanoma[J]. Front Oncol, 2020, 10: 51. DOI:10.3389/fonc.2020.00051 |
| [72] |
Kang JS. Dietary restriction of amino acids for cancer therapy[J]. Nutr Metab (Lond), 2020, 17: 20. DOI:10.1186/s12986-020-00439-x |
| [73] |
Ananieva EA, Powell JD, Hutson SM. Leucine metabolism in T cell activation:mTOR signaling and beyond[J]. Adv Nutr, 2016, 7: 798S-805S. DOI:10.3945/an.115.011221 |
| [74] |
Hashimoto H, Mccallion O, Kempkes RWM, et al. Distinct metabolic pathways mediate regulatory T cell differentiation and function[J]. Immunol Lett, 2020, 223: 53-61. DOI:10.1016/j.imlet.2020.04.011 |
| [75] |
Shafei Ma FT, Davis J. BCATc modulates crosstalk between the PI3K/Akt and the Ras/ERK pathway regulating proliferation in triple negative breast cancer[J]. Oncotarget, 2020, 11: 1971-1987. DOI:10.18632/oncotarget.27607 |
| [76] |
Tolba MF. Revolutionizing the landscape of colorectal cancer treatment:the potential role of immune checkpoint inhibitors[J]. Int J Cancer, 2020. DOI:10.1002/ijc.33056 |
| [77] |
Gaber T, Strehl C, Buttgereit F. Metabolic regulation of inflammation[J]. Nat Rev Rheumatol, 2017, 13: 267-279. DOI:10.1038/nrrheum.2017.37 |
| [78] |
Zhou Y, Su W, Liu H, et al. Fatty acid synthase is a prognostic marker and associated with immune infiltrating in gastric cancers precision medicine[J]. Biomark Med, 2020, 14: 185-199. DOI:10.2217/bmm-2019-0476 |
| [79] |
Jiang L, Fang X, Wang H, et al. Ovarian cancer-intrinsic fatty acid synthase prevents anti-tumor immunity by disrupting tumor-infiltrating dendritic cells[J]. Front Immunol, 2018, 9: 2927. DOI:10.3389/fimmu.2018.02927 |
| [80] |
Gouw AM, Margulis K, Liu NS, et al. The MYC oncogene cooperates with sterol-regulated element-binding protein to regulate lipogenesis rssential for neoplastic growth[J]. Cell Metab, 2019, 30: 556-572. DOI:10.1016/j.cmet.2019.07.012 |
| [81] |
Lin R, Zhang H, Yuan Y, et al. Fatty acid oxidation controls CD8+ tissue-resident memory T-cell survival in gastric adenocarcinoma[J]. Cancer Immunol Res, 2020, 8: 479-492. DOI:10.1158/2326-6066.CIR-19-0702 |
| [82] |
Luengo A, Gui DY, Vander Heiden MG. Targeting metabolism for cancer therapy[J]. Cell Chem Biol, 2017, 24: 1161-1180. DOI:10.1016/j.chembiol.2017.08.028 |
| [83] |
Hashemzadeh N, Adibkia K, Barar J. Indoleamine 2, 3-dioxygenase inhibitors in immunochemotherapy of breast cancer:challenges and opportunities[J]. BioImpacts, 2019, 9: 1-3. DOI:10.15171/bi.2019.01 |
| [84] |
Zakharia Y, Rixe O, Ward JH, et al. Phase 2 trial of the IDO pathway inhibitor indoximod plus checkpoint inhibition for the treatment of patients with advanced melanoma[J]. J Clin Oncol, 2018, 36: 9512. DOI:10.1200/JCO.2018.36.15_suppl.9512 |
| [85] |
Jung KH, Lorusso P, Burris H, et al. Phase I study of the indoleamine 2, 3-dioxygenase 1(IDO1) inhibitor navoximod (GDC-0919) administered with PD-L1 inhibitor (atezolizumab) in advanced solid tumors[J]. Clin Cancer Res, 2019, 25: 3220-3228. DOI:10.1158/1078-0432.CCR-18-2740 |
| [86] |
Long GV, Dummer R, Hamid O, et al. Epacadostat plus pembrolizumab versus placebo plus pembrolizumab in patients with unresectable or metastatic melanoma (ECHO-301/KEYNOTE-252):a phase 3, randomised, double-blind study[J]. Lancet Oncol, 2019, 20: 1083-1097. DOI:10.1016/S1470-2045(19)30274-8 |
| [87] |
Hornigold N, Dunn KR, Craven RA, et al. Dysregulation at multiple points of the kynurenine pathway is a ubiquitous feature of renal cancer:implications for tumour immune evasion[J]. Br J Cancer, 2020, 123: 137-147. DOI:10.1038/s41416-020-0874-y |
| [88] |
Ray A, Song Y, Du T, et al. Targeting tryptophan catabolic kynurenine pathway enhances antitumor immunity and cytotoxicity in multiple myeloma[J]. Leukemia, 2020, 34: 567-577. DOI:10.1038/s41375-019-0558-x |
| [89] |
Leone RD, Zhao L, Englert JM, et al. Glutamine blockade induces divergent metabolic programs to overcome tumor immune evasion[J]. Science, 2019, 366: 1013-1021. DOI:10.1126/science.aav2588 |
| [90] |
Oh MH, Sun IH, Zhao L, et al. Targeting glutamine metabolism enhances tumor-specific immunity by modulating suppressive myeloid cells[J]. J Clin Invest, 2020, 130: 3865-3884. DOI:10.1172/JCI131859 |
| [91] |
Raczka AM, Reynolds PA. Glutaminase inhibition in renal cell carcinoma therapy[J]. Cancer Drug Resist, 2019, 2: 356-364. DOI:10.20517/cdr.2018.004 |
| [92] |
Tripathi SC, Fahrmann JF, Vykoukal JV, et al. Targeting metabolic vulnerabilities of cancer:small molecule inhibitors in clinic[J]. Cancer Rep, 2019, 2:e1131. DOI:10.1002/cnr2.1131 |
| [93] |
Li X, Wenes M, Romero P, et al. Navigating metabolic pathways to enhance antitumour immunity and immunotherapy[J]. Nat Rev Clin Oncol, 2019, 16: 425-441. DOI:10.1038/s41571-019-0203-7 |
| [94] |
Gill KS, Fernandes P, O'donovan TR, et al. Glycolysis inhibition as a cancer treatment and its role in an anti-tumour immune response[J]. Biochim Biophys Acta, 2016, 1866: 87-105. |
| [95] |
Mezquita L, Auclin E, Ferrara R, et al. Association of the lung immune prognostic index with immune checkpoint inhibitor outcomes in patients with advanced non-small cell lung cancer[J]. JAMA Oncol, 2018, 4: 351-357. DOI:10.1001/jamaoncol.2017.4771 |
| [96] |
Feichtinger RG, Lang R. Targeting L-lactate metabolism to overcome resistance to immune therapy of melanoma and other tumor entities[J]. J Oncol, 2019, 2019: 2084195. DOI:10.1371/journal.pone.0040159 |
| [97] |
Yang W, Bai Y, Xiong Y, et al. Potentiating the antitumour response of CD8+ T cells by modulating cholesterol metabolism[J]. Nature, 2016, 531: 651-655. DOI:10.1038/nature17412 |
| [98] |
Pan J, Zhang Q, Palen K, et al. Potentiation of Kras peptide cancer vaccine by avasimibe, a cholesterol modulator[J]. EBioMedicine, 2019, 49: 72-81. DOI:10.1016/j.ebiom.2019.10.044 |
| [99] |
Wishart DS. Emerging applications of metabolomics in drug discovery and precision medicine[J]. Nat Rev Drug Discov, 2016, 15: 473-484. DOI:10.1038/nrd.2016.32 |
| [100] |
Li T, Deng P. Nuclear magnetic resonance technique in tumor metabolism[J]. Genes Dis, 2017, 4: 28-36. DOI:10.1016/j.gendis.2016.12.001 |
| [101] |
Giskeodegard GF, Madssen TS, Euceda LR, et al. NMR-based metabolomics of biofluids in cancer[J]. NMR Biomed, 2019, 32:e3927. DOI:10.1021/acs.jproteome.9b00343 |
| [102] |
Zheng H, Dong B, Ning J, et al. NMR-based metabolomics analysis identifies discriminatory metabolic disturbances in tissue and biofluid samples for progressive prostate cancer[J]. Clin Chim Acta, 2020, 501: 241-251. DOI:10.1016/j.cca.2019.10.046 |
| [103] |
Lin Y, Ma C, Bezabeh T, et al. 1H NMR-based metabolomics reveal overlapping discriminatory metabolites and metabolic pathway disturbances between colorectal tumor tissues and fecal samples[J]. Int J Cancer, 2019, 145: 1679-1689. DOI:10.1002/ijc.32190 |
| [104] |
Nicholson JK, Lindon JC. Metabonomics[J]. Nature, 2008, 455: 1054-1056. DOI:10.1038/4551054a |
| [105] |
Kibi M, Nishiumi S, Kobayashi T, et al. GC/MS and LC/MS-based tissue metabolomic analysis detected increased levels of antioxidant metabolites in colorectal cancer[J]. Kobe J Med Sci, 2019, 65: 19-27. |
| [106] |
Jing L, Guigonis JM, Borchiellini D, et al. LC-MS based metabolomic profiling for renal cell carcinoma histologic subtypes[J]. Sci Rep, 2019, 9: 15635. DOI:10.1038/s41598-019-52059-y |
| [107] |
Li H, Bullock K, Gurjao C, et al. Metabolomic adaptations and correlates of survival to immune checkpoint blockade[J]. Nat Commun, 2019, 10: 4346. DOI:10.1038/s41467-019-12361-9 |
| [108] |
Caprioli, Richard M. Imaging mass spectrometry:molecular microscopy for the new age of biology and medicine[J]. Proteomics, 2016, 16: 1607-1612. DOI:10.1002/pmic.201600133 |
| [109] |
Caprioli RM, Farmer TB, Gile J. Molecular imaging of biological samples:localization of peptides and proteins using MALDI-TOF MS[J]. Anal Chem, 1997, 69: 4751-4760. DOI:10.1021/ac970888i |
| [110] |
Takáts Z, Wiseman JM, Gologan B, et al. Mass spectrometry sampling under ambient conditions with desorption electrospray ionization[J]. Science, 2004, 306: 471-473. DOI:10.1126/science.1104404 |
| [111] |
Wang XT, Jun H, Darryl BH, et al. Metabolomic profiling of prostate cancer by matrix assisted laser desorption/ionization-Fourier transform ion cyclotron resonance mass spectrometry imaging using matrix coating assisted by an electric field (MCAEF)[J]. Biochim Biophys Acta Proteins Proteom, 2016, 1865: 755-767. |
| [112] |
Sun C, Liu W, Mu Y, et al. 1, 1'-Binaphthyl-2, 2'-diamine as a novel MALDI matrix to enhance the in situ imaging of metabolic heterogeneity in lung cancer[J]. Talanta, 2020, 209: 120557. DOI:10.1016/j.talanta.2019.120557 |
| [113] |
Sun C, Li T, Song X, et al. Spatially resolved metabolomics to discover tumor-associated metabolic alterations[J]. Proc Natl Acad Sci U S A, 2019, 116: 52-57. DOI:10.1073/pnas.1808950116 |
| [114] |
Pareek V, Tian H, Winograd N, et al. Metabolomics and mass spectrometry imaging reveal channeled de novo purine synthesis in cells[J]. Science, 2020, 368: 283-290. DOI:10.1126/science.aaz6465 |
| [115] |
Dai Z, Locasale JW. Understanding metabolism with flux analysis:from theory to application[J]. Metab Eng, 2017, 43: 94-102. DOI:10.1016/j.ymben.2016.09.005 |
| [116] |
Jang C, Chen L, Rabinowitz JD. Metabolomics and isotope tracing[J]. Cell, 2018, 173: 822-837. DOI:10.1016/j.cell.2018.03.055 |
| [117] |
Antoniewicz MR. A guide to 13C metabolic flux analysis for the cancer biologist[J]. Exp Mol Med, 2018, 50: 19. DOI:10.1038/s12276-018-0060-y |
| [118] |
Locasale JW, Grassian AR, Melman T, et al. Phosphoglycerate dehydrogenase diverts glycolytic flux and contributes to oncogenesis[J]. Nat Genet, 2011, 43: 869-874. DOI:10.1038/ng.890 |
| [119] |
Achreja A, Zhao H, Yang L, et al. Exo-MFA-A 13C metabolic flux analysis framework to dissect tumor microenvironment-secreted exosome contributions towards cancer cell metabolism[J]. Metab Eng, 2017, 43: 156-172. DOI:10.1016/j.ymben.2017.01.001 |
| [120] |
Jin N, Bi A, Lan X, et al. Identification of metabolic vulnerabilities of receptor tyrosine kinases-driven cancer[J]. Nat Commun, 2019, 10: 2701. DOI:10.1038/s41467-019-10427-2 |
| [121] |
Fernández García J, Altea Manzano P, Pranzini E, et al. Stable isotopes for tracing mammalian-cell metabolism in vivo[J]. Trends Biochem Sci, 2020, 45: 185-201. DOI:10.1016/j.tibs.2019.12.002 |
| [122] |
Radenkovic S, Vuckovic I, Lanza IR. Metabolic flux analysis:moving beyond static metabolomics[J]. Trends Biochem Sci, 2020, 45: 545-546. DOI:10.1016/j.tibs.2020.02.011 |
 2020, Vol. 55
2020, Vol. 55


