2. 北京中医药大学中药学院中药资源与鉴定系, 北京 100029
2. Department of Resources and Identification of Traditional Chinese Medicine, School of Chinese Materia Medica, Beijing University of Chinese Medicine, Beijing 100029, China
乙烯响应因子(ethylene-response factor, ERF)是属于AP2/ERF家族的亚家族, 是植物特有的一个转录因子家族, 位于乙烯信号转导途径的下游, 含有高度保守的AP2区域, 该区域是由58或59个氨基酸组成, 可形成1个α-螺旋和3个反平行链构成的β-折叠结构[1]。ERF基因与乙烯诱导基因启动子的GCC-box结合, 其过表达能够促进乙烯合成反应, 因此ERF基因是乙烯合成反应的转录调控因子[2]。最近的研究表明植物中的ERF基因在植物防御反应过程中起到重要作用。如在小麦感染白粉病早期, TaERF3通过激活防御基因的表达, 提高植物对病原菌的抗性[3]。在拟南芥中, ERF6和ERF11共同维持植物生长和抵御胁迫之间的平衡[4, 5]。在大豆中, GmERF75基因表达水平受到干旱和盐胁迫的诱导, 过表达GmERF75增强植物抗盐性[6]。野生大豆中, ERF71通过上调H+-ATP酶及改变植物生长素的积累, 增强植物对碱性胁迫的抗性[7, 8]。
白木香(Aquilaria sinensis)是我国名贵药材沉香的唯一正品药用植物来源, 目前已经被列为《濒危野生动植物种国际贸易公约》 (CITES)附录II[9]和国家濒危二级保护植物[10]。沉香是我国传统名贵中药, 具有行气止痛、温中止呕、纳气平喘等功效, 对于胸腹胀闷疼痛、胃寒呕吐呃逆、肾虚气逆喘急等有显著疗效[11]。沉香的临床应用广泛, 具有消化系统和中枢神经系统保护等药理作用。沉香是独有龙涎香和檀香混合物香味的一种香料, 融合动物界与植物界的精华, 至今无法复制这种香味, 因而极其珍贵[12]。健康的白木香并不产生沉香, 只有通过自然因素(雷劈、火烧等)或者人为因素(砍伤、接菌等)的作用才能够形成沉香, 但是沉香形成的分子机制一直没有揭示, 严重制约高效人工结香技术的建立[13]。近年来研究表明, 利用真菌提取物、盐胁迫、干旱、伤害胁迫能够诱导白木香的悬浮细胞和愈伤组织产生沉香特征性成分2-苯乙基色酮类化合物和倍半萜类化合物[14-18], 因此白木香的防御反应在沉香形成过程中起到重要作用。研究白木香中ERF的生物信息学、细胞定位、蛋白原核表达、在非生物胁迫下的表达有利于丰富ERF基因在植物防御反应中的重要作用研究, 进一步阐明白木香的结香机制。
材料与方法材料 白木香种苗是2019年6月从广州移植, 现种植于北京中医药大学中药现代研究中心温箱, 成活后于2019年6月采集白木香的根、茎、茎尖及叶提取总RNA, 检测AsERF1基因在不同器官中的特异性表达。利用白木香叶片和茎尖诱导的愈伤组织按照本课题组以前研究进行[16], 选取长势相同的白木香愈伤组织, 分别经NaCl (150 mmol·L-1)、低温(4 ℃)、CdCl2 (500 μmol·L-1)、甘露醇(200 mmol·L-1) 4种非生物胁迫处理[19, 20], 在处理后0、12、24、36、48 h提取RNA作为样品检测AsERF1基因在各种处理下的表达差异。所用菌种为大肠杆菌(Escherichia coli) DH5α、BL21购自天根生物科技有限公司; pCAMBIA1300-35S-EGFP购自武汉转导生物实验室有限公司。
白木香根、茎、叶及愈伤组织中RNA的提取和cDNA的合成 按照植物RNA快速提取试剂盒(Omega, 中国)实验操作步骤进行植物总RNA提取, 利用NanoDrop 2000C检测RNA浓度, 同时利用1.2%琼脂糖凝胶电泳检测RNA的完整性和质量。利用Sigma公司的反转录酶M-MLV将白木香总RNA反转录为第一链(cDNA), 反转录条件按照说明书进行。
白木香AsERF1基因序列全长克隆 从白木香转录组高通量测序结果中获得1个由表达序列标签拼接而成的序列, 经注释分析发现为具有完整开放阅读框的AsERF1基因, 依据两端序列利用Primer软件设计引物AsERF1-1和AsERF1-2, 序列见表 1。以白木香总RNA的反转录产物为模板, 按照下列体系对白木香中AsERF1基因进行扩增: cDNA 1 μL, 10×LA-Taq buffer 5 μL, dNTP Mix (2.5 mmol·L-1) 4 μL, LA-Taq (2.5 U·μL-1) 0.5 μL, 10 μmol引物各1 μL, 终体积为50 μL。反应程序: 94 ℃预变性5 min; 然后进行30个循环, 94 ℃ 1 min, 55 ℃ 30 s, 72 ℃ 1 min, 循环结束后72 ℃延伸反应10 min, 4 ℃保存。1%的琼脂糖凝胶电泳检测PCR产物, 凝胶成像分析系统检测, 目的片段回收和纯化按照胶回收试剂盒说明书进行。将回收后的PCR产物与pMD19-T连接, 转化到DH5α菌株, 在氨苄抗性的平板上进行筛选, 并经过菌落PCR检测后送上海英潍捷基公司测序。
| Table 1 Primer sequence |
白木香AsERF1生物信息学分析 通过在线软件ProtParam预测蛋白结构, 分析目的基因编码蛋白质的氨基酸组成、蛋白质相对分子质量、理论等电点及稳定性等参数; 通过ExPASY中的SOPMA工具分析蛋白质序列的二级结构; 利用SWISS-MODEL Workspace在线分析软件构建蛋白质三级结构模型; 利用软件TMHMM 2.0进行蛋白质跨膜结构分析; 将所获得的AsERF1基因编码的氨基酸序列在GenBank数据库中进行Blast P对比分析, 利用DNAMAN对其他物种AsERF1基因编码的氨基酸序列进行同源性分析; 通过MEGA 6.0软件构建Neighbor-joining系统进化树, 进化距离的计算采用泊松校正法, Bootstrap重复次数为1 000次。
白木香AsERF1的蛋白表达 利用引物AsERF1-3和AsERF1-4扩增AsERF1基因的全长(表 1)。利用Bam HI和Xoh I双酶切包含有目的基因的PCR产物及表达载体pET-28a, 酶切产物经过纯化后, 利用T4连接酶进行连接, 连接产物转化大肠杆菌DH5α感受态细胞, 经菌落PCR、测序后提取质粒pET28a-AsERF1, 将质粒转化大肠杆菌BL21(DE3)感受态细胞。挑取单菌落接种于含有卡那霉素的LB液体培养基中, 37 ℃活化过夜, 然后按照1:100比例加入新鲜的含有卡那霉素的LB液体培养基中, 37 ℃振荡培养至OD600为0.4~0.6, 调节温度为16 ℃, 加入IPTG后振荡培养24 h。之后4 ℃、12 000 r·min-1离心收集大肠杆菌, 悬浮于40 mmol·L-1 KPB缓冲液(pH 7.9, 含有100 mmol·L-1 NaCl, 5 mmol·L-1咪唑)中。将菌体放置于冰上, 利用超声破碎仪(Colo Parmer)破碎细胞, 之后4 ℃、12 000 r·min-1离心30 min以上。取上清液, 利用镍离子亲和色谱纯化AsERF1蛋白, 以20 mmol·L-1 KPB缓冲液(pH 7.9, 含有500 mmol·L-1 NaCl和40 mmol·L-1咪唑)洗脱目的蛋白, 利用SDS-PAGE检测重组蛋白表达。
白木香AsERF1的亚细胞定位 利用引物AsERF1-5和AsERF1-6 (表 1)扩增AsERF1基因全长, 利用BamHI和Sal I双酶切包含有目的基因AsERF1的PCR产物及双元载体pCAMBIA1300-35S-EGFP, 酶切产物纯化后, 利用T4 DNA连接酶, 将酶切PCR产物与酶切载体连接, 连接产物转入大肠杆菌DH5ɑ感受态细胞, 挑取经过菌落PCR、测序鉴定正确的单克隆, 提取质粒pCAMBIA1300-35S-EGFP-AsERF1, 将重组质粒转化农杆菌EHA105感受态细胞, 28 ℃培养2~3天后进行菌落PCR, 以确定pCAMBIA1300-35S-EGFP-AsERF1成功转入农杆菌中。将含有重组质粒的农杆菌菌液注射本生烟叶片, 2~3天后利用激光共聚焦显微镜检测本生烟叶片表皮细胞中融合蛋白的分布, 同时利用DAPI进行细胞核染色。
白木香AsERF1基因在不同组织和不同胁迫处理下的表达分析 利用实时荧光定量PCR (quantitative real-time PCR, qRT-PCR)的方法检测白木香AsERF1基因在不同组织中和不同胁迫处理下的表达情况。分析使用SYBR Green I荧光染料法, 在qRT-PCR仪上进行。选取白木香GAPDH基因作为目标基因定量表达的内参基因, 引物序列见表 1。每个样品设3个重复, 重复3次。反应体系中含有10 μL STBR Premix Ex Taq酶, 上下游引物(10 μmol·L-1)各0.4 μL, 模板0.5 μL, 总体系为20 μL。反应程序是: 95 ℃预变性3 min, 95 ℃变性30 s, 60 ℃退火/延伸30 s (每次循环后采集荧光), 40个循环后, 95 ℃变性10 s, 65~95 ℃做熔解曲线分析, 每个温度以每步0.5 ℃上升, 每个温度停留5 s。根据熔解曲线判断RT-PCR产物的特异性, 相对定量分析采用2-ΔΔCt方法进行分析。
结果与讨论 1 白木香AsERF1基因全长cDNA的克隆根据白木香愈伤组织的转录组测序结果, 以白木香愈伤组织的cDNA为模板进行扩增, 利用RT-PCR方法扩增后得到约700 bp片段, 扩增结果见图 1, 将PCR产物连接到pMD19-T载体上, 测序结果经过NCBI的BLAST比对, 确定该扩增产物是ERF基因的全长, 基因命名为AsERF1, 其序列长度为691 bp, 编码229个氨基酸。
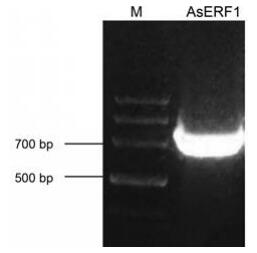
|
Figure 1 PCR amplification of AsERF1. M: DNA marker |
通过Protparam软件预测AsERF1基因编码的蛋白理化性质。推测AsERF1编码的蛋白分子式为C1120H1749N319O343S14, 相对分子质量为25 363.50, 理论等电点为7.75, 不稳定系数为63.97, 属于不稳定蛋白, 总亲水性为-0.548, 为亲水性蛋白。利用TMHMM2.0预测白木香AsERF1蛋白没有跨膜区域。
2.2 AsERF1蛋白的二级结构分析及三维结构预测运用ExPASY中的SOPMA工具预测AsERF1基因编码蛋白的二级结构, 结果显示AsERF1蛋白的二级结构是由60.70%的随机卷曲(random coil)、24.45%的α-螺旋(ɑ-helices)、7.86%的延伸链(extended strand)和6.99%的β-折叠(β-turn)组成(图 2A), 推测随机卷曲是其最多二级结构元件, 而α-螺旋、延伸链和β-折叠散布于整个蛋白中。
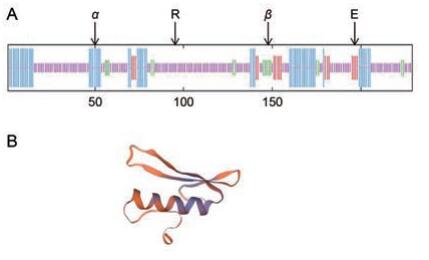
|
Figure 2 Predicted secondary and three-dimensional structure of AsERF1 protein.A. Predicted secondary structure of AsERF1 protein with SOPMA; B. The deduced three-dimensional structure of AsERF1 protein. α: α-Helices; β: β-Turn; E: Extended strand; R: Random coil |
将白木香AsERF1的氨基酸序列通过SWISS-MODEL Workspace在线分析软件建立了AsERF1的三维结构模型, 选择拟南芥中AtERF1的结构模型为模板, 对白木香AsERF1的三维结构进行预测, 结果表明, 白木香AsERF1与拟南芥中AsERF1的相似性为97.1%, 具有参考价值。具体三维结构如图 2B所示。
2.3 白木香AsERF1氨基酸序列分析和系统进化树分析将白木香AsERF1氨基酸序列与GenBank中其他植物中的ERF氨基酸序列进行比对, 通过DNAMAN软件与多种植物ERF氨基酸序列进行多序列比对分析(图 3), 发现AsERF1含有保守的AP2区域, 与其他植物ERF蛋白的AP2区域相似性非常高, 在AP2区域含有一个保守的WLG元件和一个YRG元件, 氨基酸序列符合典型的AP2/ERF转录因子特征。多重序列比对发现白木香AsERF1与毛果杨(Populus trichocarpa)、棉花(Gossypium arboretum)、可可树(Theobroma cacao)、木槿(Hibiscus syriacus)、拟南芥(Arabidopsis thaliana) ERF氨基酸序列相似性分别为60.14%、58.15%、53.20%、59.85%、27.27%, 说明AsERF1氨基酸序列除了AP2保守区域, 与其他植物ERF蛋白的氨基酸序列同源性不高。为进一步了解白木香AsERF1蛋白在植物ERF蛋白家族中的进化位置, 选取了其他植物的20条ERF蛋白序列, 利用MEGA6.0构建系统进化树(图 4)。从图 4可以看出白木香AsERF1与毛果杨ERF2蛋白的亲缘关系较近。
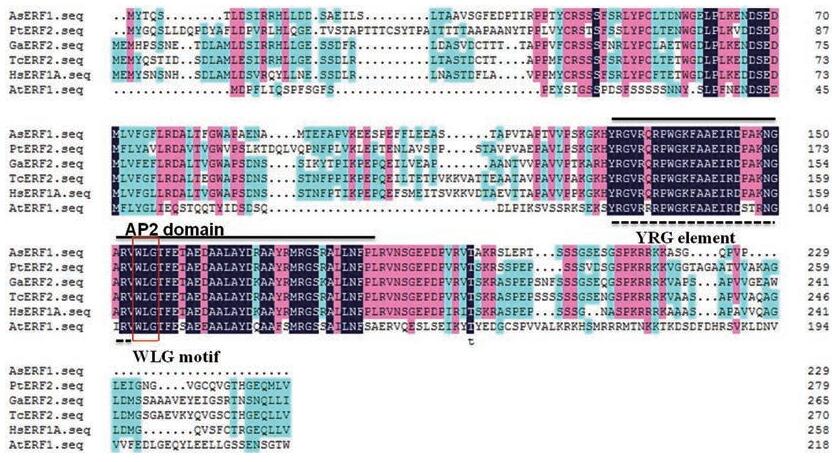
|
Figure 3 Multiple sequence alignment of AsERF1 and ERF from other plants. Black shading indicates amino acid identities, red and blue shading indicates amino acid with different similarity. The conserved AP2 domain of ERF proteins are shown with black line. The conserved YRG element and WLG motif of ERF proteins are shown with the dotted line and red box, respectively. As: Aquilaria sinensis; Pt: Populus trichocarpa; Ga: Gossypium arboretum; Tc: Theobroma cacao; Hs: Hibiscus syriacus; At: Arabidopsis thaliana |
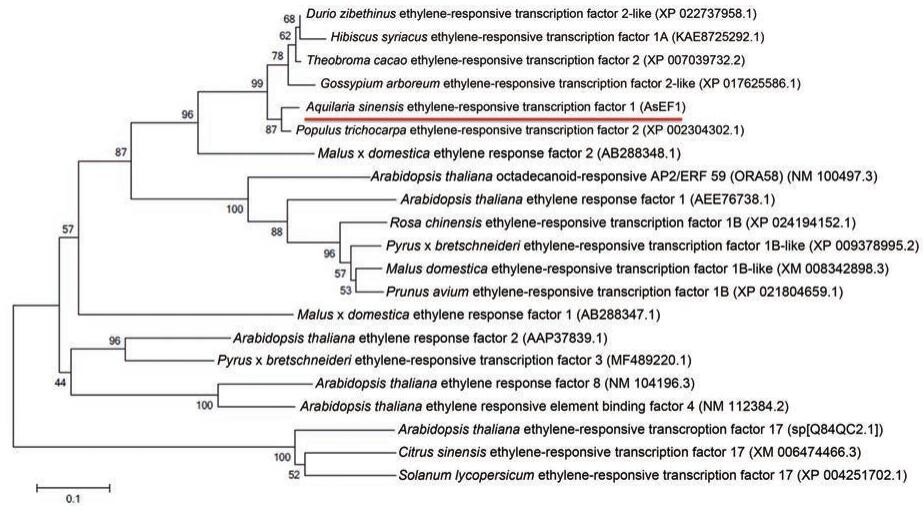
|
Figure 4 Phylogenetic analysis of ethylene-response factor (ERF) proteins from plants |
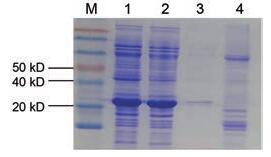
将原核表达载体pET28a-AsERF1质粒转化大肠杆菌BL21(DE3)后, 通过预实验探索及SDS-PAGE电泳检测重组蛋白是否可溶并确定最佳诱导条件, 使用最佳浓度0.8 mmol·L-1的IPTG于16 ℃诱导24 h的最佳诱导条件进行蛋白的原核表达诱导。由于重组AsERF1蛋白的氨基酸序列N端带有一个6个His (组氨酸)的标签, 通过镍离子亲和色谱纯化AsERF1蛋白, 利用SDS-PAGE检测发现在25 kD左右出现一条AsERF1蛋白纯化条带(图 5)。
|
Figure 5 SDS-PAGE analysis of recombinant AsERF1 protein. M: Marker; 1: Insoluble fraction from the induced E.coli containing pET28a-AsERF1; 2: Soluble protein from induced E. coli containing pET28a-AsERF1; 3: The purified recombinant AsERF1 protein; 4: Uninduced E. coli containing pET28a-AsERF1 |
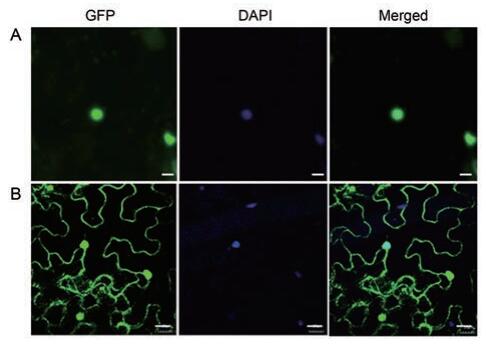
将含有pCAMBIA1300-35S-EGFP-AsERF和pCAMBIA1300-35S-EGFP农杆菌注射本生烟叶片, 进行瞬时表达。2~3天后利用激光共聚焦显微镜检测本生烟表皮细胞中融合蛋白的分布, 同时利用4, 6-二脒基-2-苯基吲哚(DAPI)进行细胞核染色, 结果显示GFP: AsERF1融合蛋白主要分布于细胞核中, 而含有gfp基因的pCAMBIA1300-35S-EGFP载体注射后荧光分布在细胞核、细胞质和细胞壁上(图 6), 这些结果表明AsERF1主要定位于植物细胞的细胞核中。
|
Figure 6 Subcellular localization of pCAMBIA1300-35S-EGFP-AsERF fusion proteins in Nicotiana benthamiana leaves. A: pCAMBIA1300-35S-EGFP-AsERF; B: pCAMBIA1300-35S-EGFP. Bars = 10 μm |
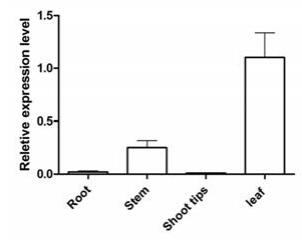
利用荧光定量PCR检测AsERF1基因的组织特异性表达, 结果显示, AsERF1在所有的组织中均有表达, 在叶中表达量最高, 茎次之, 在根和茎尖中表达量很低(图 7)。
|
Figure 7 Relative expression level of AsERF1 gene in different tissues. Repeat 3 samples, each for 3 times |
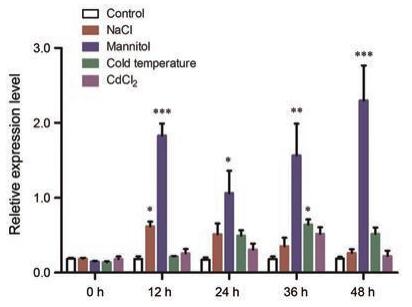
为验证白木香AsERF1基因在植物防御反应中的作用, 对白木香愈伤组织分别进行盐、干旱、低温及重金属处理, 以相同生长情况下未进行任何处理的愈伤组织作为对照, 不同时间点取样提取RNA后进行实时荧光定量PCR分析, 检测AsERF1的表达水平。盐胁迫下, AsERF1的表达水平在12 h内显著升高, 12 h表达量达到最高, 是对照该基因表达量的3.0倍, 随后表达量缓慢降低(图 8)。甘露醇诱导产生的干旱胁迫下, AsERF1的表达出现双峰现象, 在12 h内显著升高, 是对照该基因表达水平的10.1倍, 随后表达水平降低, 在48 h再次显著上升, 达到最高, 是对照该基因表达水平的12.2倍(图 8)。冷胁迫和重金属胁迫下, AsERF1的表达水平在36 h内逐渐升高, 并在36 h达到最高表达量, 分别是对照表达量的3.5倍和2.8倍, 随后逐渐降低(图 8)。这些实验结果表明盐、干旱、低温和重金属胁迫均可诱导AsERF1基因的表达, 其中干旱胁迫对AsERF1基因的表达水平影响最显著。
|
Figure 8 Relative expression level of AsERF1 gene under different abiotic stresses in Aquilaria sinensis Calli.*P < 0.05; **P < 0.01;***P < 0.001 vs Control.Repeat 3 samples, each for 3 times |
APETALA2/ethylene-responsive element binding factor (AP2/ERF)是植物中一个特有的转录因子家族, 该转录因子家族通常含有AP2/ERF保守结构域[21, 22], 基于序列同源性和AP2保守结构域将其分为5个亚家族, 即AP2、RAV、DREB、ERF和其他一些基因家族[23]。ERF转录因子在植物信号转导、生长发育和胁迫响应中发挥重要作用。目前已经在多种植物中研究了ERF家族基因的功能, 但是关于白木香中ERF基因研究的尚未见报道。本研究根据NCBI的序列和愈伤组织转录组测序结果分析, 通过RT-PCR从白木香愈伤组织中分离得到AsERF1的全长cDNA序列。AsERF1含有AP2的保守序列, 但是除了AP2保守区域, AsERF1与其他物种ERF蛋白的同源性不高。首次在大肠杆菌中表达了白木香的AsERF1蛋白, 希望能够在蛋白水平上研究白木香中AsERF1的生物学功能。通过构建原核表达载体pET28a-AsERF1在BL21(DE3)菌株中诱导表达白木香AsERF1蛋白, 重组蛋白能够以可溶性蛋白存在, 为进一步研究AsERF1的功能奠定基础。
目前发现大部分植物ERF蛋白定位于植物的细胞核中。例如大麦的ERF蛋白HvRAF定位于植物的细胞核中[24]。将土豆中ERF3构建到含有GFP的双元表达载体上, 烟草瞬时表达系统结果表明其定位于植物细胞核[25]。将含有大豆GmERF75: GFP的双元载体农杆菌转染洋葱表皮细胞, 发现融合蛋白定位于洋葱表皮细胞的细胞核中, 并根据以前报道的细胞核定位保守序列K(K/R)X(K/R)推测其核定位序列为KPVKRQRK[26, 27]。本研究将AsERF1构建到含有GFP的双元表达载体, 利用烟草瞬时表达系统实验证明AsERF1主要定位于细胞核, 根据进核蛋白核定位信号的保守序列推断AsERF1含有可能的核定位信号序列为KRRKKA。
基因在不同组织中的表达模式可为基因功能研究提供初步线索[28]。ERF在不同植物中具有不同的表达模式。在梨中, 大部分的ERF基因能够在梨成熟过程中不同阶段表达, 少部分的ERF基因在各组织中均有表达[29]。芝麻中80% ERF基因在植物各组织中均有表达, 但是表达模式各不相同[30]。大麦中的ERF基因HvRAF在各组织中均有表达, 主要分布于植物的根中[24]。本研究表明白木香AsERF1在植物各组织中均有表达, 主要表达在植物的叶中, 可能在叶生长发育过程中起到重要作用。
白木香只有在受到各种胁迫伤害产生防御反应后才能够产生沉香, 因此研究白木香响应胁迫的分子机制对于阐明沉香的结香机制具有重要意义。近期研究结果表明ERF转录因子在植物防御反应过程起到重要作用。小麦TaERF1表达水平能够被干旱、盐、低温诱导, 在拟南芥和烟草中过表达TaERF1能够提高植物对多种非生物胁迫的抗性[31]。大豆GmERF9能够被干旱和盐胁迫的诱导, 过表达GmERF9烟草具有抗旱性和抗盐性[32]。拟南芥中过表达大麦ERF基因HvRAF能够提高植物对病原菌的抗性和抗盐性[24]。烟草中ERF基因JERF1受到盐和低温胁迫的诱导, 过表达能够提高植物的抗盐和低温能力[33]。虽然植物中很多ERF在植物防御反应中的功能被研究, 但是白木香中的ERF基因功能还没报道。盐、干旱、低温和重金属胁迫是植物生长过程中最常见的非生物胁迫, 本研究发现, AsERF1能够被盐、干旱、低温及重金属胁迫诱导, 盐、干旱和低温均能够显著影响AsERF1的表达水平, 并且干旱胁迫对AsERF1基因的表达水平影响最为显著。因此AsERF1在白木香防御反应过程中起到重要作用, 很可能是植物抵御干旱胁迫的重要基因。AsERF1基因的蛋白表达、细胞定位及在非生物胁迫下的表达分析为进一步研究AsERF1在植物胁迫应答和白木香结香过程中的作用机制奠定基础。
作者贡献:李铁铮主要负责AsERF1基因克隆、原核表达、表达分析; 郑一哲主要负责AsERF1生物信息学分析、亚细胞定位和不同胁迫下的表达分析; 戎玉清负责不同胁迫下的表达分析及实验数据的整理; 魏胜利为本研究提供实验指导并对论文初稿进行修改; 王晓晖和屠鹏飞负责设计论文的实验思路, 指导学生实验, 论文撰写及修改。
利益冲突:无任何利益冲突。
| [1] |
Kuluev B, Avalbaev A, Nurgaleeva E, et al. Role of aintegumenta-like gene NtANTL in the regulation of tobacco organ growth[J]. J Plant Physiol, 2015, 189: 11-23. DOI:10.1016/j.jplph.2015.08.009 |
| [2] |
Jofuku KD, Omidyar PK, Gee Z, et al. Control of seed mass and seed yield by the floral homeotic gene APETALA2[J]. Proc Natl Acad Sci U S A, 2005, 102: 3117-3122. DOI:10.1073/pnas.0409893102 |
| [3] |
Rong W, Qi L, Wang AY, et al. The ERF transcription factor TaERF3 promotes tolerance to salt and drought stresses in wheat[J]. Plant Biotechnol J, 2014, 12: 468-479. DOI:10.1111/pbi.12153 |
| [4] |
Dubois M, Van den Broeck L, Claeys H, et al. The ethylene response factors ERF6 and ERF11 antagonistically regulate mannitol-induced growth inhibition in Arabidopsis[J]. Plant Physiol, 2015, 169: 166-179. DOI:10.1104/pp.15.00335 |
| [5] |
Koyama T, Nii H, Mitsuda N, et al. A regulatory cascade involving class Ⅱ ethylene response factor transcriptional repressors operates in the progression of leaf senescence[J]. Plant Physiol, 2013, 162: 991-1005. DOI:10.1104/pp.113.218115 |
| [6] |
Zhao MJ, Yin LJ, Liu Y, et al. The ABA-induced soybean ERF transcription factor gene GmERF75 plays a role in enhancing osmotic stress tolerance in Arabidopsis and soybean[J]. BMC Plant Biol, 2019, 19: 506. DOI:10.1186/s12870-019-2066-6 |
| [7] |
Yu Y, Duan XB, Ding XD, et al. A novel AP2/ERF family transcription factor from Glycine soja, GsERF71, is a DNA binding protein that positively regulates alkaline stress tolerance in Arabidopsis[J]. Plant Mol Biol, 2017, 94: 509-530. DOI:10.1007/s11103-017-0623-7 |
| [8] |
Yao G, Ming M, Allan AC, et al. Map-based cloning of the pear gene MYB114 identifies an interaction with other transcription factors to coordinately regulate fruit anthocyanin biosynthesis[J]. Plant J, 2017, 92: 437-451. DOI:10.1111/tpj.13666 |
| [9] |
CITES. Amendments to Appendix I and Ⅱ of CITES[C]. In Proceedings of Thirteenth Meeting of the Conference of the Parties, Bangkok, Thailand, 2004: 2-14.
|
| [10] |
State Environmental Protection Administration of China. Institute of Botany, the Chinese Academy of Sciences. List of Rare and Endangered Plants in China (Vol 1) (中国珍稀濒危保护植物名录:第一册)[M]. 1987.
|
| [11] |
Chinese Pharmacopoeia Commission.Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. Vol 1. Beijing: China Medical Science Press, 2015.
|
| [12] |
Li G, Duan LS, Yang CY, et al. Advances in studies ontechnology of agarwood formation in Aquilaria sinensis[J]. J Anhui Agric Sci (安徽农业科学), 2009, 37: 12012-12013. |
| [13] |
Zhang Z, Yang Y, Wei JH, et al. Advances in studies on mechanism of agarwood formation in Aquilaria sinensis and its hypothesis of agarwood formation induced by defense response[J]. Chin Tradit Herb Drugs (中草药), 2010, 41: 156-159. |
| [14] |
Xu Y, Zhang Z, Wang M, et al. Identification of genes related to agarwood formation:transcriptome analysis of healthy and wounded tissues of Aquilaria sinensis[J]. BMC Genomics, 2013, 14: 227-243. DOI:10.1186/1471-2164-14-227 |
| [15] |
Qi SY, He ML, Lin LD, et al. Production of 2-(2-phenylethyl) chromones in cell suspension cultures of Aquilaria sinensis[J]. Plant Cell Tiss Org, 2005, 83: 217-221. DOI:10.1007/s11240-005-5479-x |
| [16] |
Wang XH, Gao BW, Liu X, et al. Salinity stress induces the production of 2-(2-phenylethyl) chromones and regulates novel classes of responsive genes involved in signal transduction in Aquilaria sinensis Calli[J]. BMC Plant Biol, 2016, 16: 119. DOI:10.1186/s12870-016-0803-7 |
| [17] |
Wang XH, Dong XJ, Feng YY, et al. H2O2 and NADPH oxidases involve in regulation of 2-(2-phenylethyl) chromones accumulation during salt stress in Aquilaria sinensis Calli[J]. Plant Sci, 2018, 269: 1-11. DOI:10.1016/j.plantsci.2018.01.002 |
| [18] |
Dong XJ, Gao BW, Feng YY, et al. Production of 2-(2-phenylethyl) chromones in Aquilaria sinensis Calli under different treatments[J]. Plant Cell Tiss Org, 2018, 135: 53-62. DOI:10.1007/s11240-018-1442-5 |
| [19] |
Dong XJ, Feng YY, Liu X, et al. Cloning and expression analysis of S-adenosylmethionine synthetase gene from Aquilaria sinensis[J]. Acta Pharm Sin (药学学报), 2018, 53: 1743-1752. |
| [20] |
Dong XJ, Li TZ, Feng YY, et al. Cloning and expression analysis of allene oxide cyclase gene from Aquilaria sinensis[J]. Acta Pharm Sin (药学学报), 2018, 53: 467-475. |
| [21] |
Zhuang J, Chen JM, Yao QH, et al. Discovery and expression profile analysis of AP2/ERF family genes from Triticum aestivum[J]. Mol Biol Rep, 2017, 38: 745-753. |
| [22] |
Liu MY, Sun MJ, Ma ZT, et al. Genome-wide investigation of AP2/ERF gene family in tartary buckwheat (Fagopyum tatarium)[J]. BMC Plant Biol, 2019, 19: 84. DOI:10.1186/s12870-019-1681-6 |
| [23] |
Sakuma Y, Liu Q, Dubouzet JG, et al. DNA-binding specificity of the ERF/AP2 domain of Arabidopsis DREBs, transcription factors involved in dehydration- and cold-inducible gene expression[J]. Biochem Biophys Res Commun, 2002, 290: 998-1009. DOI:10.1006/bbrc.2001.6299 |
| [24] |
Jung JW, Won SY, Suh SC, et al. The barley ERF-type transcription factor HvRAF confers enhanced pathogen resistance and salt tolerance in Arabidopsis[J]. Planta, 2007, 225: 575-588. DOI:10.1007/s00425-006-0373-2 |
| [25] |
Liu MC, Pirrello JL, Kesari R, et al. A dominant repressor version of the tomato Sl-ERF.B3 gene confers ethylene hypersensitivity via feedback regulation of ethylene signaling and response components[J]. Plant J, 2013, 76: 406-419. DOI:10.1111/tpj.12305 |
| [26] |
Lange A, Mills RE, Lange CJ, et al. Classical nuclear localization signals:definition, function, and interaction with importin[J]. J Biol Chem, 2007, 282: 5101-5105. DOI:10.1074/jbc.R600026200 |
| [27] |
Kusugi S, Hasebe M, Matsumura N, et al. Six classes of nuclear localization signals specific to different binding grooves of importin[J]. J Biol Chem, 2009, 284: 478-485. DOI:10.1074/jbc.M807017200 |
| [28] |
Li YM, Zhang N, Shen X, et al. Identification and expression analysis of four CIPKs genes froma rare and medicinal plant, Dendrobium officinale[J]. Acta Pharm Sin (药学学报), 2018, 53: 304-312. |
| [29] |
Li XL, Tao ST, Wei SW, et al. The mining and evolutionary investigation of AP/ERF genes in pears (Pyrus)[J]. BMC Plant Biol, 2018, 18: 46. DOI:10.1186/s12870-018-1265-x |
| [30] |
Dossa K, Wei X, Li DH, et al. Insight into the AP2/ERF transcription factor superfamily in sesame and expression profiling of DREB subfamily under drought stress[J]. BMC Plant Biol, 2016, 16: 171. DOI:10.1186/s12870-016-0859-4 |
| [31] |
Xu ZS, Xia LQ, Ma YZ, et al. Isolation and molecular characterization of the Triticum aestivum L ethylene-responsive factor 1 TaERF1 that increases multiple stress tolerance[J]. Plant Mol Biol, 2007, 65: 719-732. DOI:10.1007/s11103-007-9237-9 |
| [32] |
Zhai Y, Shao SL, Sha W, et al. Over expression of soybean GmERF9 enhances the tolerance to drought and cold in the transgenic tobacco[J]. Plant Cell Tiss Org, 2017, 129: 607-618. |
| [33] |
Wu LJ, Chen XL, Ren HY, et al. ERF protein JERF1 that transcriptionally modulates the expression of abscisic acid biosynthesis-related gene enhances the tolerance under salinity and cold in tobacco[J]. Planta, 2007, 226: 815-825. DOI:10.1007/s00425-007-0528-9 |
 2020, Vol. 55
2020, Vol. 55


