心力衰竭(heart failure, HF)是多种原因导致的心脏, 包括心包、心肌、心内膜、心脏瓣膜、血管结构或功能障碍, 或代谢异常等引起的临床综合征[1]。HF是一种多病因、复杂性疾病, 其复杂性表现在两个方面, 首先, 几乎所有的心血管疾病, 如心肌病、高血压、心律失常、冠心病等最终都会导致HF的发生; 其次, HF属高异质性的进展性疾病, 任何触发因素, 如心肌梗死(myocardial infarction, MI)、化疗、基因突变或妊娠等, 可能使部分个体发生HF。HF是21世纪心血管疾病领域面临的最严重挑战之一, 其死亡率高于部分恶性肿瘤。《中国心血管病报告2018》的数据显示, 我国心血管病患病人数约2.9亿, 其中HF患者有450万[2]。伴随人口老龄化日益加剧, 冠心病、高血压、糖尿病、肥胖等慢性病发病的持续上升, 预计到2030年, HF患者将增加1倍。目前, HF的治疗方法虽然得到了改进和完善, 但其预后还是很差, 患者的再住院率和死亡率仍居高不下[3-5]。进一步揭示HF的发病机制、寻找特异性生物标志物、完善现有治疗方案和探索新的治疗靶点是目前HF的主要研究方向。
近年来, 组学技术在HF的研究中得到了广泛应用, 包括基因组学技术、转录组学技术、表观基因组学技术、蛋白质组学技术及代谢组学技术等。不同组学技术集各分子层面高通量检测及多元统计分析于一体, 可以较为全面地认识给定生物系统受内外因的影响所产生的生物学过程的变化, 在医药学领域应用进展显著。
本文将综合概述多种组学在HF的研究成果, 为临床HF患者早期诊断、危险度分层、治疗指导和预后判断提供客观、全面的信息支持。
1 HF研究现状及困境 1.1 HF时心肌结构和功能变化HF发生时心肌收缩力减弱, 心功能及血流动力学等多项参数均发生改变, 包括心输出量减少、射血分数降低、左室舒张末压升高、心室收缩和舒张功能出现障碍。
HF早、中期出现心肌肥厚, 即细胞内收缩成分等代偿性增多, 是心室对压力负荷过重或缺氧的一种适应性反应。HF晚期表现为进行性恶化, 成纤维细胞增殖、心肌细胞外基质(extracellular matrix, ECM)胶原增多、胶原网破坏、心肌组织纤维化、血管壁细胞增殖。此时, 心室重量增加、形态结构改变, 进而加剧心脏收缩及舒张功能障碍, 这在临床上称为心室重构过程。
1.2 HF发生的分子机制HF发病过程中, 心脏的神经内分泌调节机制发生变化, 主要包括交感神经系统的激活、肾素-血管紧张素-醛固酮系统(renin-angiotensin-aldosterone system, RAAS)的激活及其他神经内分泌变化, 如精氨酸加压素、内皮素、利钠肽类等。HF最早且最常见的变化是交感神经系统的激活, 其长期激活可使心肌β受体信号转导发生变化: β1受体下调、β1受体与兴奋性Gs蛋白脱耦联、G蛋白耦联受体激酶活性增加[6]。
此外, 线粒体融合与分裂、氧化应激、Ca2+稳态的维持也在HF发生发展过程中发挥着一定的作用。现代医学普遍认为HF是遗传、神经激素、代谢、炎症和其他作用于心脏的生化因素之间复杂相互作用的结果。
1.3 HF治疗现状HF是慢性、进展性疾病, 需从预防、治疗、预后进行全面控制, 包括药物治疗、改变生活习惯、采用机械辅助装置及心脏移植等。在药物治疗方面, β受体阻滞剂、血管紧张素转换酶抑制剂(angiotensin converting enzyme inhibitors, ACEI)、血管紧张素受体拮抗剂(angiotensin receptor blockers, ARB)可改变衰竭心肌的生物学性质, 延缓心肌重构, 从而改善患者预后; 利尿剂需用于液体潴留的HF患者; 葡萄糖-胰岛素-钾盐、曲美他嗪、左旋肉毒碱等在临床上作为辅助代谢疗法用于改善HF患者的心肌能量代谢; 中成药如麝香保心丸、芪苈强心胶囊等可改善HF病人生活质量和心功能。
循证医学研究结果显示, 药物治疗能有效降低HF死亡率。但2017年美国心脏病学会公布的大数据表明, 近10年来HF患者1年死亡率仍然在29.3%左右, 5年死亡率高达52.4%, 与20年前的数据相比, HF患者死亡率无下降趋势。导致HF死亡风险高的原因在于HF具有不同的危险因素、病因及病理生理机制[7]。目前, 仅根据左心室射血分数(left ventricular ejection fraction, LVEF)值, 分类为射血分数保留的HF (heart failure with preserved ejection fraction, HFpEF)、射血分数中间值的HF (heart failure with mid-range ejection fraction, HFmrEF)、射血分数降低的HF (heart failure with reduced ejection fraction, HFrEF), 缺乏病因诊断; 其次几乎所有患者均使用同一指南方案且都归结为目前的几个病理生理学机制, 缺乏个体化特点; 最后, 在用药方面, 遗传背景不同的患者可能存在不同的药物代谢特点。因此, 患者在当前的诊疗方式下难以获得精准治疗。
2 多种组学简介基因组学、转录组学、表观基因组学、蛋白质组学、代谢组学在HF研究中发挥着越来越重要的作用[8]。组学技术的迅速发展为HF的研究提供了新的平台(图 1)。
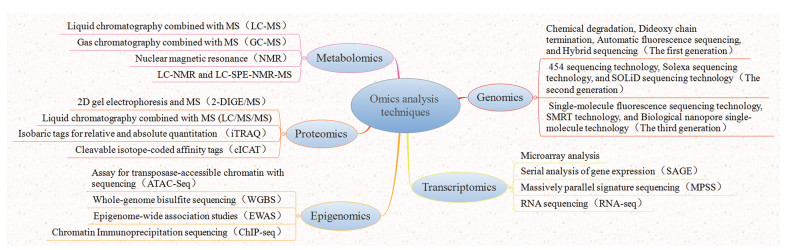
|
图 1 Multiple omics technologies |
目前, 基因组学研究方法有外显子测序技术和全基因组关联分析(genome-wide association study, GWAS)技术; 转录组学技术以RNA-seq为主, 其主要通过高通量测序研究特定组织或细胞在某个时期转录出来的mRNA的表达量; 表观基因组学研究技术有ATAC-Seq、WGBS、EWAS、ChIP-seq等; 蛋白质组分析主要使用二维凝胶电泳和液相色谱与质谱联用技术; 在代谢组学中较常用的两种工具是质谱和核磁共振波谱。HF是一种与遗传、环境及生活方式等有着密切关系的复杂性疾病。例如, 环境可对基因变异、蛋白表达及代谢状态等产生影响(图 2)。不同组学技术使人们可以从基因、蛋白及代谢物等不同层面去发现和了解HF的特征。此外, 不同组学相互间的交叉、联合分析HF病因的普遍性和特异性, 可为HF预防、临床诊疗等方面提供精准、有效的信息。
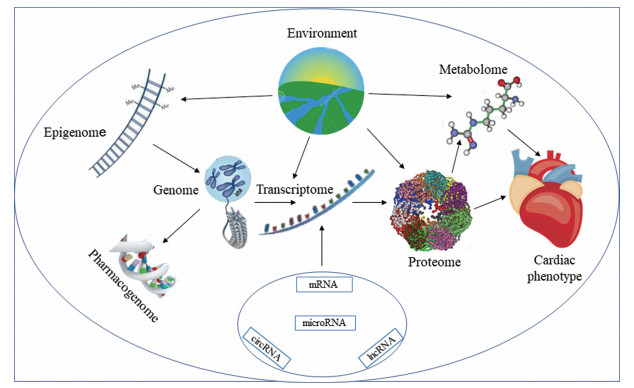
|
图 2 The relationship between different omics |
本文综述了近年来在HF多组学分析中的最新发现, 通过研究与HF相关的组学信息, 在未来有望进一步阐明与每个HF患者表型相关的组学信息, 整合来自同一HF患者的多组学数据, 进而实现基于组学的患者分型。
2.1 基因组学基因组学通过分析个体遗传变异的影响研究疾病的发生机制。HF是一种具有遗传基础的复杂性疾病, 基因组学对HF的研究使人们对该疾病遗传的理解日渐加深[9]。目前, 已有多个GWAS研究对与HF相关的遗传变异进行了鉴定(表 1)[10-19]。同时, 越来越多与HF及其病因如扩张型心肌病(dilated cardiomyopathy, DCM)和冠心病(coronary artery disease, CAD)等相关的基因变异的发现及这些基因准确的功能定位, 如ECM相关基因、线粒体相关基因等[20, 21]促进了对HF病理生理学的理解。
| 表 1 Summary of genome-wide association studies for HF (etiology). SNP: Single nucleotide polymorphisms |
目前基因组学的研究主要通过扩大样本量及使用相关表型(如血液生物标志物和左心室结构及其功能参数)来识别HF变异基因和潜在机制, 这些更稳定、更精确的指标可以称为HF的“内表型”[22]。通过GWAS所建立的HF内表型, 已用于心脏病预防和诊断、临床心脏病监控、药物治疗及指导生活方式等。
药物基因组学是目前基因组学研究与临床应用结合最紧密的领域。其研究目的是发现药物如何因药物代谢酶、药物转运体、靶点基因遗传变异而产生不同的作用[23, 24]。
2.2 转录组学转录组学是研究特定组织或细胞在某一发育阶段或功能状态下经基因转录的情况及转录调控规律的学科, 包括编码蛋白质的RNA和各种非编码RNA (ncRNA)等。人类只有2%的基因组编码蛋白质, 而约98%的人类基因组不编码蛋白质, 但能参与转录, 产生大量ncRNAs, 发挥重要的调控和结构功能[25]。很多ncRNAs都参与了HF的病理生理过程, 如microRNA (miRNA)、lncRNA和circRNA等。表 2表示与HF相关的ncRNAs及其作用[26-40]。
| 表 2 ncRNAs related to HF |
ncRNAs中研究最为广泛、功能最为典型的是miRNA[41, 42]。N末端B型利钠肽原(N-terminal pro-B-type natriuretic peptide, NT-proBNP)结合miRNA可用来检测和区分HF亚型[26]。一些miRNA簇控制心肌细胞增殖并可能对成年心肌产生有效的心肌再生作用[27]。最近研究发现lncRNA是疾病期间调节心肌重塑的分子, 参与心肌细胞肥大以及成纤维细胞增殖的调节, 可能是心血管疾病治疗的新靶点[43], 如Wisper[33]、Meg3[34]等。与lncRNA类似, 高通量测序和生物信息学分析在心肌细胞中识别出了circRNA[44]。研究发现, 一种称为MICRA的circRNA可能是有效预测心室重构的生物标志物[35]。此外, 心肌细胞、成纤维细胞和炎性细胞等不同类型细胞中的circRNA可能参与了HF的病理生理过程[45, 46]。
2.3 表观基因组学表观基因组学实质上是研究非遗传因素如何影响基因表达的变化, 而这些变化并不涉及到核酸序列的改变。表观遗传修饰包括DNA甲基化、组蛋白修饰等[47], 不同的表观遗传修饰参与了HF的发生和发展[48, 49]。
DNA甲基化是在甲基化转移酶的作用下, 在基因组CG二核苷酸的胞嘧啶5′碳位共价结合一个甲基基团, 是一个独特的表观遗传学过程, 它可以根据人体的需要和环境的不断变化对基因表达进行微调。吸烟、污染和高脂肪饮食都会改变DNA甲基化。Glezeva等[50]对DNA甲基化在HF中的作用进行了研究, 见表 3[50, 51]。
| 表 3 Epigenetics associated with HF |
组蛋白修饰主要包括乙酰化、甲基化、磷酸化等。组蛋白H3和H4富含赖氨酸和精氨酸的N-末端的修饰, 尤其是甲基化和乙酰化, 影响染色质纤维的折叠和功能状态。Stratton等[51]研究结果表明可与乙酰化的组蛋白结合的溴结构域蛋白(bromodomain-containing protein 4, BRD4)是心肌纤维化中成纤维细胞表型调控的关键分子, 通过基于P38的信号通路调控HF的表观遗传, 该研究为研发靶向抗心肌纤维化治疗的BRD4抑制剂提供了理论基础。
2.4 蛋白质组学蛋白质组学是以蛋白质组为研究对象, 从整体上直接研究基因组所表达的蛋白质和蛋白质功能。蛋白质组分析揭示了心肌ECM、心肌细胞骨架、收缩结构、能量产生和心肌细胞保护等蛋白质的调节变化, 在HF发病机制、预防及生物标志物的发现等过程中发挥了重要的作用[52]。
蛋白质生物标志物可用于反映HF的不同病理生理过程, 如表 4所示[53, 54]。其中心脏肌钙蛋白(cTn)、可溶性ST2 (sST2)、半乳糖凝集素3 (Gal-3)及生长分化因子15 (GDF-15)被推荐用于HF的诊断和预后评估。
| 表 4 Biomarkers reflecting the different pathophysiological processes of HF. ANP: Atrial natriuretic peptide; MR-proANP: Midregional pro atrial natriuretic peptide; GDF-15: Growth differentiating factor-15; NRGs: Neuregulins; CRP: C-reactive protein; TNF-α: Tumor necrosis factor-α; IL-1/6/18: Interleukin-1/6/18; PCT: Procalcitonin; ADPN: Recombinant adiponectin; MMPs: Matrix metalloproteinases; hsTn: High-sensitivity troponin; CK-MB: Creatinine kinase-MB fraction; MLCK: Myosin light chain kinase; HFABP: Heart-type fatty acid binding protein; MPO: Myeloperoxidase |
在一项突发性HF相关机制的研究中, 研究者运用蛋白组学的方法研究了HF患者及健康受试者血浆中的252种蛋白质。鉴定出的38种蛋白涉及HF的病理机制包括炎症和凋亡; ECM重塑、血管生成和生长; 血压调节和代谢[55]。此外, 蛋白质糖基化在心肌肥厚的生理和病理、心脏功能调节及HF的进展过程中发挥着一定的作用[56]。
2.5 代谢组学代谢组学作为系统生物学的一部分从代谢终产物的角度补充了基因表达和蛋白功能改变等多种信息。代谢组学因其高度可靠及可重复性被广泛应用于生物标志物识别、疾病诊断、药物机制研究、药物安全性和毒理学评价等[57]。表 5是与HF相关的代谢物[58-72]。
| 表 5 Metabolites and their metabolic pathways related to HF. PC: Phosphatidylcholines; PE: Phosphatidylethanolamine |
心肌代谢重构对HF的发展至关重要, 在HF进展过程中, 心肌对游离脂肪酸(free fatty acid, FFA)、葡萄糖、乳酸、酮体及支链氨基酸等能量代谢底物的利用发生了很大变化[73-75]。人类衰竭心脏和大多数失代偿的HF模型中的FFA代谢降低, 而葡萄糖代谢的变化仍存在争议。酮体可作为衰竭心脏代谢的底物[76-78], 有研究表明, 钠-葡萄糖共转运蛋白2 (sodium glucose cotransporter-2, SGLT-2)抑制剂依帕列净对心脏的保护作用是通过增加心肌对酮体的利用, 进而增加心肌产能, 缓解左室不良重构, 改善心功能[79]。支链氨基酸(branched chain amino acid, BCAA)对HF病理性重塑和功能障碍的进展也具有潜在的重大影响[63]。脂质组学是代谢组学的一个分支。脂质在HF的发病机制中发挥着重要作用。慢性HF患者磷脂代谢发生明显失调[69]; 基于LC-MS的脂质组学研究发现晚期HF患者循环不饱和脂肪酸减少, 神经酰胺增多, 尤其是HF患者血浆中超长链神经酰胺(C22:1和C24:1)含量显著高于对照组[70]。
不同的生物体中脂蛋白及其亚型及载脂蛋白在血液中占比不同, 影响了其对脂质的敏感性, 进而影响疾病发展及药物疗效。利用1H NMR方法, 可得到相关脂蛋白的信息(包括脂质浓度, 载脂蛋白Apo-A1、Apo-A2和Apo-B的浓度及LDL颗粒数)[80]。基于稳定同位素标记的代谢流分析结合NMR或MS信息分析细胞内代谢中间体的标记状态, 能够直观地展现代谢的整体走向, 可用于揭示疾病状态下主要变化的代谢途径和生物标志物。
2.6 多组学的联合应用高通量测序技术(high-throughput sequencing, HTS)可以全基因组的研究方式对病理条件性的遗传变异、表观遗传修饰和相关基因表达进行研究, 蛋白质组和代谢组也可以提供数百种蛋白质和代谢产物的同步分析数据。整合不同的组学方法, 找到特定病理表型下的特征分子网络, 精简分析数据, 从而使有目的的实验验证成为可能, 也为理解HF的分子表型提供了新的方法, 组学之间的联合在HF研究中的应用见表 6[81-83]。
| 表 6 The associations of omics in the study of HF |
基因组结合蛋白质组目前已被用于识别与血清NT-proBNP水平、血铜蓝蛋白水平和血浆可溶性白细胞介素-2Rα[81]水平个体间变异相关的基因。整合表观基因组和转录组分析提供了有关表观遗传变化、基因表达和剪接变体的定量信息, 可研究心脏在整个疾病进展过程中的变化以及对环境变化或治疗的反应[82]。蛋白质组学和代谢组学数据的结合可以展示一条完整的热点代谢途径或代谢子网络。
Zhou等[83]结合代谢组学、蛋白质组学的研究找到了HF诱因心房颤动的候选生物标志物, 结果发现房颤患者体内24种代谢物、16种脂质和16种蛋白质明显失调, 该研究采用组学联合方法揭示了诊断房颤患者的可能的血浆生物标志物并为房颤患者的病理研究和新的治疗靶点的确定提供了线索。图 3表示各组学在HF研究中的应用。
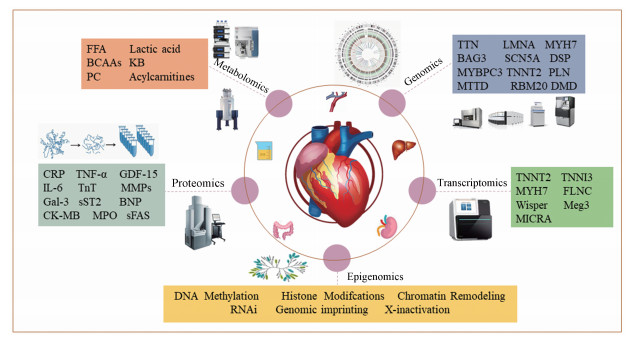
|
图 3 Omics-based analysis of heart failure. sFAS: Tumor necrosis super family 6 |
DCM和肥厚型心肌病(hypertrophic cardiomyopathy, HCM)是HF最主要的两大病因。以DCM和HCM为代表的心肌病是单基因疾病, 组学技术加速了实验动物和人类与心肌病相关遗传变异的发现。基因测序可用于心肌病风险分层以及家庭心肌病的筛查。
2019年的一项大规模的前瞻性队列人群研究[84]表明, 通过血液中白细胞的DNA甲基化水平可预测未来发生MI和CAD的风险。试验共纳入无CAD的11 461例个体, 在平均11.2年的随访期间内, 共发生1895例CAD相关不良事件和1 183例MI相关不良事件。52个CpG位点的甲基化水平与CAD或MI有关。这些CpG与在钙调节中具有关键作用的基因的表达相关, 同时与在基因组和表观基因组范围内调节的血清钙基因, 血清钙相关的CAD风险基因等均相关。试验证明了血源性DNA甲基化水平与不同人群未来发生CAD的风险有关, 为CAD、MI或HF的预防提供新的见解。
3.2 HF的诊断当前的组学研究不但与传统诊断手段提供的诊疗参数间建立起关系, 而且进一步揭示了其病理表型下的分子机制。
Rommel等[85]运用全基因组基因表达分析方法鉴定出HFpEF患者(n = 719)和对照者(n = 1 106)的17个差异表达基因。这项迄今为止最大的研究发现了可能导致HFpEF异质性的潜在机制和治疗靶点。还有研究发现利用miRNAs的特征可以区分HF患者和对照组并区分HFrEF和HFpEF[86]。同时对HFpEF患者与HFrEF患者的代谢组学研究[68, 87]表明, HFpEF患者的酰基胆碱酯酶、肌酐、甜菜碱、磷脂酰胆碱、溶血磷脂酰胆碱、酮体、鞘磷脂蛋白(C24:1)等有显著差异。此外与HF诊断及分型相关的代谢物还有假尿苷和α-酮戊二酸[61]、黄酮类、甘氨酸、2′-磷酸酯肌苷、核糖-5-磷酸酯等[59]、酰基肉碱[88]、尿胆素和鞘磷脂(30:1)[71]等。
Rosa-Garrido等[89]采用全基因组染色质构象捕获(Hi-C)和DNA测序对压力超负荷诱导心肌肥大的成人心脏组织进行分析, 评估了表观基因组在心脏发病机制中的作用, 证明染色质的整体结构重塑会增加HF发生的风险。
有研究[90]运用蛋白质组学方法首次对家族性DCM相关的CHRM2基因C722G突变情况下的蛋白质组进行了全面筛选, 并对8种可能对心功能障碍起关键作用的蛋白进行了定位。
3.3 HF药物疗效评价药物基因组学在药物疗效、不良反应及代谢等过程的研究中有着重要作用。例如, 携带一个或两个CYP2D6功能降低或缺陷型等位基因(CYP2D6*5, *10, *41等)的中间代谢者(IM)或弱代谢者(PM), 药物的血药浓度会异于正常个体, 有研究表明给予CYP2D6 PMs和非PMs同等剂量的美托洛尔, PM组的美托洛尔血浆浓度是非PM组的4.9倍, 且其心率和舒张压及平均动脉压的降低程度显著大于非PMs[91]。
ACEI/ARB是HF治疗的有效药物。ACEI/ARB的不良反应包括咳嗽、血管性水肿、低血压和高钾血症等。代谢组学结果表明, ACEI引起的咳嗽和血管神经性水肿与过量的缓激肽积聚有关[92]。ACEI/ARB药物有效性和安全性的代谢组标志物的研究有望指导药物治疗。
西药疗法在改善HF症状和降低HF死亡率方面具有良好的效果, 而对于由冠脉微循环功能障碍引起的HF的防治, 中药复方显示出了独特的疗效。以往由于中药成分的复杂性与作用靶点多样性使得在其药理、毒理方面的研究成为难题。近年来, 网络药理学的发展为中药研究提供了新的方法。采用网络药理学及多组学联合的方法对麝香保心丸[93]、芪参益气方[94]及养心氏片[95]等中药复方的作用机制的研究取得了重大进展。
基于GC-MS、LC-MS和LC-MS/MS的组学技术对给予芪苈强心的HF大鼠血液样本中的相关代谢物进行检测, 结果表明芪苈强心通过改善心肌能量代谢和调节炎性细胞因子分泌抑制心脏重构, 延缓HF进程[96], 此外, 多组学还被应用到参附汤[97]、丹参酮IIA[98]、黄芪注射液[60]等的研究中。组学方法被越来越多地用来揭示中药治疗HF的相关作用机制。
3.4 HF的预后HF患者5年存活率与恶性肿瘤相仿, 严重HF患者年平均病死率为40%~50%, 慢性HF的预后受年龄、肥胖、射血分数等多因素影响。对HF患者预后情况进行监测并采取干预措施, 有助于改善预后。
Xiao等[99]对随访1年的96例急性HF患者的研究结果表明, miR-30d可作为急性HF患者生存率的预测指标。另有研究表明, 低水平的miR-423-5p与HF较差的预后相关[100]。Hollander等[101]运用同位素标记相对绝对定量-质谱(isobaric tags for relative and absolute quantification-mass spectrometer, iTRAQ-MS)法确定了血浆中17个蛋白质生物标志物, 这些生物标志物与BNP检测相结合, 能够对患者术后恢复情况进行评估。这种简便的血液检测方法, 可用于监测HF患者的预后情况, 从而使HF患者管理更具个性化。
运用UPLC及串联MS技术测定并分析了1 084名受试者血浆中必需氨基酸及各种酰基肉碱水平为HF传统危险因素补充了代谢信息[102]。有研究对不同分期HF患者和健康受试者的血浆代谢物进行质谱分析, 结果表明由二甲基精氨酸与精氨酸的比值、丁酰肉碱总数、亚精胺总数和必需氨基酸总量组成的代谢物组, 在不依赖BNP和传统危险因素的情况下可为HF患者提供优于Gal-3的预后价值[103]。
4 总结与展望基因组学、转录组学、表观基因组学、蛋白质组学及代谢组学极大地促进了HF的诊断、治疗及预后的研究, 同时一些更精准、更有针对性的组学, 如单细胞组学、化学蛋白质组学也呈现在我们的视野中。随着不同组学技术的发展, 多组学的联合分析在疾病研究领域应运而生, 各组学数据相互补充, 将在HF患者的心脏能量代谢、心肌重塑和新的生物标志物方面带来前所未有的突破, 在精准医疗方面发挥越来越重要的作用。值得注意的是, 当前多组学的联合应用在很大程度上局限于实验模型, 为了更好地探索和整合多种组学数据模式, 使其能够运用于临床, 还需要开发复杂但易于使用的生物信息学工具, 以加速疾病的多组学研究。
| [1] |
Yancy CW, Jessup M, Bozkurt B, et al. 2017 ACC/AHA/HFSA focused update of the 2013 ACCF/AHA guideline for the management of heart failure:a report of the American College of Cardiology/American Heart Association Task Force on Clinical Practice Guidelines and the Heart Failure Society of America[J]. J Am Coll Cardiol, 2017, 70: 776-803. DOI:10.1016/j.jacc.2017.04.025 |
| [2] |
Hu SS, Yang YJ, Zheng Z, et al. Summary of the 2018 report on cardiovascular diseases in China[J]. Chin Circ J (中国循环杂志), 2019, 34: 209-220. |
| [3] |
Smith JG. Molecular epidemiology of heart failure:translational challenges and opportunities[J]. JACC Basic Transl Sci, 2017, 2: 757-769. DOI:10.1016/j.jacbts.2017.07.010 |
| [4] |
Seferović PM, Polovina M, Bauersachs J, et al. Heart failure in cardiomyopathies:a position paper from the Heart Failure Association of the European Society of Cardiology[J]. Eur J Heart Fail, 2019, 21: 553-576. DOI:10.1002/ejhf.1461 |
| [5] |
Meijers WC, de Boer RA. Common risk factors for heart failure and cancer[J]. Cardiovasc Res, 2019, 115: 844-853. DOI:10.1093/cvr/cvz035 |
| [6] |
Woo AYH, Song Y, Xiao RP, et al. Biased β2-adrenoceptor signalling in heart failure:pathophysiology and drug discovery[J]. Br J Pharmacol, 2015, 172: 5444-5456. DOI:10.1111/bph.12965 |
| [7] |
Norgard NB, Hempel C. Towards precision in HF pharmacotherapy[J]. Curr Heart Fail Rep, 2017, 14: 1-6. |
| [8] |
Raghow R. An 'Omics' perspective on cardiomyopathies and heart failure[J]. Trends Mol Med, 2016, 22: 813-827. DOI:10.1016/j.molmed.2016.07.007 |
| [9] |
Rosenbaum AN, Pereira N. Updates on the genetic paradigm in heart failure[J]. Curr Treat Options Cardiovasc Med, 2019, 21: 37. DOI:10.1007/s11936-019-0742-3 |
| [10] |
Cappola TP, Li M, He J, et al. Common variants in HSPB7 and FRMD4B associated with advanced heart failure[J]. Circ Cardiovasc Genet, 2010, 3: 147-154. DOI:10.1161/CIRCGENETICS.109.898395 |
| [11] |
Smith NL, Felix JF, Morrison AC, et al. Association of genome-wide variation with the risk of incident heart failure in adults of European and African ancestry:a prospective meta-analysis from the cohorts for heart and aging research in genomic epidemiology (CHARGE) consortium[J]. Circ Cardiovasc Genet, 2010, 3: 256-266. DOI:10.1161/CIRCGENETICS.109.895763 |
| [12] |
Morrison AC, Felix JF, Cupples LA, et al. Genomic variation associated with mortality among adults of European and African ancestry with heart failure:the cohorts for heart and aging research in genomic epidemiology consortium[J]. Circ Cardiovasc Genet, 2010, 3: 248-255. DOI:10.1161/CIRCGENETICS.109.895995 |
| [13] |
Smith JG, Felix JF, Morrison AC, et al. Discovery of genetic variation on chromosome 5q22 associated with mortality in heart failure[J]. PLoS Genet, 2016, 12: e1006034. DOI:10.1371/journal.pgen.1006034 |
| [14] |
Parsa A, Chang YPC, Kelly RJ, et al. Hypertrophy-associated polymorphisms ascertained in a founder cohort applied to heart failure risk and mortality[J]. Clin Transl Sci, 2011, 4: 17-23. DOI:10.1111/j.1752-8062.2010.00251.x |
| [15] |
Stark K, Esslinger UB, Reinhard W, et al. Genetic association study identifies HSPB7 as a risk gene for idiopathic dilated cardiomyopathy[J]. PLoS Genet, 2010, 6: e1001167. DOI:10.1371/journal.pgen.1001167 |
| [16] |
Meder B, Rühle F, Weis T, et al. A genome-wide association study identifies 6p21 as novel risk locus for dilated cardiomyopathy[J]. Eur Heart J, 2013, 35: 1069-1077. |
| [17] |
Villard E, Perret C, Gary F, et al. A genome-wide association study identifies two loci associated with heart failure due to dilated cardiomyopathy[J]. Eur Heart J, 2011, 32: 1065-1076. DOI:10.1093/eurheartj/ehr105 |
| [18] |
Verweij N, Eppinga RN, Hagemeijer Y, et al. Identification of 15 novel risk loci for coronary artery disease and genetic risk of recurrent events, atrial fibrillation and heart failure[J]. Sci Rep, 2017, 7: 2761. DOI:10.1038/s41598-017-03062-8 |
| [19] |
Howson JMM, Zhao W, Barnes DR, et al. Fifteen new risk loci for coronary artery disease highlight arterial-wall-specific mechanisms[J]. Nat Genet, 2017, 49: 1113. DOI:10.1038/ng.3874 |
| [20] |
Cresci S, Pereira NL, Ahmad F, et al. Heart failure in the era of precision medicine:a scientific statement from the American Heart Association[J]. Circ Genom Precis Med, 2019, 12: 458-485. |
| [21] |
Hannah-Shmouni F, Seidelmann SB, Sirrs S, et al. The genetic challenges and opportunities in advanced heart failure[J]. Can J Cardiol, 2015, 31: 1338-1350. DOI:10.1016/j.cjca.2015.07.735 |
| [22] |
van der Ende MY, Said MA, Van Veldhuisen DJ, et al. Genome-wide studies of heart failure and endophenotypes:lessons learned and future directions[J]. Cardiovasc Res, 2018, 114: 1209-1225. |
| [23] |
Abuzaanona A, Lanfear D. Pharmacogenomics of the natriuretic peptide system in heart failure[J]. Curr Heart Fail Rep, 2017, 14: 536-542. DOI:10.1007/s11897-017-0365-5 |
| [24] |
Mottet F, Vardeny O, De Denus S. Pharmacogenomics of heart failure:a systematic review[J]. Pharmacogenomics, 2016, 17: 1817-1858. DOI:10.2217/pgs-2016-0118 |
| [25] |
Boon RA, Jaé N, Holdt L, et al. Long noncoding RNAs:from clinical genetics to therapeutic targets?[J]. J Am Coll Cardiol, 2016, 67: 1214-1226. |
| [26] |
Wong LL, Zou R, Zhou L, et al. Combining circulating microRNA and NT-proBNP to detect and categorize heart failure subtypes[J]. J Am Coll Cardiol, 2019, 73: 1300-1313. |
| [27] |
Giacca M, Zacchigna S. Harnessing the microRNA pathway for cardiac regeneration[J]. J Mol Cell Cardiol, 2015, 89: 68-74. DOI:10.1016/j.yjmcc.2015.09.017 |
| [28] |
Ovchinnikova ES, Schmitter D, Vegter EL, et al. Signature of circulating microRNAs in patients with acute heart failure[J]. Eur J Heart Fail, 2016, 18: 414-423. DOI:10.1002/ejhf.332 |
| [29] |
Corsten MF, Dennert R, Jochems S, et al. Circulating microRNA-208b and microRNA-499 reflect myocardial damage in cardiovascular disease[J]. Circ Cardiovasc Genet, 2010, 3: 499-506. DOI:10.1161/CIRCGENETICS.110.957415 |
| [30] |
He M, Yang Z, Abdellatif M, et al. GTPase activating protein (SH3 domain) binding protein 1 regulates the processing of microRNA-1 during cardiac hypertrophy[J]. PLoS One, 2015, 10: e0145112. DOI:10.1371/journal.pone.0145112 |
| [31] |
Chen S, Puthanveetil P, Feng B, et al. Cardiac miR-133a overexpression prevents early cardiac fibrosis in diabetes[J]. J Cell Mol Med, 2014, 18: 415-421. DOI:10.1111/jcmm.12218 |
| [32] |
Ucar A, Gupta SK, Fiedler J, et al. The miRNA-212/132 family regulates both cardiac hypertrophy and cardiomyocyte autophagy[J]. Nat Commun, 2012, 3: 1078. DOI:10.1038/ncomms2090 |
| [33] |
Micheletti R, Plaisance I, Abraham BJ, et al. The long noncoding RNA Wisper controls cardiac fibrosis and remodeling[J]. Sci Transl Med, 2017, 9: eaai9118. DOI:10.1126/scitranslmed.aai9118 |
| [34] |
Piccoli MT, Gupta SK, Viereck J, et al. Inhibition of the cardiac fibroblast-enriched lncRNA Meg3 prevents cardiac fibrosis and diastolic dysfunction[J]. Circ Res, 2017, 121: 575-583. DOI:10.1161/CIRCRESAHA.117.310624 |
| [35] |
Vausort M, Salgado-Somoza A, Zhang L, et al. Myocardial infarction-associated circular RNA predicting left ventricular dysfunction[J]. J Am Coll Cardiol, 2016, 68: 1247-1248. |
| [36] |
Wang K, Long B, Liu F, et al. A circular RNA protects the heart from pathological hypertrophy and heart failure by targeting miR-223[J]. Eur Heart J, 2016, 37: 2602-2611. DOI:10.1093/eurheartj/ehv713 |
| [37] |
Khan MAF, Reckman YJ, Aufiero S, et al. RBM20 regulates circular RNA production from the titin gene[J]. Circ Res, 2016, 119: 996-1003. DOI:10.1161/CIRCRESAHA.116.309568 |
| [38] |
Du WW, Yang W, Chen Y, et al. Foxo3 circular RNA promotes cardiac senescence by modulating multiple factors associated with stress and senescence responses[J]. Eur Heart J, 2016, 38: 1402-1412. |
| [39] |
Boeckel JN, Jaé N, Heumüller AW, et al. Identification and characterization of hypoxia-regulated endothelial circular RNA[J]. Circ Res, 2015, 117: 884-890. DOI:10.1161/CIRCRESAHA.115.306319 |
| [40] |
Geng HH, Li R, Su YM, et al. The circular RNA Cdr1as promotes myocardial infarction by mediating the regulation of miR-7a on its target genes expression[J]. PLoS One, 2016, 11: e0151753. DOI:10.1371/journal.pone.0151753 |
| [41] |
Tijsen AJ, Pinto YM, Creemers EE. Circulating microRNAs as diagnostic biomarkers for cardiovascular diseases[J]. Am J Physiol Heart Circ Physiol, 2012, 303: H1085-H1095. DOI:10.1152/ajpheart.00191.2012 |
| [42] |
Wang H, Cai J. The role of microRNAs in heart failure[J]. Biochim Biophys Acta Mol Basis Dis, 2017, 1863: 2019-2030. DOI:10.1016/j.bbadis.2016.11.034 |
| [43] |
Salamon I, Jotti GS, Condorelli G. The long noncoding RNA landscape in cardiovascular disease:a brief update[J]. Curr Opin Cardiol, 2018, 33: 282-289. DOI:10.1097/HCO.0000000000000507 |
| [44] |
Werfel S, Nothjunge S, Schwarzmayr T, et al. Characterization of circular RNAs in human, mouse and rat hearts[J]. J Mol Cell Cardiol, 2016, 98: 103-107. DOI:10.1016/j.yjmcc.2016.07.007 |
| [45] |
Devaux Y, Creemers EE, Boon RA, et al. Circular RNAs in heart failure[J]. Eur J Heart Fail, 2017, 19: 701-709. DOI:10.1002/ejhf.801 |
| [46] |
Zhao G. Significance of non-coding circular RNAs and microRNAs in the pathogenesis of cardiovascular diseases[J]. J Med Genet, 2018, 55: 713-720. DOI:10.1136/jmedgenet-2018-105387 |
| [47] |
Costantino S, Libby P, Kishore R, et al. Epigenetics and precision medicine in cardiovascular patients:from basic concepts to the clinical arena[J]. Eur Heart J, 2017, 39: 4150-4158. |
| [48] |
Alexanian M, Padmanabhan A, McKinsey TA, et al. Epigenetic therapies in heart failure[J]. J Mol Cell Cardiol, 2019, 130: 197-204. DOI:10.1016/j.yjmcc.2019.04.012 |
| [49] |
Meder B, Haas J, Sedaghat-Hamedani F, et al. Epigenome-wide association study identifies cardiac gene patterning and a novel class of biomarkers for heart failure[J]. Circulation, 2017, 136: 1528-1544. DOI:10.1161/CIRCULATIONAHA.117.027355 |
| [50] |
Glezeva N, Moran B, Collier P, et al. Targeted DNA methylation profiling of human cardiac tissue reveals novel epigenetic traits and gene deregulation across different heart failure patient subtypes[J]. Circ Heart Fail, 2019, 12: e005765. |
| [51] |
Stratton MS, Bagchi RA, Felisbino MB, et al. Dynamic chromatin targeting of BRD4 stimulates cardiac fibroblast activation[J]. Circ Res, 2019, 125: 662-677. DOI:10.1161/CIRCRESAHA.119.315125 |
| [52] |
Smith JG, Gerszten RE. Emerging affinity-based proteomic technologies for large-scale plasma profiling in cardiovascular disease[J]. Circulation, 2017, 135: 1651-1664. DOI:10.1161/CIRCULATIONAHA.116.025446 |
| [53] |
Israr MZ, Heaney L M, Suzuki T. Proteomic biomarkers of heart failure[J]. Heart Fail Clin, 2018, 14: 93-107. DOI:10.1016/j.hfc.2017.08.010 |
| [54] |
Sarhene M, Wang Y, Wei J, et al. Biomarkers in heart failure:the past, current and future[J]. Heart Fail Rev, 2019, 24: 867-903. DOI:10.1007/s10741-019-09807-z |
| [55] |
Ferreira JP, Verdonschot J, Collier T, et al. Proteomic bioprofiles and mechanistic pathways of progression to heart failure:the HOMAGE study[J]. Circ Heart Fail, 2019, 12: e005897. |
| [56] |
Yang S, Chatterjee S, Cipollo J. The glycoproteomics-MS for studying glycosylation in cardiac hypertrophy and heart failure[J]. Proteomics Clin Appl, 2018, 12: 1700075. DOI:10.1002/prca.201700075 |
| [57] |
McGarrah RW, Crown SB, Zhang GF, et al. Cardiovascular metabolomics[J]. Circ Res, 2018, 122: 1238-1258. DOI:10.1161/CIRCRESAHA.117.311002 |
| [58] |
Tenori L, Hu X, Pantaleo P, et al. Metabolomic fingerprint of heart failure in humans:a nuclear magnetic resonance spectroscopy analysis[J]. Int J Cardiol, 2013, 168: e113-e115. DOI:10.1016/j.ijcard.2013.08.042 |
| [59] |
Zhang X, Liu H, Gao J, et al. Metabolic disorder in the progression of heart failure[J]. Sci China Life Sci, 2019, 62: 1153-1167. DOI:10.1007/s11427-019-9548-9 |
| [60] |
Liu Y, Xu W, Xiong Y, et al. Evaluations of the effect of HuangQi against heart failure based on comprehensive echocardiography index and metabonomics[J]. Phytomedicine, 2018, 50: 205-212. DOI:10.1016/j.phymed.2018.04.027 |
| [61] |
Dunn WB, Broadhurst DI, Deepak SM, et al. Serum metabolomics reveals many novel metabolic markers of heart failure, including pseudouridine and 2-oxoglutarate[J]. Metabolomics, 2007, 3: 413-426. DOI:10.1007/s11306-007-0063-5 |
| [62] |
Wang J, Li Z, Chen J, et al. Metabolomic identification of diagnostic plasma biomarkers in humans with chronic heart failure[J]. Mol Biosyst, 2013, 9: 2618-2626. DOI:10.1039/c3mb70227h |
| [63] |
Sun H, Olson KC, Gao C, et al. Catabolic defect of branched-chain amino acids promotes heart failure[J]. Circulation, 2016, 133: 2038-2049. DOI:10.1161/CIRCULATIONAHA.115.020226 |
| [64] |
Delles C, Rankin NJ, Boachie C, et al. Nuclear magnetic resonance-based metabolomics identifies phenylalanine as a novel predictor of incident heart failure hospitalisation:results from PROSPER and FINRISK 1997[J]. Eur J Heart Fail, 2018, 20: 663-673. DOI:10.1002/ejhf.1076 |
| [65] |
Cheng ML, Wang CH, Shiao MS, et al. Metabolic disturbances identified in plasma are associated with outcomes in patients with heart failure:diagnostic and prognostic value of metabolomics[J]. J Am Coll Cardiol, 2015, 65: 1509-1520. DOI:10.1016/j.jacc.2015.02.018 |
| [66] |
Lopaschuk GD, Ussher JR, Folmes CD L, et al. Myocardial fatty acid metabolism in health and disease[J]. Physiol Rev, 2010, 90: 207-258. DOI:10.1152/physrev.00015.2009 |
| [67] |
Doenst T, Nguyen TD, Abel ED. Cardiac metabolism in heart failure:implications beyond ATP production[J]. Circ Res, 2013, 113: 709-724. DOI:10.1161/CIRCRESAHA.113.300376 |
| [68] |
Zordoky BN, Sung MM, Ezekowitz J, et al. Metabolomic fingerprint of heart failure with preserved ejection fraction[J]. PLoS One, 2015, 10: e0124844. DOI:10.1371/journal.pone.0124844 |
| [69] |
Marcinkiewicz-Siemion M, Ciborowski M, Ptaszynska-Kopczynska K, et al. LC-MS-based serum fingerprinting reveals significant dysregulation of phospholipids in chronic heart failure[J]. J Pharm Biomed Anal, 2018, 154: 354-363. DOI:10.1016/j.jpba.2018.03.027 |
| [70] |
Brunjes DL, Dunlop M, Wu C, et al. Analysis of skeletal muscle torque capacity and circulating ceramides in patients with advanced heart failure[J]. J Card Fail, 2016, 22: 347-355. DOI:10.1016/j.cardfail.2016.02.002 |
| [71] |
Stenemo M, Ganna A, Salihovic S, et al. The metabolites urobilin and sphingomyelin (30:1) are associated with incident heart failure in the general population[J]. ESC Heart Fail, 2019, 6: 764-773. DOI:10.1002/ehf2.12453 |
| [72] |
Kang SM, Park JC, Shin MJ, et al. 1H nuclear magnetic resonance based metabolic urinary profiling of patients with ischemic heart failure[J]. Clin Biochem, 2011, 44: 293-299. DOI:10.1016/j.clinbiochem.2010.11.010 |
| [73] |
Peterzan MA, Lygate CA, Neubauer S, et al. Metabolic remodeling in hypertrophied and failing myocardium:a review[J]. Am J Physiol Heart Circ Physiol, 2017, 313: H597-H 616. DOI:10.1152/ajpheart.00731.2016 |
| [74] |
Ikegami R, Shimizu I, Yoshida Y, et al. Metabolomic analysis in heart failure[J]. Circ J, 2017, 82: 10-16. |
| [75] |
Hunter WG, Kelly JP, Iii MG, et al. Metabolic dysfunction in heart failure:diagnostic, prognostic, and pathophysiologic insights from metabolomic profiling[J]. Curr Heart Fail Rep, 2016, 13: 119-131. DOI:10.1007/s11897-016-0289-5 |
| [76] |
Bedi Jr KC, Snyder NW, Brandimarto J, et al. Evidence for intramyocardial disruption of lipid metabolism and increased myocardial ketone utilization in advanced human heart failure[J]. Circulation, 2016, 133: 706-716. DOI:10.1161/CIRCULATIONAHA.115.017545 |
| [77] |
Chen L, Song J, Hu S. Metabolic remodeling of substrate utilization during heart failure progression[J]. Heart Fail Rev, 2019, 24: 143-154. DOI:10.1007/s10741-018-9713-0 |
| [78] |
Aubert G, Martin OJ, Horton JL, et al. The failing heart relies on ketone bodies as a fuel. Clinical perspective[J]. Circulation, 2016, 133: 698-705. DOI:10.1161/CIRCULATIONAHA.115.017355 |
| [79] |
Santos-Gallego CG, Requena-Ibanez JA, San Antonio R, et al. Empagliflozin ameliorates adverse left ventricular remodeling in nondiabetic heart failure by enhancing myocardial energetics[J]. J Am Coll Cardiol, 2019, 73: 1931-1944. |
| [80] |
Bell JD, Sadler PJ, Macleod AF, et al. 1H NMR studies of human blood plasma assignment of resonances for lipoproteins[J]. FEBS Lett, 1987, 219: 239-243. DOI:10.1016/0014-5793(87)81224-3 |
| [81] |
Durda P, Sabourin J, Lange EM, et al. Plasma levels of soluble interleukin-2 receptor α:associations with clinical cardiovascular events and genome-wide association scan[J]. Arterioscler Thromb Vasc Biol, 2015, 35: 2246-2253. DOI:10.1161/ATVBAHA.115.305289 |
| [82] |
Perrino C, Barabási AL, Condorelli G, et al. Epigenomic and transcriptomic approaches in the post-genomic era:path to novel targets for diagnosis and therapy of the ischaemic heart? Position Paper of the European Society of Cardiology Working Group on Cellular Biology of the Heart[J]. Cardiovasc Res, 2017, 113: 725-736. DOI:10.1093/cvr/cvx070 |
| [83] |
Zhou J, Sun L, Chen L, et al. Comprehensive metabolomic and proteomic analyses reveal candidate biomarkers and related metabolic networks in atrial fibrillation[J]. Metabolomics, 2019, 15: 96. DOI:10.1007/s11306-019-1557-7 |
| [84] |
Agha G, Mendelson MM, Ward-Caviness CK, et al. Blood leukocyte DNA methylation predicts risk of future myocardial infarction and coronary heart disease:a longitudinal study of 11461 participants from population-based cohorts[J]. Circulation, 2019, 140: 645-657. DOI:10.1161/CIRCULATIONAHA.118.039357 |
| [85] |
Rommel KP, Kirsten H, Beser C, et al. Genome-wide gene expression analysis reveals pathomechanisms in patients with heart failure and preserved ejection fraction (HFpEF)-Insights from the Leipzig Heart Study[C]. NJ USA: Wiley, 2019.
|
| [86] |
Watson CJ, Gupta SK, O'Connell E, et al. MicroRNA signatures differentiate preserved from reduced ejection fraction heart failure[J]. Eur J Heart Fail, 2015, 17: 405-415. DOI:10.1002/ejhf.244 |
| [87] |
Zordoky B, Miranda Sung J, Ezekowitz RM, et al. Serum metabolomics reveal a distinct fingerprint of heart failure with preserved ejection fraction[J]. J Mol Cell Cardiol, 2015, 85: S1-S56. |
| [88] |
Lanfear DE, Gibbs JJ, Li J, et al. Targeted metabolomic profiling of plasma and survival in heart failure patients[J]. JACC Heart Fail, 2017, 5: 823-832. DOI:10.1016/j.jchf.2017.07.009 |
| [89] |
Rosa-Garrido M, Chapski DJ, Schmitt AD, et al. High-resolution mapping of chromatin conformation in cardiac myocytes reveals structural remodeling of the epigenome in heart failure[J]. Circulation, 2017, 136: 1613-1625. DOI:10.1161/CIRCULATIONAHA.117.029430 |
| [90] |
Hou D, Chen Y, Liu J, et al. Proteomics screen to reveal molecular changes mediated by C722G missense mutation in CHRM2 gene[J]. J Proteomics, 2013, 89: 39-50. DOI:10.1016/j.jprot.2013.05.027 |
| [91] |
Rau T, Wuttke H, Michels LM, et al. Impact of the CYP2D6 genotype on the clinical effects of metoprolol:a prospective longitudinal study[J]. Clin Pharmacol Ther, 2009, 85: 269-272. DOI:10.1038/clpt.2008.218 |
| [92] |
Flaten HK, Monte AA. The pharmacogenomic and metabolomic predictors of ACE inhibitor and angiotensin II receptor blocker effectiveness and safety[J]. Cardiovasc Drugs Ther, 2017, 31: 471-482. DOI:10.1007/s10557-017-6733-2 |
| [93] |
Fang HY, Zeng HW, Lin LM, et al. A network-based method for mechanistic investigation of Shexiang Baoxin Pill's treatment of cardiovascular diseases[J]. Sci Rep, 2017, 7: 43632. DOI:10.1038/srep43632 |
| [94] |
Wu L, Wang Y, Li Z, et al. Identifying roles of "Jun-Chen-Zuo-Shi" component herbs of QiShenYiQi formula in treating acute myocardial ischemia by network pharmacology[J]. Chin Med, 2014, 9: 24. DOI:10.1186/1749-8546-9-24 |
| [95] |
Wu RM, Jiang B, Li H, et al. A network pharmacology approach to discover action mechanisms of Yangxinshi Tablet for improving energy metabolism in chronic ischemic heart failure[J]. J Ethnopharmacol, 2020, 246: 112227. DOI:10.1016/j.jep.2019.112227 |
| [96] |
Fu J, Chang L, Harms AC, et al. A metabolomics study of Qiliqiangxin in a rat model of heart failure:a reverse pharmacology approach[J]. Sci Rep, 2018, 8: 3688. DOI:10.1038/s41598-018-22074-6 |
| [97] |
Dawei Y, Xiaoxing W, Yaping W, et al. Urinary metabolomic profiling reveals the effect of Shenfu Decoction on chronic heart failure in rats[J]. Molecules, 2015, 20: 11915-11929. DOI:10.3390/molecules200711915 |
| [98] |
Zhang X, Wang Q, Wang X, et al. Tanshinone IIA protects against heart failure post-myocardial infarction via AMPKs/mTOR-dependent autophagy pathway[J]. Biomed Pharmacother, 2019, 112: 108599. DOI:10.1016/j.biopha.2019.108599 |
| [99] |
Xiao J, Gao R, Bei Y, et al. Circulating miR-30d predicts survival in patients with acute heart failure[J]. Cell Physiol Biochem, 2017, 41: 865-874. DOI:10.1159/000459899 |
| [100] |
Seronde MF, Vausort M, Gayat E, et al. Circulating microRNAs and outcome in patients with acute heart failure[J]. PLoS One, 2015, 10: e0142237. DOI:10.1371/journal.pone.0142237 |
| [101] |
Hollander Z, Lazárová M, Lam KKY, et al. Proteomic biomarkers of recovered heart function[J]. Eur J Heart Fail, 2014, 16: 551-559. DOI:10.1002/ejhf.65 |
| [102] |
Wang CH, Cheng ML, Liu MH. Simplified plasma essential amino acid-based profiling provides metabolic information and prognostic value additive to traditional risk factors in heart failure[J]. Amino Acids, 2018, 50: 1739-1748. DOI:10.1007/s00726-018-2649-9 |
| [103] |
Wang CH, Cheng ML, Liu MH, et al. Metabolic profile provides prognostic value better than galectin-3 in patients with heart failure[J]. J Cardiol, 2017, 70: 92-98. DOI:10.1016/j.jjcc.2016.10.005 |
 2020, Vol. 55
2020, Vol. 55


