2. 中国医科大学, 辽宁 沈阳 110122;
3. 四川新绿色药业科技发展有限公司, 四川 成都 611900;
4. 国家中医药管理局"中药配方颗粒质量与疗效评价"重点实验室, 四川 成都 611900
2. China Medical University, Shenyang 110122, China;
3. Neo-Green Pharmaceutical Co., Ltd., Chengdu 611900, China;
4. Key Laboratory of Chinese Medicine Formulations Particle Mass and Clinical Evaluation, Chengdu 611900, China
白头翁为我国传统中药, 2015版《中国药典》收载其为毛莨科植物白头翁Pulsatilla chinensis (Bge.) Regel的干燥根, 具有清热解毒、凉血止痢的功效, 用于热毒血痢、阴痒带下[1]。白头翁始载于《神农本草经》[2]; 梁代医家陶弘景有“近根处有白茸, 状似人白头, 故以为名”的描述[3]。明代李时珍《本草纲目》[4]以丈人、胡使、奈何为别名, 皆状老翁之意。为了研究白头翁药材市场应用品种混乱的问题[5], 本课题组实地调查了安徽亳州、河北安国、广西玉林、四川荷花池等7家中药材市场, 利用性状鉴定及基于ITS2序列的DNA条形码鉴定技术对其中的38份药材进行了鉴定, 发现了朝鲜白头翁P. cernua (Thunb.) Bercht. et Opiz.、兴安白头翁P. dahurica (Fisch.) Spreng.、细叶白头翁P. turczaninovii Kryl. et Serg.、金县白头翁P. chinensis (Bge.) Regel var. kissii (Mandl) S. H. Li et Y. H. Huang及翻白草Potentilla discolor Bge.、火绒草Leontopodium leontopodioides (Willd.) Beauv. Bull. Soc. Bot. Genève.、大火草Anemone tomentosa (Maxim.) C. P'ei等冒充白头翁药材。Chen等[6]利用ITS2序列作为DNA条形码和特定的PCR-RFLP分析方法对30种商品白头翁药材进行鉴定, 结果显示只有2种是正品白头翁, 正品率仅为6.7%, 白头翁的基原鉴定显得十分迫切。本课题组前期研究也表明利用ITS2序列能准确鉴别白头翁药材及伪品[7]。Xing等[8]利用高通量测序技术可成功对牛蒡子药材及伪品药材的混合粉末进行鉴定, 但尚未见多种白头翁药材混合样品DNA分子鉴定的研究报道。
第二代测序技术, 又称新一代测序技术(next-generation sequencing), 已广泛应用于动植物全基因组测序、基因组重测序、转录组测序、小RNAs测序和表观基因组测序等方面[9]。随着测序技术的提高和基因组学的发展, 对单一物种DNA序列的测定已经不能满足学科的发展, 人们提出了一种新的研究思路, 即宏基因组学(metagenomics)研究策略[10, 11]。宏基因组学是研究生态群体基因功能和其相互作用的新科学领域, 以特定生态环境中微小生物遗传物质的总和作为研究对象, 基本研究策略是通过克隆、异源表达, 筛选出有用的新基因及其产物[12]。Handelsma等[13]在研究土壤微生物的文章中定义宏基因组为“微生物群落中的所有基因组的集合”。高通量测序技术通量高、速度快, 适合宏基因组的深度测序研究[14], 使宏基因组的测序分析得以进一步发展。本文对五种白头翁药材的混合粉末进行高通量测序, 采用宏基因组学物种多样性的方法进行分析, 对混合样品进行物种鉴定, 为混合中药材的基原鉴定提供新的方法和思路。
材料与方法试材 白头翁干燥根于2016年采集于辽宁省鞍山市千山、朝鲜白头翁干燥根于2016年采集于辽宁省凤城市、兴安白头翁干燥根于2016年采集于黑龙江省伊春市、细叶白头翁干燥根于2016年采集于内蒙古自治区通辽市、金县白头翁干燥根于2016年采集于辽宁省大连市大黑山, 均由辽宁中医药大学康廷国教授鉴定。
试剂 天根植物提取试剂盒, 1×TAE缓冲液(Solarbio公司), 琼脂糖(Promega公司), 溴化乙锭(Fluka公司), DNA Taq聚合酶(TaKaRa公司), 2000 bp DNA Marker (TaKaRa公司)。
仪器 PCR仪(ABI公司, 型号9700), 电泳系统(北京市六一仪器厂, 型号DYY-12), 低温冷冻离心机(德国Eppendorf, 型号5810R), 紫外凝胶成像分析仪(英国Syngene, 型号GBOXHR), 微量移液器(德国Eppendorf)。
混合粉末样品总DNA提取 取5种白头翁药材各5 g, 等比混合, 用研钵研碎, 取混合粉末100 mg作为样品。使用天根植物总DNA提取试剂盒提取样品的总DNA, 操作方法按试剂盒使用说明进行。获取混合样品总DNA, 并通过0.8%琼脂糖凝胶电泳检测DNA提取质量, 同时采用紫外分光光度计对DNA进行定量。
PCR扩增 混合样品宏基因组物种多样性分析, 目标为核糖体RNA中ITS2片段, 根据序列中的保守区域设计相应引物, 引物序列为ITS3: 5'-GCATCGATGAAGAACGCAGC-3'; ITS4: 5'-TCCTCCGCTTATTGATATGC-3'。扩增体系(25 μL): 5×反应buffer 5 μL, 5×GC buffer 5 μL, dNTP (2.5 mmol·L-1) 2 μL, 上游引物(10 μmol·L-1) 1 μL, 下游引物(10 μmol·L-1) 1 μL, DNA模板(4 μmol·L-1) 2 μL, ddH2O 8.75 μL, Q5 DNA聚合酶0.25 μL。扩增参数: 98 ℃预变性2 min, 98 ℃变性15 s, 55 ℃退火30 s, 72 ℃延伸30 s, 25~ 30循环, 72 ℃伸长5 min, 10 ℃保存。PCR扩增产物通过2%琼脂糖凝胶电泳进行检测, 并对目标片段进行切胶回收, 回收采用AXYGEN公司的凝胶回收试剂盒。
测序文库制备 使用Illumina MiSeq平台对药材DNA片段进行双端(paired-end)测序。采用滑动窗口法对FASTQ格式的双端序列逐一进行质量筛查:窗口大小为10 bp, 步长为1 bp, 从5'端第一个碱基位置开始移动, 要求窗口中碱基平均质量≥Q20 (即碱基平均测序准确率≥99%), 从第一个平均质量值低于Q20的窗口处截断序列, 并要求截断后的序列长度≥150 bp, 且不允许存在模糊碱基(ambiguous base) N。利用FLASH软件[15], 对通过质量初筛的双端序列根据重叠碱基进行配对连接:要求Read 1和Read 2两条序列的重叠碱基长度≥10 bp, 且不允许碱基错配。最后, 根据每个样本所对应的Index信息(即Barcode序列, 为序列起始处用于识别样本的一小段碱基序列), 将连接后的序列识别分配入对应样本(要求Index序列完全匹配), 从而获得每个样本的有效序列。
使用QIIME软件默认参数, 调用UCLUST这一序列比对工具[16], 对前述获得的序列按95%的序列相似度进行归并和OTU划分, 并选取每个OTU中丰度最高的序列作为该OTU的代表序列。将丰度值低于全体样本测序总量0.001% (十万分之一)的OTU去除[17], 并将去除了稀有OTU的此丰度矩阵用于后续的一系列分析。利用ITS序列的数据库: UNITE数据库[18] (Release 5.0, https://unite.ut.ee/)和NCBI (https://www.ncbi.nlm.nih.gov/)将OTU代表序列与对应数据库的模板序列相比对, 获取每个OTU所对应的分类学信息。
分类等级与进化分析 使用GraPhlAn可视化工具[19], 对样本总体在各分类水平的组成构建等级树, 发现优势类群。并使用QIIME软件将所有OTU代表序列与白头翁、朝鲜白头翁、兴安白头翁、细叶白头翁、金县白头翁的ITS2参考序列比对, 进一步进行筛选, 使用MEGA 6.0对筛选所得序列进行种水平上的进一步分析。
结果 1 测序结果对5种白头翁药材混合粉末进行宏基因组物种多样性分析, 共得到53 024条序列, 序列长度集中在147~417 bp, 如图 1。其中长度为384 bp的序列最多为29 443条, 其次是长度为383 bp和322 bp的序列分别为18 804和1 430条。
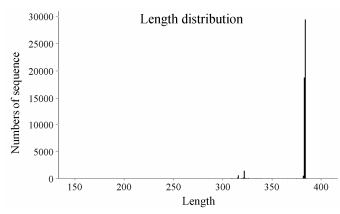
|
Figure 1 Sequence length distribution diagram. The horizontal coordinate is the length distribution of the sequence in all samples, and the vertical coordinate is the total number of sequences corresponding to each length value |
将混合粉末样品中的52 295条ITS2有效序列划分为384个OTU, 根据OTU划分和分类地位鉴定结果, 可以获得样本在各分类水平的具体组成, 分类结果见表 1、表 2。使用NCBI选取的白头翁属植物的ITS2序列与样本中所有OTU进行聚类分析, 有222个OTU与白头翁属植物的ITS2序列聚为一支, 将222个OTU与NCBI中选取的白头翁、朝鲜白头翁、兴安白头翁、细叶白头翁、金县白头翁序列进行聚类分析, 其中有179个OTU与白头翁属5种植物分别聚为一支。
| Table 1 Statistical table of species number of different classifycation levels |
| Table 2 Statistical table of sequences and classification of species |
利用GraPhlAn可视化工具[19], 对样本384个OTU在各分类水平的组成构建等级树如图 2。其中Ⅰ节点表示白头翁属植物, 白头翁属序列占有效序列总量的93.8%, 微生物序列占比小于7%, 可以看出白头翁属植物平均丰度最大, 属于优势类群。
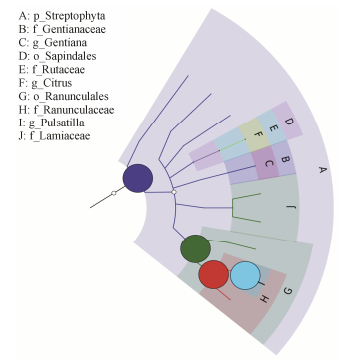
|
Figure 2 The classification level tree map of all sample based on GraphlAn. The classification tree shows the hierarchical relationships of all the taxa (represented by nodes) from phylum to genus (from inner ring to outer ring) in the sample. The size of the node corresponds to the average relative abundance of the taxa. The top 10 taxonomic units also be marked with letters in the figure (arranged in order from the outer layer to the inner layer from phylum to genus). The color of the shadow on the letter was the same as that of the corresponding node |
使用MEGA 6.0对NCBI选取本课题组前期上传的五种白头翁药材的已知ITS2序列与样品中OTU代表序列进行NJ系统分类法分析, 筛选出与混合样品中物种进化关系匹配度高的26个OTU, 其相似度达99%以上, 进一步NJ聚类分析, 结果如图 3。

|
Figure 3 Molecular phylogenetic tree of screening sequence constructed by Neighbour-Joining clustering analysis. The bootstrap scores (1 000 replicates) are shown |
编号denovo157、denovo51、denovo259、denovo69、denovo303、denovo145、denovo295的OTU总计有37 974条序列, 约占序列总数的71.6%, 与朝鲜白头翁的ITS2参考序列相似度最高, 在邻接法分类系统树里与本课题组前期成功上传至GenBank中的3条朝鲜白头翁序列聚为一支。编号denovo70、denovo112、denovo8、denovo50、denovo379、denovo356的OTU总计有1 068条序列, 与白头翁的ITS2参考序列相似度最高, 在邻接法分类系统树里与本课题组前期成功上传至GenBank中的3条白头翁序列聚为一支。编号denovo383、denovo139的OTU总计有59条序列, 与金县白头翁的ITS2参考序列相似度最高, 在邻接法分类系统树里与本课题组前期成功上传至GenBank中的3条金县白头翁序列聚为一支。编号denovo342、denovo48、denovo154、denovo2、denovo13、denovo337的OTU总计有61条序列, 与兴安白头翁的ITS2参考序列相似度最高, 在邻接法分类系统树里与本课题组前期成功上传至GenBank中的3条兴安白头翁序列聚为一支。编号denovo153、denovo260、denovo121、denovo152、denovo380的OTU总计有398条序列, 与细叶白头翁的ITS2参考序列相似度最高, 在邻接法分类系统树里与本课题组前期成功上传至GenBank中的3条细叶白头翁序列聚为一支。混合样品中5种白头翁都被鉴定出, 鉴定出的序列数量占白头翁属5种药材序列总量的80%以上。综上, 此混合样品含有白头翁、朝鲜白头翁、兴安白头翁、细叶白头翁、金县白头翁, 结果表明此方法可以对5种白头翁药材混合样品进行准确鉴定。
讨论中药分子鉴定是分子生药学的研究任务之一, 近些年来, 中药分子鉴定快速发展[20]。分子鉴定技术是指通过直接分析遗传物质DNA的多态性来推断物种内在的遗传变异而实现药材鉴定的方法[21]。这种方法已广泛应用于中成药中原料药的鉴定, 如Song等[22]运用shotgun metagenomic sequencing方法对龙胆泻肝丸中成药的鉴定, 可以检测到所有的混合物种基原信息, 为商品中成药的物种鉴定提供新思路。Cui等[23]运用荧光测序分型技术对六味地黄丸原料药进行分子鉴定, 可成功鉴别出3种原料药; 成药连翘败毒丸中的19种原料药材可以通过psbA-trnH和rbcL序列鉴定出10种[24]。Gou等[25]采用叶绿体基因trnL-F序列和当归特异鉴别标记, 成功对10份中成药中当归原料药材进行了鉴别。
中药材DNA条形码分子鉴定是以ITS2为主体条形码序列鉴定中药材的方法体系, 其中植物类中药材选用ITS2为主体序列[26]。如Chen等[27]利用ITS2序列对棘豆属药材的鉴定, Luo等[28]利用ITS2序列对秦艽正伪品的鉴定, Xin等[29]利用ITS2序列对羌活药材的鉴定等。本研究采用高通量测序技术对样品中的ITS2序列进行测序, 进而对混合粉末中的物种进行鉴定。利用此方法可鉴定出混合粉末中的5种白头翁药材。
通过高通量测序技术还有一部分OTU被鉴定为各个不同科属的微生物, 但是丰度较低。这些物种类群的来源可能为白头翁药材本身带有的微生物, 但不是白头翁药材中的主要成分。
通过NJ聚类分析, 可以看出白头翁、金县白头翁、兴安白头翁聚为一大支, 可推测三者有较近的亲缘关系。并且, 白头翁与金县白头翁进一步聚成一支, 两者亲缘关系更近。
尽管高通量测序技术有诸多的优势, 但其局限性也不容忽视[30]。与朝鲜白头翁聚为一支的denovo259含有37 653条序列, 与细叶白头翁聚为一支的denovo260仅有2条序列, 可能是由于高通量测序技术自身存在一些不足之处, 如测序过程中碱基偏好性、组装偏差等都会导致与细叶白头翁相似度较高的OTU未被分析。同时, 也可能是由于PCR技术本身的局限性导致对不同物种的DNA模板扩增效率不同, 使该物种的DNA扩增产物浓度过低, 出现该结果。
本实验主要对测序后的结果进行比对, 实现中药混合粉末的鉴定。因此, 用于比对的数据库的大小和数据的准确性对药材的鉴定结果具有十分重要的影响。随着分子标记技术的不断发展, 可以筛选出更合适的DNA进行标记, 为药材的成功鉴定提供基础, 使本方法能更广泛、更深入的运用到中药混合粉末的DNA分子鉴定中。
| [1] | Chinese Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China. Part 1(中华人民共和国药典一部)[S]. Beijing: China Medical Science Press, 2015: 104. |
| [2] | Gu GG, Yang PJ. Shennong's Herbal (神农本草经)[M]. Beijing: Xueyuan Press, 2007: 262-263. |
| [3] | Tao HJ. Bencao Jing Jizhu (本草经集注)[M]. Beijing: People's Medical Publishing House, 1994: 370-371. |
| [4] | Li SZ. Bencao Gangmu (本草纲目)[M]. Beijing: Huaxia Press, 2013: 1116. |
| [5] | Liang YM, Zhao R, Xu L, et al. Herbal textual analysis of Pulsatillae Radix and taxonomy of Pulsatilla sect in China[J]. Chin J Exp Tradit Med Form (中国实验方剂学杂志), 2017, 23: 203–209. |
| [6] | Shi YH, Zhao MM, Yao H, et al. Rapidly discriminate commercial medicinal Pulsatilla chinensis (Bge.) Regel from its adulterants using ITS2 barcoding and specific PCR-RFLP assay[J]. Sci Rep, 2017, 7: 40000. DOI:10.1038/srep40000 |
| [7] | Liang YM, Chen SY, Xu L, et al. Identification of plants and herbs of Pulsatilla genus based on ITS2 barcode[J]. J Chin Med Mater (中药材), 2017, 40: 1547–1551. |
| [8] | Xing YP, Chen SY, Xu L, et al. Study on high throughput sequencing identification of Fructus Arctii and five spurious species mix power[J]. China J Chin Mater Med (中国中药杂志), 2018, 43: 303–308. |
| [9] | Yue GD, Gao Q, Luo LH, et al. The application of high-throughput sequencing technology in plant and animal research[J]. Chin Bull Life Sci (生命科学), 2012, 42: 107–124. |
| [10] | Handelsman J. Metagenomics:application of genomics to uncultured microorganisms[J]. Microbiol Mol Biol Rev, 2004, 68: 669–685. DOI:10.1128/MMBR.68.4.669-685.2004 |
| [11] | Hugenholtz P, Tyson GW. Microbiology:metagenomics[J]. Nature, 2008, 455: 481–483. DOI:10.1038/455481a |
| [12] | Chu YL, Yang E. Research progress of metagenomics and its technology[J]. J Xi'an Jiaotong Univ (西安交通大学学报), 2008, 29: 601–608. |
| [13] | Handelsman J, Rondon MR, Brady SF, et al. Molecular biological access to the chemistry of unknown soil microbes:a new frontier for natural products[J]. Chem Biol, 1998, 5: R245–R249. DOI:10.1016/S1074-5521(98)90108-9 |
| [14] | Liu LY, Cui HF, Tian G. Application of high-throughput sequencing technology in metagenomics[J]. Chin Med Biotechnol (中国医药生物技术), 2013, 8: 196–200. |
| [15] | Magoc T, Salzberg SL. FLASH:fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27: 2957–2963. DOI:10.1093/bioinformatics/btr507 |
| [16] | Edgar RC. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26: 2460–2461. DOI:10.1093/bioinformatics/btq461 |
| [17] | Bokulich NA, Subramanian S, Faith JJ, et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nat Methods, 2013, 10: 57–59. DOI:10.1038/nmeth.2276 |
| [18] | Koljalg U, Nilsson RH, Abarenkov K, et al. Towards a unified paradigm for sequence-based identification of fungi[J]. Mol Ecol, 2013, 22: 5271–5277. DOI:10.1111/mec.12481 |
| [19] | Asnicar F, Weingart G, Tickle TL, et al. Compact graphical representation of phylogenetic data and metadata with GraPhlAn[J]. PeerJ, 2015, 1029: 1–17. |
| [20] | Huang LQ, Liu CX. Molecular Pharmacognosy (分子生药学)[M]. Beijing: Science Press, 2015: 20. |
| [21] | Shi SM, Pan MJ, Wang J, et al. Application of molecular identification techniques in Chinese materia medica[J]. Chin Tradit Herb Drugs (中草药), 2016, 47: 3121–3126. |
| [22] | Xin TY, Su C, Lin YL, et al. Precise species detection of traditional Chinese patent medicine by shotgun metagenomic sequencing[J]. Phytomedicine, 2018, 47: 40–47. DOI:10.1016/j.phymed.2018.04.048 |
| [23] | Cui ZH, Huang LQ, Yuan Y, et al. Study on fluorescence sequencing typing technology identification of raw materials in Liuwei Dihuang Pill[J]. China J Chin Mater Med (中国中药杂志), 2014, 39: 3695–3700. |
| [24] | Cui ZH, Jiang C, Li MH, et al. Molecular identification of raw materials from Lian Qiao Bai Du Wan[J]. Acta Pharm Sin (药学学报), 2013, 48: 590–596. |
| [25] | Gou H, Wang YW, Zheng Q, et al. DNA extraction and molecular identification in Angelicae Sinensis Radix preparations[J]. Chin J Exp Tradit Med Form (中国实验方剂学杂志), 2018, 24: 44–50. |
| [26] | Chen SL, Yao H, Han JP, et al. Principles for molecular identification of traditional Chinese materia medica using DNA barcoding[J]. China J Chin Mater Med (中国中药杂志), 2013, 38: 141–148. |
| [27] | Chen SY, Wu YN, Xu L, et al. DNA barcoding of Mongolian Oxytropis medicinal materials[J]. J Chin Med Mater (中药材), 2016, 39: 284–288. |
| [28] | Luo K, Ma P, Yao H, et al. Identification of Gentianae Macrophyllae Radix using the ITS2 barcodes[J]. Acta Pharm Sin (药学学报), 2012, 47: 1710–1717. |
| [29] | Xin TY, Yao H, Luo K, et al. Stability and accuracy of the identification of Notopterygii Rhizoma et Radix using the ITS/ITS2 barcodes[J]. Acta Pharm Sin (药学学报), 2012, 47: 1098–1105. |
| [30] | Wang XC, Yang ZR, Wang M, et al. High-throughput sequencing technology and its application[J]. China Biotechnol (中国生物工程杂志), 2012, 32: 109–114. |
 2018, Vol. 53
2018, Vol. 53


