2. 川北医学院基础医学院, 四川 南充 637007;
3. 国家蛋白质科学中心, 蛋白质组学国家重点实验室, 北京 102206
2. School of Basic Medical Sciences, North Sichuan Medical College, Nanchong 637007, China;
3. State Key Laboratory of Proteomics, National Center for Protein Sciences, Beijing 102206, China
赫赛汀是靶向HER2的人源单克隆抗体, 能够结合HER2胞外段亚结构域IV, 通过抗体依赖细胞介导细胞毒作用(antibody-dependent cell-mediated cytotoxicity, ADCC)和抑制信号转导发挥对肿瘤细胞的杀伤作用[1-3]。该药于1998年首次获准用于HER2表达阳性转移性乳腺癌的治疗, 2010年又获准用于HER2表达阳性晚期胃癌的治疗。作为第一个用于临床的单克隆抗体药物和信号转导抑制剂, 赫赛汀在肿瘤治疗中表现出良好的应用潜能[4]。
尽管赫赛汀能够延长乳腺癌及胃癌患者的总生存期和无进展生存期, 然而由于客观治疗有效率不高, 且起初接受赫赛汀治疗有效的多数患者, 在用药8个月后肿瘤细胞即产生明显抗药性, 导致药效降低甚至无效[5-7]。由于赫赛汀用于胃癌治疗时间尚短, 因此, 胃癌赫赛汀耐药机制研究尚未深入开展, 加之肿瘤耐药的复杂性和多样性, 导致人们对其耐药机制的认识还不够, 已报道的研究结果仍无法系统阐明胃癌赫赛汀耐药的速度和广度。因此, 探索并研究其耐药机制, 不仅为延迟或逆转肿瘤耐药治疗提供科学依据, 也为新型靶向药物的设计、开发提供理论指导。
蛋白质组学(proteomics)本质是研究生物体蛋白质的表达、翻译后再修饰、蛋白质与蛋白质相互作用等, 由此获得蛋白水平上关于疾病发生、发展的变化规律[8, 9]。以质谱为手段的蛋白质组学的快速发展是蛋白质组高通量筛选的重要保障。基于蛋白质组学技术, 可以批量研究生物体在外界刺激下, 或病理生理状态下蛋白质谱的变化, 探索并洞察蛋白质量变背后隐藏的生物学意义。近年来, 国家蛋白质科学中心秦钧教授团队发展了一种非标定量蛋白质新技术, 能够在12 h有效质谱时间内检测并定量超过7 000个蛋白质[10], 该技术已广泛应用于细胞、组织、体液等生物样本的蛋白质组研究, 为实现大样本蛋白质的快速鉴定与定量研究提供了技术保障。
本研究以赫赛汀敏感细胞N87及赫赛汀耐药细胞N87/R为研究对象, 分析母本和耐药细胞蛋白质组表达水平的差异, 以获得与赫赛汀耐药相关蛋白质谱信息。采用生物信息学技术对差异蛋白质进行基因本体(gene ontology, GO)分析、基因–疾病分析、激活通路富集及网络构建分析, 并对发生显著改变的信号通路进行生物学验证。本研究为深入研究胃癌赫赛汀耐药机制及延迟或逆转耐药治疗提供了理论基础。
材料与方法仪器 质谱仪(Obitrap FUSION)、Easy-nLC 1000 nano-液相色谱系统(Thermo公司); 自制反相C18预分离柱[填料粒径3 μm, 孔径120 Å (1 Å = 0.1 nm), 2 cm × 100 μm]、自制反相C18分析柱(填料粒径1.7 μm, 孔径120 Å, 15 cm × 150 μm, SunChrom公司)。多功能超声仪(Uibracell公司); 光学显微镜(Olympus公司)。质谱参数:母离子(precursor ions)的质量扫描范围为m/z 300~1 400, 分辨率为120 000, 扫描模式为Top-speed, 母离子选择后进入高能碰撞室(higher energy collision-induced dissociation, HCD)进行碎裂, 碰撞能为37%, 获得的子离子(daughter ions)进入线性离子阱进一步检测。母离子的肽段电荷数设为+2、+3、+4、+5和+6。Orbitrap一级扫描的自动增益控制(automatic gain control, AGC)为5e5, MS/MS二级扫描的AGC为5e3;动态排除时间设定为18 s。
材料 N87和N87/R细胞株由军事医学科学院施明教授提供; 赫赛汀(罗氏制药); 胎牛血清(FBS, Gibco公司); DMEM培养基、胰酶(Mediatech公司); 双抗(北京钮英泰克生物公司); HER2抗体(Abcam公司, #16901); ERK1/2抗体(#8544)、β-actin (#12620) (Cell Signaling Technology公司); HRP标记山羊抗兔二抗(中杉金桥, #ZB-2301);二硫苏糖醇(DTT)、吲哚-3-乙酸(IAA)、尿素、碳酸氢钠、氢氧化钠(Sigma公司); 甲醇、乙腈、甲酸、质谱级水、蛋白酶抑制剂(Thermo公司); SCH772984 (MedChem Express公司, #HY-50846); CCK-8 kit (同仁化学); Bradford蛋白定量试剂盒、高灵敏化学发光检测试剂(天根生化科技)。
细胞培养 N87和N87/R细胞培养于含10% FBS和1%双抗的完全培养液中, 根据细胞生长状态换液、清洗和传代。耐药细胞培养期间加入定量赫赛汀, 最终质量浓度为20 μg·mL-1。
细胞增殖实验 取对数期生长的母本和耐药细胞, 均匀吹打制成单细胞悬液, 种于96孔培养皿, 细胞数约为4 000个/孔, 培养过夜, 弃去旧培养液, 每孔加入新鲜培养液100 μL。耐药细胞加入质量浓度分别为0、5、10和20 μg·mL-1赫赛汀, 每个浓度设3个复孔, 培养5天, 弃去旧培养液, 加入含10% CCK-8的无血清培养液100 μL, 避光孵育3 h, 检测OD450 nm处吸光度, 实验重复3次。
提取蛋白 收集细胞, 沉淀用预冷磷酸盐缓冲液(PBS)洗涤2次; 加入4倍体积、浓度为8 mol·L-1尿素和1%的蛋白酶抑制剂, 震荡混匀, 冰上裂解10 min; 超声2 min; 4 ℃, 16 000 ×g离心10 min; 取上清, Bradford法测蛋白浓度。
还原烷基化 取蛋白样品100 μg, 加入浓度为0.5 mol·L-1, 1/100体积的DTT, 震荡混匀, 56 ℃水浴反应30 min; 加入浓度为1 mol·L-1, 1/50体积的IAA, 震荡混匀, 室温避光30 min; 加入浓度为0.5 mol·L-1, 1/100体积的DTT, 混匀, 室温避光15 min; 16 000×g离心5 min, 取上清, 待用。
FASP酶解[11] 蛋白样品加入10 kD超滤管中, 16 000 ×g离心40 min; 加入浓度为50 mmol·L-1 NH4HCO3溶液300 μL, 16 000×g离心3次, 每次40 min, 弃去滤液。另取干净超滤管, 加入浓度为50 mmol·L-1 NH4HCO3 50 μL和5 μg胰蛋白酶混合液, 于37 ℃恒温箱酶解4 h; 补加50 mmol·L-1 NH4HCO3 150 μL和5 μg胰蛋白酶, 于37 ℃旋转酶解过夜; 16 000 ×g离心40 min, 收集液体, 加入300 μL质谱水清洗1次, 离心; 合并两次洗脱液, 混匀, 真空抽干。
样品纯化及sRP柱分离 称取C18粉末, 加适量乙腈, 充分震荡后等分到Tip 400柱, 加乙腈洗脱2次, 使用乙腈体积分数分别为50%、80%、50%、0%、0%的NH4HCO3依次洗脱1次, 每次100 μL, 加入NH4CO3 80 μL洗去非特异结合肽段; 使用乙腈体积分别为6%、9%、12%、15%、18%、21%、25%、30%和35%的NH4HCO3溶液洗脱4次, 收集各组分洗脱液, 按照6%+25%、9%+30%、12%+35%、15%、18%和21%方式合并组分段; 减压抽干, 样品储存于-20 ℃, 备用。
洗脱系统、蛋白鉴定、定量及差异蛋白质的筛选 肽段样品用0.1%甲酸溶解, 充分振荡, 16 000 ×g离心10 min, 取上清, 采用Easy-nLC 1000 nano-液相系统进行分离。液相条件:流动相A (0.1%甲酸-水), 流动相B (0.1%甲酸-乙腈), 洗脱梯度为0~5 min, 95% A; 5~21 min, 95%~92% A; 21~55 min, 92%~45% A, 55~65 min, 45%~35% A, 65~75 min, 35%~5% A; 流速为600 nL·min-1, 分离后的肽段进入Obitrap FUSION质谱仪在线检测。采用搭载Mascot 2.3 (Matrix Science Inc.)的Proteome Discover 2.1 (Thermo公司)数据库进行搜库, 数据库为人类生物技术信息中心(Human National Center for Biotechnology Information, NCBI)蛋白质库(更新至04/07/2013, 包含蛋白质32 015个)。检索参数设置如下:母离子质量公差(mass tolerances)为20 ppm, 子离子为0.5 Da; 允许胰酶漏切数≤2;肽段长度为7~25个氨基酸; 蛋白质修饰为:半胱氨酸末端的脲甲基化(carbamidomethylation)为固定修饰, 蛋氨酸N-端乙酰化及氧化(N-term acetylation和oxidation)为动态修饰, 肽段水平的假阳性率(false discovery rate, FDR)设定为1%。采用IBQ方法进行蛋白质定量, 其基本原理为每个蛋白质鉴定的全部肽段曲线下的面积和(areas under the curve, AUCs)与该蛋白质理论酶切肽段数(N)之比为该蛋白质的表达量(AUCs/N)。采用FOT (fraction of total)代表蛋白质标准化后的峰值, 其定义为每个蛋白质的IBQ除以该样本中鉴定的所有蛋白质的IBQ。为了使数据具有可读性, 将FOT乘以105得到FOT5。肽段筛选标准为:每个蛋白质被检测的特异肽段数(unique peptides)≥2, 且Mascot ions scores ≥20。数据分析采用两样本的配对t检验, 若某蛋白的定量在母本和耐药组中的比值≥2且满足P < 0.05, 则认为该蛋白质为差异表达蛋白。
Western blot 按上述方法制备蛋白样品, 取等量蛋白, 加入2×上样缓冲液10 μL, 95 ℃煮5 min; 上样; 聚丙烯酰胺凝胶电泳; 湿法转膜; 用含5%脱脂牛奶的TBST室温封闭1 h; 4 ℃一抗孵育过夜; TBST漂洗5次, 每次5 min; 二抗室温孵育1 h; TBST漂洗5次, 每次5 min; 加入高灵敏化学发光检测试剂, 暗室曝光, 冲洗胶片, 扫描图像。
SCH772984对细胞增殖的影响 取对数期生长的母本和耐药细胞, 均匀吹打制成单细胞悬液, 种于96孔板, 细胞数约为6 000个/孔, 于37 ℃培养24 h, 弃去旧培养液, 每孔加入新鲜培养液100 μL, 过夜培养。用不同浓度的SCH772984 (0.1、1、10、100、400和1 600 nmol·L-1)处理细胞48 h (DMSO为参照, 浓度≤0.1%, v/v); 加入含10% CCK-8的无血清培养基100 μL, 避光孵育3 h, 测定OD450 nm处的吸光度, 实验重复3次。
结果 1 赫赛汀获得性耐药细胞模型评价细胞在赫赛汀质量浓度分别为5、10和20 μg·mL-1的培养液中连续培养5天, 测得母本和耐药细胞的相对细胞活力分别为84.27%、74.02%、69.15%和96.31%、95.72%、94.60%。与耐药组相比, 母本组细胞活力随药物浓度增加显著降低(图 1), 而耐药细胞活力无明显变化, 表明N87细胞经赫赛汀长时间诱导后产生了抗药性。
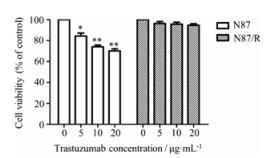
|
Figure 1 Cell viability was evaluated by CCK-8 kit. n = 3, x± s. *P < 0.05, **P < 0.01 vs control |
基于Western blot检测了母本和耐药细胞中HER2的表达水平。结果显示, 母本组和耐药组HER2均高表达, 且二者差异无统计学意义(图 2)。
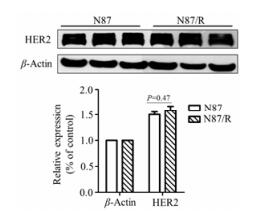
|
Figure 2 Expression of HER2 in N87 and N87/R cells was confirmed by Western blot |
首先, 在肽段水平设置了1% FDR, 12个样本共检测到蛋白质10 308个; 在此基础上, 执行了更加严格的筛选, 即在蛋白水平设置了1% FDR, 得到8 509个蛋白质; 其中7 163个蛋白质在8个样本中覆盖(这些蛋白的定量具有高可信性); 将这7 163个蛋白质用于生物信息学及统计学分析。为了明确数据整体分布, 对耐药组和母本组中所有蛋白质的变化倍数(fold change)进行分析。将耐药组与母本组6次生物重复样本中蛋白质峰面积均值的比值作为变化倍数, 再进行对数转化, 得图 3。由图得知, 超过95%的蛋白质在耐药组/母本组中的变化倍数介于上下调2倍范围内; 不足5%的蛋白质丰度波动较大(这些蛋白质具有较小的iFOT (iFOT < 1), 或者在样本中被检测的随机性较大, 均值偏差较大)。分析可知, 耐药组/对照组数据整体呈正态分布态势, 因此, 数据适合配对样本的t检验分析。根据显著性水平及均值的变化倍数绘制了火山图(volcano plot) (图 3)。差异表达蛋白质的指认标准为: fold change (N87/R/N87) ≥2且P < 0.05为上调; fold change≤0.5且P < 0.05为下调。代表上调蛋白质对应的点在火山图中注释为红色(右上侧位置), 代表下调蛋白质对应点的注释为蓝色(左上侧位置), fold change < 2或P≥0.05的蛋白质, 认为其表达未发生显著变化, 图中注释为灰色(中间和下侧位置)。筛选发现180个蛋白质在耐药组中发生了显著改变, 其中上调110个(占总蛋白的1.5%), 下调70个(占总蛋白的0.9%), 表达未显著改变的蛋白质6 983个(占总蛋白的97.6%)。
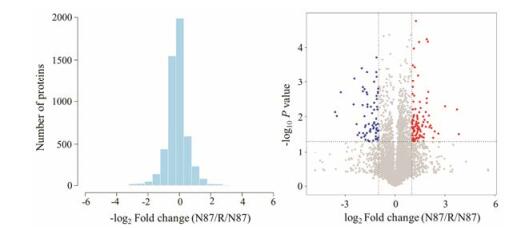
|
Figure 3 The distribution of protein abundance ratios is displayed with histogram and differentially expressed proteins are illustrated by volcano plot |
基于Web Gestalt (http://bioinfo.vanderbilt.edu/webgestalt/option.php)数据库对差异表达蛋白分别从生物过程(biological process)、细胞成分(cellular component)和分子功能(molecular function)层面进行GO注释分析(图 4)。就细胞成分而言, 上调蛋白质主要位于细胞质膜、细胞衔接处及细胞外围, 而下调蛋白质主要定位在胞外区及MHC蛋白复合物; 在分子功能方面, 上调蛋白质主要表现为调节cAMP反应元件结合蛋白活性、转录因子结合活性、环蛋白依赖性蛋白激酶活性方面, 下调蛋白质则主要参与调控清道夫受体活性、Aldo-keto还原酶活性及乙醇脱氢酶活性方面。从生物过程来看, 上调蛋白质主要体现在调控T细胞因子活性、免疫效应及有机酸的转运过程, 而下调蛋白质在外界刺激反应、上皮细胞发育及转运调控方面具有重要作用。
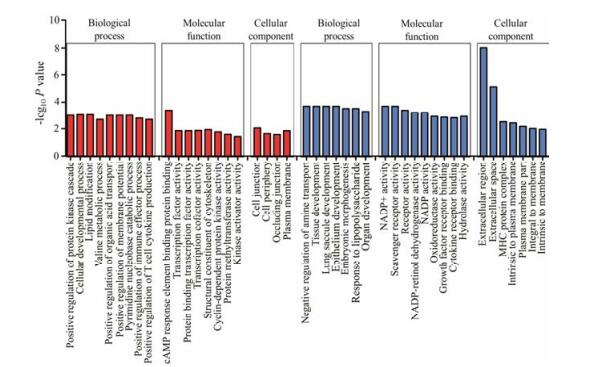
|
Figure 4 GO enrichment of up-regulated proteins (red bar) and down-regulated proteins (blue bar) involved in cell component, molecular function and biological process |
为了探索差异表达蛋白质对应的基因可能参与调控的疾病类型, 基于Web Gestalt数据库对基因–疾病关联性进行分析。结果表明, 部分基因直接或间接参与调控包括肿瘤在内的多种疾病, 以及与肿瘤转移、侵袭及恶化程度相关的多个生物学过程。依据P值筛选出相关性最高的9类, 分别为表皮癌、肿瘤转移、肿瘤侵袭、胃肠道肿瘤、肿瘤、炎症、肿瘤过程、胃溃疡及骨肉瘤。富集结果见图 5。
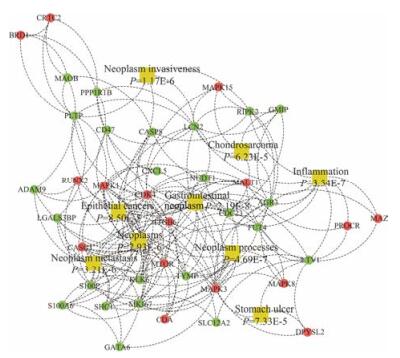
|
Figure 5 Genes associated with diseases were shown according to the enrichment analysis in Web Gestalt. Each square indicates one disease. Red and green nodes represent up/down-regulated proteins respectively |
为了进一步探明耐药细胞信号通路的变化, 利用STRING (v10.5)数据库对相互作用得分大于0.7的差异表达蛋白质进行网络构建, 进一步基于Web Gestalt数据库中Wikipathway模块, 对差异表达蛋白质执行了通路富集分析, 利用Cytoscape (v3.6.0)软件对富集结果进行了可视化展示(图 6)。筛选出显著性水平P < 1×10-4的通路有8条, 分别为IL-2、MAPK/ERK、mTOR、aurora A、NF-κB、Ret激酶、免疫调控及代谢通路。显而易见, MAPK/ERK和IL-2通路具有较小的P值(P = 1.6E-7和P = 8.1E-6), 提示激活IL-2和MAPK/ERK信号是N87细胞赫赛汀耐药的重要机制。
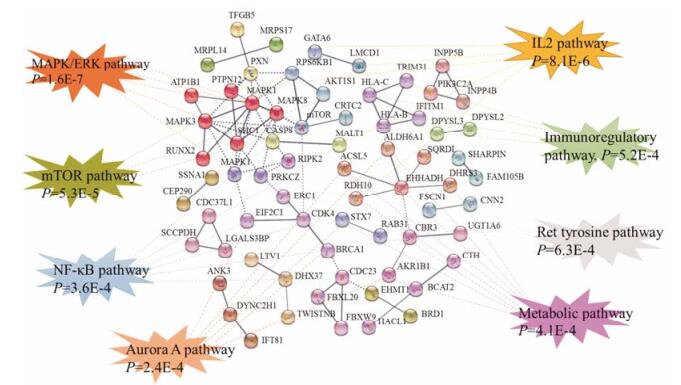
|
Figure 6 Wikipathway analysis identified major signaling pathways in which the differential proteins are involved. Each colored line indicated a different pathway |
利用STRING数据库对差异表达蛋白质的相互作用执行了网络构建, 采用Cytoscape软件对得分大于0.7的网络进行可视化展示(图 7)。分析网络得知, 具有不同相互作用关系的蛋白质分子构成了网络中的蛋白节点, 节点大小代表了相互作用关系的蛋白质数目, 节点越大表示与之关联的蛋白数越多, 反之越少。按照上述规则, 发现整个网络存在两个具有方向指向性的蛋白质集合, 分别代表了上调和下调蛋白质, 其中代表上调方向的主要节点蛋白质是MAPK1、MAPK3、MAPK8、mTOR、RPS6KB1、CDK4和EHHADH, 代表下调方向的主要节点蛋白质是SHC1、AKT1S1和CASP8。分析这些蛋白质功能发现, MAPK1、MAPK3和MAPK8属于MAPK/ ERK家族重要分子; mTOR、RPS6KB1和AKT1S1属于mTOR通路中的主要调控蛋白质; CDK4是重要的丝氨酸/苏氨酸蛋白激酶家族成员, 在细胞DNA合成和有丝分裂过程中发挥重要作用, 同时还参与细胞增殖、分化、黏附和迁移等过程, 也参与调控恶性肿瘤的发展、转移等过程[12, 13]; EHHADH在调控肿瘤代谢过程中发挥了重要作用[14]; SHC1参与细胞骨架的重塑, 对细胞损伤后修复具有重要作用[15]; CASP8是重要凋亡蛋白, 在细胞凋亡过程中, 直接参与凋亡启动及凋亡信号活化, 同时还参与细胞分化和转移等生物学过程[16, 17]。上述蛋白质分别从不同角度诠释了耐药细胞的生物学特性, 为全方位理解胃癌细胞赫赛汀耐药提供了广阔的洞察视角。
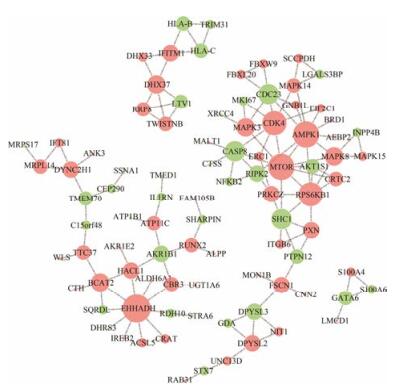
|
Figure 7 Network analysis of differential proteins was accomplished with Cytoscape software. Red nodes represent up-regulated proteins and green nodes represent down-regulated ones |
由蛋白质组数据得知, MAPK1、MAPK3、MAPK8、MAPK14和MAPK15的表达在耐药组中显著上调(图 8A), 这些蛋白质的变化趋势与激活MAPK/ERK通路的结论相符。为了进一步研究MAPK/ERK通路在母本和耐药细胞中的变化, 基于Western blot检测了MAPK/ERK通路中的关键调控蛋白ERK1/2的表达水平(图 8B, C)。结果表明, ERK1/2在耐药组中显著上调, 表明MAPK/ERK信号在耐药细胞中显著激活, 提示激活该通路是诱发胃癌赫赛汀耐药的重要机制。
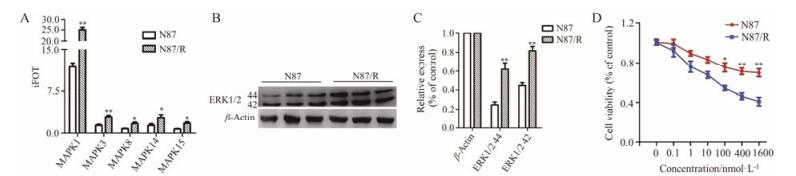
|
Figure 8 Validation of differentially expressed MAPK/ERK pathway proteins by mass spectrometry (A) and Western blot (B, C). SCH772984 preferentially inhibited the viability of N87/R cells (D). *P < 0.05, **P < 0.01 vs N87 |
另一方面, 使用MAPK/ERK通路特异性抑制剂SCH772984处理细胞, 观察药物对两组细胞活力的影响。结果表明, 与母本细胞相比, 100 nmol·L-1 SCH772984可使耐药细胞活力降低(P < 0.05);随着药物浓度的增加(400和1 600 nmol·L-1), 二者差异愈加显著(P < 0.01) (图 8D); 表明SCH772984对耐药细胞的增殖表现为偏好性抑制作用, 提示抑制MAPK/ERK通路能够降低耐药细胞的增殖。
讨论获得性耐药是导致肿瘤化疗失败的主要原因。近年来, 探索肿瘤耐药机制及其逆转耐药策略已成为肿瘤领域中的研究热点。肿瘤诱发耐药是一个极其复杂的过程, 需要启动一系列生物学过程, 包括信号通路的激活及生存信号的转变, 以及某些调控基因的突变。
HER2是当前肿瘤靶向分子治疗中研究最成熟、疗效最确切的治疗靶标之一[18-20]。赫赛汀作为第一个抗HER2的人源化单克隆抗体药物, 在转移性乳腺癌治疗中显示出良好的治疗效果[21]。近年来, 该药物在HER2表达阳性晚期胃癌治疗中备受关注, 随着药物的使用, 临床上胃癌赫赛汀耐药已成为当前面临的一项亟待解决的科学问题。文献报道的胃癌赫赛汀耐药机制包括: IL-6/STAT3/Jagged-1/Notch环路活化[22]; FGFR3过表达及其配体FGF9活化[23]; MACC1通过激活PI3K/AKT信号促使Warburg效应[24]; MUC4上调引起肾上腺β2受体激活; MUC1和MUC4上调激活STAT3信号[25, 26]; mTOR信号激活[27]。亦有文献报道, 赫赛汀耐药的N87细胞发生HER2表达上调, 过表达的HER2分子活化下游PI3K/AKT信号, 引起肿瘤细胞增殖、迁移、侵袭及恶性程度增强[28]。本研究中, 母本和耐药细胞中HER2表达无显著差异(P>0.05) (图 2), 提示本实验中诱发赫赛汀耐药的机制可能与文献报道不同。
结果显示, 多条通路在耐药细胞中显著改变, 包括IL-2、MAPK/ERK、mTOR、aurora A、Ret激酶、NF-κB、代谢和免疫调控。结合生物学实验证实, 抑制MAPK/ERK信号能够显著降低耐药细胞的增殖能力, 表明激活该通路是导致胃癌赫赛汀耐药的重要机制, 提示针对该通路的治疗有可能成为延迟或逆转赫赛汀耐药的治疗靶标。
研究表明, 活化或抑制HER2分子可触发细胞内多条复杂的信号转导, 包括Ras/Raf/MAPK、PI3K/ AKT和PLC/PKC通路等。MAPK/ERK作为HER2下游的重要信号分子, 在HER2参与调控的信号转导过程中发挥着重要作用, 通过和其他信号分子相互作用形成复杂的正反馈或负反馈参与信号转导过程, 共同调控恶性肿瘤的生物学行为[29, 30]。本实验证实, 采用赫赛汀抑制肿瘤细胞HER2分子的生物功能后, 其MAPK/ERK通路则异常活化, 提示在赫赛汀持续加压诱导下, 肿瘤细胞赖以生存的信号发生了转变, 这些信号通路的改变对肿瘤获得性耐药具有重要意义。有文献报道, MAPK/ERK通路的异常活化诱导肿瘤细胞抗凋亡能力增强, 进一步促使肿瘤的恶性行为, 因此针对该通路的药物治疗具有重要临床意义[31-33]。近年来还有一种观点认为, 刺激激活MAPK/ERK信号转导, 进一步激活下游抑癌基因, 导致肿瘤生长抑制[34], 这些不同的研究结果, 为科学家重新探索和研究该通路提出新的挑战。因此, 深入研究MAPK/ ERK通路在胃癌赫赛汀获得性耐药中的机制, 将为HER2阳性表达胃癌的靶向治疗提供科学依据, 也将为延迟或逆转胃癌赫赛汀耐药提供治疗策略。
| [1] | Roukos DH. Targeting gastric cancer with trastuzumab:new clinical practice and innovative developments to overcome resistance[J]. Ann Surg Oncol, 2010, 17: 14–17. DOI:10.1245/s10434-009-0766-0 |
| [2] | Gomez-Martín C, Lopez-Rios F, Aparicio J, et al. A critical review of HER2-positive gastric cancer evaluation and treatment:from trastuzumab, and beyond[J]. Cancer Lett, 2014, 351: 30–40. DOI:10.1016/j.canlet.2014.05.019 |
| [3] | Spector NL, Blackwell KL. Understanding the mechanisms behind trastuzumab therapy for human epidermal growth factor receptor 2 positive breast cancer[J]. J Clin Oncol, 2009, 27: 5838–5847. DOI:10.1200/JCO.2009.22.1507 |
| [4] | Tang X, Ding Q, Lin L, et al. Development of antibody drugs targeting against HER2 for cancer therapy[J]. Acta Pharm Sin (药学学报), 2012, 47: 1297–1305. |
| [5] | Pohlmann PR, Mayer IA, Mernaugh R. Resistance to trastu-zumab in breast cancer[J]. Clin Cancer Res, 2009, 15: 7479–7491. DOI:10.1158/1078-0432.CCR-09-0636 |
| [6] | Gajria D, Chandarlapaty S. HER2-amplified breast cancer:mechanisms of trastuzumab resistance and novel targeted therapies[J]. Expert Rev Anticancer Ther, 2011, 11: 263–275. DOI:10.1586/era.10.226 |
| [7] | Hudis CA. Trastuzumab-mechanism of action and use in clinical practice[J]. New Engl J Med, 2007, 357: 39–51. DOI:10.1056/NEJMra043186 |
| [8] | Li X, Wang W, Chen J. Recent progress in mass spectrometry proteomics for biomedical research[J]. Sci China Life Sci, 2017, 60: 1093–1113. DOI:10.1007/s11427-017-9175-2 |
| [9] | Abazova N, Krijgsveld J. Advances in stem cell proteomics[J]. Curr Opin Genet Dev, 2017, 46: 149–155. DOI:10.1016/j.gde.2017.07.007 |
| [10] | Ding C, Jiang J, Wei J, et al. A fast workflow for identification and quantification of proteomes[J]. Mol Cell Proteomics, 2013, 12: 2370–2380. DOI:10.1074/mcp.O112.025023 |
| [11] | Wisniewski JR, Zougman A, Nagaraj N, et al. Universal sample preparation method for proteome analysis[J]. Nat Methods, 2009, 6: 359–362. DOI:10.1038/nmeth.1322 |
| [12] | Liu F, Korc M. CDK4/6 inhibition induces epithelial-mesenchymal transition and enhances invasiveness in pancreatic cancer cells[J]. Mol Cancer Ther, 2012, 11: 2138–2148. DOI:10.1158/1535-7163.MCT-12-0562 |
| [13] | Baker SJ, Reddy EP. CDK4 a key player in the cell cycle, development, and cancer[J]. Genes Cancer, 2012, 3: 658–669. DOI:10.1177/1947601913478972 |
| [14] | Houten SM, Denis S, Argmann CA, et al. Peroxisomal L-bifunctional enzyme (EHHADH) is essential for the production of medium-chain dicarboxylic acids[J]. J Lipid Res, 2012, 53: 1296–1303. DOI:10.1194/jlr.M024463 |
| [15] | Huang H, Svoboda RA, Lazenby AJ, et al. Up-regulation of N-cadherin by collagen I activated discoidin domain receptor 1 in pancreatic cancer requires the adaptor molecule SHC1[J]. J Biol Chem, 2016, 291: 23208–23223. DOI:10.1074/jbc.M116.740605 |
| [16] | Fulda S. Caspase 8 in cancer biology and therapy[J]. Cancer Lett, 2009, 281: 128–133. DOI:10.1016/j.canlet.2008.11.023 |
| [17] | Tummers B, Green DR. Caspase-8:regulating life and death[J]. Immunol Rev, 2017, 277: 76–89. DOI:10.1111/imr.2017.277.issue-1 |
| [18] | Dent S, Oyan B, Honig A, et al. HER2-targeted therapy in breast cancer:a systematic review of neoadjuvant trials[J]. Cancer Treat Rev, 2013, 39: 622–631. DOI:10.1016/j.ctrv.2013.01.002 |
| [19] | Roskoski R. The ErbB/HER family of protein-tyrosine kinases and cancer[J]. Pharmacol Res, 2014, 79: 34–74. DOI:10.1016/j.phrs.2013.11.002 |
| [20] | Arteaga CL, Engelman JA. ERBB receptors:from oncogene discovery to basic science to mechanism-based cancer thera-peutics[J]. Cancer Cell, 2014, 25: 282–303. DOI:10.1016/j.ccr.2014.02.025 |
| [21] | Chang L, Li CH, Gao J. Progress in the study of HER2-targeted cancer therapeutic antibodies[J]. Acta Pharm Sin (药学学报), 2015, 50: 516–520. |
| [22] | Yang ZY, Guo L, Liu D, et al. Acquisition of resistance to trastuzumab in gastric cancer cells is associated with activation of IL-6/STAT3/Jagged-1/Notch positive feedback loop[J]. Oncotarget, 2015, 6: 5072–5087. |
| [23] | Piro G, Carbone C, Cataldo I, et al. An FGFR3 autocrine loop sustains acquired resistance to trastuzumab in gastric cancer patients[J]. Clin Cancer Res, 2016, 22: 6164–6175. DOI:10.1158/1078-0432.CCR-16-0178 |
| [24] | Liu J, Pan C, Guo L, et al. A new mechanism of trastuzumab resistance in gastric cancer:MACC1 promotes the Warburg effect via activation of the PI3K/AKT signaling pathway[J]. J Hematol Oncol, 2016, 9: 76. DOI:10.1186/s13045-016-0302-1 |
| [25] | Zhao C, Li H, Lin HJ, et al. Feedback activation of STAT3 as a cancer drug-resistance mechanism[J]. Trends Pharmacol Sci, 2016, 37: 47–61. DOI:10.1016/j.tips.2015.10.001 |
| [26] | Shi M, Yang ZY, Hu M, et al. Catecholamine-induced β2-adrenergic receptor activation mediates desensitization of gastric cancer cells to trastuzumab by upregulating MUC4 expression[J]. J Immunol, 2013, 190: 5600–5608. DOI:10.4049/jimmunol.1202364 |
| [27] | Liu WH, Chang JX, Liu MW, et al. Quantitative proteomics profiling reveals activation of mTOR pathway in trastuzumab resistance[J]. Oncotarget, 2017, 8: 45793–45806. |
| [28] | Zuo Q, Liu J, Zhang J, et al. Development of trastuzumab-resistant human gastric carcinoma cell lines and mechanisms of drug resistance[J]. Sci Rep, 2015, 5: 11634. DOI:10.1038/srep11634 |
| [29] | Zhao YP, Shen SJ, Guo JC, et al. Mitogen-activated protein kinases and chemoresistance in pancreatic cancer cells[J]. J Surg Res, 2006, 136: 325–335. DOI:10.1016/j.jss.2006.06.031 |
| [30] | Haagenson KK, Wu GS. The role of MAP kinases and MAP kinase phosphatase-1 in resistance to breast cancer treatment[J]. Cancer Metast Rev, 2010, 29: 143–149. DOI:10.1007/s10555-010-9208-5 |
| [31] | Mccubrey JA, Steelman LS, Chappell WH, et al. Roles of the Raf/MEK/ERK pathway in cell growth, malignant trans-formation and drug resistance[J]. Biochim Biophys Acta, 2007, 1773: 1263–1284. DOI:10.1016/j.bbamcr.2006.10.001 |
| [32] | Housman G, Byler S, Heerboth S, et al. Drug resistance in cancer:an overview[J]. Cancers, 2014, 6: 1769–1792. DOI:10.3390/cancers6031769 |
| [33] | Ochi N, Takigawa N, Harada D, et al. Src mediates ERK reactivation in gefitinib resistance in non-small cell lung cancer[J]. Exp Cell Res, 2014, 322: 168–177. DOI:10.1016/j.yexcr.2014.01.007 |
| [34] | Yang XL, Lin FJ, Guo YJ, et al. Gemcitabine resistance in breast cancer cells regulated by PI3K/AKT mediated cellular proliferation exerts negative feedback via the MER/MAPK and mTOR pathways[J]. Onco Targets Ther, 2014, 7: 1033–1042. |
 2018, Vol. 53
2018, Vol. 53


