中药黄芩为临床常用药材, 来源于唇形科多年生草本植物黄芩Scutellaria baicalensis Georgi的干燥根, 其主要活性成分为黄芩苷、黄芩素等黄酮类化合物。研究表明, 高温、光照、缺水等适度的环境胁迫均能促进黄芩活性成分的积累[1-4], 且与植物内源激
素赤霉素和生长素的调控密切相关。光照条件下, 悬浮细胞中黄芩苷的含量随生长素IAA (indole-3-acetic acid)含量的降低而降低; 水分胁迫下, 黄芩中黄芩素的含量随着生长素IAA含量的升高而升高, 且外源赤霉素(gibberellin A3, GA3)影响黄芩中内源生长素及活性成分的含量[5-7]。
转录因子在调控苯丙烷类次生代谢产物积累与植物抗逆反应中发挥重要作用, 然而苯丙烷类化合物基因调控网络中转录因子之间的相互作用和协同作用却鲜见报道。前期研究发现, 黄芩中MYB蛋白可以激活类黄酮合成途径下游关键酶基因的表达, 多种转录因子(eg: ARF bHLH等)参与调控过程。ARF (auxin response factor)是一类较新发现的转录因子家族, 对生长素响应基因的表达起到激活或抑制作用, 广泛参与调控生长素及其他植物激素信号转导途径[8]。同时, ARF转录因子参与植物多途径信号的协同作用, 如番茄中的SlARF7基因是生长素和赤霉素信号途径的关键结合位点[9], 拟南芥中的MYB77转录因子通过与ARF7协同作用, 控制环境变化对于侧根生长发育的影响[10]。
近年来, 对于ARF转录因子的功能研究逐渐增多, 主要集中在拟南芥、烟草、水稻、丹参等植物中[11-13], 黄芩中ARF转录因子的功能及理化性质却未见报道。本文通过黄芩ARF转录因子的生物信息学分析, 同时比较ARF与黄芩次生代谢产物合成相关基因的表达, 为进一步研究黄芩中ARF转录因子的生物学功能奠定基础, 为完善黄芩中黄酮类成分的分子调控网络提供依据。
材料与方法 材料黄芩种子为中国中医科学院中药资源中心实验室保存, 将黄芩种子于湿润的滤纸上萌发, 萌发苗两周后移栽至花盆中, 25 ℃、16 h光照室温条件下培养。以100 μmol·L-1 GA3喷施黄芩叶片, 并分别于喷施后的0、1、2、3 h取样, -80 ℃冰箱保存备用。夏季黄芩花期时, 于同一植株不同部位分别取花、根、茎、叶, 清水清洗后以滤纸除去多余水分, -80 ℃冰箱保存备用。
黄芩ARF基因序列获取从TAIR数据库中下载拟南芥ARF核酸序列, 以此作为探针, 通过BLAST比对, 在黄芩转录组数据库中搜寻同源序列, 识别标准为e≤10-15, Score≥100, 匹配数b = 1。
生物信息学分析通过ORF Finder在线工具(http://www.ncbi.nlm.nih.gov/projects/gorf/)预测ARF基因序列的开放阅读框序列。运用在线软件Protparam (expasy.ch/tools/protparam.html)预测ARF编码蛋白的理化性质; 采用ExPASyPROSITE (http://www.expasy.ch/prosite)和CDD (http://www.ncbi.mlm.nih.gov/Structure/cdd/wrpsb.cgi)进行蛋白质结构域分析; 使用WOLF PSORT (http://wolfpsort.org/)进行蛋白质亚细胞定位信号的预测; 使用SignalP 4.1 Server (http:www.cbs.dtu.dk/services/SignalP)进行分泌蛋白的预测; 使用CFSSP (http://swissmodel.expasy.org/)进行蛋白质二级结构的分析; 使用MEGA 5.0构建系统进化树(Neighbor-joining tree, NJ tree) (bootstrap值设为1 000)。
RNA提取和cDNA合成样品总RNA采用植物总RNA提取试剂盒(TIANGEN公司)提取, 经1%琼脂糖凝胶电泳检测确定RNA完整性后, 用核酸定量仪测定RNA浓度, 按照cDNA合成试剂盒(TaKaRa公司)说明书进行反转录。
基因克隆在黄芩ARF基因序列的基础上设计PCR引物, 由生工生物工程(北京)有限公司合成, 见表 1。反应体系按照Trans Start® Fast Pfu DNA Polymerase高保真酶(全式金公司)说明书配制, 50 μL反应体系, 包括5×Trans Start® Fast Pfu Buffer 10 μL、dNTP 4 μL、正反向引物各1 μL、Trans Start® Fast Pfu DNA Polymerase高保真酶1 μL、cDNA模板1μL和ddH2O 32 μL。反应程序为95 ℃预变性1 min; 95 ℃变性20 s, 55 ℃退火20 s, 72 ℃延伸1 min, 40个循环; 72 ℃复性5 min。
| Table 1 Sequences of primers used inARF cloning |
依照黄芩ARF序列设计Real-time荧光定量PCR引物, 扩增产物长度在100~250 bp之间, 由生工生物工程(北京)有限公司合成, 见表 2。反应体系按照SYBR Premix Ex TaqTM Kit (TaKaRa公司)说明书配制, 10 μL反应体系, 包括正反向引物各0.5 μL、SYBR Premix Ex Taq 5μL、cDNA模板1 μL、ddH2O 3 μL。每个反应重复3次(包括阴性对照), 在Light Cycler 480 Ⅱ型实时荧光定量PCR仪(Roche公司)上进行, 结束后进行熔解曲线分析, 扩增程序如下: 94 ℃预变性2 min; 94 ℃变性5 s, 60 ℃退火延伸34 s (每次循环后采集荧光信号), 40个循环; 94 ℃变性10 s, 65~95 ℃做熔解曲线分析, 每个温度以每步0.5 ℃上升, 停留5 s, 获得每个基因的Ct值进行相对定量分析。
| Table 2 Real-time PCR primer sequences |
100 μmol·L-1 GA3处理下黄芩基因表达量的计算是以0 h作为参比基准, 计算不同基因在GA3处理下相对表达量的变化; 不同部位黄芩基因表达量的计算是以茎作为基准参比部位。各基因表达量以内参基因Sb18S作为标准进行相对定量[14], 目的基因表达量采用2-ΔΔCt法计算, 计算公式为2-ΔΔCt = 2-[(Ct目的基因-Ct管家基因)待测组-(Ct目的基因-Ct管家基因)对照组]。
基因表达相关性分析利用SPSS 23.0软件对黄芩ARF基因及与黄芩黄酮类合成相关基因的表达进行Pearson相关性分析, |r|>0.7说明两者呈现高度线性相关, 0.7≥|r|≥0.5说明两者呈现中度线性相关, 0.5≥|r|≥0.3则两者呈现低度相关, |r|<0.3两者相关性极低, P≤0.05为显著水平, P≤0.01为极显著水平[15]。
结果 1 黄芩ARF转录因子特性分析通过BLAST比对并对所得基因片段进行拼接, 从黄芩转录组中共得到24个ARF基因序列。ORF Finder预测表明, ARF1、ARF3、ARF4、ARF6、ARF8、ARF12、ARF18、ARF20、ARF24为全长cDNA, 其余基因读码框均不完整, 对全长基因进行克隆并测序验证, 产物电泳图如图 1所示。使用ProtParam预测具有全长cDNA的9个黄芩ARF转录因子的理化性质, 结果如表 3所示。使用CDD数据库分析结构域, 结果表明ARF1、ARF3、ARF4、ARF6、ARF8、ARF12、ARF18、ARF20、ARF24均具有ARF家族所特有的Aux-IAA结构域。

|
Figure 1 Electrophoresis chart of S. baicalensis ARF PCR products. M: DL2000 DNA marker; 1-9: PCR products of ARF1, ARF3, ARF4, ARF6, ARF8, ARF12, ARF18, ARF20 and ARF24 |
| Table 3 The characteristics of S. baicalensis ARFs |
使用WOLF PSORT对黄芩含有全长cDNA的9个转录因子进行蛋白质亚细胞定位的信号预测, 结果表明, ARF4、ARF8、ARF18、ARF24被定位至细胞核中, ARF1定位于线粒体和细胞核中, ARF3定位于细胞质、叶绿体及细胞核中, ARF6定位于线粒体、细胞质及细胞核中, ARF12定位于叶绿体和线粒体中, ARF20定位于细胞核及细胞质中。SignalP 4.1 Server软件分析表明, 黄芩含有全长cDNA的9个ARF转录因子均为非分泌蛋白。
2 黄芩ARF转录因子二级结构分析使用CFSSP软件对黄芩中含有全长cDNA的9个黄芩ARF转录因子进行二级结构预测, α-螺旋(H)、β-折叠(E)、β-转角(T)所占比例如图 2所示。对于大部分黄芩ARF转录因子而言, α-螺旋在二级结构中所占比例最高, β-转角所占比例相对较低, 但ARF20中β-折叠在二级解构中所占比例最高, 同时α-螺旋也占有很高比例, β-转角所占比例仍旧较低。
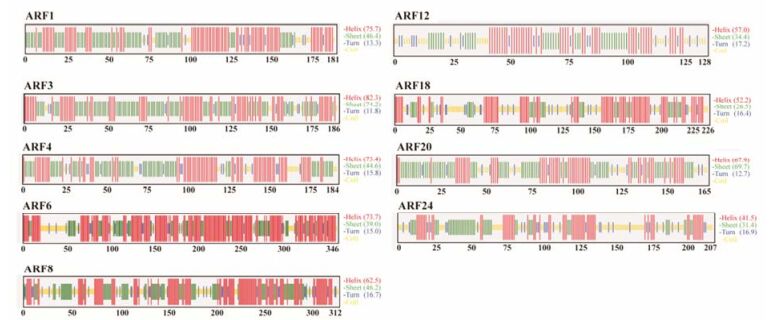
|
Figure 2 Secondary structure predictions of ARF TFs |
选取黄芩中具全长cDNA的9个ARF转录因子氨基酸序列与拟南芥、烟草、水稻和葡萄中的ARF转录因子序列, 使用MEGA5.0 Neighbor-joining法构建黄芩ARF转录因子系统进化树, 见图 3。系统进化树结果表明, 黄芩中的ARF8、ARF12与拟南芥中的At5g20730、At1g19220及烟草中的A4A49_25919、水稻中的OsARF1、OsARF16等聚为一支, 可能作为转录激活因子参与调控生长素应答基因的表达及促进根系发育[16]; 黄芩中的ARF20与烟草中的A4A49_ 40149聚为一支, 可能与生长素信号通路的激活有关[17]; 黄芩中的ARF1、ARF3、ARF4、ARF6、ARF18、ARF24与拟南芥中的At2g18390、At2g47170聚为一支, 可能与植物种子的发育与蛋白质的运输有关[18, 19]。

|
Figure 3 Analysis of the phylogenetic relationships of ARF gene members in S. baicalensis, Arabidopsisthaliana, Oryza sativa subsp. Japonica, Vitis vinifera and Nicotiana attenuate. Reddot: S. baicalensis; At: A. thaliana. GenBank Number of AtARFs: At5g20730-NP_568400.2, At1g19220-NP_173356, At1g59750-NP_849830, At5g62000-NM_125438, At2g18390-NP_179430, At2g47170-NP_001324310.1; A4A49: N. attenuate. GenBank Number of NaARFs: A4A49_25919-OIS99983.1, A4A49_26861-OIS98057.1, A4A49_40149-OIT19819.1; VITISV: V vinifera. GenBank Number of VvARFs: VITISV_024151-CAN63853.1, VITISV_030510-CAN80533.1, VITISV_000577-CAN70219.1; Os: O. sativa. GenBank Number of OsARFs: OsARF1-XP_015617187.1, OsARF10-XP_015644138.1, OsARF16-XP_015622435.1 |
为了进一步了解黄芩ARF基因的表达模式和表达水平, 通过Realtime-PCR分别分析了含全长cDNA的9个黄芩ARF基因以及与黄芩黄酮类合成相关的CHS、PAL1、PAL2、PAL3、MYB2和MYB8基因在100 μmol·L-1 GA3处理下0、1、2和3 h的表达量, 以及比较其在根、茎、叶和花4个部位之间表达量的差异。
100 μmol·L-1 GA3处理下, ARF1、ARF3、ARF4、ARF12和PAL2基因表达量在处理后1 h显著升高, ARF6、ARF18和CHS在处理后表达量首先下降, 3 h后又逐渐升高, ARF8和ARF20处理后1 h表达量升高, 随后又逐渐降低, 3 h后表达量再次升高, 见图 4。经简单相关及偏相关分析后发现100 μmol·L-1 GA3处理下, ARF3、ARF12和PAL2相对表达水平呈显著正相关(r = 0.900 6、0.910 8); ARF20与PAL1相对表达水平显著负相关(r = -0.853 6)。
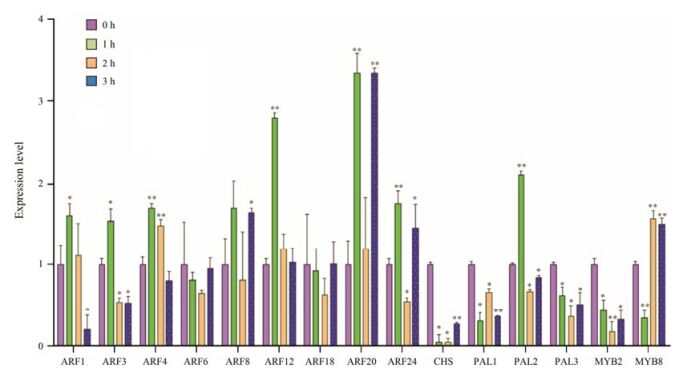
|
Figure 4 Effect of GA3 treatment on ARF and flavonoids biosynthetic related gene expression in S. baicalensis (n = 3). Expression in 0 h after treatment was set as 1 (*P ≤ 0.05, **P ≤ 0.01) |
黄芩中含完整读码框的9个ARF转录因子在花、茎、叶和根4个部位中的表达量差异明显, 由图 5可见, 黄芩大部分相关基因在花中均有最高的表达量, 高于茎和叶中的表达量。经简单相关及偏相关分析后发现ARF1、ARF3、ARF4、ARF6、ARF12、ARF18和ARF24均与PAL1在黄芩不同部位中相对表达水平显著正相关(r = 0.929 1、0.817 5、0.998 7、0.983 0、0.936 2、0.849 8、0.978 9)。
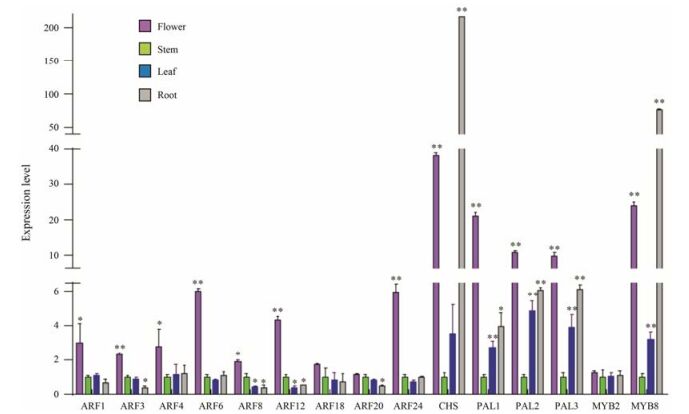
|
Figure 5 The expression levels of ARF and flavonoid biosynthetic related gene expression in S. baicalensis from different organs (n = 3). Expression in the stem was set as 1 (*P ≤ 0.05, **P ≤ 0.01) |
ARF转录因子是一类较新发现的转录因子家族, 广泛参与植物体内生长素等激素信号传导途径。本项目组前期研究证明, 温度、光照、缺水等适度环境胁迫能够增加黄芩有效成分的积累, 且黄芩活性成分的积累与PAL、CHS等关键酶基因有关。本文基于已获得的黄芩cDNA文库序列, 用生物信息学的方法结合基因表达分析, 研究黄芩中ARF转录因子的功能及与类黄酮物质合成相关基因之间的关系。本文共获得了9个含全长cDNA的ARF基因, 分别命名为ARF1、ARF3、ARF4、ARF6、ARF8、ARF12、ARF18、ARF20和ARF24。基因表达分析结果表明, 黄芩中ARF基因与黄酮次生代谢相关基因PAL关系密切, 苯丙氨酸解氨酶(PAL)能够催化苯丙烷类代谢第1步反应, 是黄芩苷合成途径中的关键酶[20]。GA3处理条件下, 黄芩中ARF3、ARF12基因与PAL2基因在表达水平上显示出较强的协同性, 据此推测黄芩中ARF基因可能参与调控黄酮类化合物的次生代谢。系统进化树结果显示, 黄芩中的ARF12与拟南芥中的At5g20730、At1g19220等聚为一支, At5g20730、At1g19220在拟南芥中参与激活生长素应答反应与促进根系发育[21], 且生长素在黄芩中参与调控黄芩素和黄芩苷的积累, 由此推断ARF12可能参与调控黄芩中的生长素信号通路, 进而影响黄芩中活性成分的积累。本文研究结果为进一步了解转录因子调控黄芩活性成分生物合成分子机制奠定基础, 并为完善黄芩中黄酮类活性成分分子调控网络提供了依据。
| [1] | Yuan Y, Hao JD, Yang B, et al. Climate change affected the best producing area of Chinese herbal medicine Scutellaria baicalensis Georgi[J]. J Tradit Med (Russia), 2010, 3s: 241. |
| [2] | Yuan Y, Qi L, Yang J, et al. A Scutellaria baicalensis R2R3-MYB gene, SbMYB8, regulates flavonoid biosynthesis and improves drought stress tolerance in transgenic tobacco[J]. Plant Cell Tiss Org, 2015, 120: 961–969. DOI:10.1007/s11240-014-0650-x |
| [3] | Yuan Y, Wu C, Liu Y, et al. The Scutellaria baicalensis R2R3-MYB transcription factors modulates flavonoid biosynthesis by regulating GA metabolism in transgenic tobacco plants[J]. PLoS One, 2013, 8: e77275. DOI:10.1371/journal.pone.0077275 |
| [4] | Yuan Y, Zhou JH, Huang LQ. Experimental verification and prospect on stress effect of Dao-di herbs Scutellaria baicalensis[J]. China J Chin Mater Med (中国中药杂志), 2016, 41: 139–143. |
| [5] | Chen SQ, Huang LQ, Yuan Y, et al. Effects of light on the correlation between endogenous hormones and active ingredients in suspension cells of Scutellaria baicalensis Georgi[J]. Chin J Exp Tradit Med Form (中国实验方剂学杂志), 2010, 16: 72–74. |
| [6] | Qin SS, Chen SQ, Huang LQ, et al. Effects of water stress on the correlation between endogenous hormones and active ingredients in Scutellaria baicalensis Georgi[J]. Chin J Exp Tradit Med Form (中国实验方剂学杂志), 2010, 16: 99–101. |
| [7] | Yuan Y, Liu Y, Wu C, et al. Water deficit affected flavonoid accumulation by regulating hormone metabolism in Scutellaria baicalensis Georgi roots[J]. PLoS One, 2012, 7: e42946. DOI:10.1371/journal.pone.0042946 |
| [8] | Wei H, Cui B, Ren Y, et al. Research progresses on auxin response factors[J]. J Integr Plant Biol, 2006, 48: 622–627. DOI:10.1111/jipb.2006.48.issue-6 |
| [9] | Li J, Dai X, Zhao Y. A role for auxin response factor 19 in auxin and ethylene signaling in Arabidopsis[J]. Plant Physiol, 2006, 140: 899. DOI:10.1104/pp.105.070987 |
| [10] | Shin R, Burch AY, Huppert KA, et al. The Arabidopsis transcription factor MYB77 modulates auxin signal transduction[J]. Plant Cell, 2007, 19: 2440–2453. DOI:10.1105/tpc.107.050963 |
| [11] | Li SB, Xie ZZ, Hu CG, et al. A review of auxin response factors (ARFs) in plants[J]. Front Plant Sci, 2016, 7: 47. |
| [12] | Hagen G, Guilfoyle T. Auxin-responsive gene expression:genes, promoters and regulatory factors[J]. Plant Mol Biol, 2002, 49: 373–385. DOI:10.1023/A:1015207114117 |
| [13] | Xu Z, Ji A, Song J, et al. Genome-wide analysis of auxin response factor gene family members in medicinal model plant Salvia miltiorrhiza[J]. Biol Open, 2016, 5: 848–857. DOI:10.1242/bio.017178 |
| [14] | Hui X, Park NI, Li X, et al. Molecular cloning and characterization of phenylalanine ammonia-lyase, cinnamate 4-hydroxylase and genes involved in flavone biosynthesis in Scutellaria baicalensis[J]. Bioresour Technol, 2010, 101: 9715–9722. DOI:10.1016/j.biortech.2010.07.083 |
| [15] | Hazra A, Gogtay N. Biostatistics series module 6:correlation and linear regression[J]. Indian J Dermatol, 2016, 61: 593. DOI:10.4103/0019-5154.193662 |
| [16] | Ito J, Fukaki H, Onoda M, et al. Auxin-dependent compositional change in mediator in ARF7-and ARF19-mediated transcription[J]. Proc Natl Acad Sci U S A, 2016, 113: 6562–6567. DOI:10.1073/pnas.1600739113 |
| [17] | Lee MH, Sano H. Attenuation of the hypersensitive response by an ATPase associated with various cellular activities (AAA) protein through suppression of a small GTPase, ADP ribosylation factor, in tobacco plants[J]. Plant J, 2007, 51: 127–139. DOI:10.1111/j.1365-313X.2007.03124.x |
| [18] | Okushima Y, Overvoorde PJ, Arima K, et al. Functional genomic analysis of the AUXIN RESPONSE FACTOR gene family members in Arabidopsis thaliana:unique and overlapping functions of ARF7 and ARF19[J]. Plant Cell, 2005, 17: 444–463. DOI:10.1105/tpc.104.028316 |
| [19] | Mcelver J, Meinke D. The TITAN5 gene of Arabidopsis encodes a protein related to the ADP ribosylation factor family of GTP binding proteins[J]. Plant Cell, 2000, 12: 1379–1392. DOI:10.1105/tpc.12.8.1379 |
| [20] | Wang W, Chen JK, Zhou TS. CHS and UBGAT expression and baicalin accumulation in the roots of Scutellaria baicalensis Georgi during cultivation seasons[J]. J Fudan Univ (Nat Sci) (复旦学报自然科学版), 2006, 45: 674–678. |
| [21] | Lee MH, Min MK, Lee YJ, et al. ADP-ribosylation factor 1 of Arabidopsis plays a critical role in intracellular trafficking and maintenance of endoplasmic reticulum morphology in Arabidopsis[J]. Plant Physiol, 2002, 129: 1507–1520. DOI:10.1104/pp.003624 |
 2017, Vol. 52
2017, Vol. 52


