2. 呼吸疾病诊疗与新药研发河南省协同创新中心, 河南 郑州 450046
2. Collaborative Innovation Center for Respiratory Disease Diagnosis and Treatment and Chinese Medicine Development of Henan Province, Zhengzhou 450046, China
垂序商陆(Phytolacca americana L.)为商陆科(Phytolaccaceae)多年生草本植物, 干燥根及其炮制品分别为生商陆和醋商陆入药, 是我国传统中药, 被2015年版《中国药典》收录为商陆药材来源[1]。商陆有逐水消肿、通利二便的功效; 用于治疗水肿胀满、二便不通, 外治痈肿疮毒等[1], 具有重要的药用价值。目前已从商陆中分离得到商陆皂苷类、黄酮类、酚酸类、甾醇类以及多糖类等多种化学成分, 其中商陆皂苷是商陆的特征性化学成分, 是其主要的药效物质基础, 具有利尿、免疫抑制、抗炎等显著的生理活性, 已成为研究热点[2]。至今已从商陆中分离得到33种商陆皂苷, 且均为齐墩果烷型[2], 由于商陆皂苷甲(esculentoside A, EsA)在商陆皂苷中含量较高, 2015年版《中国药典》收录的商陆药材项将其列为对照品[1], 通过测定EsA在商陆药材中的含量对商陆质量进行控制, 所以商陆皂苷甲(EsA)是商陆皂苷中的主要成分。现代药理学研究发现商陆具有利尿、抗菌、抗病毒、抗炎、抗肿瘤等活性; 临床上多用其治疗乙型肝炎、银屑病、过敏性紫癜等疑难疾病[2]。对于商陆的抗炎作用, 主要集中在抗炎活性极强的EsA上, 而且EsA也具有显著的免疫抑制活性。Zhang等[3]发现EsA能够减弱CCl4和GalN/LPS诱导的急性肝损伤, 而且这种保护机制与EsA的抗炎、抗氧化胁迫作用有关, 可能是通过抑制细胞产生TNF和IL-1来实现的。
作为商陆主要药效物质基础的EsA, 从其化学结构分析属于三萜皂苷(齐墩果烷型五环三萜), 关于三萜皂苷生物合成途径研究较为深入是人参皂苷, 目前已克隆了近50个与人参皂苷生物合成相关的基因并对其中20个基因进行了功能验证[4,5]。虽然商陆在化学成分、药理作用等方面已有较为深入的研究, 但关于EsA生物合成途径的报道较少, 不仅上游萜类骨架合成的相关酶类未见报道, 而且参与商陆皂苷元母核合成后修饰的酶类, 如细胞色素P450 (cytochrome P450, CYP450) 和糖基转移酶(uridine diphosphate glycosyltransferase, UGT)等的基因克隆和功能分析也未见报道。高通量测序技术具有测序通量大、价格低、时间短等优势, 极大降低了测序所需成本和时间, 广泛应用于药用植物次生代谢途径功能基因的挖掘方面[6]。目前在人参[7]、丹参[8]、三七[9]、金银花[10]等药用植物中已经利用高通量测序技术进行转录组研究, 获得了一批与药用植物药效成分生物合成及调控相关的基因[6]。本研究利用高通量测序技术对商陆幼苗进行转录组测序, 以期获得一批与EsA生物合成相关的基因, 为初步阐明EsA生物合成途径奠定基础, 也为今后利用生物技术对商陆进行遗传改良提供候选基因。
材料与方法材料 商陆种子采自河南省伏牛山区, 经过浓硫酸(98%)和酒精(70%)表面消毒, 各15 min, 再用无菌水漂洗3次, 将种子种到1/2 MS培养基上, 在人工智能培养箱中生长。培养条件为16 h、23 ℃光照, 8 h、20 ℃黑暗, 光照强度150 μE·m-2·s-1, 约3周后, 种子萌发长成幼苗, 经河南中医药大学董诚明教授鉴定为商陆科植物垂序商陆(Phytolacca americana L.), 在无菌条件下取商陆幼苗样品, 放入液氮中速冻。
商陆皂苷甲含量测定 根据药典中所收录的HPLC-ELSD含量测定方法, 稍作调整, 对无菌的商陆幼苗根、茎、叶中商陆皂苷甲进行含量测定[1]。仪器与试药: Waters e2695 Separations Module, Waters 2424 ELS Detector, Waters Empower高效色谱工作站, MILLIPORE超纯水发生器; 商陆皂苷甲(国家标准物质, 供含量测定用, 批号: 111922-201102, 纯度为92.2%)购自中国食品药品检定研究院。取商陆幼苗根、茎、叶冷冻干燥2天后, 称重, 分别打粉或研磨粉碎, 过40目筛, 备用。色谱条件及检测条件:色谱柱为ODS (Inertsil ODS-SP, 5 μm, 4.6 mm × 250 mm, GL. Sciences Inc.); 流动相:甲醇-水(65:35);流速: 1 mL·min-1; 柱温: 25 ℃; ELSD条件:漂移管温度74 ℃, 载气流速: 2.98 L·min-1。该条件下, 信噪比最佳。商陆皂苷甲色谱峰理论塔板数不低于2 000。标准品溶液制备:精密称取商陆皂苷甲5.0 mg, 置于10 mL容量瓶中, 加入6 mL甲醇并超声溶解10 min, 定容至10 mL, 制成浓度为0.5 mg·mL-1的对照品溶液。从0.5 mg·mL-1的对照品溶液中精确吸取1 mL至5 mL容量瓶中, 加入甲醇定容至5 mL, 以0.22 μm微孔滤膜过滤两次, 制成浓度为0.1 mg·mL-1的对照品溶液。供试品溶液制备:精密称量商陆根粉末0.5 g, 商陆茎粉末0.5 g, 商陆叶粉末0.5 g, 分别放入3个10 mL容量瓶中, 6 mL 70%甲醇溶解, 超声提取30 min, 定容至10 mL, 以0.22 μm微孔滤膜过滤两次, HPLC用。线性关系考察与样品测定:设定0.1 mg·mL-1的对照品溶液5、10、15、20 μL, 0.5 mg·mL-1的对照品溶液10、20 μL自动进样, 按照上述色谱条件, 用液相色谱仪进行测定, 所得峰面积与浓度进行线性回归。回归方程为y = 364 668x -262 439, R = 0.997 6。在上述色谱条件下, 设定供试品20 μL自动进样, 按外标峰面积法计算样品中商陆皂苷甲的含量。
RNA提取与检测 从商陆幼苗根、茎、叶不同组织混合样品中提取总RNA, 分别用Nanodrop和Aglient 2100检测RNA样品的浓度、纯度和完整性等, 保证RNA质量满足建库要求进行转录组测序。
RNA文库构建及文库质控 样品检测合格后, 进行文库构建, ① 用带有Oligo(dT)的磁珠富集商陆幼苗mRNA; ② 加入fragmentation buffer将mRNA打断成短片段; ③ 以mRNA为模板, 用六碱基随机引物(random hexamers)合成一链cDNA, 然后加入缓冲液、dNTPs、RNase H和DNA polymerase Ⅰ合成第二条cDNA链, 利用AMPure XP beads纯化cDNA; ④ 纯化的双链cDNA先进行末端修复、加A尾并连接测序接头, 再用AMPure XP beads进行片段大小选择; ⑤ 最后通过PCR富集得到cDNA文库。文库构建完成后, 先使用Qubit2.0进行初步定量, 随后使用Agilent 2 100对文库的插入片段大小进行检测, 以保证文库质量。
转录组测序 文库检测合格后, 用Illumina HiSeq 4000进行高通量测序, 利用双末端测序(Paired-End)法, 测序读长为PE150。测序得到的原始图像数据文件经碱基识别转化为原始数据(raw data), 对原始数据进行数据过滤, 去除接头、重复序列、低质量的序列, 获得高质量的clean data。使用Trinity软件[11]对clean data进行拼接, 通过序列之间的overlap拼接得到重叠群(contigs), 然后进一步组装得到转录本(transcripts), 最后获得单基因簇(unigenes)。
Unigene功能注释 使用BLAST软件将unigene序列与Nr (Non-redundant Protein Sequence Database in GenBank)、Swiss-Prot (Swiss-Prot Protein Sequence Database)、GO (Gene Ontology)、COG (Clusters of Orthologous Groups)、Pfam (Protein family)、KOG (euKaryotic Orthologous Groups)、KEGG (Kyoto Encyclopedia of Genes and Genomes)数据库比对, 获得unigene的注释信息, GO功能注释基于Nr和Pfam两部分的蛋白注释结果, 在Blast2GO软件上进行分析, 通过上述数据库对商陆的转录组数据进行功能基因注释, 找到参与EsA生物合成途径的关键基因。使用MEGA5软件相邻连接法(neighbor-joining)构建系统进化树, bootstrap检验的重复次数为1 000次
商陆转录组SSR分析 利用MISA软件对筛选得到的1 kb以上的unigenes做SSR位点分析, 筛选的标准为:单核苷酸重复≥10 bp、二核苷酸重复≥ 12 bp、三核苷酸重复≥15 bp、四核苷酸重复≥20 bp、五核苷酸重复≥25 bp、六核苷酸重复≥30 bp, 同时单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸最少重复次数分别为10、6、5、5、4、4, 复合型SSR至少包含2个SSR位点, 且2个位点之间距离小于100 bp, 对获得的SSR种类、数量等基本信息进行统计分析。
结果与分析 1 商陆皂苷甲含量测定取商陆幼苗根、茎、叶的样品, 按“材料与方法”项下的操作要求制备供试样品溶液, 每个样品平行制备3份溶液, 按“材料与方法”中的色谱条件, 供试样品20 μL自动进样, 按外标峰面积法计算样品中商陆皂苷甲的含量, HPLC色谱图见图 1。从图 1中可看出, 商陆幼苗不同组织部位中EsA的含量差异较大, 根中含量较高为0.77%, 茎中较低为0.37%, 而叶中未检出。
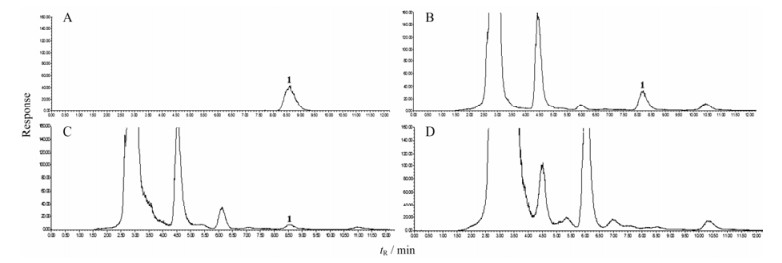
|
Figure 1 HPLC chromatograms of esculentoside A (EsA) standard and different tissues of P. americana. A: EsA standard; B: Roots; C: Stems; D: Leaves; 1: EsA |
商陆幼苗RNA样品经Nanodrop、Agilent 2100检测, OD260/OD280、OD260/OD230等各项指标均合格(表 1), 所提取的RNA质量较好, 符合转录组测序文库构建要求。
| Table 1 The quality of the RNA sample |
采用Illumina HiSeq 4000高通量测序平台对商陆转录组进行测序, 共获得32 470 764个reads片段, 包含了9 602 387 872 bp的序列信息, 经过测序质量控制, 得到9.60 Gb clean data, Q30碱基百分比到达90.12%, GC%含量平均值为46.28%。商陆转录组的原始数据(Raw data)已上传至NCBI的SRA数据库, 登录号为SRP105831。使用Trinity软件对商陆转录组进行组装, 共得到6 859 608条contigs, 其中长度200~300 bp的contigs序列有6 717 497条, 占总数的97.93%, 300~2 000 bp的占1.94%, 2 000 bp以上的占0.13% (表 2), 可见contigs序列的分布以200~300 bp为主, 其分布特征符合Illumina测序的预期结果, 可为后续数据组装提供良好的数据。在contigs数据的基础上, 再进行组装获得128 964条Transcripts, 序列总长度为176 102 380 bp, 平均长度为1 365.52 bp, N50为2 097 bp (表 2)。对得到的Transcripts序列进一步组装获得63 957条unigenes, 序列总长度为63 241 729 bp, 平均长度为988.82 bp, N50为1 541 bp (表 2)。
| Table 2 Summary of tanscriptome data assembly from P. americana |
通过选择BLAST参数E-value不大于1×10-5和HMMER参数E-value不大于1×10-10, 最终获得24 517个(38.33%)有注释信息的unigenes (表 3), 其中能被Nr数据库注释的有24 176个(37.80%); 能被Swiss-Prot数据库注释的有15 161个(23.70%); 被Pfam数据库注释的有16 428个(25.69%); 能被KOG数据库注释的有13 755个(21.51%); 能被KEGG数据库注释的有8 252个(12.90%); 能被GO数据库注释的有13 172个(20.60%); 能被COG数据库注释的有6 945个(10.86%); 有39 440个unigenes (61.7%)不能被已有的数据库注释。
| Table 3 Summary statistics of unigenes functional annotation for P. americana tanscriptome |
Unigenes在Nr数据库相似序列匹配的近缘物种中, 甜菜(Beta vulgaris)所占比例最高(14 654条, 60.64%), 其次是葡萄(Vitis vinifera, 1 492条, 6.17%)、北美云杉(Picea sitchensis, 485条, 2.01%)、美花烟草(Nicotiana sylvestris, 341条, 1.41%)、莲(Nelumbo nucifera, 329条, 1.36%)、绒毛烟草(Nicotiana tomentosiformis, 290条, 1.20%)、可可(Theobroma cacao, 286条, 1.18%)、甜橙(Citrus sinensis, 223条, 0.92%)、桃(Prunus persica, 201条, 0.83%)、蓖麻(Ricinus communis, 200条, 0.83%)和其他物种(5 663条, 23.44%)。
5 GO分类GO (Gene Ontology)数据库是一个国际标准化的基因功能分类体系, 提供了一套动态更新的标准词汇表来全面描述生物体中基因和基因产物的功能属性, 为了对商陆的转录组数据进行功能分析, 将unigenes进行GO注释, 然后将注释成功的unigenes再进行GO分类。共有13 172个unigenes获得至少一个注释结果, 这些unigenes被分为细胞组份(cellular component, CC)、分子功能(molecular function, MF)、生物学过程(biological process, BP) 3个大类, 51个小类(图 2), 其中25 542个unigenes被注释为细胞组份, 15 767个unigenes被注释为分子功能; 36 872个unigenes被注释为生物学过程。
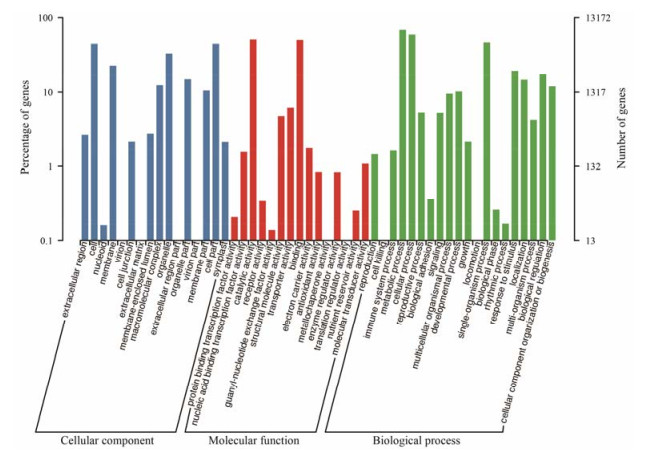
|
Figure 2 Gene ontology classification of unigenes |
COG (clusters of orthologous groups)数据库是对基因产物进行同源分类的数据库, 是一个较早的识别直系同源基因的数据库, 通过对多种生物的蛋白质序列大量比较而来。将商陆unigenes与COG数据库进行比对, 预测unigenes功能并进行分类统计。研究结果表明, 商陆unigenes根据其功能大致可分为25类。Unigenes涉及的COG功能类别比较全面, 其中, 一般功能预测类基因最多(1 806个); 其次是复制、重组和修复类基因(1 042个), 翻译、核糖体结构和生物合成类基因(878个), 转录类基因(871个), 信号转导类基因(771个), 翻译后修饰、分子伴侣类基因(706个); 而细胞运动类基因(3个)和细胞核结构类基因(1个)较少; 未发现胞外结构类基因。
7 KEGG分类为了分析商陆转录组数据中unigenes所参与的代谢路径, 将获得KO注释的unigenes进行KEGG代谢通路分析。共有8 252个unigenes归入127个代谢通路, 包含unigenes最多的代谢通路是核糖体途径(ko03010), 共有679个; 其次是氨基酸生物合成途径(ko01230), 共有285个。在商陆的转录组数据中有53个unigenes映射到萜类骨架合成通路上(ko00900), 有8个unigenes映射到倍半萜和三萜的生物合成通路上(ko00909), 共编码三萜类化合物合成途径中的19个关键酶(表 4), 包括羟甲基戊二酰辅酶A合成酶(HMGS)、羟甲基戊二酰辅酶A还原酶(HMGR)等6个甲羟戊酸途径(MVA途径)的酶; 1-脱氧-D-木酮糖-5-磷酸合成酶(DXS)、1-脱氧-D-木酮糖-5-磷酸还原异构酶(DXR)等6个甲基赤藓醇磷酸途径(MEP途径)的关键酶; 以及牻牛儿基焦磷酸合成酶(GPPS)、法尼基焦磷酸合酶(FPPS)、牻牛儿基牻牛儿基焦磷酸合酶(GGPPS)等3个催化萜类骨架直接前体生成的异戊烯基转移酶; 鲨烯合酶(SQS)、鲨烯环氧酶(SE)、β-香树脂合成酶(β-AS)等3个催化生成齐墩果烷型五环三萜前体的关键酶, 这19个关键酶参与EsA可能的生物合成途径如图 3所示。
| Table 4 Candidate genes involved in esculentoside A biosythesis pathway |

|
Figure 3 The possible biosynthesis pathway of esculentoside A (The dashed arrows indicate the supposed reaction, the functions of enzymes in dashed boxes are not yet elucidated) |
根据KEGG代谢通路分析结果, 有13条代谢通路中409条unigenes可能参与商陆其他次生代谢途径(图 4)。其中苯丙素类生物合成途径(ko00940) 所占比例最大, 达34.72%;其次是类黄酮生物合成(ko00941) 和N聚糖生物合成(ko00510) 分别占11.25%;泛醌和萜醌类物质生物合成(ko00130) 占7.82%;二萜生物合成(ko00904) 占6.60%;而单萜生物合成(ko00902) 和花青素生物合成(ko00942) 最少, 分别占0.98%和0.24%。商陆中多条次生代谢途径及相关基因的发现, 表明其次生代谢生物合成途径的复杂性, 以及商陆化学成分的多样性, 为后续从商陆中分离新化合物提供了线索, 也为阐明商陆功效物质基础提供了理论依据。
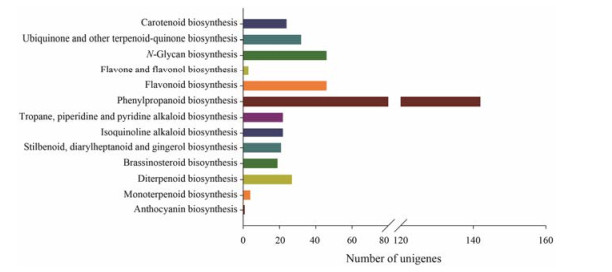
|
Figure 4 Unigenes related to other secondary metabolism from tanscriptome of P. americana |
根据推导的商陆MVA途径基因的氨基酸序列, 和来源于不同物种相同基因家族的氨基酸序列, 采用相邻连接法(neighbor-joining)构建系统进化树(图 5)。从系统进化树中可以看出, 商陆MVA途径的蛋白都归属被子植物的双子叶植物分支, 与甜菜(Beta vulgaris)的亲缘关系较近, 处于同一分支上。这一结果也与商陆转录组unigenes功能注释时, 在Nr数据库相似序列匹配的近缘物种中甜菜占的比例最高相一致。
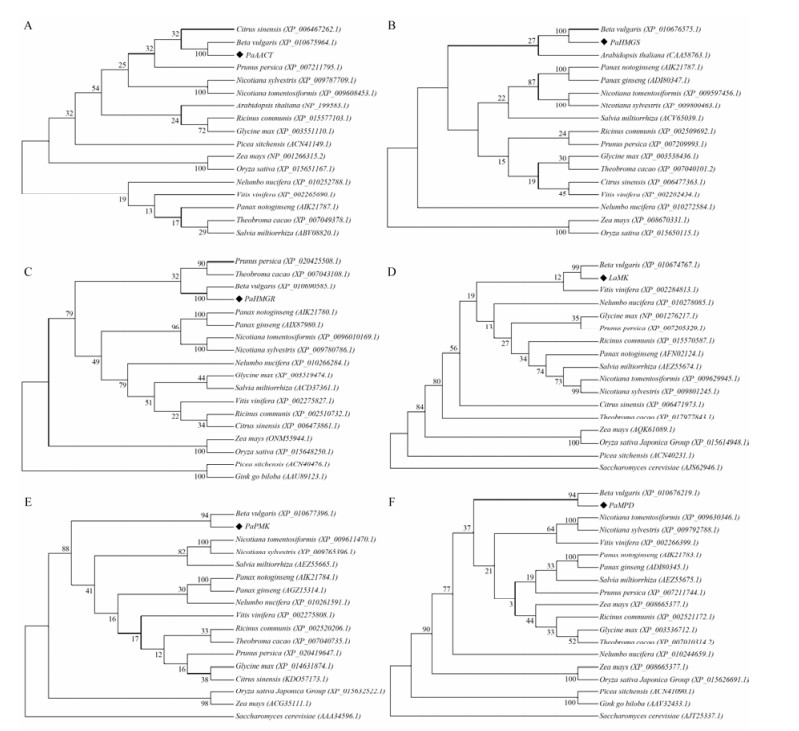
|
Figure 5 Phylogenetic trees analysis of MVA pathway enzymes. A: Phylogenetic tree of AACTs; B: Phylogenetic tree of HMGSs; C: Phylogenetic tree of HMGRs; D: Phylogenetic tree of MKs; E: Phylogenetic tree of PMKs; F: Phylogenetic tree of MPDs |
目前已从商陆中分离得到33种商陆皂苷, 都属于三萜皂苷(齐墩果烷型五环三萜)[2], 植物合成三萜化合物的骨架后, 都会在三萜骨架上进行后修饰, 如氧化、羟基化、糖基化、甲基化、乙酰化等多种反应, 形成多种三萜皂苷, 这些后修饰反应大幅增加了萜类化合物的种类及其结构的多样性[12]。
EsA生物合成途径中的后修饰反应, 推测主要包括骨架的氧化/羟基化和糖基化, 分别由不同超基因家族编码的CYP450和UGT进行催化。氧化是植物次生代谢产物后修饰中最常见的方式, 其中绝大部分依赖细胞色素P450 (CYP450) 的催化, 植物CYP450具有广泛的催化活性, 其作用特点是在底物分子中加入一个氧原子, 从而参与萜类、苯丙烷类、生物碱类等多种次生代谢产物的生物合成。在三萜皂苷的生物合成中, 细胞色素P450主要催化三萜骨架惰性甲基和亚甲基的氧化[13]。通过分析商陆转录组数据在Swiss-Prot数据库的注释结果, 共找到130条可能的CYP450基因, 隶属于24个CYP450家族(表 5)。属于CYP71家族的unigenes最多, 有17.69%;其次是CYP72、CYP86和CYP90, 分别为9.23%、8.46%和7.69%;而CYP73、CYP77、CYP83、CYP84、CYP85和CYP703家族成员最少, 仅各有1个unigene。
| Table 5 Summary of CYP450 genes in the transcriptome of P. americana |
糖基化是植物次生代谢过程中广泛的一种修饰反应, 尿苷二磷酸-糖基转移酶(UDP-glycosyl transferases, UGT)能够催化尿苷二磷酸上连接的活性糖转移到多种受体, 如可以转移到三萜皂苷的苷元上, 增加三萜的水溶性, 改善其化学稳定性和生物活性, 因此糖基化在植物三萜皂苷的生物合成中非常重要[14]。根据商陆转录组数据在Swiss-Prot数据库的注释结果, 共找到属于14个UGT亚家族的46个UGTs, 其中包括1个UGT73、2个UGT74、2个UGT75、6个UGT76、1个UGT79、6个UGT80、5个UGT85、6个UGT86、1个UGT87、8个UGT89、1个UGT90、1个UGT91、2个UGT92和4个UGT709。商陆转录组中大量CYP450和UGTs的发现为进一步分析具体CYP450和UGT的催化功能, 阐明商陆EsA生物合成途径的下游阶段后修饰的氧化和糖基化反应奠定基础。
10 SSR分析利用MISA软件对筛选得到的1 kb以上的unigenes做SSR分析, 共检出含7 924个SSR。如表 6所示, 商陆转录组SSR种类丰富, 单核苷酸重复至六核苷酸重复类型均存在, 但各类型出现的频率具有较大的差异。商陆转录组SSR种类中单核苷酸重复最多, 占SSR总数的50.92%, 其次是二核苷酸重复和三核苷酸重复, 分别占SSR总数的24.41%和19.13%。在检出SSR中, 共发现76种重复基元, 其中A/T在单核苷酸重复基元出现最多, 有3 638个, 占SSR总数的45.91%; AG/TC、CT/GA、AT/TA等3种类型在二核苷酸重复基元出现最多, 分别有775个(9.78%)、727个(9.17%)和303个(3.82%); 在三核苷酸重复基元中, CCA/GGT和CTT/GAA出现最多, 分别为111个(1.40%)和96个(1.21%)。对这些SSR的鉴定, 将为进一步筛选和开发商陆SSR标记奠定了基础, 对商陆遗传多样性分析、分子标记辅助育种等提供帮助。
| Table 6 Distribution of SSRs with the numbers of repeat motifs in transcriptome of P. americana |
商陆作为我国传统中药, EsA是商陆的主要药效成分, 关于其化学成分和药理作用方面的研究已有良好的基础, 但是商陆的基因组信息未知, 严重限制了EsA生物合成途径及相关功能基因的研究。Neller等[15]对茉莉酸(jasmonate acid, JA)处理前后商陆叶片的转录组变化进行研究, 发现差异表达基因多集中在逆境相关基因和抗病毒蛋白基因方面, 没有发现次生代谢途径相关的基因。本研究利用Illumina HiSeq 4000高通量测序技术对商陆幼苗进行转录组测序, 得到9.60 Gb的clean data, 组装后获得63 957条unigenes, 平均长度988.82 bp, N50为1 541 bp, 其中24 176条unigenes能被Nr数据库注释, 转录组测序数据饱和度检验显示, 检测到的基因数目趋于饱和。这些结果表明本次商陆转录数据组装效果较好, 获得了商陆大量的基因序列信息, 可满足后续数据分析的要求, Illumina HiSeq 4000高通量测序技术可作为批量挖掘EsA生物合成途径功能基因的有效工具。
由于在商陆幼苗的根、茎、叶中EsA含量差异较大, EsA主要存在于商陆的根中, 这与商陆以根作为入药部位一致。根据商陆转录组数据的KEGG分析结果结合其他植物中三萜皂苷的生物合成途径, EsA的生物合成途径大致可分为萜类前体物质的合成、三萜皂苷骨架的合成和后修饰3个阶段。上游阶段通过位于细胞质的MVA途径[16]和位于质体中的MEP途径[17], 合成萜类共同的前体物质异戊烯焦磷酸(IPP)和IPP的双键异构体二甲基烯丙基焦磷酸(DMAPP); 中游阶段通过SQS、SE、β-AS等关键酶催化生成β-香树脂(齐墩果烷型五环三萜); 下游阶段主要是经CYP450和UGT催化各种复杂的修饰反应生成不同类型的商陆皂苷, 但是下游阶段有哪些CYP450和UGT参与EsA的生物合成目前并不清楚。在商陆的转录组数据中有53个unigenes映射到萜类骨架合成通路上(ko00900), 有8个unigenes映射到三萜合成通路上(ko00909), 包括6个MVA途径的酶, 6个MEP途径的酶, 以及GPPS、FPPS、GGPPS等3个催化萜类骨架生成的的异戊烯基转移酶, SQS、SE、β-AS等3个催化生成β-香树脂(齐墩果烷型五环三萜)的关键酶。
三萜皂苷的生物合成途径研究较为深入的是人参皂苷, 目前关于人参皂苷生物合成途径的基本框架及关键酶的研究取得较大进展, 已从人参属植物中(人参、西洋参、竹节参、三七等)克隆得到了近50个参与人参皂苷生物合成途径的基因, 并对其中20个基因进行了功能研究, 为今后利用合成生物学生产人参皂苷奠定了基础[5]。人参皂苷的生物合成途径由20多步连续的酶促反应组成, 其中的关键酶有羟甲基戊二酰辅酶A还原酶(HMGR)、法尼基焦磷酸合酶(FPPS)、鲨烯合酶(SQS)、鲨烯环氧酶(SE)、达玛烯二醇Ⅱ合成酶(DS)、β-香树脂合成酶(β-AS)、CYP450和UGT等。HMGR是人参皂苷生物合成途径的第一个限速酶, 与动物中只含有1个HMGR基因不同, 在植物中HMGR基因属于多基因家族, 在人参中有2个HMGR基因(PgHMGR1和PgHMGR2), 使用HMGR特异性的抑制剂美维诺林(mevinolin)竞争性抑制HMGR的酶活性, 会显着降低人参不定根中人参皂苷的总含量, 而超表达PgHMGR1基因则会在人参中积累较多三萜类成分[18]。在商陆的转录组数据中有5个unigenes被注释为HMGR, 其中3个具有完整的开放阅读框(ORF), 其中c62237与PgHMGR1氨基酸序列一致性为47.09%, 可能具有HMGR的活性, 所以可使用HMGR抑制剂或者超表达商陆HMGR基因, 研究商陆皂苷含量变化和HMGR活性及表达量变化之间的关系, 最终确定商陆HMGR基因在商陆皂苷生物合成途径中的功能。人参中的DS催化2, 3-氧化鲨烯生成达玛烯二醇Ⅱ, 然后在CYP450 (CYP716A47、CYP716A53等)和UGT (UGT71A27、UGT74AE2等)的催化下生成达玛烷型人参皂苷(Rb1、Rb2、Rc、Rd、Rg1、Rg2等)[19], 这类人参皂苷属于四环三萜型皂苷, 是人参皂苷的主要成分; 而β-AS催化2, 3-氧化鲨烯生成β-香树脂, 然后在CYP450 (CYP716A52) 催化下生产齐墩果酸, 进一步在UGT的催化下生成齐墩果烷型人参皂苷(Ro)[20], 这类人参皂苷属于五环三萜型皂苷。虽然在人参皂苷生物合成途径中已有部分CYP450和UGT基因进行了功能研究, 但CYP450和UGT都属于超基因家族, 目前仍有大量基因家族成员功能未知。从化学结构分析EsA属于齐墩果烷型五环三萜, 根据商陆转录组数据的分析结果, 在商陆中2, 3-氧化鲨烯经β-AS催化生成β-香树脂后, β-香树脂可能在CYP450的催化下生产齐墩果酸, 进一步经UGT的催化生成齐墩果烷型商陆皂苷EsA, 但是有哪些CYP450和UGT基因参与这一过程, 目前并不清楚, 可通过与已知功能的CYP450或UGT基因进行序列比对, 设计简并引物扩增商陆的CYP450和UGT基因, 或者通过转录组分析对茉莉酸甲酯(MeJA)处理前后的商陆根进行比较转录组学研究, 筛选参与EsA生物合成途径的CYP450和UGT基因[21]。根据本研究获得的商陆转录组数据在Swiss-Prot数据库的注释结果, 共获得130条unigenes可能具有CYP450催化功能和46条unigenes可能具有UGT糖基化功能, 以及参与商陆其他次生代谢途径的417条unigenes。本研究获得的参与商陆次生代谢途径的unigenes, 为研究EsA生物合成途径及调控机制、克隆关键基因及功能分析奠定了基础, 也为商陆药材品质的形成提供理论依据。
| [1] | Chinese Pharmacopoeia Commission. Chinese Pharmacopoeia: Vol 1(中国药典: 一部)[S]. Beijing: China Medical Science Press, 2015: 324-325. |
| [2] | Wang PC, Wang QH, Zhao S, et al. Research progress on chemical constituents, pharmacological effects, and clinical applications of Phytolaccae Radix[J]. Chin Tradit Herb Drugs (中草药), 2014, 45: 2722–2731. |
| [3] | Zhang F, Wang X, Qiu X, et al. The protective effect of esculentoside A on experimental acute liver injury in mice[J]. PLoS One, 2014, 9: e113107. DOI:10.1371/journal.pone.0113107 |
| [4] | Yang JL, Gao LL, Zhu P. Advances in the biosynthesis research of ginsenosides[J]. Acta Pharm Sin (药学学报), 2013, 48: 170–178. |
| [5] | Kim YJ, Zhang D, Yang DC. Biosynthesis and biotechnological production of ginsenosides[J]. Biotechnol Adv, 2015, 33: 717–735. DOI:10.1016/j.biotechadv.2015.03.001 |
| [6] | Wang XY, Song JY, Xie CX, et al. RNA-Seq and genuine traditional Chinese medicine[J]. Acta Pharm Sin (药学学报), 2014, 49: 1650–1657. |
| [7] | Chen S, Luo H, Li Y, et al. 454 EST analysis detects genes putatively involved in ginsenoside biosynthesis in Panax ginseng[J]. Plant Cell Rep, 2011, 30: 1593–1601. DOI:10.1007/s00299-011-1070-6 |
| [8] | Hua WP H, Zhang Y, Song J, et al. De novo transcriptome sequencing in Salvia miltiorrhiza to identify genes involved in the biosynthesis of active ingredients[J]. Genomics, 2011, 98: 272–279. DOI:10.1016/j.ygeno.2011.03.012 |
| [9] | Luo HM, Sun C, Sun YZ, et al. Analysis of the transcriptome of Panax notoginseng root uncovers putative triterpene saponinbiosynthetic genes and genetic markers[J]. BMC Genomics, 2011, 12: 1–15. DOI:10.1186/1471-2164-12-1 |
| [10] | He L, Xu XL, Li Y, et al. Transcriptome analysis of buds and leaves using 454 pyrosequencing to discover genes associated with the biosynthesis of active ingredients in Lonicera japonica Thunb[J]. PLoS One, 2013, 8: e62922. DOI:10.1371/journal.pone.0062922 |
| [11] | Grabherr MG, Haas BJ, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nat Biotechnol, 2011, 29: 644–652. DOI:10.1038/nbt.1883 |
| [12] | Li JL, Luo XD, Zhao PJ, et al. Post-modification enzymes involved in the biosynthesis of plant terpenoids[J]. Acta Bot Yunnan (云南植物研究), 2009, 31: 461–468. |
| [13] | Tu J, Zhu P, Cheng KD. Heterologous expression systems of plant cytochrome P450[J]. Chin Biotechnol (中国生物工程杂志), 2003, 23: 32–37. |
| [14] | Luo Y, Liu XG, Zhou ZQ. Research progress on methods for isolating the gene of plant glycosyltransferase, and its biological functions[J]. Biotechnol Bull (生物技术通报), 2016, 32: 34–39. |
| [15] | Neller KCM, Klenov A, Hudak KA. The pokeweed leaf mRNA transcriptome and its regulation by jasmonic acid[J]. Front Plant Sci, 2016, 7: 283. |
| [16] | Newman JD, Chappell J. Isoprenoid biosynthesis in plants:carbon partitioning within the cytoplasmic pathway[J]. Crit Rev Biochem Mol Biol, 1999, 34: 95–106. DOI:10.1080/10409239991209228 |
| [17] | Lichtenthaler HK. The 1-deoxy-D-xylulose-5-phosphate pathway of isoprenoid biosynthesis in plants[J]. Annu Rev Plant Physiol Plant Mol Biol, 2003, 50: 47–65. |
| [18] | Kim YJ, Lee OR, Oh JY, et al. Functional analysis of 3-hydroxy-3-methylglutaryl coenzyme a reductase encoding genes in triterpene saponin-producing ginseng[J]. Plant Physiol, 2014, 165: 373–387. DOI:10.1104/pp.113.222596 |
| [19] | Han JY, Kim HJ, Kwon YS, et al. The Cyt P450 enzyme CYP716A47 catalyzes the formation of protopanaxadiol from dammarenediol-Ⅱ during ginsenoside biosynthesis in Panax ginseng[J]. Plant Cell Physiol, 2011, 52: 2062–2073. DOI:10.1093/pcp/pcr150 |
| [20] | Han JY, Kim MJ, Ban YW, et al. The involvement of β-amyrin 28-oxidase (CYP716A52v2) in oleanane-type ginsenoside biosynthesis in Panax ginseng[J]. Plant Cell Physiol, 2013, 54: 2034–2046. DOI:10.1093/pcp/pct141 |
| [21] | Liang HC, Wang QH, Gong T, et al. The basic strategies and research advances in the studies on glycosyltransferases involved in ginsenoside biosynthesis[J]. Acta Pharm Sin (药学学报), 2015, 50: 148–153. |
 2017, Vol. 52
2017, Vol. 52


