2. 中国医学科学院、北京协和医学院医药生物技术研究所, 北京 100050
2. Institute of Medicinal Biotechnology, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100050, China
表观遗传是指在基因的DNA序列没有发生改变的情况下, 翻译后修饰模式发生可遗传的变化, 最终导致新表型的出现[1]。当前研究者普遍认为, 复杂的表观遗传密码发生改变是导致疾病发展的主要机制之一。组蛋白能发生多种翻译后修饰, 其中染色质修饰酶(包括写入器“writer”和抹去器“eraser”)能给组蛋白加上或者擦除各种修饰, 然后由充当阅读器“reader”角色的一类蛋白阅读这些化学修饰, 帮助执行染色质重构和转录调控功能, 其中BET蛋白家族就是充当染色质阅读器“reader”功能。
1 Bromodomain and extraterminal (BET)蛋白的结构和功能BET蛋白家族是bromodomain蛋白超家族的一个亚类。Bromodomain是一种高度保守的约含110个氨基酸的溴蛋白质功能结构域。Bromodomain包括4个α螺旋片层, 形成疏水腔, 识别乙酰化的赖氨酸。人类基因中有40余种不同的蛋白质编码60多种bromodomain。根据序列的同源性, bromodomain蛋白家族可分成8个亚类, 其中BET蛋白家族是一个特殊群体, 其特点是N末端包含2个保守的bromodomain (BD1和BD2), C末端包含一个ET (extraterminal)结构域。该家族包括4个成员, 分别是BRD2、BRD3、BRD4和BRDT。BET家族蛋白可以结合乙酰化的组蛋白或者非组蛋白, 发挥与细胞生长和周期相关的基因转录调节功能[2, 3]。
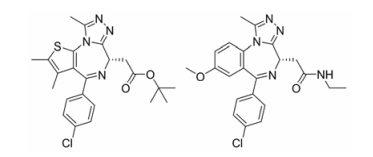
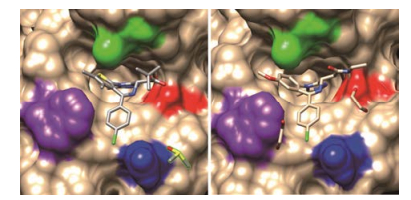
2 BET抑制剂在疾病中的研究进展 2.1 BET抑制剂在肿瘤中的研究进展2010年, 2个研究组[4, 5]分别报道了BET中bromodomain的小分子抑制剂JQ1和I-BET (GSK525762A), 二者具有相似的化学结构和靶点抑制模式(图 1和图 2)。值得注意的是, JQ1与I-BET对BET家族BD1和BD2结构域的亲和力远远超过其他bromodomain超家族成员[6]。这2种BET抑制剂都能嵌入bromodomain口袋中, 与乙酰化的肽竞争性结合, 从而引起BET蛋白从染色质中游离。目前, 已发现和设计了更多的BET小分子抑制剂, 其在肿瘤治疗中的潜在应用价值也得到广泛关注。在中线癌体内外模型中的研究表明, BET抑制剂在临床前肿瘤模型中呈现良好疗效[5]。此外研究发现, BET抑制剂在血液肿瘤小鼠模型中呈现出良好的疗效[7-9]。除了血液癌症, BET抑制剂对一些实体瘤同样敏感。例如, JQ1能抑制非小细胞肺癌和神经母细胞瘤细胞系增殖, 对小鼠甲状腺癌、卵巢癌和前列腺癌均有抑制作用[10-12]; OTX015对白血病、淋巴瘤、骨髓瘤和恶性胶质瘤都有抑制活性[13-15]。BET抑制剂的抗癌活性在特定情况下与Myc转录相关[7-9, 16]。而在另外一些情况下, 这些效果则不依赖于Myc。其中, BET抑制剂抗卵巢癌的机制依赖于FOXM1的下调; 而其对三阴乳腺癌的抑制与负责调控有丝分裂的极光激酶有关[11, 17]。鉴于BET抑制剂在临床前研究中效果良好, 一些Ⅰ期临床试验已经启动, 进一步评估BET抑制剂在癌症患者中的疗效和安全性。其中化合物OTX-015和GSK-525762正在开展Ⅱ期临床试验。
|
Figure 1 Chemical structures of JQ1 (left panel) and I-BET (right panel) |
|
Figure 2 Mode of the binding of JQ1 (PDB: 3MXF, left panel) and I-BET (PDB: 3P5O, right panel) to human BRD4 |
近几年, BET抑制剂在炎症中的研究也获得一定进展。体内实验单次注射I-BET即可抑制脂多糖(LPS)诱导的致死性感染性休克[4]。这种治疗作用已经证实与BRD2、BRD3和BRD4在炎症基因转录诱导中起重要作用相关[4, 18]。I-BET能引起BRD2、BRD3和BRD4蛋白从IL6启动子脱离, 注射I-BET能阻滞内毒素诱导的小鼠死亡[4]。在小鼠眼部炎症疾病模型中的最新研究也证实了JQ1和GSK151可以减少实验性葡萄膜炎[19]。本课题组[20]最近的研究发现JQ1对幽门螺杆菌诱导的炎症也具有调节作用。这可能是JQ1通过抑制BRD4与RelA的相互作用, 抑制了BRD4与RelA复合物被募集到特定基因的启动子上, 从而影响了炎症基因的转录。
2.3 BET抑制剂在其他疾病中的研究进展除了影响癌症和免疫过程, 药理学方法抑制BET也能影响其他器官系统。如在心血管系统中, JQ1通过改变与心肌细胞肥大相关的BRD4依赖的转录从而预防心脏衰竭[21, 22]。JQ1还能抑制四氯化碳诱导的小鼠肝纤维化[23]。在生殖器官中, BET抑制剂通过靶向睾丸特异性BET蛋白BRDT从而抑制精子发生[24]。BET抑制剂对代谢性疾病也有一定作用, 处于临床Ⅲ期试验的化合物RVX-208 (apabetalone, Reserlogix公司)可用于治疗动脉粥样硬化[25, 26]。JQ1还可以通过影响STAT5的转录活性, 抑制PPARγ和C/EBPα等基因的表达从而干扰祖细胞产生脂肪组织[27]。此外, BET抑制剂也影响骨代谢平衡。一些小分子BET抑制剂可以抑制炎性疾病如关节炎、牙周炎、骨肿瘤和骨质疏松中的骨质破坏[28-31]。以BET蛋白为靶标, 可能获得治疗包括骨质疏松症在内的骨代谢平衡紊乱疾病的候选药物[32]。总之, 这些研究表明靶向BET蛋白对哺乳动物的生理与病理生理学呈现多种不同效应(表 1)[4, 5, 7-17, 21, 22, 25-27, 33-35], 值得进行深入的临床评估。
| Table 1 Several bromodomain and extraterminal (BET) inhibitors |
使用染色质免疫沉淀结合DNA测序(ChIP-seq)的技术, 获得了BRD4在全基因组中的富集数据。在各种正常和转化的细胞中, BRD4基本与所有活性启动子和相当一部分增强子有关[21, 36, 37]。BRD4在全基因组上的富集模式与一些组蛋白乙酰化标记分子(例如H4K5、H4K8、H3K9和H3K27) 具有相似性, 这与bromodomain介导的募集机制一致[36, 37]。
对JQ1敏感型基因的分析发现, 这些基因在邻近的增强子区域富集有高水平的BRD4。例如, 在多发性骨髓瘤细胞系MM1.S中, Myc基因座增强子区即有高水平的BRD4富集, IgH增强子区域易位可驱动大量Myc表达, 促进疾病的发展。在这个特定的细胞系中, JQ1能显著抑制Myc转录[8]。值得注意的是, BRD4在IgH增强子上的富集水平超过其在Myc启动子上的10倍, 这表明增强子可能是BRD4依赖的转录活化的主要来源[8]。BRD4在特定的细胞类型中富集大量增强子元件, 但据观察, 在MM1.S细胞只有一小部分增强子(约300个)在IgH区域表现出高水平的BRD4富集[37], 这些元件被称为“超级增强子”。在超级增强子上, BRD4富集水平和跨度非常明显, 可以延伸跨越上万碱基[37, 38]。由超级增强子调节的基因对BET抑制尤为敏感[37]。基于在MM1.S细胞以及LY1淋巴瘤细胞上全基因组的分析发现, 邻近超级增强子的基因与邻近典型增强子的基因相比, 前者经JQ1处理后mRNA下调更为明显[37, 39]。
虽然BRD4在启动子附近区域促进RNA聚合酶延伸的作用已经得到确认, 但BRD4在增强子区域的调节功能尚不清楚。研究表明, BRD4结合到增强子上时, 可以与去甲基化酶JMJD6发生相互作用[40]。BRD4募集JMJD6, 促进两种不同的底物脱甲基化: 7SK RNA的5'甲基磷酸帽和组蛋白H4R3me1、H4R3me2, 其中后者是与转录抑制有关的染色质标记[41]。因为7SK依赖其甲基磷酸帽逃避核酸外切降解[42], 募集JMJD6会导致7SK的降解, 利于BRD4促进P-TEFb的局部激活[40]。远距离的染色质环型相互作用使得增强子结合BRD4/JMJD6/P-TEFb复合物以促进RNA聚合酶Ⅱ在靶启动子近侧区被解除抑制[43]。此外, BRD4和JMJD6分别敲低可引起全基因表达发生类似的变化, 这与两种蛋白在同一种复合物中发挥作用的推测一致[44]。
3.2 BRD4与转录因子直接作用增强子DNA元件一般是由丛集的转录因子结合位点组成, 这些转录因子结合位点形成募集转录调控复合物的平台[45]。BRD4在增强子中存在表明其功能可能与结合这些位点的转录因子有关。
最近的一项生化筛选评估了BRD4是否直接与各种纯化因子相互作用[46]。除了确认已知的BRD4与P-TEFb的相互作用外, 该筛选还发现与BRD4相互作用的转录因子包括p53、YY1、c-Jun、AP2、C/EBPα、C/EBPβ和Myc/Max异源二聚体[46]。在这些实验中BRD4未能与多数被测转录因子结合, 表明BRD4对转录因子具有结合特异性。这些相互作用的发生可能并不依赖于乙酰化, 因为转录因子在大肠杆菌中表达并得以纯化。在后续的生化筛选中, 研究人员还发现p53基因的C端调节结构域和BRD4中bromodomain之外的两个不同区域相互作用。其中一种相互作用, 需要BRD4被酪蛋白激酶Ⅱ (CK2) 预先磷酸化。在没有磷酸化的情况下, BRD4与p53形成的复合物并不能有效结合DNA。一旦被CK2磷酸化, BRD4的第二结合面再与p53结合, 所形成的复合物可以结合DNA并促进转录激活。BRD4的磷酸化能被自身的第二个bromodomain所抑制, 从而允许其与乙酰化的染色质相互作用。这一重要研究证明了BRD4具有通过不依赖bromodomain的方式与转录因子相互作用的功能[46]。
除了识别乙酰化组蛋白, BRD4的bromodomain也可以与转录因子的特定乙酰化区域相互作用。转录因子TWIST具有螺旋-环-螺旋结构, 在癌症发展过程中可以促进EMT[47]。TWIST可以被TIP60乙酰转移酶乙酰化, 进而特异性结合BRD4的BD2结构域[47]。研究人员提出了一个假设模型, 认为BRD4同时与乙酰化组蛋白(通过BD1) 和乙酰化TWIST (通过BD2) 的增强子区域相关联, 促进P-TEFb介导的转录激活。BRD4与TWIST的相互作用揭示了BRD4在上皮癌生物学中的作用, 这也可能是与BET抑制剂在乳腺癌异种移植模型中呈现治疗效果的机制之一[47]。
转录因子NF-κB是由p65和p50形成的异源二聚体, 在免疫和癌症的病理发展中具有重要作用[48]。当配体结合到细胞表面受体(例如, LPS结合到TLR4受体或者TNFα结合到TNFR1受体)后, 可激活下游信号反应, 引起NF-κB转位进入细胞核内并快速激活炎症相关的基因转录[48]。利用I-BET处理巨噬细胞后, 发现其能减缓LPS诱导的炎症相关基因的表达, 表明I-BET对NF-κB通路具有调节作用[4]。NF-κB亚基RelA的赖氨酸310位点能被p300乙酰转移酶乙酰化, 促进其与BRD4两个bromodomain的相互作用[49]。
本课题组[49, 50]发现, BRD4可以与赖氨酸310位点乙酰化的RelA结合, 且这种结合有助于肿瘤中NF-κB的持续性组成型激活。BET抑制剂对肿瘤的调节可能与抑制两者结合有关。通过靶向NF-κB和BRD4之间的相互作用可能是治疗NF-κB所致癌症的一种方法。JQ1就是通过抑制BRD4与赖氨酸310位点乙酰化的RelA相互作用, 削弱癌蛋白Tax介导的NF-κB的转录激活, 从而抑制人T细胞白血病病毒1 (HTLV-1) 诱发的T细胞白血病[34]。对JQ1处理的心肌细胞中基因表达分析发现, NF-κB靶基因整体表达下调, 进一步表明BET抑制剂可抑制NF-κB功能[21]。以上研究提示BET抑制剂的抗肿瘤和抗炎效应可能是通过抑制NF-κB而实现的。
3.3 BRD4在调节病毒性基因表达中的作用HIV以转录沉默状态潜伏于细胞中是其耐药的主要机制, 因此通过药理学方法重新激活HIV的转录是永久性清除病毒库的一种策略。研究表明, BRD4能调节HIV转录的多个过程[51, 52]。一系列报道指出BET抑制剂能在潜伏感染的细胞中激活HIV转录和复制[53-56]。BRD4和病毒转录激活蛋白Tat分别能竞争性结合P-TEFb[52]。BRD4被JQ1抑制后, 大量的P-TEFb得以释放并与Tat相互作用以激活HIV基因组的转录延长[54, 56]。JQ1抑制HIV转录的机制可能也涉及对BRD2的抑制, 因为BRD2能负性调节HIV的潜伏[53]。这些研究强调BET抑制剂通过改变细胞中或宿主基因中可用的P-TEFb含量, 从而间接产生转录效应[2]。
病毒编码的DNA结合蛋白也能与BRD4相互作用, 调节病毒生命周期[57]。例如, HPV能编码一种DNA结合蛋白称为E2, 它直接与BRD4相互作用, 从而调节病毒基因的表达[58, 59]。在这种特定情况下, BRD4以乙酰化依赖的方式阻断起始前复合物的组装, 发挥共抑制因子的功能[58]。HPV利用E2-BRD4的相互作用将病毒的游离基因拴到有丝分裂过程中分开的染色体上, 确保病毒在细胞分裂后持续存在[59]。基于BRD4是大量病毒的宿主细胞因子, 有必要在慢性和急性感染模型中进一步观察BET抑制剂的疗效和安全性。
4 前景与展望由于BRD4是疾病相关基因调控网络的重要组成部分, BET蛋白近年来在一系列疾病模型中成为治疗靶点。但BET抑制剂对众多转录通路的调节使得研究者质疑其在人体中的耐受性, 毕竟药物耐受性是临床试验评估的一部分。Nature也发表了几篇关于BET抑制剂的癌症耐药性机制探讨的文章[60-62], 希望可以借此改善其临床反应。将来研究的一个重要目标是判断选择性干预BET蛋白功能能否改善治疗效果。从药理学角度特异性靶向BET蛋白的BD1或BD2, 有望获得更为特异的效应。据报道, 一种BET抑制剂能优先结合BD2而非BD1, 而该化合物与JQ1相比产生了最小的转录效应[63]。基于不同BET蛋白在基因组中的富集模式不同, 针对某个BET家族成员的选择性抑制剂也在预期中[64]。BRD4作为一个新颖的案例, 体现了利用药理学方法靶向通用调节子, 进而诱导特异生物学效应的可能性。
| [1] | Holliday R. The inheritance of epigenetic defects[J]. Science, 1987, 238: 163–170. DOI:10.1126/science.3310230 |
| [2] | Bartholomeeusen K, Xiang Y, Fujinaga K, et al. Bromodomain and extraterminal (BET) bromodomain inhibition activate transcription via transient release of positive transcrip-tion elongation factor b (P-TEFb) from 7SK small nuclear ribonucleoprotein[J]. J Biol Chem, 2012, 287: 36609–36616. DOI:10.1074/jbc.M112.410746 |
| [3] | Stonestrom AJ, Hsu SC, Werner MT, et al. Erythropoiesis provides a BRD's eye view of BET protein function[J]. Drug Discov Today Technol, 2016, 19: 23–28. DOI:10.1016/j.ddtec.2016.05.004 |
| [4] | Nicodeme E, Jeffrey KL, Schaefer U, et al. Suppression of inflammation by a synthetic histone mimic[J]. Nature, 2010, 468: 1119–1123. DOI:10.1038/nature09589 |
| [5] | Filippakopoulos P, Qi J, Picaud S, et al. Selective inhibition of BET bromodomains[J]. Nature, 2010, 468: 1067–1073. DOI:10.1038/nature09504 |
| [6] | Prinjha RK, Witherington J, Lee K. Place your BETs:the therapeutic potential of bromodomains[J]. Trends Pharmacol Sci, 2012, 33: 146–153. DOI:10.1016/j.tips.2011.12.002 |
| [7] | Dawson MA, Prinjha RK, Dittmann A, et al. Inhibition of BET recruitment to chromatin as an effective treatment for MLL-fusion leukaemia[J]. Nature, 2011, 478: 529–533. DOI:10.1038/nature10509 |
| [8] | Delmore JE, Issa GC, Lemieux ME, et al. BET bromodomain inhibition as a therapeutic strategy to target c-Myc[J]. Cell, 2011, 146: 904–917. DOI:10.1016/j.cell.2011.08.017 |
| [9] | Zuber J, Shi J, Wang E, et al. RNAi screen identifies Brd4 as a therapeutic target in acute myeloid leukaemia[J]. Nature, 2011, 478: 524–528. DOI:10.1038/nature10334 |
| [10] | Puissant A, Frumm SM, Alexe G, et al. Targeting MYCN in neuroblastoma by BET bromodomain inhibition[J]. Cancer Discov, 2013, 3: 308–323. DOI:10.1158/2159-8290.CD-12-0418 |
| [11] | Zhang ZF, Ma PF, Jing Y, et al. BET bromodomain inhibition as a therapeutic strategy in ovarian cancer by downregulating FoxM1[J]. Theranostics, 2016, 6: 219–230. DOI:10.7150/thno.13178 |
| [12] | Asangani IA, Dommeti VL, Wang X, et al. Therapeutic targeting of BET bromodomain proteins in castration-resistant prostate cancer[J]. Nature, 2014, 510: 278–282. DOI:10.1038/nature13229 |
| [13] | Sahai V, Redig AJ, Collier KA, et al. Targeting BET bromodomain proteins in solid tumors[J]. Oncotarget, 2016, 7: 53997–54009. DOI:10.18632/oncotarget.v7i33 |
| [14] | Berenguer-Daize C, Astorgues-Xerri L, Odore E, et al. OTX015(MK-8628), a novel BET inhibitor, displays in vitro and in vivo antitumor effects alone and in combination with conventional therapies in glioblastoma models[J]. Int J Cancer, 2016, 139: 2047–2055. DOI:10.1002/ijc.v139.9 |
| [15] | Amorim S, Stathis A, Gleeson M, et al. Bromodomain inhibitor OTX015 in patients with lymphoma or multiple myeloma:a dose-escalation, open-label, pharmacokinetic, phase 1 study[J]. Lancet Haematol, 2016, 3: e196–e204. DOI:10.1016/S2352-3026(16)00021-1 |
| [16] | Mertz JA, Conery AR, Bryant BM, et al. Targeting MYC dependence in cancer by inhibiting BET bromodomains[J]. Proc Natl Acad Sci U S A, 2011, 108: 16669–16674. DOI:10.1073/pnas.1108190108 |
| [17] | Sahni JM, Gayle SS, Bonk KL, et al. Bromodomain and extraterminal protein inhibition blocks growth of triplenegative breast cancers through the suppression of Aurora kinases[J]. J Biol Chem, 2016, 291: 23756–23768. DOI:10.1074/jbc.M116.738666 |
| [18] | Belkina AC, Nikolajczyk BS, Denis GV. BET protein function is required for inflammation:Brd2 genetic disruption and BET inhibitor JQ1 impair mouse macrophage inflammatory responses[J]. J Immunol, 2013, 190: 3670–3678. DOI:10.4049/jimmunol.1202838 |
| [19] | Eskandarpour M, Alexander R, Adamson P, et al. Pharmacological inhibition of bromodomain proteins suppresses retinal inflammatory disease and downregulates retinal Th17 cells[J]. J Immunol, 2017, 198: 1093–1103. DOI:10.4049/jimmunol.1600735 |
| [20] | Chen J, Wang Z, Hu X, et al. BET inhibition attenuates Helicobacter pylori-induced inflammatory response by suppressing inflammatory gene transcription and enhancer activation[J]. J Immunol, 2016, 196: 4132–4142. DOI:10.4049/jimmunol.1502261 |
| [21] | Anand P, Brown JD, Lin CY, et al. BET bromodomains mediate transcriptional pause release in heart failure[J]. Cell, 2013, 154: 569–582. DOI:10.1016/j.cell.2013.07.013 |
| [22] | Spiltoir JI, Stratton MS, Cavasin MA, et al. BET acetyllysine binding proteins control pathological cardiac hypertrophy[J]. J Mol Cell Cardiol, 2013, 63: 175–179. DOI:10.1016/j.yjmcc.2013.07.017 |
| [23] | Ding N, Hah N, Yu RT, et al. BRD4 is a novel therapeutic target for liver fibrosis[J]. Proc Natl Acad Sci U S A, 2015, 112: 15713–15718. |
| [24] | Matzuk MM, McKeown MR, Filippakopoulos P, et al. Small-molecule inhibition of BRDT for male contraception[J]. Cell, 2012, 150: 673–684. DOI:10.1016/j.cell.2012.06.045 |
| [25] | Gilham D, Wasiak S, Tsujikawa LM, et al. RVX-208, a BETinhibitor for treating atherosclerotic cardiovascular disease, raises ApoA-I/HDL and represses pathways that contribute to cardiovascular disease[J]. Atherosclerosis, 2016, 247: 48–57. DOI:10.1016/j.atherosclerosis.2016.01.036 |
| [26] | Nicholls SJ, Puri R, Wolski K, et al. Effect of the BET protein inhibitor, RVX-208, on progression of coronary atherosclerosis:results of the phase 2b, randomized, doubleblind, multicenter, assure trial[J]. Am J Cardiovasc Drugs, 2016, 16: 55–65. DOI:10.1007/s40256-015-0146-z |
| [27] | Goupille O, Penglong T, Kadri Z, et al. Inhibition of the acetyl lysine-binding pocket of bromodomain and extraterminal domain proteins interferes with adipogenesis[J]. Biochem Biophys Res Commun, 2016, 472: 624–630. DOI:10.1016/j.bbrc.2016.03.013 |
| [28] | Lamoureux F, Baud'huin M, Rodriguez Calleja L, et al. Selective inhibition of BET bromodomain epigenetic signalling interferes with the bone-associated tumour vicious cycle[J]. Nat Commun, 2014, 5: 3511. |
| [29] | Park-Min KH, Lim E, Lee MJ, et al. Inhibition of osteo-clastogenesis and inflammatory bone resorption by targeting BET proteins and epigenetic regulation[J]. Nat Commun, 2014, 5: 5418. DOI:10.1038/ncomms6418 |
| [30] | Meng S, Zhang L, Tang Y, et al. BET inhibitor JQ1 blocks inflammation and bone destruction[J]. J Dent Res, 2014, 93: 657–662. DOI:10.1177/0022034514534261 |
| [31] | Gjoksi B, Ghayor C, Siegenthaler B, et al. The epigenetically active small chemical N-methyl pyrrolidone (NMP) prevents estrogen depletion induced osteoporosis[J]. Bone, 2015, 78: 114–121. DOI:10.1016/j.bone.2015.05.004 |
| [32] | Baud'huin M, Lamoureux F, Jacques C, et al. Inhibition of BET proteins and epigenetic signaling as a potential treatment for osteoporosis[J]. Bone, 2017, 94: 10–21. DOI:10.1016/j.bone.2016.09.020 |
| [33] | Lockwood WW, Zejnullahu K, Bradner JE, et al. Sensitivity of human lung adenocarcinoma cell lines to targeted inhibition of BET epigenetic signaling proteins[J]. Proc Natl Acad Sci U S A, 2012, 109: 19408–19413. DOI:10.1073/pnas.1216363109 |
| [34] | Wu X, Qi J, Bradner JE, et al. Bromodomain and extraterminal (BET) protein inhibition suppresses human T cell leukemia virus 1(HTLV-1) Tax protein-mediated tumorigenesis by inhibiting nuclear factor κB (NF-κB) signaling[J]. J Biol Chem, 2013, 288: 36094–36105. DOI:10.1074/jbc.M113.485029 |
| [35] | Chung CW, Coste H, White JH, et al. Discovery and characterization of small molecule inhibitors of the BET family bromodomains[J]. J Med Chem, 2011, 54: 3827–3838. DOI:10.1021/jm200108t |
| [36] | Zhang W, Prakash C, Sum C, et al. Bromodomain-containing protein 4(BRD4) regulates RNA polymerase Ⅱ serine 2 phosphorylation in human CD4+ T cells[J]. J Biol Chem, 2012, 287: 43137–43155. DOI:10.1074/jbc.M112.413047 |
| [37] | Lovén J, Hoke HA, Lin CY, et al. Selective inhibition of tumor oncogenes by disruption of super-enhancers[J]. Cell, 2013, 153: 320–334. DOI:10.1016/j.cell.2013.03.036 |
| [38] | Whyte WA, Orlando DA, Hnisz D, et al. Master transcription factors and mediator establish super-enhancers at key cell identity genes[J]. Cell, 2013, 153: 307–319. DOI:10.1016/j.cell.2013.03.035 |
| [39] | Chapuy B, McKeown MR, Lin CY, et al. Discovery and characterization of super-enhancer-associated dependencies in diffuse large B cell lymphoma[J]. Cancer Cell, 2013, 24: 777–790. DOI:10.1016/j.ccr.2013.11.003 |
| [40] | Liu W, Ma Q, Wong K, et al. Brd4 and JMJD6-associated anti-pause enhancers in regulation of transcriptional pause release[J]. Cell, 2013, 155: 1581–1595. DOI:10.1016/j.cell.2013.10.056 |
| [41] | Zhao Q, Rank G, Tan YT, et al. PRMT5-mediated methylation of histone H4R3 recruits DNMT3A, coupling histone and DNA methylation in gene silencing[J]. Nat Struct Mol Biol, 2009, 16: 304–311. DOI:10.1038/nsmb.1568 |
| [42] | Jeronimo C, Forget D, Bouchard A, et al. Systematic analysis of the protein interaction network for the human transcription machinery reveals the identity of the 7SK capping enzyme[J]. Mol Cell, 2007, 27: 262–274. DOI:10.1016/j.molcel.2007.06.027 |
| [43] | Hargreaves DC, Horng T, Medzhitov R. Control of inducible gene expression by signal-dependent transcriptional elongation[J]. Cell, 2009, 138: 129–145. DOI:10.1016/j.cell.2009.05.047 |
| [44] | Dey A, Chitsaz F, Abbasi A, et al. The double bromodomain protein Brd4 binds to acetylated chromatin during interphase and mitosis[J]. Proc Natl Acad Sci U S A, 2003, 100: 8758–8763. DOI:10.1073/pnas.1433065100 |
| [45] | Calo E, Wysocka J. Modification of enhancer chromatin:what, how, and why?[J]. Mol Cell, 2013, 49: 825–837. DOI:10.1016/j.molcel.2013.01.038 |
| [46] | Wu SY, Lee AY, Lai HT, et al. Phospho switch triggers Brd4 chromatin binding and activator recruitment for gene-specific targeting[J]. Mol Cell, 2013, 49: 843–857. DOI:10.1016/j.molcel.2012.12.006 |
| [47] | Shi J, Wang Y, Zeng L, et al. Disrupting the interaction of BRD4 with diacetylated Twist suppresses tumorigenesis in basal-like breast cancer[J]. Cancer Cell, 2014, 25: 210–225. DOI:10.1016/j.ccr.2014.01.028 |
| [48] | Hayden MS, Ghosh S. NF-κB, the first quarter-century:remarkable progress and outstanding questions[J]. Genes Dev, 2012, 26: 203–234. DOI:10.1101/gad.183434.111 |
| [49] | Huang B, Yang XD, Zhou MM, et al. Brd4 coactivates transcriptional activation of NF-κB via specific binding to acetylated RelA[J]. Mol Cell Biol, 2009, 29: 1375–1387. DOI:10.1128/MCB.01365-08 |
| [50] | Zou Z, Huang B, Wu X, et al. Brd4 maintains constitutively active NF-κB in cancer cells by binding to acetylated RelA[J]. Oncogene, 2014, 33: 2395–2404. DOI:10.1038/onc.2013.179 |
| [51] | Jang MK, Mochizuki K, Zhou M, et al. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase Ⅱ-dependent transcription[J]. Mol Cell, 2005, 19: 523–534. DOI:10.1016/j.molcel.2005.06.027 |
| [52] | Yang Z, Yik JHN, Chen R, et al. Recruitment of P-TEFb for stimulation of transcriptional elongation by the bromodomain protein Brd4[J]. Mol Cell, 2005, 19: 535–545. DOI:10.1016/j.molcel.2005.06.029 |
| [53] | Boehm D, Calvanese V, Dar RD, et al. BET bromodomaintargeting compounds reactivate HIV from latency via a Tatindependent mechanism[J]. Cell Cycle, 2013, 12: 452–462. DOI:10.4161/cc.23309 |
| [54] | Li Z, Guo J, Wu Y, et al. The BET bromodomain inhibitor JQ1 activates HIV latency through antagonizing Brd4 inhibition of Tat-transactivation[J]. Nucleic Acids Res, 2013, 41: 277–287. DOI:10.1093/nar/gks976 |
| [55] | Banerjee C, Archin N, Michaels D, et al. BET bromodomain inhibition as a novel strategy for reactivation of HIV-1[J]. J Leukoc Biol, 2012, 92: 1147–1154. DOI:10.1189/jlb.0312165 |
| [56] | Zhu J, Gaiha GD, John SP, et al. Reactivation of latent HIV-1by inhibition of BRD4[J]. Cell Rep, 2012, 2: 807–816. DOI:10.1016/j.celrep.2012.09.008 |
| [57] | Weidner-Glunde M, Ottinger M, Schulz TF. What do viruses BET on?[J]. Front Biosci, 2010, 15: 537–549. DOI:10.2741/3632 |
| [58] | Wu SY, Lee AY, Hou SY, et al. Brd4 links chromatin targeting to HPV transcriptional silencing[J]. Genes Dev, 2006, 20: 2383–2396. DOI:10.1101/gad.1448206 |
| [59] | You J, Croyle JL, Nishimura A, et al. Interaction of the bovine papillomavirus E2 protein with Brd4 tethers the viral DNA to host mitotic chromosomes[J]. Cell, 2004, 117: 349–360. DOI:10.1016/S0092-8674(04)00402-7 |
| [60] | Fong CY, Gilan O, Lam EYN, et al. BET inhibitor resistance emerges from leukaemia stem cells[J]. Nature, 2015, 525: 538–542. DOI:10.1038/nature14888 |
| [61] | Rathert P, Roth M, Neumann T, et al. Transcriptional plasticity promotes primary and acquired resistance to BET inhibition[J]. Nature, 2015, 525: 543–547. DOI:10.1038/nature14898 |
| [62] | Shu S, Lin CY, He HH, et al. Response and resistance to BET bromodomain inhibitors in triple-negative breast cancer[J]. Nature, 2016, 529: 413–417. DOI:10.1038/nature16508 |
| [63] | Picaud S, Wells C, Felletar I, et al. RVX-208, an inhibitor of BET transcriptional regulators with selectivity for the second bromodomain[J]. Proc Natl Acad Sci U S A, 2013, 110: 19754–19759. DOI:10.1073/pnas.1310658110 |
| [64] | Anders L, Guenther MG, Qi J, et al. Genome-wide localization of small molecules[J]. Nat Biotechnol, 2014, 32: 92–96. |
 2017, Vol. 52
2017, Vol. 52


