2. 中药鉴定与安全性检测评估北京市重点实验室, 中国中医科学院中药研究所, 北京 100700;
3. 华南农业大学林学与风景园林学院, 广东 广州 510642
2. Key Laboratory of Beijing for Identification and Safety Evaluation of Chinese Medicine, Institute of Chinese Materia Medica, China Academy of Chinese Medical Sciences, Beijing 100700, China;
3. College of Forestry and Landscape Architecture, South China Agricultural University, Guangzhou 510642, China
2015版《中国药典》规定桃仁为蔷薇科植物桃Prunus persica (L.) Batsch或山桃P. davidiana (Carr.) Franch.的干燥成熟种子, 具有活血祛瘀、润肠通便、止咳平喘的功效; 苦杏仁为蔷薇科植物山杏Prunus armeniaca L. var. ansu Maxim.、东北杏P. mandshurica (Maxim.) Koehne、西伯利亚杏P. sibirica L.或杏P. armeniaca L.的干燥成熟种子, 具有降气止咳平喘、润肠通便的功效[1]。在外形上两者颇为相似, 色泽、大小均具有较高的相似度, 尤其是去皮后的桃仁和苦杏仁更加难以区分。在药效上, 两者功效亦较为相近, 但是在临床上应用范围却不同, 且两者互为混伪品。药理学研究证明苦杏仁具有小毒, 苦杏仁掺杂到桃仁中存在一定风险, 需谨慎用药[2]。因此, 建立一种准确鉴定桃仁和苦杏仁的分子鉴定体系, 以及苦杏仁对桃仁进行掺伪鉴定, 以控制桃仁的药材质量, 对桃仁以及中药材的用药安全都具有十分重要的意义。目前, 中药材鉴定技术主要依靠显微鉴定、理化鉴定及化学分析, 但是这些传统技术局限于研究人员的专业知识和主观判断, 存在一定的局限性, 因此, 迫切需要寻求一种准确、客观和快捷的新方法来解决混伪品及掺伪品的问题。
高分辨率熔解曲线 (high resolution melting, HRM) 技术作为本草基因组学的重要组成部分[3], 是一种较为前沿的实时定量PCR技术[4, 5], 在核苷酸差异检测中具有快速、灵敏和准确等突出的优点, 在基因突变、基因分型和遗传多样性等研究中发展迅速[6-10]。DNA条形码 (DNA barcoding) 技术具有较强的通用性, 作为物种鉴定的新技术, 颇受国内外研究者关注, 近几年迅速成为物种分类和鉴定的研究热点。该技术通过筛选确定通用条形码, 建立条形码数据库和鉴定平台进而对物种进行鉴定。中国学者通过大样本量系统性研究, 提出internal transcribed spacer 2 (ITS2) 序列可以作为植物物种鉴别的通用条形码用于鉴定药用植物和近缘种[11]。目前以ITS2序列为主体的《中药材DNA条形码分子鉴定指导原则》已被纳入2015版《中国药典》第四部。近几年来, 国内外学者将HRM技术结合DNA条形码应用于药用植物及中成药产品鉴定, 表现出较强的鉴定能力[12, 13]。本研究运用HRM技术, 基于植物DNA条形码ITS2序列对苦杏仁掺桃仁进行定性、定量检测分析, 研究HRM技术对桃仁中药材基原物种鉴定的适用性和对苦杏仁混品定量的灵敏性, 以期为桃仁中药材及其产品真实性进行快速准确的鉴定, 为其生产过程、市场监管和临床用药安全提供科学依据和保障。
材料与方法材料 本研究通过实地采集桃仁和苦杏仁各基原植物, 实验材料经中国中医科学院中药研究所孙伟副研究员鉴定, 凭证标本保存于湖北中医药大学药学院, 详细信息见表 1。另外从药材市场收集到5份桃仁药材, 并分别对其编号为Com 1~5。
| Table 1 Sample information and number of Persicae semen and Armeniacae semen amarum used in this study |
试剂 植物基因组DNA提取试剂盒购于天根生化科技 (北京) 有限公司。2× HRM PCR master mix试剂盒购于凯杰 (德国) 生物技术有限公司。其他试剂均为国产分析纯试剂。
样品DNA提取 取桃仁和苦杏仁各基原植物叶片30 mg, 或刮去种子外表皮取药材内部约50 mg, 用研磨仪 (Sceintz-48, 宁波新芝) 研磨2 min (50次/秒) 后, 利用植物基因组DNA提取试剂盒 (Tiangen Biotech Co., China) 提取总DNA。采用NanoDrop 2000 (Thermo Scientific, USA) 测定OD260/OD280比值及DNA浓度。利用Qubit 3.0分光光度计 (Invitrogen Co., USA) 将DNA浓度稀释到50~60 ng·μL-1并保存于-20 ℃备用。
为了检验HRM方法对中药掺杂的灵敏性和可行性, 根据参考文献设计了一系列的苦杏仁对桃仁的掺杂比例, 分别为0、1%、5%、7%、10%、15%、30%、50%和100%[14]。药材充分研磨成粉末状后按不同比例混合均匀, 每个比例的总量为100 mg, 每个比例做3次物理重复。
PCR扩增 PCR扩增和高分辨率熔解曲线分析在Rotor-Gene Q MDx (QIAGEN GmbH, Hilden, Ger many) 荧光PCR仪上进行, 反应试剂来自QIAGEN®HRM PCR master mix试剂盒, 序列扩增引物采用ITS2通用引物 (ITS2F: 5'-ATG CGA TAC TTG GTG TGA AT-3', ITS3R: 5'-GAC GCT TCT CCA GAC TAC AAT-3')。在实验过程中, 对反应体系进行了优化实验, 结果发现最佳的反应体系为25 μL: 50~ 60 ng基因组DNA 1.0 μL, 2× HRM PCR master mix 12.5 μL, 上下游引物 (10 μmol·L-1) 各1.0 μL, 补足ddH2O至25 μL。最佳反应条件是采用72孔反应模块, 扩增程序为94 ℃预变性3 min, 94 ℃变性30 s, 56 ℃退火30 s, 72 ℃延伸45 s, 循环40次, 每个循环结束时收集荧光信号数据。扩增产物的熔解步骤在PCR循环结束后立即进行, 程序为:熔解温度从70 ℃逐渐升温至90 ℃, 每0.15 ℃收集一次荧光信号。HRM-PCR扩增产物使用ABI 3730XL测序仪 (Applied Biosystems Co., USA) 进行双向测序。
数据分析高分辨率熔解曲线分析用QIAGEN公司的Rotor-Gene Q (version 2.3.1.49) 软件进行。运用CodonCode Aligner V 6.0.5 (CodonCode Co., USA) 对获得的序列进行校对拼接, 并去除低质量序列及引物区; 使用基于隐马尔可夫模型的HMMer注释方法[15]去除两端5.8S和28S区段, 获得ITS2间隔区序列。
结果与分析 1 HRM技术在中药材桃仁和苦杏仁中的鉴定应用图 1 (图 1a和1b) 显示桃仁与苦杏仁两种药材能够很好地区分开, 且桃与山桃能够分开, 但杏、西伯利亚杏、东北杏、山杏区分不为明显。标准化的随温度变化的熔解曲线 (normalized and shifted melting curve) 可以展示分型间的差异 (图 1a), 也可派生出其他曲线形式 (normalized and temp-shifted difference plot) (图 1b) 将其间差异进行放大, 更方便观察物种及基因型间的差别。通过基因型 (genotypes) 设置, 以基原物种桃P. persica为基因型 (图 1b), 并设置基因型置信值GCPs (genotype confidence percentages) ≥90%, 结果显示, 桃仁和苦杏仁能更为明显地鉴定区分。
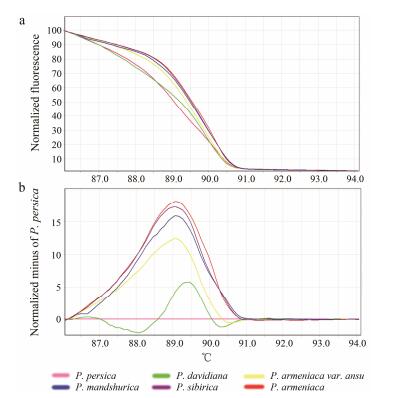
|
Figure 1 Melting curve profiles of amplicons obtained by HRM analysis of species of Persicae semen and Armeniacae semen amarum with the ITS2 type indicated. (a) Normalized melting profiles of three species with the ITS2 type indicated. (b) Difference curves of three species with one haplotype of P. persica as the reference genotype. Color-coded legend of species corresponded to the colors of the melting curves. |
经调查发现桃仁的主要来源为桃, 苦杏仁为杏, 故本文将测序获得的桃和杏的ITS2序列经BLASTN比对分析, 结果表明桃仁 (桃) 和苦杏仁 (杏) 的核苷酸序列之间存在较大的种间差异, 相似度为95.3% (图 2)。由此可知, 尽管在形态学上两种药材基原难以区分, 但两者的ITS2序列的差异位点可以为进一步建立桃仁药材HRM鉴定方法提供依据。用HRM技术检测7个不同比例的杏与桃混合样品, 结果显示, 7个不同比例的混合样品产生了7个不同的熔解曲线, 且各曲线差异显著 (图 3)。以桃P. persica设为基因型作为参照, 并设置基因型置信值GCPs≥90%, 与杏对桃不同比例的混合样本产生相对荧光值的差值为纵坐标而生成派生熔解曲线。图 3结果显示, 不同比例的杏与桃样品之间的基因型置信值具有一定的规律相关性, 且线性回归模型相关系数为r = 0.96 (图 4)。根据图 3和图 4的结果, 可以推断HRM技术对杏样品的最低检出限为1%。将HRM-PCR产物进行Sanger测序后发现, 混合样品的序列峰图都出现套峰现象, 无法通过DNA Barcoding的方法进行鉴定。而HRM技术在混合样品检测上却具有非常高的灵敏度, 通过HRM曲线的可视化结果可以直观判断某样品是否为混合样品, 并且可以根据曲线的峰型推算出混伪品的掺入量。

|
Figure 2 Sequence alignment of Prunus persica and P. armeniaca |
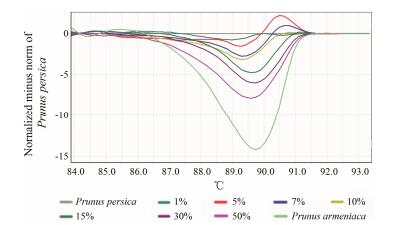
|
Figure 3 Prunus persica mixed with the adulterations of P. armeniaca detected by HRM analysis. Different graphs of HRM curves of the ITS2 region primer assay. HRM data are presented as graphs displaying the fluorescence of two amplicons and are applied to reference mixtures containing 1%, 5%, 7%, 10%, 15%, 30%, and 50% of seed from pure P. armeniaca in pure P. persica. The difference graph of seven proportions using P. persica as the reference genotype is shown. Color-coded legend of proportions corresponded to the colors of the melting curves |
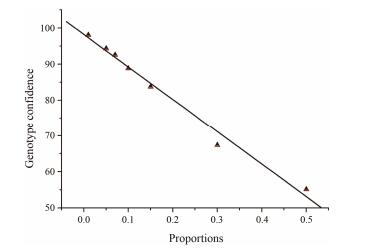
|
Figure 4 Pure Prunus persica was mixed with P. armeniaca in progressive proportions (1%, 5%, 7%, 10%, 15%, 30%, 50%). The results are presented as a linear regression with r = 0.96 |
利用HRM技术结合ITS2条形码序列对药材市场收集到的5份样品进行检测, 检测结果如图 5所示, 研究发现Com 1、2曲线与山桃P. davidiana接近; Com 5曲线与桃P. persica接近; Com 3曲线与东北杏P. mandshurica接近; Com 4曲线在另一侧, 没有与已知物种的曲线所对应, 经测序比对发现为麦李P. glandulosa。为了确保HRM的实验结果的准确性, 将HRM-PCR产物进行DNA条形码测序验证。将获得的ITS2序列在中药材DNA条形码鉴定系统 (www.tcmbarcode.cn) 和GenBank数据库中应用BLAST (basic local alignment search tool) 方法进行结果判定, 结果中相似性最高的序列对应物种为查询序列最接近的物种, 结果见表 2。比对鉴定结果表明, 与HRM结果相吻合, 从另一方面说明HRM技术具有快速识别正伪品、可疑物种等优势, 是DNA条形码技术有效的补充。
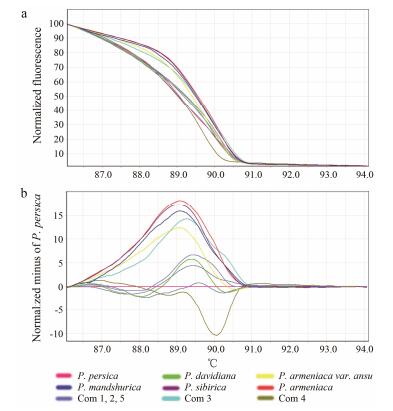
|
Figure 5 Melting curve profiles of amplicons obtained by HRM analysis of species of Persicae semen, Armeniacae semen amarum and commercial herbal products with the ITS2 type indicated. (a) Normalized melting profiles of three species with the ITS2 type indicated. (b) Difference curves of three species with one haplotype of P. persica as the reference genotype. Color-coded legend of species corresponded to the colors of the melting curves |
| Table 2 The results of ITS2 sequences in this study |
近年来, 中药中毒事件时有发生, 中药准确安全使用的情况备受人们关注, DNA条形码技术的兴起为检测中药材的替代品和掺假药材提供新的鉴定方法。2010年, Chen等[11]对753个属的4 800种植物6 600份样品, 7种候选DNA条形码序列 (psbA-trnH、martK、rbcL、rpoC1、ycf5、ITS2和ITS) 进行筛选, 发现ITS2序列在种级水平上的鉴定率高达92.7%。ITS2序列现已被广泛应用于中草药中, 例如ITS2序列能够很好地对羌活及其混伪品, 以及对含有马兜铃酸等药用植物[16, 17]进行准确鉴定并区分。2015版《中国药典》第四部已将以ITS2序列为主, psbA-trnH序列为辅作为植物类药材核心条形码序列, 列入“中药材DNA条形码分子鉴定指导原则”。
DNA条形码技术对评估中国药材市场出售的中草药具有重要的潜力。药材市场出售的药材产品经常未标记或加工后难以辨认, 如粉末、细小种子或其他干燥药材, 给中药鉴定带来很大困难, 再如本研究中的桃仁和苦杏仁, 若将两者混合后难以肉眼区分, 人为或无意掺杂对中药安全用药造成极大威胁。Lin等[18]采用ITS2序列已成功对桃仁及其近缘混伪品进行鉴定区分, 其中也包含苦杏仁。桃仁和苦杏仁均为《中国药典》收载的品种, 同为常用药, 但各自临床应用范围不同, 且不可相互替换使用。而苦杏仁作为毒性药材, 如果不正确使用 (滥用或误用) 可能会导致严重的健康问题。本研究也表明HRM技术能够提供便捷、快速、高灵敏度的方法对苦杏仁进行量化[19], 相信在不久的将来此技术也将会在中医药中广泛应用。
本文通过大量物种及样本筛选, 以及不断学习和总结经验[20-22], 最终选取形态、大小、质地相近的桃仁与苦杏仁作为研究对象。HRM结果显示, 桃仁和苦杏仁能够很好的区分, 并可以准确鉴定到各自的物种。然而实际上, 只单单采用测序往往无法识别混合样品中出现套峰的杂质, 不能很好地阐明两个或多个物种共同存在而产生的套峰。HRM技术对检测桃与杏混合产生的7种不同比例的样本混合物, 能够检测到其最低混合限度1:100, 为以后市场药材检测提供一定的技术基础。HRM技术可以很好地补充DNA条形码, 并提供一个快速识别 (时间 < 2 h)、闭管操作 (操作过程无需开盖)、灵敏度高 (能够识别单碱基差异)、无损样品 (产物可直接测序) 及低成本的检测技术, 也是本草基因组学重要的组成部分。将HRM技术结合DNA条形码ITS2序列, 对《中国药典》种子类药材桃仁的准确鉴定和定量将起到一定的作用, 保证了药材在临床上的安全准确应用。
| [1] | Chinese Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. 2015 ed. Vol 1. Beijing: China Medical Science Press, 2015: 201-278. |
| [2] | Ye HZ, Cai JZ. Identification and application if Armeniacae Semen amarum and Persicae Semen[J]. Strait Pharm J, 2008, 20: 85–86. |
| [3] | Chen SL, Song JY. Herbgenomics[J]. China J Chin Mater Med (中国中药杂志), 2016, 41: 3881–3889. |
| [4] | Wittwer CT. High resolution DNA melting analysis:advancements and limitations[J]. Hum Mutat, 2009, 30: 857–859. DOI:10.1002/humu.v30:6 |
| [5] | Vietina M, Agrimonti C, Marmiroli N. Detection of plant oil DNA using high resolution melting (HRM) post PCR analysis:a tool for disclosure of olive oil adulteration[J]. Food Chem, 2013, 141: 3820–3826. DOI:10.1016/j.foodchem.2013.06.075 |
| [6] | Krypuy M, Newnham GM, Thomas DM, et al. High resolution melting analysis for the rapid and sensitive detection of mutations in clinical samples:KRAS codon 12 and 13 mutations in non-small cell lung cancer[J]. BMC Cancer, 2006, 6: 295. DOI:10.1186/1471-2407-6-295 |
| [7] | Lin CY, Li MJ, Chang JG, et al. High-resolution melting analyses for genetic variants in ARID5B and IKZF1 with childhood acute lymphoblastic leukemia susceptibility loci in Taiwan[J]. Blood Cells Mol Dis, 2014, 52: 140–145. DOI:10.1016/j.bcmd.2013.10.003 |
| [8] | Madesis P, Ganopoulos I, Anagnostis A, et al. The application of Bar-HRM (Barcode DNA-High Resolution Melting) analysis for authenticity testing and quantitative detection of bean crops (Leguminosae) without prior DNA purification[J]. Food Control, 2012, 25: 576–582. DOI:10.1016/j.foodcont.2011.11.034 |
| [9] | Sakaridis I, Ganopoulos I, Argiriou A, et al. A fast and accurate method for controlling the correct labeling of prod-ucts containing buffalo meat using High Resolution Melting (HRM) analysis[J]. Meat Sci, 2013, 94: 84–88. DOI:10.1016/j.meatsci.2012.12.017 |
| [10] | Druml B, Cichna-Markl M. High resolution melting (HRM) analysis of DNA-its role and potential in food analysis[J]. Food Chem, 2014, 158: 245–254. DOI:10.1016/j.foodchem.2014.02.111 |
| [11] | Chen SL, Yao H, Han JP, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plan species[J]. PLoS One, 2010, 5: e8613. DOI:10.1371/journal.pone.0008613 |
| [12] | Osathanunkul M, Madesis P, De BH. Bar-HRM for authentication of plant-based medicines:evaluation of three medicinal products derived from Acanthaceae species[J]. PLoS One, 2015, 10: e0128476. DOI:10.1371/journal.pone.0128476 |
| [13] | Osathanunkul M, Suwannapoom C, Ounjai S, et al. Refining DNA barcoding coupled high resolution melting for discrimination of 12 closely related croton species[J]. PLoS One, 2015, 10: e0138888. DOI:10.1371/journal.pone.0138888 |
| [14] | Ganopoulos I, Madesis P, Tsaftaris A. Universal ITS2 barcoding DNA region coupled with high-resolution melting (HRM) analysis for seed authentication and adulteration testing in leguminous forage and pasture species[J]. Plant Mol Biol Rep, 2012, 30: 1322–1328. DOI:10.1007/s11105-012-0453-3 |
| [15] | Keller A, Schleicher T, Schultz J, et al. 5.8S-28S rRNA interaction and HMM-based ITS2 annotation[J]. Gene, 2009, 430: 50–57. DOI:10.1016/j.gene.2008.10.012 |
| [16] | Xin TY, Yao H, Luo K, et al. Stability and accuracy of the identification of Notopterygii Rhizoma et Radix using the ITS/ITS2 barcodes[J]. Acta Pharm Sin (药学学报), 2012, 47: 1098–105. |
| [17] | Wu L, Sun W, Wang B, et al. An integrated system for identifying the hidden assassins in traditional medicines containing aristolochic acids[J]. Sci Rep, 2015, 5: 11318. DOI:10.1038/srep11318 |
| [18] | Lin YH, Liu X, Hu ZG, et al. Molecular identification of Persicae semen and its sibling species based on ITS2 sequence[J]. World Sci Technol Mod Tradit Chin Med Mater Med (世界科学技术-中医药现代化), 2013, 15: 429–434. |
| [19] | Bosmali I, Ganopoulos I, Madesis P, et al. Microsatellite and DNA-barcode regions typing combined with High Resolution Melting (HRM) analysis for food forensic uses:a case study on lentils (Lens culinaris)[J]. Food Res Int, 2012, 46: 141–147. DOI:10.1016/j.foodres.2011.12.013 |
| [20] | Chen K, Jiang C, Yuan Y, et al. High resolution melting and its application in traditional Chinese medicine[J]. Acta Pharm Sin (药学学报), 2015, 50: 1581–1588. |
| [21] | Li JJ, Song M, Xiong C, et al. Application of barcode high-resolution melting for rapid authentication of the medicinal plant Psammosilene tunicoides[J]. Biotechnol Biotechnol Equip, 2016. DOI:10.1080/13102818.2016.1181988 |
| [22] | Sun W, Li JJ, Xiong C, et al. The potential power of Bar-HRM technology in herbal medicine identification[J]. Front Plant Sci, 2016. DOI:10.3389/fp1s.2016.00367 |
 2017, Vol. 52
2017, Vol. 52


