2. 中国农业科学院特产研究所, 吉林 长春 130112;
3. 中国科学院北京基因组研究所, 北京 100101
2. Institute of Special Animal and Plant, Chinese Academy of Agricultural Sciences, Changchun 130112, China;
3. Beijing Institute of Genomics, Chinese Academy of Sciences, Beijing 100101, China
人参 (Panax ginseng C. A. Mey) 为五加科多年生宿根草本植物, 在我国已有5 000多年的用药历史[1], 具有大补元气、复脉固脱、补脾益肺、生精养血、安神益智的功效[2]。人参皂苷 (ginsenoside) 是人参的主要活性成分[3], 广泛作用于心脑血管系统、中枢神经系统、免疫系统等方面[4, 5], 是一类极具药用价值的化学物质。大量研究表明, 植物激素可以调控人参皂苷的合成[6, 7]、提高光合速率与抗氧化酶系统活性[8, 9]、促进根的生长发育[10], 其种类与时空分布显著影响人参的发育及次生代谢。生长素是第一个被发现的植物激素, 在植物生长发育过程中如顶端优势、花序和叶序发育、胚的发育、主根发育、侧根和不定根发生、向性生长及维管组织的分化等扮演着重要角色[11]。已有研究表明, 生长素可以促进人参皂苷合成, 对人参根的生长也有重要调节作用[10, 12], 因此探索生长素对人参生长发育的调控机制将有助于人参品种的改良。
生长素对植物生长发育的调节主要依赖于极性运输形成的生长素浓度梯度[11], PIN (pin-formed) 基因是公认的生长素极性运输基因[13]。拟南芥PIN蛋白家族有PIN1~PIN8共8个成员, 广泛作用于多个生长发育阶段。其中, PIN2主要作用于植物根的向地性[14], PIN3主要作用于茎和根的向性生长[15], PIN6主要作用于根与花的发育[16, 17]。本文首次研究了人参生长素极性运输基因PIN在不同部位的表达情况, 为进一步挖掘PIN基因的功能、解析生长素分布对人参皂苷合成及人参生长发育的调控奠定理论基础。
材料与方法材料 取人参种子, 用70%乙醇水溶液浸泡3 min, 然后用1%次氯酸钠浸泡30 min, 灭菌去离子水洗3遍, 将已成熟的胚状体从种子中转移出来, 并在含有1%蔗糖的1/2 MS固体培养基中25 ± 2 ℃培养3周 (16 h光照/8 h黑暗), 萌发后的材料转移到分化培养基 (MS + 3 mg·L-1 6-BA + 5 mg·L-1 NAA + 1 mg·L-1 IAA) 中诱导成苗。组培苗的叶柄切成1.0 cm长, 作为诱导人参不定根的外植体。将外植体在1/2 MS (含3.0 mg·L-1 IBA) 固体培养基中25 ± 2 ℃暗培养4周, 待外植体形成不定根后将其分离, 并继代到1/2 MS液体培养基 (含3.0 mg·L-1 IBA) 中25 ± 2 ℃暗培养。3周继代一次, 12周后, 不定根生长稳定快速, 可作为后续的实验材料使用。
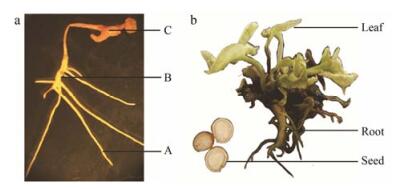
总RNA提取与单链cDNA合成 人参不定根分为根尖1 cm (A)、有分支部位 (B)、棕黄色老根 (C) 3个部位 (图 1a), 人参组培苗分为根、叶及种子3个部位 (图 1b)。取人参组培苗及不定根不同部位的样品用液氮研磨粉碎, 根据TRIzol Plant RNA Kit (Life Technologies) 说明书提取总RNA, 用1%琼脂糖凝胶电泳 (Bio-Rad) 确定RNA完整性, 核酸/蛋白定量仪 (NanoDrop 2000, Thermo) 测定RNA含量。cDNA合成按M-MLV RTase cDNA Synthesis Kit (大连宝生物工程有限公司) 说明书进行并作为PCR反应的模板。
|
Figure 1 Three parts of adventitious roots (a) and three parts culture seedlings (b) of P. ginseng. A: Root tips; B: Branching roots; C: Old roots |
引物的设计与合成 根据拟南芥PIN基因核酸序列信息, 在人参基因组中进行同源序列比对分析, 获得人参PIN基因序列, 运用PCR扩增并测序确认了3条人参PIN基因, 根据这3条序列信息设计并验证了3对荧光定量PCR引物PIN2-F/R、PIN3-F/R和PIN6-F/R; 以人参IF3G1基因为内参, 序列同文献[18]。引物由上海生工生物工程技术服务有限公司合成, 见表 1。
| Table 1 Primers of real-time PCR |
实时荧光定量PCR 利用实时荧光定量PCR仪 (Applied Biosystems 7500) 检测上述基因在不定根不同部位的表达模式。实时荧光定量PCR反应体系为SYBR Premix Ex Taq酶 (大连宝生物工程有限公司) 5 μL、上下游引物 (10 μmol·L-1) 各0.5 μL、ROX 0.25 μL、cDNA模板 (100 mg·L-1) 1 μL, 加水补足至10 μL, 每个反应体系至少重复3次。扩增程序如下: 95 ℃预变性3 min; 95 ℃变性5 s, 60 ℃退火延伸34 s (每次循环后采集荧光信号), 40个循环; 95 ℃变性10 s, 65~95 ℃做熔解曲线分析, 每个温度以每步0.5 ℃上升, 停留5 s, 获得每个基因的Ct值进行相对表达量分析。
基因相对表达量的计算 人参不定根基因表达的计算是以不定根C部位作为基准参比部位, 计算5个基因在A、B、C部位的相对表达量; 人参组培苗基因表达的计算是以种子作为基准参比部位。利用美国ABI公司SDS V2.3软件对PCR过程的数据进行收集和分析, 然后计算样品基因相对表达量, 计算公式为: 2-ΔΔCt = 2-[(Ct目的基因-Ct管家基因) 待测组-(Ct目的基因-Ct管家基因) 对照组]。
人参转录组热图聚类分析 将NCBI SRA (Se quence Read Archive) 库中4年生果期吉林人参不同组织部位转录组数据 (SRR2952867~SRR2952880) 进行重头拼接, 构建完整的转录本库, 运用已获得的3条人参PIN基因序列进行比对, 筛选出5条PIN2、4条PIN3和1条PIN6同源基因。运用Heml软件 (Heatmap Illustrator, version 1.0) 对基因表达进行聚类分析, 分析这10个PIN家族基因在吉林人参不同组织部位的表达模式。
基因表达相关性分析 利用SPSS 20.0软件对人参不同组织部位的5个基因表达进行Pearson相关性分析, |r| > 0.7说明两者呈现高度线性相关, 0.7≥ |r| ≥ 0.5说明两者呈现中度线性相关, 0.5≥ |r| ≥0.3则两者呈现低度相关, |r| < 0.3两者相关性极低, P≤0.05为显著水平, P≤0.01为极显著水平[19]。
结果与讨论 1 人参PIN基因的生物信息学分析根据拟南芥PIN家族基因的序列信息从人参基因组搜寻人参同源的PIN基因序列, 经过PCR扩增及测序, 筛选到3条PIN基因序列。通过BLASTn比对分析, 对这3条序列进行命名, 以核苷酸相似度最高的序列为准 (Query cover、E-value、Identities综合判断)。与Daucus carota subsp. sativus的PIN2 (GenBank号为XM_017388497.1) 相似度最高的人参PIN序列命名为PgPIN2 (Query cover = 92%, Identi ties = 82%, E-value = 0.0), 与Camellia sinensis的PIN3 (GenBank号为KP896474.1) 相似度最高的人参PIN序列分别命名为PgPIN3 (Query cover = 63%, Identi ties = 79%, E-value = 0.0), 与Vitis vinifera的PIN6 (GenBank号为XM_010666578.2) 相似度最高的人参PIN序列命名为PgPIN6 (Query cover = 91%, Identi ties = 77%, E-value = 0.0)。
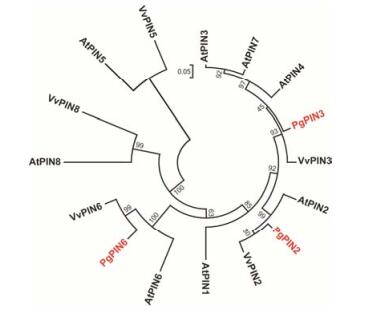
运用MEGA5.05构建人参与拟南芥、葡萄PIN的系统发育树, 结果显示: PgPIN6与拟南芥、葡萄PIN6聚为一支, PgPIN2与拟南芥、葡萄PIN2聚为一支; PgPIN3与葡萄VvPIN3、拟南芥AtPIN3、AtPIN4、AtPIN7聚为一支。研究表明, 拟南芥AtPIN2主要与根向地性生长有关[14], AtPIN3主要在茎和根的向性生长中起作用[15], 而AtPIN6主要与植物蜜腺、雄蕊、初生根与不定根的生长发育相关[16, 17], 系统发育树分析表明, PgPIN2、PgPIN6与拟南芥PIN相似度高, PgPIN3与拟南芥的PIN3有一定差别 (图 2)。因此, 有必要对人参PIN家族基因的表达进行测定, 为进一步挖掘PgPIN2、PgPIN3、PgPIN6在人参中的功能奠定基础。
|
Figure 2 Phylogenetic analysis of protein sequences of P. ginseng PgPINs, Arabidopsis thaliana AtPINs and Vitis vinifera VvPINs. GenBank Number of AtPINs: AtPIN1-NP_177500.1, AtPIN2-NP_ 568848.1, AtPIN3-NP_177250.1, AtPIN4-NP_565261.1, AtPIN5-NP_197157.4, AtPIN6-NP_177836.1, AtPIN7-NP_001077584.1, AtPIN8-NP_197014.1; GenBank Number of VvPINs: VvPIN2-XP_ 002266059.1, VvPIN3-XP_002284302.1, VvPIN5-XP_002279191.1, VvPIN6-XP_010664880.1, VvPIN8-XP_002282480.1 |
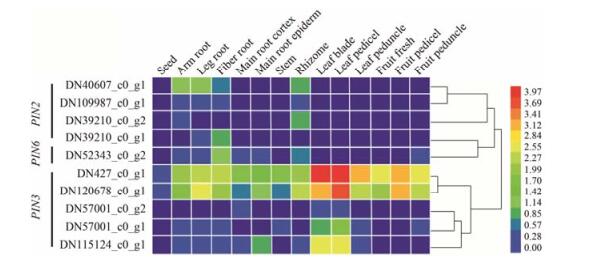
为了系统研究人参不同组织部位PIN家族基因的表达模式, 下载、拼装了NCBI SRA (Sequence Read Archive) 数据库中4年生果期吉林人参不同组织部位的转录组数据 (SRR2952867~SRR2952880)。通过序列同源比对, 从中鉴定出4条PgPIN2的unigene、5条PgPIN3的unigene、1条PgPIN6的unigene。基因表达热图聚类分析发现, 转录组中5条PgPIN3基因在叶 (叶片、总叶柄、小叶柄)、支根表达比较高, 其中DN427_c0_g1与DN120678_c0_g1具有相似的表达模式; PgPIN2基因表达模式类似, 均在艼、支根、须根、芦头表达较高; PgPIN6主要在须根、支根、艼、芦头中表达较高 (图 3)。从结果可以推测, PgPIN3对人参植株地上部分的生长具有较重要的意义, PgPIN2及PgPIN6对人参植株地下部分的生长具有重要意义。
|
Figure 3 Heatmap and cluster analysis of PgPINs in different organs of four-year-old Jilin ginseng at the fruit ripening stage |
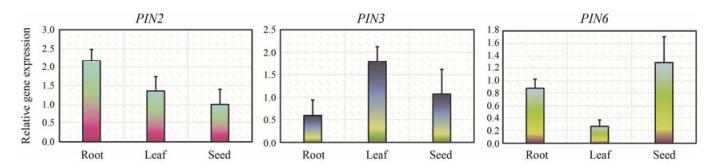
分析了PgPIN在人参组培苗中的叶、根及最初诱导组培苗的种子这3个部位的表达模式 (图 4)。PgPIN2在人参组培苗的根中表达最高, 说明PgPIN2很可能对人参根的生长具有重要作用; PgPIN3在人参组培苗叶、种子、根中的表达依次降低, 表明PgPIN3可能对人参地上部分的极性生长具有调控作用, 这与转录组数据分析相一致; PgPIN6在人参组培苗根和种子中表达高, 而叶中表达很低, 表明PgPIN6主要对人参的地下部位生长具有作用, 而对地上部位的发育作用较小。
|
Figure 4 Expression analysis of PgPINs in different parts of P. ginseng culture seedlings (n≥3) |
为了进一步了解PgPIN2、PgPIN3和PgPIN6的功能, 利用实时荧光定量PCR分析了PgPIN在人参不定根3个部位表达情况 (图 5)。结果显示, PgPIN2在人参不定根B段 (不定根分支处) 表达较低, 而在不定根A段与C段没有显著差异; PgPIN3与PgPIN6在人参不定根A段 (不定根根尖处) 显著高于B段和C段 (P < 0.01), 表现出显著的表达偏好性, 暗示这两个基因可能在不定根的生长发育及向性生长具有相应的功能。
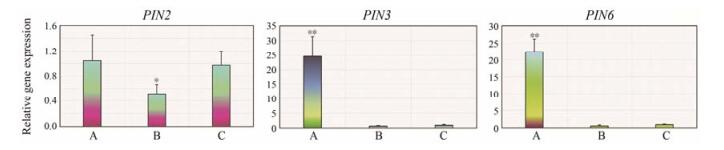
|
Figure 5 Expression analysis of PgPINs in different parts of adventitious root of P. ginseng (n≥3). A: Root tips; B: Branching roots; C: Old roots |
有研究表明, PIN蛋白在细胞水平的生物学功能相同, 即将生长素从胞内输出胞外或输入某种细胞器内, 但PIN家族基因的表达既有组织和器官的特异性又有重叠[13], 因此有必要对这几个基因表达的相关性进行分析。通过Pearson相关性分析可以发现, 在人参不定根中, PgPIN3及PgPIN6之间的表达呈显著正相关 (r > 0.9, P < 0.01), 两者主要在人参不定根的根尖中表达; 在人参组培苗中, 三者表达相关性并不显著。从结果可以看出, 在人参组培苗的不同器官中PgPIN2、PgPIN3与PgPIN6的表达相差较大, 在人参不定根不同部位中PgPIN3与PgPIN6的表达具有一定程度的重叠 (表 2)。
| Table 2 Pearson correlation analysis of PgPINs |
生长素是植物体内唯一具有极性运输特点的激素, 它参与植物的形态建成、向性反应、调控组织的伸长生长及维管束分化、参与胚胎发育、光形态建成等[13]。PIN是一类编码生长素极性运输载体元件的基因家族, 参与植物器官的发生发育、根的向地性及其他向性反应、根的伸长生长及早期的胚胎发育等过程[13, 20]。本研究首次对人参的PIN基因进行生物信息学预测分析及表达研究, 这也是人参属植物第一次报道该基因。
通过序列同源比对和RT-PCR验证, 筛选出3条人参PIN基因序列PgPIN2、PgPIN3及PgPIN6。系统发育分析显示人参的PgPIN与拟南芥及葡萄相应的PIN基因聚在一起, 表明人参PgPIN具有较高的功能保守性。检测了人参不同材料及组织部位PIN基因的表达模式, 并结合拟南芥中PIN的同源基因分析PgPIN相应的功能。拟南芥AtPIN2在根中特异表达, 参与根的向地性反应并调节生长素在根部分生组织中的不对称分布[14, 21], PgPIN2主要在人参地下非主根部位 (艼、支根、须根、芦头) 中表达, 这些部位存在较多比例的原分生组织和初生分生组织, 表明PgPIN2很可能与生长素在人参根部分生组织的极性运输有关。拟南芥AtPIN3能够响应重力刺激, 控制细胞中生长素的侧向分配, 调控植物的向性生长[15], PgPIN3在人参不定根根尖、人参组培苗叶中表达较高, 而PgPIN3在吉林人参中的叶子、果实及根中都有表达, 说明PIN3对人参各器官的极性生长都具有重要意义。AtPIN6能够调控拟南芥蜜腺、雄蕊、初生根、侧根与不定根的生长发育[16, 17], PgPIN6在不定根根尖及人参的地下部分表达明显, 说明人参的PgPIN6可能主要用于调控根的生长发育。
本研究的开展为发掘人参向性生长提供基础数据, 为人参根的形态构建提供新思路, 为人参优良品种的选育奠定基础。同时, 不同器官对人参PIN基因的表达模式有明显影响, 因此, PIN基因也可以作为潜在鉴定人参不同部位入药的标记基因。人参PgPIN基因如何调控人参不同器官的生长发育, 它们之间存在怎样的关系均有待进一步研究。
| [1] | Hemmerly TE. A ginseng farm in Lawrence County, Tennessee[J]. Econ Bot, 1977, 31: 160–162. DOI:10.1007/BF02866586 |
| [2] | Chinese Pharmacopoeia Committee. Pharmacopoeia of People's Republic of China (中华人民共和国药典)[S]. Vol 1. Beijing: China Medical Science Press, 2015: 8. |
| [3] | Im D, Nah S. Yin and Yang of ginseng pharmacology:ginsenosides vs gintonin[J]. Acta Pharmacol Sin, 2013, 34: 1367–1373. DOI:10.1038/aps.2013.100 |
| [4] | Kang S, Min H. Ginseng, the 'immunity boost':the effects of Panax ginseng on immune system[J]. J Ginseng Res, 2012, 36: 354–368. DOI:10.5142/jgr.2012.36.4.354 |
| [5] | Zhao Y, Zhang F, Qu S, et al. The main component of pharmacological research about ginseng in the last three years[J]. Asia-Pacific Tradit Med (亚太传统医药), 2012, 8: 171–174. |
| [6] | Oh JY, Kim YJ, Jang MG, et al. Investigation of ginsenosides in different tissues after elicitor treatment in Panax ginseng[J]. J Ginseng Res, 2014, 38: 270–277. DOI:10.1016/j.jgr.2014.04.004 |
| [7] | Yu LL. The Related Gene Expression of the Ginsenosides Synthesis Regulated by Elicitor in Ginseng Hair Roots (诱导子调控下人参发状根皂苷合成相关基因表达的研究)[D]. Changchun: Jilin Agricultural University, 2014. |
| [8] | Cao WL. Effect of salicylic acid on photosynthesis, physio-biochemistry and quality of Panax ginseng under full sun shine in spring[J]. China J Chin Mater Med (中国中药杂志), 2015, 40: 3553–3559. |
| [9] | Zhang Y, Wang Y, Jiang S, et al. Cloning of the differential expression fragment from ginseng culture induced by salicylic acid[J]. Gen Appl Biol (基因组学与应用生物学), 2009, 28: 245–250. |
| [10] | Washida D, Shimomura K, Takido M, et al. Auxins af-fected ginsenoside production and growth of hairy roots in Panax hybrid[J]. Biol Pharm Bull, 2004, 27: 657–660. DOI:10.1248/bpb.27.657 |
| [11] | Ren Y, Dai S, Liu W. Auxin transport and its roles in signal transduction and plant development[J]. Biotechnol Bull (生物技术通报), 2012, 3: 9–16. |
| [12] | Xiu J, Lian M, Piao C, et al. Effect of auxins and cytokinins on cell growth and ginsenoside production of Panax ginseng during suspension culture[J]. J Agric Sci Yanbian Univ (延边大学农学学报), 2011, 33: 83–88. |
| [13] | Liu SP, Wang L, Wang JR, et al. PIN gene family in higher plants[J]. Plant Physiol Commun (植物生理学通讯), 2009, 45: 833–841. |
| [14] | Chen R, Hilson P, Sedbrook J, et al. The Arabidopsis thaliana AGRAVITROPIC 1 gene encodes a component of the polar-auxin-transport efflux carrier[J]. Proc Natl Acad Sci U S A, 1998, 95: 15112–15117. DOI:10.1073/pnas.95.25.15112 |
| [15] | Friml J, Wiśniewska J, Benková E, et al. Lateral relocation of auxin efflux regulator PIN3 mediates tropism in Arabidopsis[J]. Nature, 2002, 415: 806–809. DOI:10.1038/415806a |
| [16] | Bender RL, Fekete ML, Klinkenberg PM, et al. PIN6 is required for nectary auxin response and short stamen development[J]. Plant J, 2013, 74: 893–904. DOI:10.1111/tpj.2013.74.issue-6 |
| [17] | Cazzonelli CI, Vanstraelen M, Simon S, et al. Role of the Arabidopsis PIN6 auxin transporter in auxin homeostasis and auxin-mediated development[J]. PLoS One, 2013, 8: e70069. DOI:10.1371/journal.pone.0070069 |
| [18] | Wang M, Lu S. Validation of suitable reference genes for quantitative gene expression analysis in Panax ginseng[J]. Front Plant Sci, 2016, 6: 1259. |
| [19] | Hazra A, Gogtay N. Biostatistics series module 6:correlation and linear regression[J]. Indian J Dermatol, 2016, 61: 593–601. DOI:10.4103/0019-5154.193662 |
| [20] | Zou C, Men S. Research advances in auxin efflux carrier PIN proteins[J]. Chin J Cell Biol (中国细胞生物学学报), 2013, 35: 574–582. |
| [21] | Blakeslee JJ, Peer WA, Murphy AS. Auxin transport[J]. Curr Opin Plant Biol, 2005, 8: 494–500. DOI:10.1016/j.pbi.2005.07.014 |
 2017, Vol. 52
2017, Vol. 52


