2. 中国医学科学院、北京协和医学院医药生物技术研究所, 北京 100050;
3. 淮海工学院海洋学院, 江苏 连云港 222000
2. Institute of Medicinal Biotechnology, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100050, China;
3. HuaiHai Insititute of Technology, Lianyungang 222000, China
2012年之前, 全球近40年无全新结构和作用机制的抗菌药品上市, 其中新型可药性靶标的发现相对缺乏是限制抗菌新药发展的瓶颈之一。因此, 针对全新靶点的抗菌新药的研发迫在眉睫。
细菌细胞壁是位于细胞最外的一层厚实、坚韧的外被, 具有固定细胞外形和保护细胞不受损伤等多种生理功能。细菌细胞壁由一些化学成分不同的物质组成, 如肽聚糖、磷壁酸、脂多糖、磷脂、外膜蛋白等, 在这些组成成分中对细胞壁的生理功能起主要作用的是肽聚糖, 除少数细菌如产甲烷细菌这类古细菌的细胞壁是由其他化学物质组成外, 几乎所有细菌的细胞壁都含有肽聚糖, 在G+细菌中肽聚糖含量高达30%~95%, G-细菌也有5%~20%的含量。由于哺乳动物细胞内不存在与细菌类似的细胞壁结构, 也不存在合成细胞壁的各种生物通路, 因此, 与细胞壁合成相关的通路一直都是抗菌药物研究的热点。
肽聚糖是细菌细胞壁重要的组成部分, 是细菌生存所必须的, 它为细菌提供刚性结构使得细菌可在低渗环境中生存。近年来, 随着肽聚糖在细菌内的生物合成通路被逐步解析, 肽聚糖生物合成所涉及的每一个酶也成为发现新型抗生素的重要靶点[1, 2]。
在以往抗生素发现的进程中, 研究重点主要集中于肽聚糖生物合成的后半部[3-5]。然而, 最近几年研究人员也开始关注胞内肽聚糖合成所涉及的靶标酶尤其是Mur酶系[2, 6-8]。
MurA-F催化6步酶促反应最终形成肽聚糖的先导物尿苷-5'-二磷酸 (UDP)-N-乙酰胞壁酸五肽 (图 1)。MurA和MurB以UDP-N-乙酰葡萄糖胺 (UDP-GlcNAc) 为底物催化形成UDP-N-乙酰胞壁酸 (UDP-MurNAc)。随后, Mur连接酶 (MurC-F) 先后将L-Ala、D-Glu、meso-diaminopimelic acid (革兰阴性菌) 或L-Lys (革兰阳性菌) 和二肽D-Ala-D-Ala连入UDP-MurNAc形成UDP-MurNAc-五肽[9, 10]。
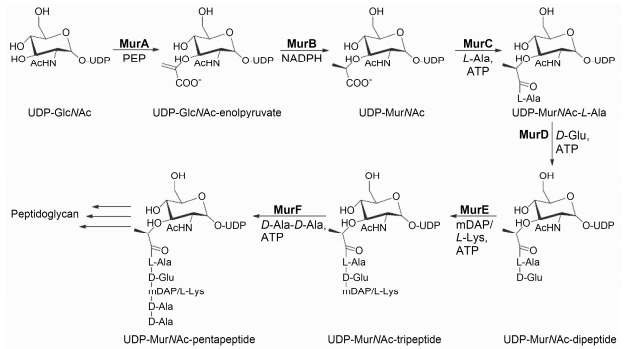
|
Figure 1 Cytoplasmic steps of peptidoglycan biosynthesis catalyzed by the Mur enzymes |
目前对肽聚糖生物合成通路中酶的底物专一性、动力学、催化机制和结构生物学研究的逐渐深入[6, 9-15]为抗结核药物的研究提供了重要的基础和保障。本文对Mur酶近10年所发现的小分子抑制剂的研究进展进行了综述和总结, 为本领域研究人员提供挖掘抑制剂的前沿方向, 为发现活性更为优异的药物先导物打下坚实的基础。
1 MurA抑制剂众所周知, 广谱抗生素fosfomycin (化合物1, 图 2) 是MurA的抑制剂之一, 它能与MurA的活性位点 (Cys115) 形成共价键。Fosfomycin对MurA的抑制存在时间依赖性, 并且在底物UDP-GlcNAc存在的情况下抑制活性升高[16]。结核分支杆菌、沙眼衣原体和伯氏疏螺旋体等对于fosfomycin具有固有耐药性, 因为它们相应的Cys突变为Asp[17]。另外, 细菌降低渗透压、修饰活性中心和产生相应的酶钝化fosfomycin可产生对fosfomycin的耐药性[17], 因此, 当前迫切需要研发具有不同化学结构和作用机制的抑制剂。
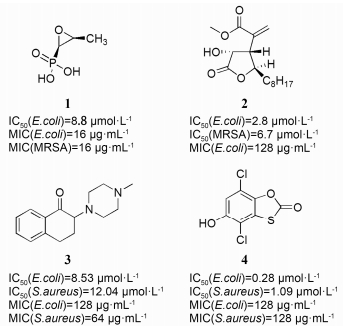
|
Figure 2 Structures and biological activities of some structurally diverse inhibitors of MurA |
最近, Chang等[18]报道了从Neosartorya fischeri分离得到的燕麦曲菌素衍生物 (化合物2, 图 2), 该类化合物是MRSA和fosfomycin抵抗的MurA的有效抑制剂。通过对Novartis化合物库进行高通量筛选, 2-aminotetralone衍生物和benzothioxalone系列化合物被鉴定出来。2-Aminotetralone衍生物 (化合物3, 图 2), 对来自于E. coli和S. aureus的MurA的IC50在微摩尔级别, 并且显示出良好的抑菌活性, MIC在8至128 μg·mL-1之间。Benzothioxalone系列化合物 (化合物4, 图 2) 对MurA的IC50在0.25到18.54 μmol·L-1之间, 一些此类抑制剂对S. aureus的MIC在4和128 μg·mL-1之间。
2 MurB抑制剂至今为止, 只报道了少数几种MurB的抑制剂。4-Thiazolidinone衍生物 (化合物5, 图 3) 是第一类被报道的MurB的抑制剂, 它是模拟烯醇式丙酮酸-UDP-GlcNAc中的二磷酸胞苷部分设计的[19]。其杂环生物电子等排体, imidazolinone衍生物 (化合物6, 图 3) 显示出对MurB高效的抑制性, 并对S.aureus显示出优异的抗菌活性[20]。
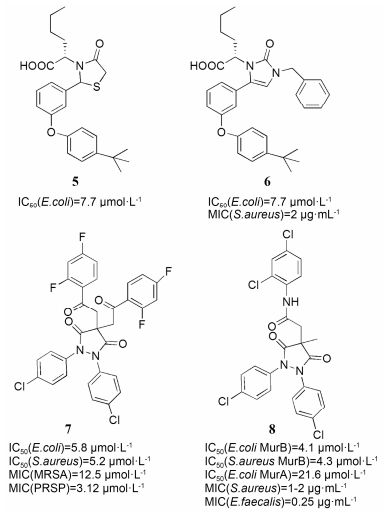
|
Figure 3 Structures and biological activities of the MurB inhibitors, and the dual MurA/MurB inhibitors |
Alkyl pyrazolidinedione衍生物 (化合物7, 图 3) 对来源于S. aureus和E. coli的MurB具有良好的抑制性, 同时对革兰阳性细菌尤其是耐青霉素的Streptococcus pneumoniae (PRSP) 显示出中等的抗菌活性[21]。另外, 一系列pyrazolidinedione衍生物 (化合物8, 图 3) 在低微摩尔浓度下对来源于S. aureus和E. coli的MurB能够产生抑制作用, 同时对MurA和MurC也具有中等的抑制性[22, 23]。
Mur连接酶MurC、MurD、MurE和MurF具有3个相同的活性结构域[24, 25]: N-端结构域1、中心结构域2和C-结构域3 (图 4)。结构域1结合核苷底物。例如, 来自大肠中的酶, 由5个平行的β-折叠被α-螺旋包围组成, 其中α-螺旋在MurE中为2个, 在MurF中为3个, MurF的中心结构域包含6个平行的β-折叠, MurC则为7个。MurD和MurE中β-折叠被7个α-螺旋环绕, MurC中β-折叠被4个α-螺旋环绕, MurF中β-折叠被8个α-螺旋环绕, 这个结构域的两侧被更小的反平行的3β-折叠包围; 结构域3包含6个β-折叠, 其中1个反平行的、5个平行的, 这6个β-折叠被5个α-螺旋包围。这个结构域包含罗斯曼二核苷酸折叠并结合氨基酸基质。这些特点可被用来设计同时靶向多个酶的多靶点抑制剂, 使与靶标相关抵抗的发展的可能性降到最低。
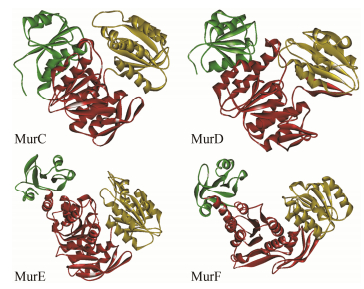
|
Figure 4 Representation of the tridimensional structures of MurC, MurD, MurE and MurF. Domain 1 of the Mur ligases is represented in green, domain 2 in red and domain 3 in yellow |
第一类MurC抑制剂是phosphinate transitionstate衍生物, 其抑制作用最强的化合物 (化合物9, 图 5) 的IC50为49 nmol·L-1。研究发现该类化合物的UDP部分是活性必须基团[26]。
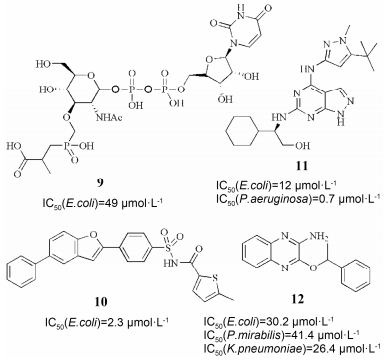
|
Figure 5 Structures and biological activities of the MurC inhibitors |
2004年, AstraZeneca公司通过高通量筛选化合物库获得一系列benzofuran acyl-sulfonamides MurC抑制剂。活性最高的衍生物 (化合物10, 图 5) 对E. coli来源的MurC显示出时间依赖性的、部分可逆的低微摩尔浓度 (IC50, 2.3 μmol·L-1) 的抑制作用。最近, 该公司又筛选获得了一个pyrazolopyrimidine化合物 (化合物11, 图 5), 该化合物对E. coli和P. aeruginosa来源的MurC的IC50均在纳摩尔级别。
Pfizer公司报道了另一个有潜力的MurC的抑制剂。他们利用对E. coli来源的MurC进行高通量筛选获得一个可逆的小分子抑制剂 (化合物12, 图 5), 其IC50为30.2 μmol·L-1, 实验表明化合物12与ATP竞争性结合MurC, 其Ki为8.2 μmol·L-1。然而遗憾的是化合物12仅能抑制部分来源于与E. coli紧密相关菌株的MurC (如Proteus mirabilis, IC50: 41.4 μmol·L-1; Klebsiella pneumoniae, IC50: 26.4 μmol·L-1), 对于来源于其他革兰阴性菌 (H. influenza, Acinetobacter baylyi, P. aeruginosa) 的MurC无明显抑制作用, 另外化合物12对E. coli的生长无明显抑制作用[27]。
4 MurD抑制剂MurD催化D-Glu和UDP-MurNAc-L-Ala之间的酰胺键形成UDP-MurNAc-L-Ala-D-Glu。由于MurD对D-氨基酸基质表现出非常高的特异性, 使得它被认为是发现选择性抗菌剂的最有希望的新靶标之一。
以MurD为靶点的药物筛选研究已有多篇文献报道[9, 28]。通过计算机辅助设计获得的化合物13 (IC50: 0.7 μmol·L-1) 是活力最好的大环类MurD抑制剂[29]。通过虚拟筛选获得9H-xanthene衍生物和polycyclic杂环类抑制剂。化合物14是其中最有代表性的, 它对E. coli来源的MurD的IC50为10 μmol·L-1[30]。Benzene-1, 3-dicarboxylic acid衍生物也是通过虚拟筛选获得的, 化合物15 (图 6) 是其中活性最高的, (IC50: 270 μmol·L-1), 另外, 化合物15对E. coli来源的MurE也有抑制作用 (IC50: 32 μmol·L-1)[31]。
下一代MurD抑制剂是带有谷氨酸结构基元的抑制剂, 最早的含谷氨酸的抑制剂是phosphinate transition-state类似物[32]。这个系列的抑制剂依然是至今已报道过的MurD抑制剂中活性最强的 (例如化合物16, 图 6)。随着谷氨酸部分被确证是发挥MurD抑制活性的关键部分, 对此类抑制剂其他部分改造的研究也在进行着, 引入磺胺基后得到了另一系列的MurD抑制剂。和次磷酸盐结构一样, 磺胺基的引入是为了模拟MurD四面体过渡态[33-35]。早期的萘磺胺类是N-substituted glutamic acid衍生物[34, 35], 化合物17 (包含D-Glu) 和化合物18 (包含L-Glu) 均可抑制E. coli来源的MurD, 包含L-Glu对映体 (化合物18) 的效价要比D-Glu衍生物 (化合物17) 低的多[34]。对早期萘磺胺结构进一步优化得到的新的抑制剂, 其中活性最好的化合物19对MurD的IC50可达85 μmol·L-1。通过NMR和考虑配体柔性和配体-酶特殊相互作用的分子动力学研究对萘磺胺和MurD之间的相互作用进一步分析解释了其抑制活性不高的潜在原因[36]。通过用更加刚性的环取代D-Glu得到了化合物20和21 (图 6), 与原始结构的化合物相比, 对MurD的抑制活性有所提高, 因此证实了构象限制的优势。化合物20与MurD共结晶结构揭示两个羧酸基团与母体化合物的D-Glu占据了完全一样的结合位点。
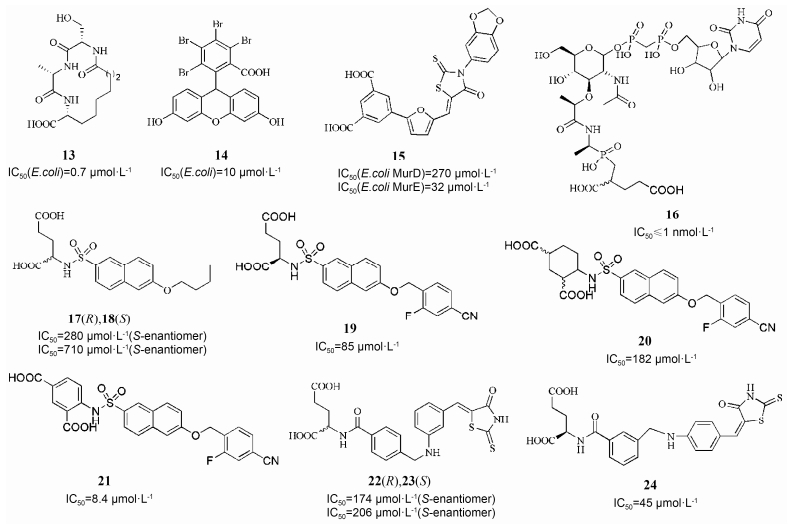
|
Figure 6 Structures and biological activities of some diverse inhibitors of MurD and dual MurD/MurE inhibitors |
Benzylidene-2, 4-thiazolidin-dione和2-thioxothiazolidin-4-onesubstituted glutamic acids代表着另一大类MurD抑制剂。研究发现这类MurD抑制剂当D-Glu (化合物22, 图 6) 被L-Glu (化合物23, 图 6) 取代后活性并未消失[37]。进一步研究发现, 当对取代的芳香环进行改变后, E. coli来源的MurD抑制活性 (化合物24, 图 6) IC50为45 μmol·L-1[38]。一些benzylidene-2, 4-thiazolidin-dione和2-thioxothiazolidin-4-one类抑制剂与MurD复合物晶体结构的成功解析是这类抑制剂发展的一个里程碑, 并且揭示了它们的结合模式, 这两类抑制剂的结合模式非常相似。
来自不同菌种的MurD酶具有保守的催化活性必须的氨基酸残基。然而, 它们整个氨基酸序列的相似性非常小。因此, 只有少数化合物能够同时抑制不同种属的MurD酶也就不足为奇了。例如, 化合物21和24能够在微摩尔水平抑制来自S. aureus, S. pneumoniae, B. burgdorferi和M. tuberculosis的MurD酶。
至今已有多个多肽和非肽类抑制剂被报道。两个九肽 (序列分别为CPAHWPHPC和CSAWSNKFC) 显示出对E. coli来源的MurD良好的抑制活性[39]。
5 MurE抑制剂第一个被发现的MurE的抑制剂是亚磷酸盐 (化合物25, IC50: 1.1 μmol·L-1)[40]。类似于上述MurD的磺胺类抑制剂也可以抑制MurE的活性 (化合物26, 图 7; IC50: 181 μmol·L-1)[31]。序列为CQANLRSQC的环肽对S. aureus来源的MurE有抑制活性[28]。MurE的多肽类抑制剂也被发现, 例如MurEp1对MurE的IC50为500 μmol·L-1[41]。3-Methoxynordomesticine (化合物27, 图 7) 是M. tuberculosis来源的MurE抑制剂。另一个最近发现的可抑制M. tuberculosis来源MurE的抑制剂是化合物28 (IC50: 75 μmol·L-1)[42, 43]。
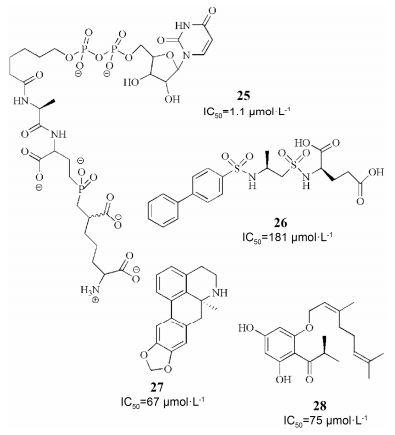
|
Figure 7 Structures and biological activities of the MurE inhibitor |
最初, 利用亲和选择筛选技术获得了两个非常有前景的MurF抑制剂 (化合物29, 图 8; IC50: 1 μmol·L-1)[44]。它们与MurF的复合晶体结构证实了结合模型, 这些数据也为后来的结构优化及最终获得活性最高的化合物30 (IC50: 22 nmol·L-1)[45, 46]奠定了基础。不幸的是, 这一系列化合物均未显示出抗菌活性, 而且对E. coli AcrAB外排泵突变菌株也未显示出活性。这些实验结果全都表明MurF可能不是肽聚糖生物合成的限速步骤[47]。
通过MurF的酶结合实验筛选获得一系列的8-hydroxyquinolines化合物, 其可抑制E. coli来源的MurF活性, 其中化合物31 (图 8) 显示出显著的抗菌活性[48]。此外, 通过在这些化合物基础上构建的一个药效团模型获得两个diarylquinolines化合物 (例如化合物32, 图 8), 可通过破坏细胞壁的生物合成抑制MurF活性。化合物32还对Enterococcus faecalis, Enterococcus faecium和S. aureus有抑菌活性, MIC是8 μg·mL-1 [49]。
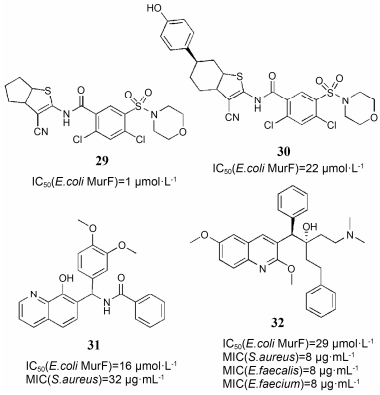
|
Figure 8 Structures and biological activities of the MurF inhibitors |
研究人员已认识到多靶点药物的治疗效果要比选择性单靶点药物的要高。但是设计多靶点药物仍面临着众多挑战, 例如保留类药特性和平衡各靶点之间的亲和作用[50, 51]。针对多靶点Mur酶抑制剂的研究已获得了一些具有适度多靶点抑制活性的化合物。
Wyeth等[52]研究发现萘基特窗酸是Mur酶的多靶点抑制剂, 对MurA-E, 显示出IC50在微摩尔级别的抑制活性。化合物33 (图 9) 显示出对9个Mur酶均衡的抑制作用, 而且对E. coli和S. aureus具有显著的抗菌活性, 其MIC值分别为2 μg·mL-1和1~2 μg·mL-1。
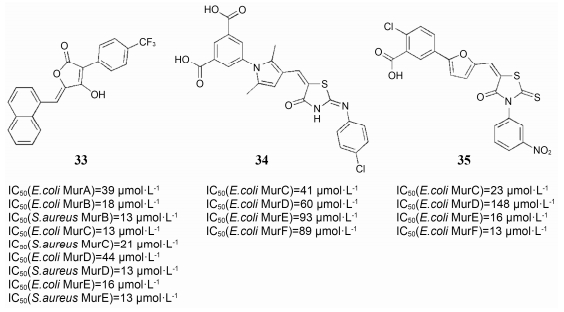
|
图 9 Multiple inhibitors of the Mur enzymes |
最近, Perdih等[53]通过虚拟筛选发现benzene-1, 3-dicarboxylic acid 2, 5-dimethylpyrrole类衍生物具有MurD/MurE双靶点抑制活性, 并在此基础上经过结构优化发现了5个抑制水平在微摩尔级别的MurC-MurF的多靶点抑制剂 (例如化合物34)。Perdih等[54]还报道了furan-based benzene-1, 3-dicarboxylic acid类衍生物对MurC-MurF的多靶点抑制活性, 其中, 化合物35还对S. aureus有抑菌活性, MIC是32 μg·mL-1。
8 结语由于耐药菌的广泛出现, 使得新药的研究迫在眉睫[55, 56]。本篇综述以尚未开发的抗菌药物靶标——Mur酶为切入点详细介绍了很多已报道的Mur酶抑制剂。Mur酶被用于抗菌抑制剂靶标研究的原因如下: ①它是细菌生存所必需的; ② X射线晶体结构的获得, 显示了跨菌种的相似性, 并为以结构为基础的药物设计提供了可能; ③它们与哺乳动物的酶没有同源性; ④它们的复合底物和生化实验较易获得或实现; ⑤迄今已研制出很多的抑制剂, 证明Mur酶具有成药性[6, 9, 14]。
以MurA为靶点的抗菌药物的有效性, 已通过在临床上使用的小分子抗生素fosfomycin被证实。但是, 还有一些与其他Mur酶相关的问题在将来抑制剂的开发过程中需要牢记和克服[57]。至今为止还未有具有抗菌活性的MurB-F抑制剂明确作用模式的报道。主要原因是大多数抑制剂不能透过细胞壁或者被外排泵快速泵出。另外, Mur系列酶在体内可能会形成复杂的多酶复合物, 并阻止抑制剂到达其活性位点。同时Mur系列酶容易对代谢通路中的产物产生反馈抑制。概括地说要获得具有良好抗菌活性的Mur酶抑制剂还有很长的路要走[57]。目前, 具有纳摩尔级别抑制活性和类药性的抑制剂仅局限于MurF, 不可逆抑制剂仅在MurA中发现, 针对其他Mur酶的抑制剂还有待于进一步的设计开发。
众所周知, 作用于单靶点的抑制剂容易由于细菌结合位点的突变而失去活性。为了避免此种情况, 同时作用于多靶点的抑制剂被开发出来。尽管现阶段多靶点Mur酶抑制剂还处于概念验证阶段, 但相信随着进一步的结构优化和发展, 最终会产生具有良好抗菌活性的多靶点Mur酶抑制剂[50, 51]。
| [1] | Green DW. The bacterial cell wall as a source of antibacte-rial targets[J]. Expert Opin Ther Targets, 2002, 6: 1–20. DOI:10.1517/14728222.6.1.1 |
| [2] | Gautam A, Vyas R, Tewari R. Peptidoglycan biosynthesis machinery:a rich sourceof drug targets[J]. Crit Rev Biotechnol, 2011, 31: 295–336. DOI:10.3109/07388551.2010.525498 |
| [3] | Sauvage E, Kerff F, Terrak M, et al. The penicillin-binding proteins:structure and role in peptidoglycan biosynthesis[J]. FEMS Microbiol Rev, 2008, 32: 234–258. DOI:10.1111/j.1574-6976.2008.00105.x |
| [4] | Kong KF, Schneper L, Mathee K. Beta-lactam antibiotics:from antibiosis to resistance and bacteriology[J]. APMIS, 2010, 118: 1–36. |
| [5] | Schneider T, Sahl HG. An oldie but a goodie-cell wall biosynthesis as antibiotic target pathway[J]. Int J Med Microbiol, 2010, 300: 161–169. DOI:10.1016/j.ijmm.2009.10.005 |
| [6] | Bugg TDH, Braddick D, Dowson CG, et al. Bacterial cell wall assembly:still an attractive antibacterial target[J]. Trends Biotechnol, 2011, 29: 167–173. DOI:10.1016/j.tibtech.2010.12.006 |
| [7] | Coates ARM, Halls G, Hu Y. Novel classes of antibiotics or more of the same?[J]. Brit J Pharmacol, 2011, 163: 184–194. DOI:10.1111/j.1476-5381.2011.01250.x |
| [8] | Wise R, Blaser M, Carrs O, et al. The urgent need for new antibacterial agents[J]. J Antimicrob Chemother, 2011, 66: 1939–1940. DOI:10.1093/jac/dkr261 |
| [9] | Barreteau H, Kovač A, Boniface A, et al. Cytoplasmic steps of peptidoglycan biosynthesis[J]. FEMS Microbiol Rev, 2008, 32: 168–207. DOI:10.1111/j.1574-6976.2008.00104.x |
| [10] | El Zoeiby A, Sanschagrin F, Levesque RC. Structure and function of the Mur enzymes:development of novel inhibi-tors[J]. Mol Microbiol, 2003, 47: 1–12. |
| [11] | Vollmer W, Blanot D, De Pedro MA. Peptidoglycan struc-ture and architecture[J]. FEMS Microbiol Rev, 2008, 32: 149–167. DOI:10.1111/j.1574-6976.2007.00094.x |
| [12] | Katz AH, Caufield CE. Structure-based design approaches to cell wall biosynthesis inhibitors[J]. Curr Pharm Design, 2003, 9: 857–866. DOI:10.2174/1381612033455305 |
| [13] | Van Heijenoort J. Recent advances in the formation of the bacterial peptidoglycan monomer unit[J]. Nat Prod Reports, 2001, 18: 503–519. DOI:10.1039/a804532a |
| [14] | Smith CA. Structure, function and dynamics in the mur family of bacterial cell wall ligases[J]. J Mol Biol, 2006, 362: 640–655. DOI:10.1016/j.jmb.2006.07.066 |
| [15] | Lovering AL, Safadi SS, Strynadka NCJ. Structural perspective of peptidoglycan biosynthesis and assembly[J]. Ann Rev Biochem, 2012, 81: 451–478. DOI:10.1146/annurev-biochem-061809-112742 |
| [16] | Marquardt JL, Brown ED, Lane WS, et al. Kinetics, stoi-chiometry, and identification of the reactive thiolate in the inactivation of UDP-GlcNAc enolpyruvoyl transferase by the antibiotic fosfomycin[J]. Biochemistry, 1994, 33: 10646–10651. DOI:10.1021/bi00201a011 |
| [17] | Castañeda-García A, Blázquez J, Rodríguez-Rojas A. Molecular mechanisms and clinical impact of acquired and intrinsic fosfomycin resistance[J]. Antibiotics, 2013, 2: 217–236. DOI:10.3390/antibiotics2020217 |
| [18] | Chang CM, Chern J, Chen MY, et al. Avenaciolides:poten-tial MurA-targeted inhibitors against peptidoglycan biosynthesis in methicillin-resistant Staphylococcus aureus (MRSA)[J]. J Am Chem Soc, 2014, 137: 267–275. |
| [19] | Bachelier A, Mayer R, Klein CD. Sesquiterpene lactones are potent and irreversible inhibitors of the antibacterial target enzyme MurA[J]. Bioorg Med Chem Lett, 2006, 16: 5605–5609. DOI:10.1016/j.bmcl.2006.08.021 |
| [20] | Steinbach A, Scheidig AJ, Klein CD. The unusual binding mode of cnicin to the antibacterial target enzyme MurA revealed by X-ray crystallography[J]. J Med Chem, 2008, 51: 5143–5147. DOI:10.1021/jm800609p |
| [21] | Kutterer KMK, Davis JM, Singh G, et al. 4-Alkyl and 4, 4'-dialkyl 1, 2-bis (4-chlorophenyl) pyrazolidine-3, 5-dione derivatives as new inhibitors of bacterial cell wall biosynthesis[J]. Bioorg Med Chem Lett, 2005, 15: 2527–2531. DOI:10.1016/j.bmcl.2005.03.058 |
| [22] | Gilbert AM, Failli A, Shumsky J, et al. Pyrazolidine-3, 5-diones and 5-hydroxy-1H-pyrazol-3(2H)-ones, inhibitors of UDP-N-acetylenolpyruvyl glucosamine reductase[J]. J Med Chem, 2006, 49: 6027–6036. DOI:10.1021/jm060499t |
| [23] | Yang Y, Severin A, Chopra R, et al. 3, 5-Dioxopyrazolidines, novel inhibitors of UDP-N-acetylenolpyruvyl glucosamine reductase (MurB) with activity against Gram-positive bacteria[J]. Antimicrob Agents Chemother, 2006, 50: 556–564. DOI:10.1128/AAC.50.2.556-564.2006 |
| [24] | Smith CA. Structure, function and dynamics in themur family of bacterial cell wall ligases[J]. J Mol Biol, 2006, 362: 640–655. DOI:10.1016/j.jmb.2006.07.066 |
| [25] | Kouidmi I, Levesque RC, Paradis-Bleau C. The biology of Mur ligases as an antibacterial target[J]. Mol Microbiol, 2014, 94: 242–253. DOI:10.1111/mmi.2014.94.issue-2 |
| [26] | Reck F, Marmor S, Fisher S, et al. Inhibitors of the bacte-rial cell wall biosynthesis enzyme MurC[J]. Bioorg Med Chem Lett, 2001, 11: 1451–1454. DOI:10.1016/S0960-894X(01)00251-7 |
| [27] | Zawadzke LE, Norcia M, Desbonnet CR, et al. Identifica-tion of an inhibitor of the MurC enzyme, which catalyzes an essential step in the peptidoglycan precursor synthesis pathway[J]. Assay Drug Develop Technol, 2008, 6: 95–103. DOI:10.1089/adt.2007.114 |
| [28] | Šink R, Barreteau H, Patin D, et al. MurD enzymes:some recent developments[J]. Biomol Concepts, 2013, 4: 539–556. |
| [29] | Horton JR, Bostock JM, Chopra I, et al. Macrocyclic inhibitors of the bacterial cell wall biosynthesis enzyme Mur D[J]. Bioorg Med Chem Lett, 2003, 13: 1557–1560. DOI:10.1016/S0960-894X(03)00176-8 |
| [30] | Turk S, Kovač A, Boniface A, et al. Discovery of new inhibitors of the bacterial peptidoglycan biosynthesis enzymes MurD and MurF by structure-based virtual screening[J]. Bioorg Med Chem, 2009, 17: 1884–1889. DOI:10.1016/j.bmc.2009.01.052 |
| [31] | Perdih A, Kovač A, Wolber G, et al. Discovery of novel benzene 1, 3-dicarboxylic acid inhibitors of bacterial MurD and MurE ligases by structure-based virtual screening ap-proach[J]. Bioorg Med Chem Lett, 2009, 19: 2668–2673. DOI:10.1016/j.bmcl.2009.03.141 |
| [32] | Tanner ME, Vaganay S, van Heijenoort J, et al. Phosphi-nate inhibitors of the D-glutamic acid-adding enzyme of peptidoglycan biosynthesis[J]. J Org Chem, 1996, 61: 1756–1760. DOI:10.1021/jo951780a |
| [33] | Humljan J, Kotnik M, Boniface A, et al. A new approach towards peptidosulfonamides:synthesis of potential inhibitors of bacterial peptidoglycan biosynthesis enzymes MurD and MurE[J]. Tetrahedron, 2006, 62: 10980–10988. DOI:10.1016/j.tet.2006.08.030 |
| [34] | Kotnik M, Humljan J, Contreras-Martel C, et al. Structural and functional characterization of enantiomeric glutamic acid derivatives as potential transition state analogue inhibitors of MurD ligase[J]. J Mol Biol, 2007, 370: 107–115. DOI:10.1016/j.jmb.2007.04.048 |
| [35] | Humljan J, Kotnik M, Contreras-Martel C, et al. Novel naphthalene-N-sulfonyl-D-glutamic acid derivatives as inhibittors of MurD, a key peptidoglycan biosynthesis enzyme[J]. J Med Chem, 2008, 51: 7486–7494. DOI:10.1021/jm800762u |
| [36] | Simcic M, Hodošček M, Humljan J, et al. NMR and molecular dynamics study of the binding mode of naphtha-lene-N-sulfonyl-D-glutamic acid derivatives:novel MurD ligase inhibitors[J]. J Med Chem, 2009, 52: 2899–2908. DOI:10.1021/jm900117n |
| [37] | Tomašić T, Zidar N, Rupnik V, et al. Synthesis and bio-logical evaluation of new glutamic acid-based inhibitors of MurD ligase[J]. Bioorg Med Chem Lett, 2009, 19: 153–157. DOI:10.1016/j.bmcl.2008.10.129 |
| [38] | Zidar N, TomašićT, Šink R, et al. Discovery of novel 5-benzylidenerhodanine and 5-benzylidenethiazolidine-2, 4-dione inhibitors of MurD ligase[J]. J Med Chem, 2010, 53: 6584–6594. DOI:10.1021/jm100285g |
| [39] | Bratkovič T, Lunder M, Urleb U, et al. Peptide inhibitors of MurD and MurE, essential enzymes of bacterial cell wall biosynthesis[J]. J Basic Microbiol, 2008, 48: 202–206. DOI:10.1002/(ISSN)1521-4028 |
| [40] | Zeng B, Wong KK, Pompliano DL, et al. A phosphinate inhibitor of the meso-diaminopimelic acid-adding enzyme (MurE) of peptidoglycan biosynthesis[J]. J Org Chem, 1998, 63: 10081–10085. DOI:10.1021/jo981895p |
| [41] | Paradis-Bleau C, Lloyd A, Sanschagrin F, et al. Pseudomo-nas aeruginosa MurE amide ligase:enzyme kinetics and peptide inhibitor[J]. Biochem J, 2009, 421: 263–272. DOI:10.1042/BJ20081395 |
| [42] | Guzman JD, Gupta A, Evangelopoulos D, et al. Anti-tubercular screening of natural products from Colombian plants:3-methoxynordomesticine, an inhibitor of MurE ligase of Mycobacterium tuberculosis[J]. J Antimicrob Chemother, 2010, 65: 2101–2107. DOI:10.1093/jac/dkq313 |
| [43] | Shiu WKP, Malkinson JP, Rahman MM, et al. A new plant-derived antibacterial is an inhibitor of efflux pumps in Staphylococcus aureus[J]. Int J Antimicrob Agents, 2013, 42: 513–518. DOI:10.1016/j.ijantimicag.2013.08.007 |
| [44] | Comess KM, Schurdak ME, Voorbach MJ, et al. An ultra efficient affinity-based high-throughout screening process:application to bacterial cell wall biosynthesis enzyme MurF[J]. J Biomol Screening, 2006, 11: 743–754. DOI:10.1177/1087057106289971 |
| [45] | Gu YG, Florjancic AS, Clark RF, et al. Structure-activity relationships of novel potent MurF inhibitors[J]. Bioorg Med Chem Lett, 2004, 14: 267–270. DOI:10.1016/j.bmcl.2003.09.073 |
| [46] | Stamper GF, Longenecker KL, Fry EH, et al. Struc-ture-based optimization of MurF inhibitors[J]. Chem Biol Drug Design, 2006, 67: 58–65. DOI:10.1111/jpp.2006.67.issue-1 |
| [47] | Hrast M, Anderluh M, Knez D, et al. Design, synthesis and evaluation of second generation MurF inhibitors based on a cyanothiophene scaffold[J]. Eur J Med Chem, 2014, 73: 83–96. DOI:10.1016/j.ejmech.2013.11.031 |
| [48] | Baum EZ, Crespo-Carbone SM, Klinger A, et al. A MurF inhibitor that disrupts cell wall biosynthesis in Escherichia coli[J]. Antimicrob Agents Chemother, 2007, 51: 4420–4426. DOI:10.1128/AAC.00845-07 |
| [49] | Baum EZ, Crespo-Carbone SM, Foleno BD, et al. MurF inhibitors with antibacterial activity:effect on muropeptide levels[J]. Antimicrob Agents Chemother, 2009, 53: 3240–3247. DOI:10.1128/AAC.00166-09 |
| [50] | Silver LL. Multi-targeting by monotherapeutic antibacteri-als[J]. Nat Rev Drug Discov, 2007, 6: 41–55. DOI:10.1038/nrd2202 |
| [51] | Silver LL. Polypharmacology as an Emerging Trend in Antibacterial Discovery[M]//Peter JU. Polypharmacology in Drug Discovery. Hoboken:John Wiley and Sons, 2012:167-202. |
| [52] | Mansour TS, Caufield CE, Rasmussen B, et al. Naph-thyltetronic acids as multi-target inhibitors of bacterial peptidoglycan biosynthesis[J]. ChemMedChem, 2007, 2: 1414–1417. DOI:10.1002/(ISSN)1860-7187 |
| [53] | Perdih A, Hrast M, Barreteau H, et al. Benzene-1, 3-dicar-boxylic acid 2, 5-dimethylpyrrole derivatives as multiple inhibitors of bacterial Mur ligases (MurC-MurF)[J]. Bioorg Med Chem, 2014, 22: 4124–4134. DOI:10.1016/j.bmc.2014.05.058 |
| [54] | Perdih A, Hrast M, Pureber K, et al. Furan-based benzene mono-and dicarboxylic acid derivatives as multiple inhibi-tors of the bacterial Mur ligases (MurC-MurF):experimental and computational characterization[J]. J Computer-aided Mol Design, 2015, 29: 541–560. DOI:10.1007/s10822-015-9843-6 |
| [55] | Bax RP, Anderson R, Crew J, et al. Antibiotic resistance-what can we do?[J]. Nat Med, 1998, 4: 545–546. DOI:10.1038/nm0598-545 |
| [56] | Projan SJ, Youngman PJ. Antimicrobials:new solutions badly needed[J]. Curr Opin Microbiol, 2002, 5: 463–465. DOI:10.1016/S1369-5274(02)00364-8 |
| [57] | Silver LL. Challenges of antibacterial discovery[J]. Clin Microbiol Rev, 2011, 24: 71–109. DOI:10.1128/CMR.00030-10 |
 2017, Vol. 52
2017, Vol. 52


