2. 江苏理工学院电气信息工程学院, 生物信息与医药工程研究所, 江苏 常州 213001
2. Institute of Bioinformatics and Medical Engineering, School of Electrical and Information Engineering, Jiangsu University of Technology, Changzhou 213001, China
凝集素(lectin) 一般是指从各种植物、动物中提取出来能够凝集细胞或沉降复合糖质, 非免疫起源的蛋白质或糖蛋白, 很少具有酶活性[1]。食(药) 用真菌中的凝集素主要分布在真菌的菌丝体、菌核、菌盖和菌柄中[2], 凝集素可能在大型真菌的休眠、生长和形态发生中发生变化, 凝集素可能在大型真菌受寄生感染后的形态改变过程中行使一定的功能[3]。目前, 大型真菌的凝集素主要应用于真菌的分类学、胚胎学以及细菌学研究[4]。
根据蛋白氨基酸序列的同源性及其在进化上的关系, 可以将凝集素分为7个家族。其中一个家族为Ⅱ型核糖体失活蛋白(RIP), 即Ⅱ型RIPs, 由两条链组成: A链(RTA) 和B链(RTB), 其中A链结构类似于Ⅰ型RIPs, 具有一般的酶活性, 蛋白的分子质量较大, 约35 kDa左右; B链对半乳糖结构具有特定的凝集素活性[5]。目前所有的研究表明, 已发现的Ⅰ型RIPs对真菌、动物病毒都具有抗菌活性[6], 而仅有部分Ⅱ型RIPs表现出此性质。
食(药) 用真菌中分布着大量的凝集素, 猪苓菌核隶属于多孔菌科的大型真菌, 是我国最重要的药用真菌资源之一, 其生长发育必须依赖蜜环菌提供营养物质; 在猪苓与蜜环菌形成共生关系的过程中, 蜜环菌菌索会侵入猪苓菌核, 诱发猪苓菌核的防御反应。Ⅱ型RIPs作为凝集素家族的一个成员, 探明其在猪苓防御体系中的作用机制是十分重要的。
在本课题组前期构建的猪苓菌核转录组文库中发现1个差异表达的凝集素蛋白家族基因(Ⅱ型核糖体失活蛋白A链基因) 的全长开放阅读框(ORF)。本研究中利用RT-PCR技术克隆得到猪苓菌核中该基因的开放阅读框全长序列, 并就该基因的结构特征和功能进行初步分析, 对该基因在蜜环菌侵染后的转录模式进行了检测, 并构建了猪苓PuRIP基因原核表达载体, 进行了重组蛋白的表达与纯化, 为制备多克隆抗体提供了材料, 以期为进一步揭示Ⅱ型核糖体失活蛋白的功能以及探讨猪苓的防御分子机制奠定基础。
材料与方法猪苓 猪苓菌核于2014年8月采自山西古县党家山村。
总RNA提取及cDNA合成 根据天根生化科技(北京) 有限公司的植物总RNA提取试剂盒说明书提取总RNA, 利用1.2%琼脂糖电泳检测RNA的完整性, 使用分光光度计NanoDrop 2000 (Thermo Scien tific, 美国) 测定RNA浓度。应用M-MLV Reverse Transcriptase kit (艾德莱, 中国) 反转录合成猪苓cDNA第一链并存储于-20 ℃保存备用。
基因的克隆 在本课题组已测得的猪苓转录组数据库中找到1个编码RIP A链蛋白的全长基因unigene序列, 利用primer 3在线软件分别从起始密码子与终止密码子开始设计1对引物: PuRIPP1 5'-ATG ACT CTT TCT CGC GGA AT -3', PuRIPP2 5'-TCA GAA GTA GGC AAG GTA G -3', 用RT-PCR来获取RIP基因的全长开放阅读框。以猪苓cDNA为模板进行PCR, 总反应体系为25 µL, 包括: 0.5 µL dNTPs (10 mmol·L-1)、1 µL cDNA、12.5 µL Phanta Max buffer、0.5 µL Phanta Max Super-Fidelity DNA Polymerase、引物各1 µL (10 mmol·L-1) 和8.5 µL ddH2O。扩增反应条件为: 94 ℃ 5 min; 94 ℃ 30 s, 60 ℃ 30 s, 72 ℃ 90 s, 30个循环; 72 ℃延伸7 min。将获得的目的条带通过TaKaRa Agarose Gel DNA Purification Kit Ver.4.0割胶回收纯化, 与pMD18-T Vector连接后转化DH5α感受态细胞, 经蓝白斑筛选, 挑取白色菌落进行培养, 由苏州金唯智公司进行序列测定。获得测序结果后通过GenBank进行BLAST搜索比对。
生物信息学分析 利用ORF Finder程序(http://www.ncbi.nih.gov/gorf/gorf.htmL) 进行开放阅读框预测, 氨基酸序列在NCBI的保守域数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml) 进行分析以寻找其保守域。利用Blast程序(http://www.ncbi.nlm.nih.gov/blast/) 对该基因进行序列同源性比对, 利用ExPASy的ProtParam工具(http://www.expasy.org/tools/protparam.html) 进行蛋白质理化性质(等电点、分子量) 分析, 采用DNASTAR 6.0进行氨基酸序列比对分析; 借助MEGA 6.0构建系统进化树。
实时荧光定量PCR分析 利用实时荧光定量PCR技术分析RIP A链基因的表达模式, 利用primer 5软件设计引物: qPuRIPP1 5'-CCT CAC CTC AGC ACA CAG TA-3', qPuRIPP2 5'-ATT TCT CCG CTC CCC ATT CA-3', 用猪苓β-tublin基因作为内参基因。分别取被蜜环菌侵染的菌核(CT) 以及未被蜜环菌侵染的菌核(CK), 提取RNA后用M-MLV Reverse Transcriptase kit (艾德莱, 中国) 反转录获得cDNA, 用Roche LightCycler® 480Ⅱ对目的基因的转录水平进行检测。使用SYBR® Premix Ex TaqTM试剂盒(艾德莱, 中国) 进行qPCR反应, 反应体系为15 µL, 其中包括7.5 μL 2× SYBR® Premix ExTaqTM Master Mix、0.5 μL引物(10 μmol·L-1)、2 μL cDNA模板以及4.5 μL ddH2O。反应程序设定如下:预变性95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 32 s, 40个循环, 同时绘制熔解曲线。每个反应重复3次, 实验重复3次。
原核表达载体pET-15b-RIP的构建 以稀释100倍的RIP基因的pMD18-T质粒为模板, 用以下引物进行PCR扩增, 5'-GGT TCC GCG GGA TCC ATG ACT CTT TCT CGC GGA AT -3', P2: 5'-GGC CGC TCG CAT ATG TCA GAA GTA GGC AAG GTA G -3', 划线部分为与pET15b相同的同源臂。PCR反应体系如下: 10×Buffer 2.5 μL、MgCl2 (25 mmol·L-1) 2.0 μL、dNTP (10 mmol·L-1) 0.5 μL、引物1 (10 μmol·L-1) 0.5 μL、引物2 (10 μmol·L-1) 0.5 μL、质粒0.5 μL、DNA polymerase 0.5 μL。PCR反应程序: 94 ℃ 3 min; 94 ℃ 50 s; 60 ℃ 1 min; 72 ℃ 1 min; 30循环; 72 ℃ 10 min。PCR产物经1.8%琼脂糖凝胶电泳, 回收目的条带。pET15b载体质粒分别用BamHI和NdeI进行双酶切, 经琼脂糖凝胶电泳后, 回收目的片段, 在PCR离心管中分别加入: pET-15b酶切回收片段100 ng; 5× In-Fusion HD Enzyme 2.0 μL; 纯化的RIP基因片段50 ng。瞬时离心, 在PCR仪中50 ℃孵育15 min, 然后置于冰上。转化E.coli TOP10感受态细胞, 随机挑选克隆, 提取质粒, 送北京金唯智公司进行测序。阅读框正确的质粒即为融合表达载体pET-15b-RIP。
PuRIP蛋白的原核表达 将含有重组质粒pET-15b-RIP的E. coli BL21 (DE3) 阳性克隆振荡培养过夜, 菌液以1:100比例稀释于含有Amp (100 μg·mL-1) 的LB液体培养基中, 37 ℃培养至OD600 ≈ 0.8时, 加入IPTG至终浓度为0.1 mmol·L-1, 转入15 ℃摇床以200 r·min-1振荡培养24 h, 离心收集菌体。
重组蛋白的纯化 冰水浴超声破碎菌体, 收集上清液。利用Ni-NTA His-band Colum纯化蛋白, 用10倍柱体积的10 mmol·L-1咪唑平衡Ni-NTA His-band Colum, 将裂解上清液上柱、淋洗, 上样完毕后, 依次用10倍柱体积的咪唑(10、20、50、80和100 mmol·L-1) 过柱洗去杂蛋白。用3倍柱体积的300 mmol·L-1咪唑洗脱目的蛋白, 收集至2 mL离心管中。利用SDS-PAGE电泳及后续的Western blotting分析蛋白的纯度。
Western blotting鉴定表达蛋白 按照Western blotting方法, 将纯化的蛋白进行SDS-PAGE电泳, 200 mA恒流转移2 h到PVDF膜上, 1×TBST洗膜3次, 每次10 min, 用5%的脱脂牛奶4 ℃低温封闭4 h, 用鼠源的His单克隆抗体于4 ℃孵育过夜。次日, 用1×TBST洗膜3次, 每次10 min, 鼠二抗(1:1 000) 室温孵育1 h后, 用1×TBST洗膜3次, 每次10 min, ECL显色, 化学发光成像仪(Bio-Rad Gel Doc XR+) 成像, 并保存。
结果 1 猪苓RIP基因的克隆以未被蜜环菌侵染的猪苓菌核的cDNA为模板, 利用上文中的引物通过RT-PCR方法获得猪苓的RIP基因片段, 目的片段经回收、克隆、测序后, 分离得到猪苓RIP基因完整的ORF序列(图 1)。与转录组中拼接的序列进行比对后, 发现其与文库中的unigene序列的ORF序列一致, 因此可以证明已成功获得RIP基因的全长ORF。BLAST分析表明其为RIP家族基因的同源基因。
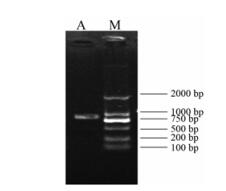
|
Figure 1 PCR product of RIP protein gene. A: RIP gene; M: DL2000 |
ProtParam程序预测RIP基因编码氨基酸序列分子质量为32.33 kDa, 理论pI值为5.58。推测该蛋白分子式为C1457H2165N379O435S12。核苷酸序列比对结果表明, 该基因核苷酸编码的氨基酸序列与硬柄小皮伞(Marasmius oreades) 同源性为52%, 与宽鳞多孔菌(Polyporus squamosus) 同源性为45%;将推测的氨基酸序列提交到NCBI的保守域数据库(Conserved Domain Database, CDD) 进行搜索, 结果如图 2所示, 该基因的核苷酸序列具有RICIN超家族蛋白的保守结构域。推测克隆的该基因为猪苓菌核的RIP基因。NCBI登陆号为: KU179190, 用SignalP 4.1 Server检测发现该蛋白无信号肽区域(图 3)。用TMHMM 2.0分析发现该蛋白并无跨膜区域, 但是该蛋白定位于细胞膜外, 属于胞外蛋白(图 4)。
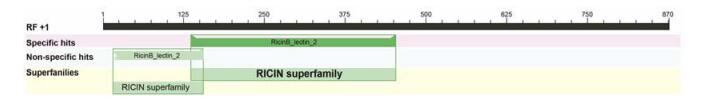
|
Figure 2 Conserved protein domains of RIP protein |
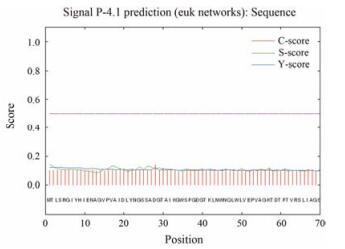
|
Figure 3 Signal peptide analysis of the RIP |
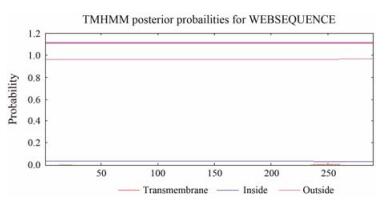
|
Figure 4 The transmembrane domain of RIP analysis by TMHMM |
利用DNASTAR 6.0软件将此基因编码的蛋白序列与其他真菌RIP蛋白序列进行比对表明, 猪苓中的凝集素蛋白基因与其他真菌中的RIP蛋白具有很高的相似度(图 5)。

|
Figure 5 Multiple sequence alignment of RIP and RIP proteins from other fungi. The amino acids in the dark were the same |
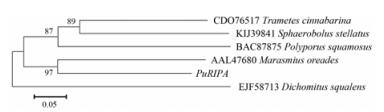
为了研究RIP基因编码蛋白的分子进化关系, 根据猪苓凝集素推导的氨基酸序列及其他真菌凝集素的氨基酸序列构建了系统进化树, 所用的软件是ClusterX 2.0及MEGA 6.0。结果如图 6所示, 所建的系统进化树主要分为两大分支, 所分离得到的猪苓RIP蛋白与硬柄小皮伞(Marasmius oreades) RIP蛋白亲缘关系最近。污叉丝孔菌(Dichomitus squalens) RIP蛋白作为外类群。
|
Figure 6 Phylogenetic relationship of RIP in different fungal species. The number at each branch points is the percentage supported by bootstrap. Bar, 5% sequence divergence |
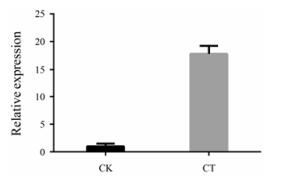
通过实时定量PCR检测RIP基因在不同猪苓菌核部位的转录水平。结果表明, RIP基因在未被蜜环菌侵染及被蜜环菌侵染的猪苓菌核部位均有表达。荧光定量PCR结果表明, 该基因在被蜜环菌侵染部位的表达量要显著高于未被蜜环侵染的猪苓菌核, 为未被侵染部分的17.96倍(图 7)。
|
Figure 7 qRT-PCR analysis of the RIP protein expression. CK: Controlled medullar tissue; CT: Treated medullar tissue |
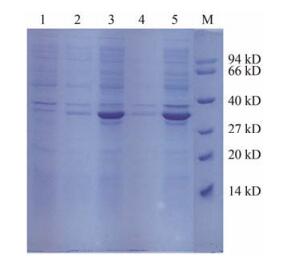
将表达载体pET15b转入BL21 (DE3) 中作为对照, 同时诱导含有pET15b-RIP和pET15b的BL21 (DE3)。在15 ℃摇床中以200 r·min-1振荡培养24 h后, 收集菌体, 菌体超声破碎后, 分别收集上清液和沉淀, 经10% SDS-PAGE电泳分析。如图 8所示, 含pET15b空载体的质粒无特异蛋白出现, 含有重组质粒pET15b-RIP的菌体经IPTG诱导后, 在32 kDa处出现1条特异蛋白条带。而且, 在菌体沉淀中几乎看不到特异蛋白的出现, 说明所表达的蛋白几乎都存在于上清液中。
|
Figure 8 Expression profile analysis of His-PuRIPA fusion protein by SDS-PAGE. M: Low molecular protein maker; 1: E. coli BL21 (DE3) transformed with pET-15b; 2: None IPTG-induced E. coli BL21 (DE3) transformed with pET15b-RIP-vector; 3: The soluble expressed proteins of pET15b-RIP-vector induced with 0.1 mmol·L-1 IPTG 24 h at 15 ℃; 4: Insoluble expressed proteins of pET15b-RIP-vector induced with 0.1 mmol·L-1 IPTG 24 h at 15 ℃; 5: The total expressed proteins of pET15b-RIP-vector induced with 0.1 mmol·L-1 IPTG 24 h at 15 ℃ |
重组工程菌经诱导表达, 超声破碎菌体后收集上清液, 上清液经Ni-NTA His-band Colum纯化后, 纯化后的His-PuRIPA融合蛋白, 待使用Western blotting方法检测。
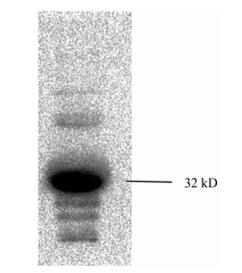
6 重组蛋白的鉴定为了更准确鉴定所纯化的蛋白是His-PuRIPA的融合蛋白, 利用His标签蛋白的相应抗体进行检测。经检测证实该融合蛋白可与His单克隆抗体发生特异性结合, 结果如图 9所示。大约在32 kDa位置有清晰的目的条带。同时, 在Western blotting的结果中出现了许多非特异性条带, 可能是由于二抗试剂的非特异结合所造成的结果。
|
Figure 9 His-PuRIPA fusion protein verification by Western blotting. His-PuRIPA fusion protein marked with 32 kDa |
凝集素蛋白起源古老, 在漫长的进化史中, 编码类凝集素的基因不断发生基因复制事件[7]。因此, 凝集素具有多种生物学功能。本研究从野生猪苓菌核中克隆到RIP A链基因, 并对该基因的序列和表达模式进行了比较详细的研究。
本研究利用RT-PCR技术, 扩增得到了猪苓菌核Ⅱ型核糖体失活蛋白(RIP) A链基因的全长序列。经与NCBI数据库进行BlastX分析, 它与硬柄小皮伞(Marasmius oreades)、宽鳞多孔菌(Polyporus squamosus) 同源性分别为52%和45%。
猪苓Ⅱ型核糖体失活蛋白由290个氨基酸残基组成, 其不具有信号肽及跨膜结构, 其分子量大小为32.33 kDa, 与之前文献[8]报道的Ⅱ型核糖体失活蛋白的性质一致。
Ⅱ型核糖体失活蛋白在植物中研究较多, 有研究对蓖麻的全基因数据组进行搜索, 结果发现其基因组中含有7个Ⅱ型RIP基因[9]。通过搜索水稻的全基因组发现水稻的基因组中含有31个Ⅰ型RIP基因[10]。不同RIP基因家族中的基因, 其表达常常表现出组织特异性和发育阶段的特异性, 并且可应答不同的胁迫环境, 参与植物体的抗逆境过程。有研究表明, 假眼小绿叶蝉取食茶树叶的过程中, 可以诱导其Ⅱ型RIP基因的表达, 并且随着取食时间的延长, RIP基因的表达量逐渐增加[11]。烟草在生长过程中常常受到草花叶病毒(tobacco mosaic virus, TMV) 的侵染, 但将麻疯树中叶子中的新型RIPs在烟草中过表达后, 可以有效增加烟草对TMV、水稻纹枯病菌(Rhizoctonia solani) 的抗性, 减少TMV和其他真菌对其的侵染[12]。本研究利用定量qPCR技术检测猪苓Ⅱ型核糖体失活蛋白基因在猪苓经蜜环菌侵染后表达趋势。结果显示:在蜜环菌侵染时的猪苓菌核中, Ⅱ型核糖体失活蛋白基因表达量都呈现很高趋势, 显著大于未被蜜环菌侵染的猪苓菌核。说明蜜环菌侵染造成的机械伤害显著诱导RIP基因的表达, 提示该基因可能参与了猪苓防御过程。此外, 高质量His-PuRIP融合蛋白的获得, 为今后PuRIP多克隆抗体制备提供了科学依据; Ⅱ型核糖体失活蛋白基因的克隆与表达分析以及蛋白的获得, 不仅为猪苓凝集素基因家族增添了新成员, 而且为进一步研究Ⅱ型核糖体失活蛋白基因在猪苓抗逆中的作用奠定了基础。
| [1] | Sommer R, Exner TE, Titz A. A biophysical study with carbohydrate derivatives explains the molecular basis of monosaccharide selectivity of the Pseudomonas aeruginosa Lectin LecB[J]. PLoS One, 2014, 9: e112822.. DOI:10.1371/journal.pone.0112822 |
| [2] | Wohlschlager T, Butschi A, Grassi P, et al. Methylated glycans as conserved targets of animal and fungal innate defense[J]. Proc Natl Acad Sci USA, 2014, 111: E2787–E2796. DOI:10.1073/pnas.1401176111 |
| [3] | Huysamen C, Brown GD. The fungal pattern recognition receptor, Dectin-1, and the associated cluster of C-type lectinlike receptors[J]. FEMS Microbiol Lett, 2009, 290: 121–128. |
| [4] | Khan F, Khan MI. Fungal lectins:current molecular and biochemical perspectives[J]. Int J Biol Chem, 2011, 5: 1–20. DOI:10.3923/ijbc.2011.1.20 |
| [5] | Mundy J, Leah R, Boston R, et al. Genes encoding ribosomeinactivating proteins[J]. Plant Mol Biol Rep, 1994, 12: S60–S62. DOI:10.1007/BF02671573 |
| [6] | Parikh BA, Tumer NE. Antiviral activity of ribosome inactivating proteins in medicine[J]. Mini Rev Med Chem, 2004, 4: 523–543. DOI:10.2174/1389557043403800 |
| [7] | Virgilio MD, Lombardi A, Caliandro R, et al. Ribosomeinactivating proteins:from plant defense to tumor attack[J]. Toxins, 2010, 2: 2699–2737. DOI:10.3390/toxins2112699 |
| [8] | Sharma N, Park SW, Vepachedu R, et al. Isolation and characterization of an RIP (ribosome-inactivating protein)-like protein from tobacco with dual enzymatic activity[J]. Plant Physiol, 2004, 134: 171–181. DOI:10.1104/pp.103.030205 |
| [9] | Leshin J, Danielsen M, Credle JJ, et al. Characterization of ricin toxin family members from Ricinus communis[J]. Toxicon, 2010, 55: 658–661. DOI:10.1016/j.toxicon.2009.09.002 |
| [10] | Jiang SY, Ramamoorthy R, Bhalla R, et al. Genome-wide survey of the RIP domain family in Oryza sativa and their expression profiles under various abiotic and biotic stresses[J]. Plant Mol Biol, 2008, 67: 603–614. DOI:10.1007/s11103-008-9342-4 |
| [11] | Yuan HY, Ma N, Yang HM, et al. Cloning and expression of Ⅱtype ribosome inactivating protein genes CsRIP1 and CsRIP2 from Camellia sinensis[J]. Sci Sil Sin (林业科学), 2015, 51: 147–153. |
| [12] | Huang MX, Hou P, Wei Q, et al. A ribosome-inactivating protein (curcin 2) induced from Jatropha curcas can reduce viral and fungal infection in transgenic tobacco[J]. Plant Growth Regul, 2008, 54: 115–123. DOI:10.1007/s10725-007-9234-7 |
 2017, Vol. 52
2017, Vol. 52


