铁皮石斛(Dendrobium officinale Kimura et Migo)为兰科石斛属植物, 具有生津益胃、润肺止咳、滋阴清热、抗癌防老、增强免疫力等多种药用功效, 是我国传统的名贵中药材[1], 主要分布于云南、贵州、广西、湖南、江西等地区[2]。传统中药材主要来源于铁皮石斛野生资源, 但野生铁皮石斛对生长环境要求十分严格, 自然条件下繁殖率低, 再加上人类掠夺式的采挖, 导致铁皮石斛野生资源急剧紧缺[3, 4]。为了保护和满足市场需求, 铁皮石斛人工种植规模不断扩大, 现已成为解决铁皮石斛资源紧缺的主要途径[5]。目前, 在人工种植过程中, 铁皮石斛种质来源不一, 有些直接来自野生居群, 有些则来自野生居群杂交后代。复杂的种源导致铁皮石斛品质差异较大, 铁皮石斛优质种源的创新迫在眉睫。在改善和培育优质铁皮石斛过程中, 明确种质来源和选择优质亲本十分重要。目前, 传统形态学方法很难将铁皮石斛野生居群、亲本及其杂交后代严格鉴别开来[6]。因此, 亟需建立一种有效可靠的方法用于铁皮石斛野生居群的鉴定和杂交种质的鉴别与质量控制研究。
目标区域扩增多态性(target region amplification polymorphism, TRAP)是一种新型DNA分子标记, 最早由Hu和Vick[7]于2003年提出, 主要根据目的基因的cDNA或EST序列设计固定引物, 并结合随机引物对目标区域进行多态性扩增, 从而产生围绕目的基因的多态性标记, 极易将性状与分子标记相联系。由于TRAP标记是与目的基因紧密连锁的分子标记, 更适合用于基因型与表型之间连锁关系的分析, 可以极大地提高基因型与表型选择的一致性, 被广泛应用于居群间的遗传多样性、亲缘关系、居群的鉴定及杂交种质的鉴别研究[8-11]。然而, 有关TRAP分子标记用于铁皮石斛居群间的鉴定与杂交种质的鉴别尚未见报道。
由于铁皮石斛大量多糖含量及微量联卞类、酚酸类、菲类、氨基酸、生物碱含量是评估其药用价值的重要指标[12]。然而, 不同居群间铁皮石斛多糖和生物碱含量存在较大差异, 探究铁皮石斛多糖和生物碱相关基因的多态性差异对明确其药用品质差异具有重要作用[13, 14]。由于磷酸烯醇式丙酮酸羧化酶和尿苷二磷酸葡萄糖焦磷酸化酶均是多糖合成代谢的关键酶, 控制多糖类次生代谢产物的合成[15, 16]。此外, 苯丙氨酸解氨酶是生物碱合成过程中苯丙烷类物质代谢反应的限速酶之一, 该酶的活性间接影响铁皮石斛中生物碱含量[17]。因此, 本实验根据磷酸烯醇式丙酮酸羧化酶基因(PEPC)、尿苷二磷酸葡萄糖焦磷酸化酶基因(UGP)和苯丙氨酸解氨酶基因(PAL)分别设计3对固定引物, 并结合18个随机引物共组合成54对TRAP引物, 旨在建立一种稳定性强、重复性好的TRAP分子标记用于铁皮石斛野生居群间的鉴定与杂交后代种质的鉴别, 提高种质质量控制的效率, 为改善铁皮石斛品质提供理论依据。
材料与方法材料 本实验所用材料采自云南广南(GN)、江西南丰(NF)、贵州三都(SD)、广西西林(XL)、湖南邵阳(SY)和广西天峨(TE)产地的野生铁皮石斛(表 1), 上述材料均由南京师范大学丁小余教授鉴定, 保存于南京师范大学生命科学学院植物资源与环境研究所。
| Table 1 Location and sampling size of fourD. officinale used in this study |
杂交后代的获得:以来自GN铁皮石斛样本为母本, NF和SD为父本, 得到正交H1;以NF和SD铁皮石斛样本为母本, GN为父本, 得到反交H2。将种子接种到种子萌发培养基中, 进行组织培养。当杂交后代幼苗长至4~5 cm高时, 分别采集12株杂交后代幼苗的叶片和亲本幼嫩叶片用硅胶干燥, -20 ℃保存。
基因组DNA的提取与鉴别 采用改良的CTAB法[18]提取铁皮石斛叶片基因组DNA, 用1%琼脂糖凝胶电泳鉴别DNA的完整性, 再利用紫外分光光度计鉴别DNA浓度, 并将其浓度稀释至20 ng·μL-1, -20 ℃保存备用。
引物设计 采用引物设计软件Primer 5.0[19], 根据铁皮石斛多糖与生物碱合成代谢的相关途径, 选择多糖合成相关基因磷酸烯醇式丙酮酸羧化酶(PEPC)和尿苷二磷酸葡萄糖焦磷酸化酶(UGP), 以及生物碱合成相关基因苯丙氨酸解氨酶(PAL)作为目的基因, 根据其cDNA序列, 设计3对固定引物。18个随机引物参考Li和Quiros[20]的设计, 引物均由上海生工生物技术公司合成。引物序列见表 2、3。
| Table 2 Information of the TRAP-fixed primers |
| Table 3 Information of the TRAP-random primers |
TRAP-PCR反应体系的优化 采用L16(44)正交实验设计对dNTP、Mg2+、Taq DNA聚合酶、固定引物与随机引物浓度配比进行4因素4水平筛选, 建立TRAP-PCR最佳反应体系, 方案设计见表 4、5。
| Table 4 Factors and levels of TRAP-PCR system. Note: Arbitrary primers: fixed primers (1 to 4) are 0.5:1, 0.75:1, 1:1, 1.25:1 (2 μL of fixed primers, increase the concentration of arbitrary primers) |
| Table 5 Orthogonal design for TRAP-PCR [L16(44)] |
模板DNA浓度的优化 采用上述最佳反应体系, 以PEPC-F/Em1为TRAP引物, 将20 µL反应体系中的模板DNA浓度分别设置8个浓度梯度, 模板DNA加入量分别为5、10、20、30、40、50、60和70 ng, 对模板浓度进行优化。
引物筛选 利用上述优化后的TRAP-PCR反应体系, 以GN和NF两亲本及其杂交后代DNA为模板, 用于筛选出扩增条带数多, 重复性好, 并能明显区分不同居群、亲本及其杂交后代的多态性引物, 用于居群间的鉴定和杂交后代的鉴别。固定与随机引物组合方式为:上游固定引物与下游随机引物、下游固定引物与上游随机引物随机组合, 共形成54对可供筛选的TRAP引物。
亲本及其杂交后代的TRAP扩增反应 利用优化后的TRAP反应体系和筛选出的TRAP引物对铁皮石斛亲本及其杂交后代基因组DNA进行多态性扩增。参照Wang等[11]扩增程序, 本实验扩增程序为94 ℃预变性5 min, 开始5个循环(94 ℃变性45 s, 35 ℃退火45 s, 72 ℃延伸1 min), 再进行35个循环(94 ℃变性45 s, 每对固定引物的最佳退火温度退火45 s, 72 ℃延伸1 min), 最后72 ℃延伸10 min, 反应结束后, 扩增产物置于4 ℃冰箱中保存。采用琼脂糖凝胶电泳检测模板DNA浓度的优化结果, 采用8%非变性聚丙烯酰胺凝胶电泳对扩增条带进行分离, 并采用银染法进行鉴别[21], 用凝胶成像系统观察结果并进行拍照记录。
数据分析 根据8%非聚丙烯酰胺凝胶电泳图, 在相同位置上有清晰条带的记为1, 无条带或条带模糊的记为0, 将得到二元矩阵作为TRAP分析的原始数据。首先, 根据记录结果, 统计亲本条带在杂交后代H1和H2中的分布情况和比例, 包括双亲共有带、偏母型带、偏父型带、杂合型带、双亲缺失型带、母本缺失型带和父本缺失型带。其次, 使用PopGen3.2分析软件计算亲本与后代间的遗传距离和遗传一致度[22]。最后使用NTSYS-pc2.1软件, 根据计算出来的遗传一致度, 进行UPGMA聚类分析[23]。
结果 1 TRAP-PCR的扩增TRAP-PCR体系优化的正交实验结果如图 1所示, 参照Li等[24]正交设计直观分析法, 对扩增条带特异性、条带数目以及条带强弱进行分析, 将16个正交设计处理组分为1~9分进行计分统计, 分数越高, 表示扩增效果越好。1~16泳道分数分别为7、9、5、8、7、3、6、4、3、7、4、7、4、5、9和7, 其中处理组2和15得分最高, 扩增条带清晰, 数目较多, 扩增效果最好。但2和15处理组中所用dNTP、Mg2+和引物用量不同, 综合考虑扩增效果和成本, 最终选择2号处理组为TRAP-PCR最佳反应体系, 即20 µL反应体系中包括: 10xbuffer 2.0 μL、dNTP (2.5 mmol·L-1) 1.0 μL、Mg2+ (25 mmol·L-1) 1.2 μL、Taq DNA聚合酶0.9 U和引物(2 mmol·L-1)浓度比为0.75:1 (即随机引物1.5 μL, 固定引物2 μL)。通过验证实验, 所获得的TRAP最佳反应体系扩增效果更好, 可用于铁皮石斛杂交后代鉴别研究。
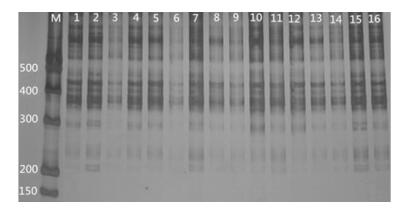 | Figure 1 The result of TRAP-PCR orthogonal design. M: DL 500 DNA Marker; Lane 1−16: Treatments of orthogonal design |
采用最佳TRAP-PCR反应体系, 对模板DNA浓度进行优化(图 2)。扩增结果显示, 当模板DNA加入量为5 ng时, 只扩增出一个条带且亮度暗; 当模板加入量为10 ng时, 基本能扩增出主要条带, 但亮度较暗; 当模板加入量为20、30、40、50、60、70 ng时, 均能扩增出有效条带, 彼此间差异不大, 可知在一定范围内DNA浓度对PCR扩增没有显著影响。因此, 在保证扩增条带稳定的基础上, 为了节省模板DNA和方便取样, 本研究选用20 ng DNA加入量为适宜的模板浓度。
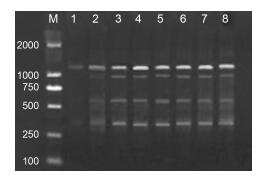 | Figure 2 The results of TRAP-PCR with different concentrations of template DNA. The adding template DNA concentrations (1 to 8) are 5, 10, 20, 30, 40, 50, 60, 70 ng |
经过大量的引物筛选, 从54对引物中筛选出7对TRAP引物, 即PEPC-F/Em1、PEPC-F/Em6、PEPC-R/Me2、PEPC-R/Me6、UGP-F/Em2、PAL-F/Em7和PAL-R/ Me8, 可将铁皮石斛6个野生居群较好地鉴别开, 且具有较好的重复性, 其中引物PEPC-F/Em1和UGP-F/ Em2对各个居群的扩增结果见图 3。
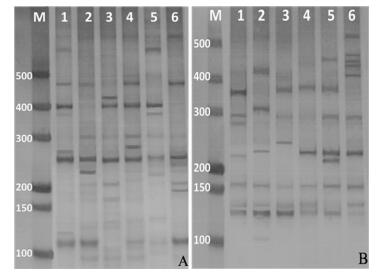 | Figure 3 Amplification results of TRAP primers in six popula tions. A: PEPC-F/Em1; B: UGP-F/Em2; Lane 1−6: Populations of GN, NF, SD, XL, SY, TE. GN: Guangnan; NF: Nanfeng; SD: Sandu; XL: Xilin; SY: Shaoyang; TE: Tian'e |
采用筛选出的7对引物对4种杂交组合的亲本及杂交后代进行扩增, 分别扩增出188、178、188和177个条带, 共得到7种类型的条带分布, 分别为双亲共有型、偏母型、偏父型、双亲缺失型、母本缺失型、父本缺失型和杂合型, 这些条带分布类型可将亲本及其杂交后代有效鉴别开(表 6, 图 4)。
| Table 6 Statistical analysis of TRAP bands of hybrid |
 | Figure 4 Amplification results of PAL-F1E7 primer in parents (GN and NF) and their hybrids. A: The result of H1(GN♀NF♂); Lane 1−12: Samples in H1. B: The result of H2(NF♀GN♂); Lane 1−12: Samples in H2; ↗ Right arrow the markers of father; ↖ Left arrow the makers of mother; a b c show the unique markers of hybrids |
在杂交组合GN×NF中, 铁皮石斛6个野生居群间, 居群SD和SY的遗传一致性最大, 为0.644 7, 亲缘关系最近; 亲缘关系最远的是居群GN和TE, 遗传一致性为0.538 5。在亲本及杂交后代间, 杂交后代之间的遗传一致度最大(0.854 4), 遗传距离最小(0.157 3), 两亲本间的遗传一致度最小, 为0.610 6, 亲缘关系最远(表 7)。
| Table 7 Nei's genetic identity (above diagonal) and genetic distance (below diagonal) of the hybrids and their parents (GN and NF) generated. GN: Guangnan; NF: Nanfeng; SD: Sandu; XL: Xilin; SY: Shaoyang; TE: Tian'e |
在杂交组合GN×SD中, 6个铁皮石斛野生居群间, 亲缘关系最近的是居群SY和NF (0.645 5);居群SD和XL的遗传一致度最小, 为0.483 9, 彼此间亲缘关系最远。在亲本及杂交后代中, 杂交后代间的遗传一致度最大(0.828 2), 遗传距离则最小(0.188 6), 两亲本的遗传一致度最小(0.544 3), 亲缘关系最远(表 8)。
| Table 8 Nei's genetic identity (above diagonal) and genetic distance (below diagonal) of the hybrids and their parents (GN and SD) generated |
聚类分析结果显示, 在4种杂交组合中, 当以GN居群铁皮石斛作为亲本时, 杂交后代均先与亲本GN聚为一支, 再与另一亲本聚为一支, 且6个铁皮石斛野生居群间差异较大, 能够明显区分开(图 5)。
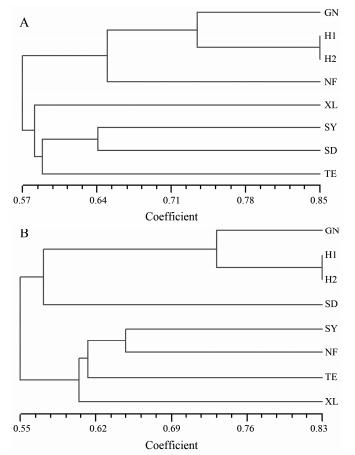 | Figure 5 Dendrogram of the hybrids and their parents by cluster analysis (UPGMA). A: The hybrids and their parents (GN and NF); B: The hybrids and their parents (GN and SD) |
随着分子生物学迅速发展, 分子标记技术已广泛用于石斛属植物的居群鉴定及种质鉴别[25-29]。Ding等[29]利用RAPD分子标记、Xie等[26]采用SSR分子标记、Li等[28]利用AFLP分子标记对铁皮石斛不同野生居群进行有效鉴别。这些分子标记虽然可用于种质鉴别, 但其扩增的多态性片段均是随机分布于基因组中, 基因水平上的多态性与表型差异性之间的关联性较低[30]。然而, TRAP标记是对目标基因进行多态性扩增, 从而获得与目的基因紧密连锁的分子标记, 更适合用于基因型与表型之间连锁关系的分析, 有助于优良品质的保存与控制, 已成功应用于居群亲缘关系、居群间的鉴别、种质鉴别等方面的研究[8]。
Hu等[8]根据编码富含亮氨酸重复(LRR)类似蛋白基因序列, 利用TRAP分子标记成功鉴别出向日葵属16个野生种及其2个杂交种。Wang等[11]利用TRAP分子标记对4个产地的丹参进行鉴别, 筛选出6对引物能够有效地将4个不同产地的丹参区分开, 为丹参的鉴别提供分子水平上的证据。本实验中, 根据铁皮石斛多糖合成途径中PEPC和UGP, 以及生物碱合成代谢过程中PAL开发TRAP分子标记, 确定了铁皮石斛TRAP反应的最佳条件, 保证了在此条件下TRAP分子标记的稳定性和可重复性[29]。利用筛选出的7对TRAP引物可将铁皮石斛的6个野生居群进行准确鉴别, 对铁皮石斛野生资源的利用进行严格控制, 选择优质居群进行人工培育, 更好地保护好珍稀濒危铁皮石斛的野生资源。本实验所采用的铁皮石斛野生居群的个体材料经过重复、稳定的实验体系验证, 基本保持一致。
杂交育种是培育优质新品种的主要育种手段, 而选配亲本是杂交育种的核心内容[31]。本实验通过TRAP分析, 在杂交后代中均能检测到父母特征条带和新产生条带, 能够有效地将亲本及其杂交后代鉴别开来。由于不同亲本基因在后代基因库中所占比例不同, 双亲对后代群体的遗传贡献是不均等的[32]。本实验中, 正交组合中子代先与母本聚类, 再与父本聚类; 而反交组合中子代先与父本聚类后再与母本聚类, 两者共同体现了子代与云南广南(GN)铁皮石斛的亲缘关系较近。这可能是因为云南广南居群的铁皮石斛遗传给后代的遗传物质较多, 也可能是遗传基因重组、缺失、插入以及突变等原因所致[33]。因此, 在杂交育种中, 应重视理想优质亲本的选择与控制。本实验利用筛选出的7对TRAP引物对亲本及其杂交后代进行鉴别, 实现了杂交后代的早期鉴定和提高种质质量控制的效率, 为铁皮石斛优质亲本的选择提供理论依据。
由于不同居群铁皮石斛多糖和生物碱含量存在较大差异, 探究铁皮石斛多糖和生物碱相关基因的多态性差异对明确其主要药用成分差异具有重要作用[13, 14]。而在多糖合成过程中, 磷酸烯醇式丙酮酸羧化酶和尿苷二磷酸葡萄糖焦磷酸化酶均是多糖合成代谢的关键酶[15, 16]; 生物碱合成代谢途径中, 苯丙氨酸解氨酶是苯丙烷类物质代谢反应的限速酶之一, 该酶的活性间接影响铁皮石斛中生物碱含量[17]。基因水平上的多态性差异可以反映相关酶的表达差异[34], 因此, 根据PEPC、UGP和PAL开发TRAP分子标记, 从而产生围绕目的基因的多态性片段, 从而间接反映多糖、生物碱等药用成分的含量差异, 可有效用于铁皮石斛杂交种质的质量控制。本实验采用的TRAP分子标记不仅能够将铁皮石斛不同野生居群鉴别开, 还能有效用于杂交后代的早期鉴别, 提高良种选育的效率。该标记所需材料较少, 鉴别结果可靠、稳定, 操作简单、重复性好, 有利于对实验材料进行快速鉴别[35]。
| [1] | Chinese Pharmacopoeia Committee. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. Part 1. Beijing:China Medical Science Press, 2015:282-283. |
| [2] | Tsi ZH. Flora of China[M]. Beijing: Science Press, 1999 . |
| [3] | Vellupillai M, Swarup S, Goh CJ. Histological and protein changes during early stages of seed germination in the Orchid, Dendrobium crumenatum[J]. J Hort Sci, 1997, 72 :941–948. |
| [4] | Ding G, Zhang D, Ding X, et al. Genetic variation and conservation of the endangered Chinese endemic herb Dendrobium officinale based on SRAP analysis[J]. Plant Syst Evol, 2008, 276 :149–156. DOI:10.1007/s00606-008-0068-1 |
| [5] | Si JP, Yu QX, Song XS, et al. Artificial cultivation modes for Dendrobium officinale[J]. China J Chin Mater Med (中国中药杂志), 2013, 38 :481–484. |
| [6] | Ding XY, Xu LS, Wang ZT, et al. Studies on population difference of Dendrobium officinale Ⅰ differences in morphological structure[J]. Chin Tradit Herb Drugs (中草药), 2001, 32 :828–831. |
| [7] | Hu J, Vick BA. Target region amplification polymorphism:a novel marker technique for plant genotyping[J]. Plant Mol Biol Rep, 2003, 21 :289–294. DOI:10.1007/BF02772804 |
| [8] | Hu JG, Miller J, Xu S, et al. TRAP (target region amplification polymorphism) technique and its application to crop genomics[C]//Plant and Animal Genome VX Conference Abstracts. San Diego:Plant and Animal Genome XⅡ Conference, 2004:47. |
| [9] | Hu J, Mou B, Vick BA. Genetic diversity of 38 spinach (Spinacia oleracea L.) germplasm accessions and 10 commercial hybrids assessed by TRAP markers[J]. Genet Resour Crop Evol, 2007, 54 :1667–1674. DOI:10.1007/s10722-006-9175-4 |
| [10] | Carmo CD, Santos DB, Alves LB, et al. Development of TRAP (target region amplification polymorphism) as new tool for molecular genetic analysis in Cassava[J]. Plant Mol Biol Rep, 2015, 33 :1953–1966. DOI:10.1007/s11105-015-0887-5 |
| [11] | Wang QH, Zhang BL. TRAP analysis of Salvia miltiorrhiza Bge from different places of production[J]. Acta Pharm Sin (药学学报), 2009, 44 :927–930. |
| [12] | Zhang GN, Bi ZM, Wang ZT, et al. Advances in studies on chemical constituents from plants of Dendrobium Sw[J]. Chin Tradit Herb Drugs (中草药), 2003, 34 :5–8. |
| [13] | Zhou GF, Lv GY. Analysis of monosaccharide compositions and contents in polysaccharide of Dendrobium officinale from different sources and growth years by pre-column derivatization-HPLC[J]. Chin Pharm J (中国药学杂志), 2011, 46 :626–629. |
| [14] | Zhu Y, Zhang A, Bowei HE, et al. Quantitive variation of total alkaloids contents in Dendrobium officinale[J]. China J Chin Mater Med (中国中药杂志), 2010, 35 :2388–2391. |
| [15] | Hatch MD. C4 photosynthesis:a unique blend of modified biochemistry, anatomy and ultrastructure[J]. Biochim Biophys Acta, 1987, 895 :81–106. DOI:10.1016/S0304-4173(87)80009-5 |
| [16] | Eimert K, Villand P, Kilian A, et al. Cloning and characterization of several cDNAs for UDP-glucose pyrophosphorylase from barley (Hordeum vulgare) tissues[J]. Gene, 1996, 170 :227–232. DOI:10.1016/0378-1119(95)00873-X |
| [17] | Facchini PJ, Johnson AG, Poupart J, et al. Uncoupled defense gene expression and antimicrobial alkaloid accumlation in elicited opium poppy cell cultures[J]. Plant Physiol, 1996, 111 :687–697. DOI:10.1104/pp.111.3.687 |
| [18] | Doyle JJ. Isolation of plant DNA from fresh tissue[J]. Focus, 1990, 12 :13–15. |
| [19] | Lalitha S. Primer Premier 5[J]. Biotech Software Internet Rep, 2000, 1 :270–272. DOI:10.1089/152791600459894 |
| [20] | Li G, Quiros CF. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J]. Theor Appl Genet, 2001, 103 :455–461. DOI:10.1007/s001220100570 |
| [21] | Benbouza H, Jacquemin JM, Baudoin JP, et al. Optimization of a reliable, fast, cheap and sensitive silver sraining method to detect SSR markers in polyacrylamide gels[J]. Biotechnol Agron Soc Environ, 2006, 10 :77–81. |
| [22] | Yeh FC, Yang RC, Boyle TBJ, et al. POPGENE 3.2:The user-friendly shareware for population genetic analysis[M].3.2 ed. Edmonton: University of Alberta, 1999 . |
| [23] | Rohlf FJ. NTSYS-pc:Numerical taxonomy and multivariate analysis system, version 2.1[EB/OL]. New York:Applied Biostatistics Inc., 2000. http://www.exetersoftware.com/cat/ntsyspc/ntsyspc.html. |
| [24] | Li YX, Li TH. Optimization of SSR reaction system of peach[J]. J Chin Agric Univ (中国农业大学学报), 2005, 10 :57–61. |
| [25] | Ding G, Li X, Ding X, et al. Genetic diversity across natural population of Dendrobium officinale, the endangered medicinal herb endemic to China, revealed by ISSR and RAPD markers[J]. Russ J Genet, 2009, 45 :327–334. DOI:10.1134/S1022795409030119 |
| [26] | Xie ML, Hou BW, Han L, et al. Development of microsatellites of Dendrobium officinale and its application in purity identification of germplasm[J]. Acta Pharm Sin (药学学报), 2010, 45 :667–672. |
| [27] | Feng SG, Lu JJ, Gao L, et al. Molecular phylogeny analysis and species identification of Dendrobium (Orchidaceae) in China[J]. Biochem Genet, 2014, 52 :127–136. DOI:10.1007/s10528-013-9633-6 |
| [28] | Li X, Ding X, Chu B, et al. Genetic diversity analysis and conservation of the endangered Chinese endemic herb Dendrobium officinale Kimura et Migo (Orchidaceae) based on AFLP[J]. Genetica, 2008, 133 :159–166. DOI:10.1007/s10709-007-9196-8 |
| [29] | Ding G, Ding XY, Shen J, et al. Genetic diversity and molecular authentication of wild populations of Dendrobium officinale by RAPD[J]. Acta Pharm Sin (药学学报), 2005, 40 :1028–1032. |
| [30] | Creste S, Accoroni KAG, Pinto LR, et al. Genetic variability among sugarcane genotypes based on polymorphisms in sucrose metabolism and drought tolerance genes[J]. Euphytica, 2010, 172 :435–446. DOI:10.1007/s10681-009-0078-2 |
| [31] | Oettler G, Tams SH, Utz HF, et al. Prospects for hybrid breeding in winter triticale[J]. Crop Sci, 2005, 45 :1476–1482. DOI:10.2135/cropsci2004.0462 |
| [32] | Yang N, Wu CX. Unequal genetic contributions of parents to offsprings and their optimal control[J]. Acta Genet Sin (遗传学报), 1993, 20 :294–299. |
| [33] | Chi XF, Jia ZY, Li CT, et al. Analysis of genetic structure in obverse and inverse cross of purse red carp and german mirror by microsatellite markers[J]. J Dalian Ocean Univ (大连海洋大学学报I), 2010, 25 :450–455. |
| [34] | Liu Y, Chen HH, Wen H, et al. Enhancing the accumulation of B-amyrin in Saccharomyces cerevisiae by co-expression of Glycyrrhiza uralensis squalene 1 and B-amyrin synthase genes[J]. Acta Pharm Sin (药学学报), 2014, 49 :734–741. |
| [35] | Guo WZ, Ma GJ, Zhu YC, et al. Molecular tagging and mapping of quantitative trait loci for lint percentage and morphological marker genes in upland cotton[J]. Plant Biol, 2006, 48 :320–326. |
 2016, Vol. 51
2016, Vol. 51


