脂质不仅是细胞的保护屏障,也是细胞内信号分子的前体物质,调节膜运输,并且参与细胞信号转导[1, 2]。脂质的代谢产物,如花生四烯酸类成分,参与炎症的发生,有报道认为炎症反应与脂类代谢紊乱息息相关[3, 4]。细胞膜甘油磷脂代谢紊乱会导致各种炎症性疾病的发生[5],如动脉粥样硬化[6]、2型糖尿 病[7]、阿尔茨海默症[8]和癌症[9]等。
甘油磷脂是细胞膜脂质的主要组成成分,是细胞膜不可缺少的组成构架,作为调节和控制细胞功能与疾病的重要参与者。甘油磷脂的结构由甘油骨架、两条脂肪酸链 (sn-1、sn-2) 和磷酸头部组成[10]。在哺乳动物细胞中,磷脂主要包括磷脂酸 (PA)、磷脂酰胆碱 (PC)、磷脂酰乙醇胺 (PE)、磷脂酰肌醇 (PI)、甘油磷脂酰甘油 (PG) 以及磷脂酰丝氨酸 (PS)。由于不同的磷脂酸头部、脂肪酸链及其含有的不饱和键数目,使得甘油磷脂的种类繁多,这就为磷脂成分的鉴定增添了不少难度。
电喷雾质谱 (ESI-MS),可在短时间内对不同种类的脂质成分进行快速分析,这为脂质的整体分析鉴定提供了核心技术支持[11]。色谱质谱技术的联合 使用极大地推动磷脂组学的发展,尤其是软电离离子化和高分辨质谱分析技术在脂质组学的分析应用。ESI-MS具有高分辨率、高灵敏度、高通量、样品 前处理过程简单、可实现大批量自动化进样的特点,适合对磷脂混合物的定性定量研究[12, 13]。1997年,Brügger等[14]第一次利用ESI/MS技术对从仓鼠卵巢细胞中提取的几种磷脂进行分析, 包括PC、PE、PG、PI和PA。Lorenzen等[15]运用UHPLC-ESI-MS对黄 色黏球菌 (Myxococcus xanthus) 的脂质成分进行 了全面系统的分析。Wu等[16]通过UHPLC-QTOF/MS技术寻找非甾体抗炎药物干预RAW264细胞炎症状态下甘油磷脂的动态变化。Cífková等[17]结合HILIC- HPLC-MS和多变量数据分析方法对人类乳腺癌周围的正常组织的脂质进行分析。Zeng等[18]运用UHPLC- QTOF/MS分析T2DM模型大鼠的血浆磷脂代谢,寻找2型糖尿病生物标记物。Koivusalo等[19]运用ESI-MS对磷脂定量分析发现磷脂的酰基链长度、不饱和度以及进样浓度和仪器的响应有密切的关系。
本实验采用液质联用技术对RAW264.7细胞甘油磷脂成分进行分析,旨在建立一种基于液质联用技术的快速、简便、可靠的RAW264.7巨噬细胞甘油磷脂类成分表征方法。
材料与方法实验仪器及试剂 超高效液相色谱/四级杆飞行时间串联质谱仪 (Acquity UHPLC-QTOF/MS,美国Waters公司); BEH C18柱 (2.1 mm× 50 mm,1.7 μm,Waters); Masslynx 4.1数据处理系统 (Waters); HHS型电热恒温水浴锅 (上海博迅实业有限公司); KDC- 220HR高速冷冻离心机 (科大创新股份有限公司); TDL80-2B低速离心机 (上海安亭科学仪器厂); HF90 CO2培养箱 (上海力申科学仪器有限公司); 显微镜 (江南永新光学有限公司); SW-CJ-1FD超净台 (苏净安泰); WH-2微型涡旋混合仪 (上海沪西分析仪器厂)。
异丙醇 (色谱纯,德国Merck公司); 甲醇、乙腈 (色谱纯,上海安谱实验科技股份有限公司); 乙酸 (色谱纯,美国Aladdin公司); 亮氨酸脑啡肽 (美国Sigma公司); 纯水 (屈臣氏); RAW264.7巨噬细胞 (购自中国科学院细胞库); 胎牛血清 (FBS,Gibco); 高糖培养基 (DMEM,Gibco); 0.25% 胰蛋白酶 (Gibco); 双抗(北京康贝源科技有限责任公司); 磷酸盐缓冲溶液 (PBS,美国Hyclone公司); DMSO (美国Sigma公司); 甘油磷脂对照品: 1-棕榈酰基-2-油酰基-sn-丙三氧基-3-磷脂胆碱 > 99% {PC (16∶0/18∶1); [M+H]+ = 760}、1-棕榈酰基-2-羟基-sn-丙三氧基-3-磷脂胆碱>99% {PC (16∶0/0∶0); [M+H]+ = 496}、1-硬脂酰基- 2-羟基-sn-丙三氧基-3-磷脂乙醇胺 > 99% {PE (18∶0/ 0∶0); [M+H]+ = 482}、1-油酰基-2-羟基-sn-丙三氧基- 3-磷脂胆碱 > 99% {PC (18∶1/0∶0); [M+H]+ = 522} (Avanti polar lipids)。
细胞培养 细胞复苏后接种于培养皿中,培养基为DMEM∶FBS∶双抗=89∶10∶1,置于37 ℃、5% CO2以及饱和湿度的孵育箱中培养,每两天更换培养基,以1∶3的比例进行传代。
甘油磷脂对照品的配制 精密称定各甘油磷脂对照品,用甲醇溶解配制成10 μg·mL−1的对照品溶液。
甘油磷脂的提取 采用修饰后的Bligh-Dyer[20, 21]法提取甘油磷脂。磷脂的前处理过程在低温下 (4 ℃) 操作。提取溶剂采用甲醇−0.1 mol·L−1盐酸−氯仿 (1∶1∶1),涡旋静置分层时,保留有机相,弃去水相以除去非脂成分。具体提取过程如下: ① 将细胞 (100 mm培养皿) 置于冰袋上操作; ② 吸取冷1×PBS 5 mL清洗贴壁细胞两次; ③ 加入冷1×PBS 1 mL,将贴壁细胞完全刮落,吹匀,转移至离心管中; ④ 600×g,4 ℃,离心5 min,弃去上清液; ⑤ 迅速置于液氮中使其结冰粒,即可进行后续操作; ⑥ 加入冷的0.1 mol·L−1盐酸−甲醇(1∶1,各400 μL),再加入氯仿400 μL; ⑦ 涡旋1 min,16 000×g、4 ℃离心8 min; ⑧ 取下层有机相,氮气吹干; ⑨ 加入甲醇−氯仿 (9∶1) 80 μL,复溶,转移至微量内衬管进行UHPLC-QTOF/MS分析。
甘油磷脂的UHPLC-QTOF/MS分析 色谱柱为Acquity UHPLC BEH C18 (2.1 mm× 50 mm,1.7 μm)。流动相A: 水 (含10 mmol·L−1乙酸铵和0.25% 乙酸),流动相B: 乙腈 (含10 mmol·L−1乙酸铵和0.25% 乙酸)。梯度洗脱程序0~4 min,61%~81.4% B; 4~ 20 min,81.4%~90% B; 20~21 min,90%~100% B; 21~24 min,100% B。流速: 0.3 mL·min−1; 进样量: 5 μL; 柱温: 50 ℃; 梯度洗脱曲线 = 7。
质谱条件: 采用浓度为500 ng·mL−1亮氨酸−脑啡肽进行实时校正质量数。离子源为ESI源,分别在正负离子模式下进行质谱信号采集,亮氨酸−脑啡肽在ESI+、ESI− 模式下的m/z分别为 [M+H]+ = 556.277 1,[M−H]− = 554.261 5。质量数扫描离子范围设定为450~950 Da。
质谱参数设定如下: 毛细管电压: 3.0 kV,锥孔电压: 30 V,提取锥孔电压: 1.0 V,脱溶剂温度: 300 ℃,离子源温度: 100 ℃,离子能量: 1.0 V,碰撞能量: 6.0 V,反向锥孔气流: 50 L·h−1,脱溶剂气流: 500 L·h−1。
结果与讨论 1 RAW264.7巨噬细胞甘油磷脂成分的UHPLC- QTOF/MS分析鉴定在正、负离子模式下,得到RAW264.7细胞甘油磷脂的总离子流图,如图 1所示。对总离子流图中每个峰进行推断分析,每个色谱峰含有多个甘油磷脂成分的质谱信息,保留时间相近,但能够通过质谱系统自主地选择目标离子进行MS/MS分析。根据化合物的精确分子量及其元素组成推断其分子式,限定误差范围为5 ppm以内,并结合特征性碎片,通过Lipid Maps磷脂数据库进行检索比对,推断得到RAW264.7巨噬细胞甘油磷脂成分 (表 1)。
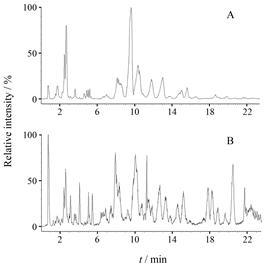
|
Figure 1 Total-ion-current chromatograms of glycerophos;pholipids in RAW264.7 cells (A: ESI+,B: ESI−) |
| Table 1 Identification of glycerophospholipids in RAW264.7 macrophage |
在鉴定出的82个甘油磷脂成分中,包括PC类、PE类、PG类和PI类。PC类在正离子模式下,容易生成一个特征性的碎片离子,即m/z 184[22]。在负离子模式下,由于流动相里含有乙酸,PC类容易形成 [M+ CH3COO]−,也容易失去一个甲基形成 [M−CH3]−[23],此外,PC (16∶0/0∶0) 的脂肪酸链断裂形成碎片峰m/z 255,代表C16∶0脂肪酸链。
PE类在正离子模式下会形成准分子离子峰 [M+H]+,还可断裂极性的头部形成PE类在ESI+下二级 串联质谱裂解的特征峰 [M+H−141]+[24]。在负离子模式下,PE类会形成准分子离子峰 [M−H]−,还可裂解产生PE类物质在负离子模式下的特征峰m/z 140 ([C2H7O4NP]−) 和m/z 196([C5H11O5NP]−)[25],此外,PE (18∶0/0∶0) 的脂肪酸链断裂形成碎片峰m/z 283,代表C18∶0脂肪酸链。
PI类在负离子模式下,会形成准分子离子峰 [M−H]−,此外,其极性头部的不同断裂还会产生一些特征峰,如果断裂sn-1位置的链,将产生m/z 223 ([C6H8O7P]−) 和m/z 297 ([C9H14O9P]−)[26],如果sn-2位置的脂肪酸链断裂形成乙烯酮结构,将产生m/z 241 ([C6H10O8P]−)、m/z 223 ([C6H8O7P]−) 和m/z 315 ([C9H16O10P]−),这些特征性的离子碎片即可判别生物样品中是否存在PI类物质[26, 27]。另外,PI (16∶0/ 18∶1) 的两条脂肪酸链分别断裂形成m/z 253和m/z 281,代表C16∶1和C18∶1两条脂肪酸链,且m/z 281的信号强度比m/z 253强,说明C18∶1键合在甘油骨架的sn-2位[28]。
PG类在负离子模式下断裂一条脂肪酸链形成[M−H−R2COOH]−,在此基础上,还会继续断裂形成[M−H−R2COOH−74]−[29],另外,PG类容易裂解产生m/z 171 ([C3H8O6P]−) 和m/z 227 ([C6H12O7P]−) 的特征峰[25]。
本实验通过运用UHPLC-QTOF/MS技术并结合各类甘油磷脂类裂解规律,共推断出82个甘油磷脂成分,其中包括57个磷脂酰胆碱 (PCs)、21磷脂酰乙醇胺 (PEs)、3个磷脂酰甘油 (PGs) 和1个磷脂酰肌醇 (PI)。本实验表明UHPLC-QTOF/MS能够有效地分离推断RAW264.7巨噬细胞甘油磷脂成分。因此,本文建立了一种基于UHPLC-QTOF/MS技术的快速、简便、可靠的RAW264.7巨噬细胞甘油磷脂类成分快速表征方法,为以RAW264.7巨噬细胞作为研究对象的脂质组学研究提供依据。
| [1] | Bou khalil M, Hou W, Zhou H, et al. Lipidomics era: accom-plishments and challenges[J]. Mass Spectrom Rev , 2010, 29 :877–929. DOI:10.1002/mas.v29:6 |
| [2] | Watson AD. Thematic review series: systems biology approaches to metabolic and cardiovascular disorders[J]. J Lipid Res , 2006, 47 :2101–21111. DOI:10.1194/jlr.R600022-JLR200 |
| [3] | Schleimer RP, Davidson DA, Lichtenstein LM, et al. Selective inhibition of arachidonic acid metabolite release from human lung tissue by antiinflammatory steroids[J]. J Immunol , 1986, 136 :3006–3011. |
| [4] | Ogletree ML, Begley CJ, King GA, et al. Influence of steroidal and nonsteroidal anti-inflammatory agents on the accumulation of arachidonic acid metabolites in plasma and lung lymph after endotoxemia in awake sheep[J]. Am Rev Respir Dis , 1986, 133 :55–61. DOI:10.1164/arrd.1986.133.1.55 |
| [5] | Astudillo AM, Balgoma D, Balboa MA, et al. Dynamics of arachidonic acid mobilization by inflammatory cells[J]. Biochim Biophys Acta , 2012, 1821 :249–256. DOI:10.1016/j.bbalip.2011.11.006 |
| [6] | Krauss RM. Phospholipid transfer protein and atherosclerosis: genetic studies take aim at a moving target[J]. Circulation , 2010, 122 :452–454. DOI:10.1161/CIRCULATIONAHA.110.966572 |
| [7] | Zhu C, Liang Q, Hu P, et al. Phospholipidomic identification of potential plasma biomarkers associated with type 2 diabetes mellitus and diabetic nephropathy[J]. Talanta , 2011, 85 :1711–1720. DOI:10.1016/j.talanta.2011.05.036 |
| [8] | Igarashi M, Ma K, Gao F, et al. Disturbed choline plasmalogen and phospholipid fatty acid concentrations in Alzheimer disease prefrontal cortex[J]. J Alzheimer's Dis , 2011, 24 :507–517. |
| [9] | Jeong RU, Lim S, Kim MO, et al. Effect of D-allose on prostate cancer cell lines: phospholipid profiling by nanoflow liquid chromatography–tandem mass spectrometry[J]. Anal Bioanal Chem , 2011, 401 :689–698. DOI:10.1007/s00216-011-5113-1 |
| [10] | Brouwers JF. Liquid chromatographic–mass spectrometric analysis of phospholipids. Chromatography, ionization and quantification. Biochim Biophys[M]. 2011 : 763 -775. |
| [11] | Zhu C, Hu P, Liang QL, et al. Recent advance in lipidomics[J]. Chin J Anal Chem (分析化学) , 2009, 37 :1390–1396. DOI:10.1016/S1872-2040(08)60132-2 |
| [12] | Nguyen HP, Schug KA. The advantages of ESI-MS detection in conjunction with hilic mode separations: fundamentals and applications[J]. J Sep Sci , 2008, 31 :1465–1480. DOI:10.1002/(ISSN)1615-9314 |
| [13] | Schneiter R, Brügger B, Sandhoff R, et al. Electrospray ionization tandem mass spectrometry (ESI-MS/MS) analysis of the lipid molecular species composition of yeast subcellular membranes reveals acyl chain-based sorting/remodeling of distinct molecular species en route to the plasma membrane[J]. J Cell Biol , 1999, 146 :741–754. DOI:10.1083/jcb.146.4.741 |
| [14] | Brügger B, Erben G, Sandhoff R, et al. Quantitative analysis of biological membrane lipids at the low picomole level by nano-electrospray ionization tandem mass spectrometry[J]. Proc Natl Acad Sci USA , 1997, 94 :2339–2344. DOI:10.1073/pnas.94.6.2339 |
| [15] | Lorenzen W, Bozhüyük KAJ, Cortina NS, et al. A comprehensive insight into the lipid composition of Myxococcus xanthus by UPLC-ESI-MS[J]. J Lipid Res , 2014, 55 :2620–2633. DOI:10.1194/jlr.M054593 |
| [16] | Wu X, Zhao L, Peng H, et al. Search for potential biomarkers by UPLC/Q-TOF-MS analysis of dynamic changes of glyc-erophospholipid constituents of RAW264.7 cells treated with NSAID[J]. Chromatographia , 2015, 78 :211–220. DOI:10.1007/s10337-014-2822-6 |
| [17] | Cífková E, Holčapek M, Lísa M, et al. Determination of lipidomic differences between human breast cancer and surrounding normal tissues using HILIC-HPLC/ESI-MS and Multivariate Data Analysis[J]. Anal Bioanal Chem , 2014, 407 :991–1002. |
| [18] | Zeng P, Zang Y, Rui W, et al. UPLC-QTOF/MS analysis of phospholipids metabolite profiling in plasma of T2DM rat[J]. Acta Pharm Sin (药学学报) , 2015, 50 :882–886. |
| [19] | Koivusalo M, Haimi P, Heikinheimo L, et al. Quantitative determination of phospholipid compositions by ESI-MS/MS: effects of acyl chain length, unsaturation, and lipid concentration on instrument response[J]. J Lipid Res , 2001, 42 :72–663. |
| [20] | Ivanova PT, Milne SB, Byrne MO, et al. Glycerophospholipid identification and quantitation by electrospray ionization mass spectrometry[J]. Methods Enzymol , 2007, 432 :21–57. DOI:10.1016/S0076-6879(07)32002-8 |
| [21] | Bligh EG, Dyer WJ. A rapid method of total lipid extraction and purification[J]. Can J Biochem Physiol , 1959, 37 :911–917. DOI:10.1139/o59-099 |
| [22] | Haroldsen PE, Gaskell SJ. Quantitative analysis of platelet activating factor using fast atom bombardment/tandem mass spectrometry[J]. Biomed Environ Mass Spectrom , 1989, 18 :439–444. DOI:10.1002/(ISSN)1096-9888 |
| [23] | Harrison KA, Murphy RC. Negative electrospray ionization of glycerophosphocholine lipids: formation of[J]. Biol Mass Spectrom , 1995, 30 :1772–1773. DOI:10.1002/(ISSN)1096-9888 |
| [24] | Brouwers JF, Vernooij EA, Tielens AG, et al. Rapid separation and identification of phosphatidylethanolamine molecular species[J]. J Lipid Res , 1999, 40 :164–169. |
| [25] | Yan X, Li HY, Xu JL, et al. Analysis of phospholipids in microalga Nitzschia closterium By UPLC-Q-TOF-MS[J]. Chin J Oceanol Limnol , 2010, 28 :106–112. DOI:10.1007/s00343-010-9263-3 |
| [26] | Melissa P, Murphy RC. Electrospray mass spectrometry of phospholipids[J]. Mass Spectrom Rev , 2003, 22 :332–364. DOI:10.1002/(ISSN)1098-2787 |
| [27] | Taguchi R, Hayakawa J, Takeuchi Y, et al. Two-dimensional analysis of phospholipids by capillary liquid chromatography/ electrospray ionization mass spectrometry[J]. J Mass Spectrom , 2010, 43 :2297–2310. |
| [28] | Wang Q, Fu L, Zhang Y, et al. Rapid identification of phospholipids in rat plasma by UPLC/Q-TOF MS[J]. J Instrum Anal (分析测试学报) , 2014, 33 :256–263. |
| [29] | Larsen A, Uran S, Jacobsen PB, et al. Collision-induced dissociation of glycero phospholipids using electrospray ion-trap mass spectrometry[J]. Rapid Commun Mass Spectrom , 2001, 15 :8–2393. DOI:10.1002/(ISSN)1097-0231 |
 2016, Vol. 51
2016, Vol. 51


