2. 四川省烟草质量监督检测站, 四川 成都 610041
2. Sichuan Tobacco Quality Supervision and Testing Station, Chengdu 610041, China
他莫昔芬 (tamoxifen,TAM) 属于三苯乙烯非甾体类抗雌激素受体拮抗剂,在临床上广泛用于乳腺癌和卵巢癌治疗[1],尤其适用于雌激素受体阳性 (ER+) 和孕酮受体阳性 (PR+) 的乳腺癌患者的治疗,目前在全球120多个国家都有应用。TAM的疗效和安全性研究国内外均有大量报道,早期乳腺癌临床试验协作组 (EBCTCG) 推荐乳腺癌患者一般至少进行为期5年TAM辅助治疗。绝经前/后患有原发性乳腺癌的患者,初次确诊后采用TAM进行内分泌辅助治疗能够显著降低15年复发和死亡率[2]。然而,个体差异使得其30%~50% 的乳腺癌患者进行TAM辅助治疗的预后较差或治疗失败[3, 4],有研究[5]表明TAM在代谢与转运过程中代谢酶和转运体的基因多态性对其疗效及不良反应产生显著影响。
TAM在体内通过Ⅰ相代谢酶产生活性更高的代谢物,主要是细胞色素酶P450 (cytochrome P450s,CYP450s),参与TAM代谢的CYP450s有CYP2D6、CYP3A4和CYP2C9等[6, 7]; 再通过Ⅱ相代谢酶磺基转移酶(sulfotransferases,SULTs) 和UDP-葡糖醛酸转移酶(UDP-glucuronosyltransferases,UGTs) 去活或生成水溶性更好的结合物排出体外[8]。研究[9-13]表明,代谢酶的基因多态性会影响TAM在体内代谢为活性代谢物从而影响其临床治疗效果。此外,体内参与TAM及其活性代谢物转运的转运体的多态性也会影响TAM及其活性代谢物在细胞或血液中活性代谢物的浓度,从而影响其药效发挥,最终影响其临床效果[5]。个体化差异一直都是导致药物疗效降低或不良反应增加的主要原因,也是个体化用药的难点与热点,药物基因组学的研究为药物的个体化用药提供了可以选择的手段[14]。本文总结了近年来的相关研究,深入分析了TAM主要代谢酶和转运体的基因多态性对乳腺癌患者进行TAM治疗的药动学、药效学以及临床预后效果影响,以期为TAM临床合理有效应用提供理论支撑,并为深入研究TAM药物基因组学提供参考。
1 他莫昔芬的代谢过程参与TAM的体内代谢过程的Ⅰ相和Ⅱ相代谢酶较多,其中最为主要的Ⅰ相代谢酶是CYP2D6和CYP3A4。TAM在肝脏内主要的代谢途径有两条[6]: 一条是约40% 的TAM经由CYP3A4和CYP3A5代谢去甲基化生成N-去二甲基他莫昔芬 (N-DES-TAM),然后一部分经CYP3A4和CYP3A5的作用下继续去甲基生成N,N-去二甲基他莫昔芬,另外一部分经CYP2D6代谢羟基化生成活性更高的N-去甲基-4-羟基他莫昔芬 (endoxifen,EDF),临床的结果显示去甲基化他莫昔芬与雌激素受体的亲合力较弱,而EDF的抗雌激素活性达到原药的30倍; 另一条代谢途径是通过CYP2D6的代谢羟基化作用产生抗雌激素活性更强的4-羟基他莫昔芬 (4-OH-TAM),4-OH-TAM也可以在CYP3A4的去甲基作用下生成EDF。4-OH- TAM和EDF有顺和反 (E/Z) 两种构型,临床研究的结果显示Z-4-OH-TAM和Z-EDF是主要的发挥抗雌激素药效的代谢物,因为,与原药TAM相比,4-OH- TAM与ER的亲合作用要高100倍,对乳腺癌细胞的雌激素抑制作用高30~100倍[15-18]。在CYP3A4和CYP3A5作用下,TAM还可以代谢生成α-OH-TAM和3-OH-TAM,但这两种代谢物的生成量较低[19]。
参与TAM代谢的Ⅱ相代谢酶主要为SULTs和UGTs,TAM的代谢产物经肝脏中SULTs和UGTs代谢,转化成无活性、可溶解的物质,最终被排出体外[8]。体外实验数据表明,UGT2B7是主要的介导4-OH-TAM和EDF反式O-葡糖苷酸化的肝细胞酶[20]。但是并非所有的SULT作用都是生成无活性的结合物,有研究表明羟基化磺基转移酶2A1 (hSULT2A1) 能够催化活化TAM,生成α-磺酰基-TAM,这一代谢物能够与DNA结合,从而导致TAM具有潜在的致癌性,其中4-OH-TAM、N-DES-TAM和EDF都是hSULT2A1的底物[21]。
药物转运体是位于细胞膜上的功能性膜蛋白,影响药物吸收、分布和排泄等过程,按转运方向的不同大致分为摄入性和外排性转运体两大类[22]: 已知的摄入性转运体主要有阴离子多态转运体 (organic anion-transporting polypeptides,OATP; 基因符号: SLCO),有机阴离子转运体 (organic cation transporters,OCT; 基因符号: SLC22A); 外排性转运体主要有: 乳腺癌耐药蛋白 (breast cancer resistance protein,BCRP; 基因符号: ABCG2)、多药抗性相关蛋白 (multidrug resistance-associated protein,MRP; 基因符号: ABCC)、P糖蛋白 (P-glycoprotein,P-gp; 基因符号: ABCB1)、多药及毒素外排转运蛋白 (multidrug and toxin extrusion protein,MATE; 基因符号: SLC47A) 等(图 1)。目前研究表明,外排性转运体MRP、BCRP、P-gp和摄入性转运体OAT在TAM及其代谢产物的转运方面发挥了重要作用,因而相关基因多态性可能影响TAM的药效学和药动学。
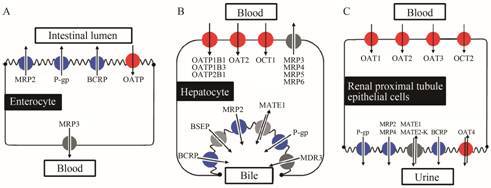
|
Figure 1 Transporters expressed in human intestinal epithelium (A),human hepatocytes (B) and human renal proximal tubule epithelial cells (C). Efflux transporters discussed in this review are colored in blue,uptake proteins in red |
代谢酶基因多态性及其对药物作用的影响是药物基因组学研究的重要内容,部分研究成果已进行广泛的临床转化,如根据CYP2D6基因型指导TAM的临床应用等,但大部分的研究均处于实验室研究阶段,绝大部分研究结果缺乏系统的回顾性及前瞻性研究确证。TAM药物基因组学的研究一直都受到广泛关注,主要是通过研究TAM主要的代谢酶及其基因多态性来阐明TAM的代谢过程以及代谢物的药理作用。
2.1 CYP2D6肝脏中CYP2D6含量仅占肝微粒体 CYP450总量的2%,却参与约25% 的临床用药的代谢过程[23]。许多作用于神经系统和心血管系统的药物都由此酶所代谢。目前已知的CYP2D6等位基因的变异达到100多种,主要分为单核苷酸变异 (single nucleotide polymorphism,SNPs)、大片段基因的丢失以及多基因拷贝等,这些变异使CYP2D6基因呈现出多态性,并决定其代谢表型的多态性。按CYP2D6代谢表型多态性可将人群分为4种类型[24, 25]: 弱代谢者 (poor metabolizer,PM; 包含CYP2D63,4,5,6)、中等代谢者 (intermediate metabolizer,IM; 包含CYP2D610,41)、快代谢者 (extensive metabolizer,EM) 和超快代谢者 (ultra rapid metabolizer,UM)。其中,含两个以上正常功能等位基因的为UMs,含两个正常功能等位基因或一个正常功能和一个无效等位基因的为EMs,含两个功能降低等位基因或一个功能降低和一个无效等位基因的为IMs,含两个无效等位基因的为PMs。CYP2D6代谢的表型分布存在明显的种族差异,PM主要以欧洲人为主,IM在亚洲人中居多,UMs在高加索人中较为常见 (10%~15%) 而亚洲人较为少见[5, 26]。中国大陆的最为常见PMs等位基因主要是CYP2D6*5 (7.2%) 和CYP2D6*14 (2.0%),其他东方人中常见PM等位基因 (CYP2D6*10) 在大陆出现的频率较低[27]。
CYP2D6的基因型与表型之间的关系已经研究得较为透彻[28],CYP2D6是主要的将TAM代谢成活性更高的4-OH-TAM和EDF的代谢酶,而PMs和IMs患者代谢TAM的能力减弱,使得体内TAM转化4-OH-TAM和EDF比率降低,从而使得其抗雌激素和孕激素的能力减弱; 反之UMs患者可能会导致TAM治疗过程中的不良反应增加,主要是增加热潮红和子宫内膜瘤等风险。CYP2D6的基因多态性会导致5%~10% 白人体内CYP2D6酶功能的缺失,在亚洲人和非洲人中出现的概率为1%~2%[29]。目前,研究最多的CYP2D6代谢表型为PM,早在2003年,Stearns等[16]首先将CYP2D6的基因多态性与EDF的血药浓度联系起来,他的研究结果显示PM会导致其体内EDF的血药浓度显著降低,存在wt/*等位基因 (一个野生等位基因一个多态等位基因) 的妇女体内EDF的浓度会降低64% (95% CI = 39%~89%)。此后一系列研究都关注于CYP2D6基因多态性对TAM临床终点指标影响[11, 30-37]。
也有一些研究开始针对PM和IM患者,在治疗窗口内增加TAM用药剂量以期达到EDF应有的血 药浓度。Dezentje等[11]研究显示TAM的增加会导致PM和IM体内活性代谢物EDF的血药浓度显著增加 (P < 0.001; P = 0.002),用药剂量控制在每天120 mg以下,不会增加TAM的不良反应,在PM体内用药剂量的增加可以使得EDF的浓度达到EM正常浓度的24%~81%。当前,美国食品药品监督管理局 (FDA) 认可在进行TAM治疗前测试CYP2D6多态性对准确用药进行指导,FDA咨询委员会药物科学和临床药理学分会也推荐在TAM的药物标签上标明基因分型影响[38]。针对波兰的乳腺癌女性患者的一项临床研究也显示CYP2D6分析并不能直接预测接受TAM辅助治疗患者血液中Z-EDF浓度是否达到治疗窗口,因为CYP2D6基因多态性导致Z-EDF浓度的显著变化 (27%,P < 1×10-16),这一变化也预示在波兰的乳腺癌患者中直接代谢生成Z-EDF的比例达到51 % (P < 1×10-43)[39]。前期的一系列针对CYP2D6基因多态性的研究 (表 1)[11, 30, 32, 33, 35, 37, 39-57]都提示,CYP2D6多态性可能会影响TAM体内活性代谢物的含量水平,从而从药动学上影响TAM的治疗过程,但是量-效关系还有待进一步确证。在TAM治疗前进行CYP2D6的基因分型能够在一定程度上解决TAM治疗存在较大个体差异的问题,饮食和环境因素的影响也需纳入考虑[12]。
近期,也有较多的研究者关注CYP2D6基因多 态性对乳腺癌患者进行TAM辅助治疗的预后效果的影响 (表 1)。由于CYP2D6存在显著的种族差异,种族间N-DES-TAM/Z-EDF比率与CYP2D6基因多态 性显著相关 (R2: 0.53,P < 1×10-77),无远处复发生存 (DRFS) 的提升与N-DES-TAM/Z-EDF比率呈负相关 (P = 0.036) 与CYP2D6活性评分呈正相关 (HR = 0.62; 95% CI,0.43~0.91; P = 0.013),患者体内血液中低EDF浓度 (< 14 nmol·L-1) 可能比高EDF浓度 (> 35 nmol·L-1) 患者的DRFS短 (单变量P = 0.03; 多变量 HR = 1.94; 95% CI,1.04~4.14; P = 0.064)[30]。携带CYP2D6*10 T/T基因的乳腺癌患者的平均无病生存期 (DFS) 显著短于携带CYP2D6*10 C/T基因型的患者 (P = 0.036),但是其DFS时间与携带CYP2D6*10 C/C基因型的DFS差异不显著 (P = 0.316),研究者也得出CYP2D6*10/*10可能与较短的DFS显著相关[33]。针对日本乳腺癌患者 (n = 345) 的一项临床研究也显示携带CYP2D6 10q22(rs1050937) 突变基因可能与接受TAM辅助治疗的预后效果相关,携带突变基因的数量可能在无复发生存率 (RFS) 上具有积累效应[40]。较多研究都得出CYP2D6基因多态性对乳腺癌患者进行TAM辅助治疗的预后效果的影响并不显著,包括多项结局指标。Mwinyi等[41]研究发现,所有乳腺癌患者的平均无复发时间 (RFT) 要高于UM和EM携带患者,但这种影响不显著。CYP2D6基因多态性与DFS无显著关联[32]。出现这种矛盾性的结论主要源于基因分型不全面或者治疗过程中同时服用了CYP2D6抑制剂[34]。
2.2 CYP3A4/5CYP3A4是肝脏及肠壁中含量最丰富的P450同工酶,约占肝脏CYP总量的30%,超过40% 临床药物主要经CYP3A4代谢[58]。同时也是TAM的主要代谢酶,参与TAM的去甲基作用生成抗雌激素作用较弱的N-DES-TAM,后者与ER的亲合力与TAM相当[59]。由于N-DES-TAM会进一步代谢成为ER亲合力更强的DEF,因此CYP3A4的基因多 态性也可能会影响TAM的抗雌激素作用[13]。部分研究显示,CYP3A4单核苷酸基因多态性会影响年轻女性 (n = 729,绝经前) 尿内雌酮葡糖苷酸浓度变化,这可能会增加患乳腺癌的风险[60]。CYP3A4*1B突变可提高CYP3A4的转录活性[61],增加活性产物的暴露量,携带该等位基因的年轻女性 (年龄小于45岁) 乳腺癌患者比携带CYP3A4*1A纯合子的年轻女性出现卵巢功能早期衰退的时间显著延后,接受TAM治疗的也与卵巢功能早期衰退存在关联[62]。存在这种现象的原因一方面可能是肿瘤患者的生存率和卵巢功能衰退本身的机制比药物代谢更复杂,需要进行更深入的研究; 另一方面可能由于某些研究的结果为假阳性或出现统计学I类错误。
目前研究已发现CYP3A4的某些突变可改变酶功能,从而影响TAM的药动学和药效学作用,但研究结论存在争议。携带CYP3A4*22等位基因的患者接受TAM治疗时体内EDF稳态血药浓度会随季节变化,体内EDF的浓度在冬季比一年内的平均浓度低20%,推测CYP3A4*22代谢TAM的作用可能与体内维生素D水平和日照强度存在一定的关系[44]。近期的一项研究结果也表明,与携带CYP3A4*1/*1的患者相比,CYP3A4*22突变可以使得患者体内4-OH-TAM和TAM的血药浓度分别增加47% 和53% (n = 178)[63]。ter Heine等[64]研究表明,CYP3A4的活性会显著影响EDF的血药浓度 (P < 0.001),加入CYP2D6探针药物 (右美沙芬) 后,个体间EDF的血药浓度差异从55% 下降到25% (n = 40)。CYP3A4的基因多态性对TAM的不良反应也会产生影响,携带CYP3A4*22突变基因的乳腺癌患者,接受TAM治疗产生热潮红的概率降低 (OR: 8.87,P < 0.01),且与血液中EDF稳态血药浓度呈负相关[65]。目前还未见报道CYP3A4基因多态性与乳腺癌患者接受TAM治疗的临床预后效果的影响。
| Table 1 The influence of genetic polymorphisms of CYP2D6 on the plasma concentrations of active tamoxifen (TAM) metabolites and clinical outcomes for breast cancer patients treated with TAM. CYP: Cytochrome P450; M: Tamoxifen metabolite; M1: N-DES-TAM; M2: 4-OH-TAM; M3: EDF; Css: Steady-state concentration; MR: Metabolic ratio; EM: Extensive metabolizer; IM: Intermediate metabolizer; PM: Poor metabolizer; UM: Ultrarapid metabolizer; NS: Not significant; MRN-DES-TAM/EDF: Metabolic ratio of plasma N-DES-TAM over plasma EDF; wt/wt: Two wild type alleles; wt/*: One wild type allele and one polymorphic all; */*: Two polymorphic alleles; RFT: Relapse-free time; DFS: Disease-free survival; BCSS: Breast cancer-specific survival; iDFS: Invasive disease-free survival; TTR: Time to recurrence; BCFi: Breast cancer-free interval; DDFS: Disease-free survival; DRFS: Distant recurrence-free survival; TTP: Time to tumor; PFS: Progression-free survival; OS: Overall survival; RFS: Recurrence-free survival; HR: Hazard ratio; CI: Confidence interval |
CYP3A5较为常见的突变基因类型为CYP3A5*3和CYP3A5*6,在瑞士进行的一项临床研究显示[66],入组的677位绝经后的乳腺癌患者中,大约40.7% 的患者携带CYP3A5*3突变型基因,而仅有9.1% 患者携带CYP3A5*6突变型基因。由于CYP3A5参与少 量α-OH-TAM和部分N-DES-TAM (占51%~61%) 的代谢过程,而N-DES-TAM会进一步代谢成活性代谢物,因此,CYP3A5基因多态性可能会间接影响TAM的药动学过程。有研究报道CYP3A5基因多态性会影响N-DES-TAM的血药浓度[19],但也有研究认为CYP3A5基因多态性对TAM的药动学过程并无影响。Tucker等[66]的研究表明CYP3A5的这两种基因突变对患者体内TAM及其代谢物以及不良反应都没有显著影响 (n = 98,绝经的乳腺癌患者) ,但是携带CYP3A5*6等位基因患者肿瘤 (3.6 ± 0.98 cm) 的平均大小要显著高于未携带此基因型患者 (2.0 ± 0.18 cm) (P < 0.02)。一项随机临床试验的数据显示CYP3A5基因多态性对TAM药效学产生影响,携带CYP3A5*3等位基因的乳腺癌患者接受两年期TAM治疗可增 加复发的风险,但是这种差异不显著 (HR = 2.84,95% CI = 0.68~11.99,P = 0.15); 与纯合子相比,携带CYP3A5*3等位基因的乳腺癌患者接受5年期TAM治疗的RFS风险率显著降低 (HR = 0.20,95% CI = 0.07~0.55,P = 0.002),RFS的显著增加也表明延长TAM的治疗期会使得携带CYP3A5*3等位基因患者获益 (HR = 0.13,CI = 0.02~0.86,P = 0.03)[67]。
2.3 CYP2C19和CYP2C9由于CYP2C19和CYP2C9也参与少量TAM代谢为4-OH-TAM及N-DES-TAM的过程,其代谢活性改变在一定程度上可能会影响体内TAM活性代谢物水平以及不良反应[68]。CYP2C19主要突变基因型为CYP2C19*2,该突变可使酶功能缺失,此缺陷基因存在明显的种族差异,中国人中CYP2C19*2突变基因携带者显著高于白人(1.7%~3.0%,P < 0.01); 中国的不同民族之间也存在差异,缺陷等位基因CYP2C19*2在汉族 (32.4%)、回族 (29.7%) 的携带概率显著高于蒙古族的携带概率 (18.2%,P < 0.01),CYP2C19*2/*3在汉族和回族中的携带概率也显著高于白人的携带率 (分别是11.1%~16.3% 和0~0.2%,P < 0.01)[69]。研究显示携带CYP2C19*2和/或*3等位基因的乳腺癌患者血液中EDF的浓度显著降低,携带CYP2C9*2和/或*3等位基因的乳腺癌患者血液中Z-4-OH-TAM的浓度显著降低[30],但是CYP2C9对血液内EDF的浓度的影响不显著[70]。
CYP2C19*17是CYP2C19的高活性等位基因,研究报道携带该等位基因的乳腺癌患者接受TAM治疗的预后效果 (HR,0.45; 95% CI,0.21~0.92; P = 0.03) 显著优于CYP2C19*1,*2和*3携带者[71]。因此,建议乳腺癌患者在接受TAM治疗前,有必要进行CYP2C19酶活性测试[46]。然而,CYP2C19*17对TAM治疗预后效果的影响存在一定争议,一项长达14年的临床研究结果表明,携带CYP2C19*17突变基因的乳腺癌患者 (n = 256,95%为白人) 与不携带该基因的乳腺癌患者接受TAM治疗的DFS差异并不显著 (P = 0.667)[72]。
目前,研究CYP2C9基因多态性对TAM治疗乳腺癌患者的药效学及不良反应的报道较少,可能源于CYP2C9不是其主要的代谢酶,其活性的降低可能会诱导其他主要代谢酶的活性从而使得其影响减弱。
2.4 UGTsUGTs是人体Ⅱ相反应中最重要的酶 之一,利用葡糖醛酸为糖基供体催化广泛的内源性和外源性化学物质进行结合反应,增加其极性而利于排出体外。体外研究实验表明: 在人肝微粒中,具有纯合子UGT2B7 Tyr(268)Tyr 等位基因的人肝微粒代谢TAM时E-4-OH-TAM和E-EDF的O-葡糖醛酸作用的比率分别比具有UGT2B7 His(268)His基因型的低28% (P < 0.001) 和27% (P = 0.002)[20],这表明 参与TAM代谢的二相代谢酶UGTs的多态性 (包括UGT2B7和UGT1A8) 可能导致TAM在不同个体治疗过程中的差异。重组UGT1A4参与了TAM及其代谢物的N-葡糖醛酸作用,是UGTs中最为活跃的酶之一,UGT1A4的SNPs可能会显著降低TAM代谢物在肝脏中的葡糖醛酸化作用[73]。
携带UGT1A448Val纯合子的患者,接受TAM 辅助治疗时葡糖醛酸代谢物的含量水平显著低于携带wt/wt或wt/48Val的患者 (P = 0.037和P = 0.031); 携带UGT2B7268Tyr纯合子的女性患者4-OH-TAM/ 4-OH-TAM-O-葡糖苷酸和4-OH-TAM/4-OH-TAM-N-葡糖苷酸代谢比率也显著低于携带杂合子wt/wt或wt/48Val的女性 (P = 0.005和P = 0.003); 表明携带突变基因UGT1A448Val,UGT2B7268Tyr或UGT2B17nodel由于能够更好的代谢TAM导致其血液中活性代谢物的增加可能在TAM辅助治疗中获益更多,但此结果可能需要进一步临床试验的确证[9]。Ahern等[74]研究显示UGTs (包含3个UGTs: UGT2B15、UGT2B7和UGT1A8) 的基因多态性与接受TAM辅助治疗乳腺癌患者的复发率并无关联。另一项临床研究也显示UGT2B15的多态性对于接受TAM治疗的乳腺癌患者的RFS并无显著影响[67]。得出这些结果可能的解释为TAM活性代谢物-葡糖醛酸加合物在肝脏循环过程中被肠道内活性微生物分泌中葡糖醛酸酶水解断开了,从而以活性代谢物的形式再吸收,这一过程抵消了基因多态性造成的活性代谢物浓度的波动; 另一个可能原因是UGTs也是雌激素的代谢酶,活性增加或者降低对雌激素同样产生影响,从而使得其对TAM的治疗效果影响不大。
2.5 SULTsSULTs主要介导羟基化-TAM的磺化作用,使其失活然后排出体外。早期研究发现SULT1A1*2突变可能会导致其酶活性的降低[75],由于SULT是TAM重要的二相代谢酶,Wegman等[67]研究了此突 变对乳腺癌患者接受TAM辅助治疗效果的影响,与携带SULT1A1*1基因的患者相比,携带SULT1A1*2的患者接受TAM治疗后乳腺癌的复发率降低 (相 对风险率 = 0.48,95% CI = 0.21~1.12,P = 0.074,n = 226)。由于此项临床试验的入组病例相对较少,紧接着这一研究团队又设计了一项入组病例 (n = 677,绝经后的乳腺癌患者) 更多的临床试验研究SULT1A1 (SULT1A1*1和*2) 多态性对绝经后的乳腺癌患者接受TAM辅助治疗效果的关联,结果表明SULT1A1的多态性与乳腺癌患者接受TAM治疗的RFS无显著关联。携带SULT1A2*2和SULT1A2*3等位基因的乳腺癌患者在接受TAM治疗时体内4-OH-TAM和EDF的血药浓度显著增高 (P = 0.025和P = 0.006,n = 135,ER+乳腺癌患者)[47],SULT1A1多态性与接受TAM治疗的西班牙乳腺癌患者体内血液中高EDF的相关性较弱,对其影响不显著 (SULT1A1 (*1/*1: 36比49%; *1/*2: 46比39%,*2/*2: 18比12%; P = 0.152 2)[70]。
TAM治疗的个体差异性导致的复发概率达到50%,而TAM代谢酶多态性对这种差异性的影响只占了一部分,建议综合评价其他影响TAM治疗失败的标志物,包括吸收、分布和排泄过程中的转运体的过表达或基因突变,或是考虑进行更合理细化的分组等[76]。
3 转运体基因多态性 3.1 P-gp(ABCB1) 前期的研究表明P-gp可能会影响接受TAM治疗患者体内活性代谢物EDF,4-OH- TAM的积累,从而导致先天性或获得性的乳腺癌治疗过程中的TAM耐药[77, 78]。P-gp的多态性可能与 乳腺癌患者接受TAM治疗的预后效果相关,携带ABCB1 C3435T纯合子的乳腺癌患者术后复发的时间更短,且术后复发的风险也会显著增加 (P = 0.002); 同时携带ABCB1 C3435T纯合子和CYP2D6 IM基 因的乳腺癌患者复发的平均时间仅有12个月 (95% CI = 0.79~23.2),而不携带此基因的患者为48个月 (95% CI = 14.7~81.2)[31]。Sensorn等[79]针对泰国早期乳腺癌患者的研究也显示,转运体基因多态性对接受TAM辅助治疗预后效果 (DFS) 的影响显著; 携带ABCB1 3435 CT杂合子的患者的DFS显著短于携带3435 CC纯合子的患者 (P = 0.041),而携带3435 CC纯合子患者与携带野生型3435 CC基因的患者的DFS差异不显著; 与携带ABCB1 3435 CC基因的患者相比,携带ABCB1 3435 CT基因的患者术后复发的风险要高5倍(P = 0.043; HR = 5.11; 95% CI: 1.05~24.74)。近期的研究也显示携带至少一个ABCB1 rs1045642等位基因的乳腺癌患者与携带野生纯合子患者具有更长的事件生存期 (n = 71,绝经前的ER+乳腺癌患者),ABCB1的多态性确实会导致绝经前的乳腺癌患者接受TAM替代治疗的预后效果,因此在进行TAM治疗的过程中,除了考虑CYP2D6的基因多态性,应该也将此因素纳入考虑[80]。
3.2 MRP(ABCC) TAM不仅是MRP2的底物,并且能诱导灵长类动物 (猕猴) MRP2基因的表达,导致猕猴肝脏中MRP2的mRNA和蛋白显著增加[81]。TAM的获得性耐药是乳腺癌患者进行TAM治疗失败的重要原因,而MRPs的过表达是乳腺癌细胞从药物敏感细胞向耐药乳腺癌细胞转变的主要原因[82]。转运体基因的51个单倍型标记-SNPs中,携带ABCC2 rs3740065 AA突变基因的患者比GG基因型的乳腺癌患者具有更短的RFS (n = 282,ER+/PR+,接受TAM治疗; HR= 10.64; 95% CI,1.44~78.88)[35]。另一项针对ABCC的SNP对TAM治疗效果的影响研究也显示,携带ABCC2 rs10509373突变基因也与日本乳腺癌患者 (n = 345) 接受TAM辅助治疗的预后效果 (RFS) 显著相关 (P = 1.26×10-10)[40]。另一项针对ER+/PR+的乳腺癌Ⅰ期和Ⅱ期患者 (n = 516,绝经前) 的研究显示MRP1过表达不会导致接受TAM治疗的乳腺癌患者术后RFS和OS期缩短 (RFS: HR = 0.99; 95% CI,0.74~1.31; P = 0.9; OS: HR = 0.68; 95% CI,0.40~1.15; P = 0.1)[83]。
3.3 BCRP(ABCG2) 细胞核中ERβ的表达与BCRP呈正相关,而ERβ是癌症抑制因子,不管是内源性或者是外源性的通过ERβ上调BCRP的表达都可能会导致ERα-/PR-乳腺癌细胞的增殖,但是这一过程可以通过服用TAM得到抑制[84]。
3.4 OATP(SLCO) 有机阴离子转运体OATP1B1是较为常见的存在于肝细胞膜上的摄入性转运体。体外实验显示OATP1B1的基因突变388GG和521CC会增加MCF-7的凋亡率,并且会抑制OATP1B1酶蛋白活性,从而导致其摄入TAM的能力减弱[85]。OATP1B1的两个突变基因OATP1B1 A388G和OATP1B1 T521C参与了TAM的跨膜转运,最近的一项临床试验表明,与携带野生纯合子乳腺癌患者相比,OATP1B1 T521C突变基因者携带的总生存时间 (OS) 存在显著差异 (P = 0.034),但是OATP1B1 A388G突变基因的差异不显著 (P = 0.388)[57]。
4 展望TAM在治疗乳腺癌过程中发挥着重要作用,尤其是对于ER+/PR+的乳腺癌患者,但其疗效及不良反应个体差异较大,导致其在临床应用时给药剂量难以把握。如前所述,有一些报道显示代谢酶CYP2D6/ CYP3A4和转运体P-gp/MRP与乳腺癌患者接受TAM治疗的预后效果显著相关,但研究结论不一。可能原因包括: ① 研究设计的缺陷,如缺乏系统性的回顾性研究和设计合理的前瞻性研究; ② 研究群体的异质性,如不同肿瘤患者及其氧化应激状态代谢水平有所差异; ③ 不同研究给药剂量和疗程存在差异等; ④ 入组自愿者的依从性问题也会导致疗效的差异,同时服用CYP2D6或者其他代谢酶或转运体的抑制剂等; ⑤ 环境因素的影响,如吸烟或环境烟气可能会对TAM的治疗过程产生影响。
根据CYP2D6的基因型来调整TAM的给药剂量得到了FDA的认可,研究也显示适当TAM给药剂量可以使得携带CYP2D6 PM基因的患者体内EDF和4-OH-TAM的浓度增加而不增加不良反应。尽管,有一些研究对CYP2D6基因多态性会对TAM治疗乳腺癌的临床效果产生显著影响还存在疑虑,近期由国际他莫昔芬药物基因组学联盟协会组织的一项大型的系统评价研究也显示CYP2D6与iDFS显著相关[86]。其他代谢酶和转运体多态性虽然对TAM治疗过程产生了影响,但是此类研究基本还处于实验室研究阶段,并未向临床应用进一步推进,因此以后的研究可以综合考虑TAM代谢酶和转运体多态性对其治疗效果的影响,而不是只针对CYP2D6进行基因分型来调整剂量,这需要大量的研究以及系统评价作为基础。同时,网络药理学的研究进展提示,相关代谢酶和转运体的上游调控因子的基因多态性也可能对TAM疗效产生影响; 并且在机体病理状态下,免疫反应及炎症反应通路的多种因子如TNF和NF-κB等的遗传多态性也可能会对TAM的药物反应造成影响,因此后续研究也可以从TAM重要代谢酶和转运体上游调控因子以及反应通路的多态性进行研究,深入了解TAM治疗过程中多态性对其药动学和药效学过程或临床预后效果的影响。
同时,进一步的TAM药物基因组学研究,需要在全基因组关联研究及系统的功能研究的基础上,明确有临床意义的遗传变异,并通过系统的设计合理的前瞻性研究确证,使得研究结果可以很好的指导TAM临床应用,真正实现基于“基因处方”的个体化用药。
| [1] | Jordan VC. Tamoxifen: a most unlikely pioneering medicine[J]. Nat Rev Drug Discov , 2003, 2 :205–213. DOI:10.1038/nrd1031 |
| [2] | EBCTCG. Effects of chemotherapy and hormonal therapy for early breast cancer on recurrence and 15-year survival: an overview of the randomised trials[J]. Lancet , 2005, 365 :1687–1717. DOI:10.1016/S0140-6736(05)66544-0 |
| [3] | EBCTCG. Effects of adjuvant tamoxifen and of cytotoxic therapy on mortality in early breast cancer. An overview of 61 randomized trials among 28,896 women. Early Breast Cancer Trialists' Collaborative Group[J]. N Engl J Med , 1989, 319 :1681–1692. |
| [4] | EBCTCG. Tamoxifen for early breast cancer[J]. Cochrane Database Syst Rev , 2001, 1 :Cd000486. |
| [5] | Kiyotani K, Mushiroda T, Nakamura Y, et al. Pharmacoge-nomics of tamoxifen: roles of drug metabolizing enzymes and transporters[J]. Drug Metab Pharmacokinet , 2012, 27 :122–131. DOI:10.2133/dmpk.DMPK-11-RV-084 |
| [6] | Dehal SS, Kupfer D. CYP2D6 catalyzes tamoxifen 4-hydroxylation in human liver[J]. Cancer Res , 1997, 57 :3402–3406. |
| [7] | Jordan VC. New insights into the metabolism of tamoxifen and its role in the treatment and prevention of breast cancer[J]. Steroids , 2007, 72 :829–842. DOI:10.1016/j.steroids.2007.07.009 |
| [8] | Falany JL, Pilloff DE, Leyh TS, et al. Sulfation of raloxifene and 4-hydroxytamoxifen by human cytosolic sulfotransferases[J]. Drug Metab Dispos , 2006, 34 :361–368. |
| [9] | Romero-Lorca A, Novillo A, Gaibar M, et al. Impacts of the glucuronidase genotypes UGT1A4, UGT2B7, UGT2B15 and UGT2B17 on tamoxifen metabolism in breast cancer patients[J]. PLoS One , 2015, 10 :e0132269. DOI:10.1371/journal.pone.0132269 |
| [10] | Johnson JA, Hamadeh IS, Langaee TY. Loss of heterozygosity at the CYP2D6 locus in breast cancer: implications for tamoxifen pharmacogenetic studies[J]. J Natl Cancer Inst , 2015 . |
| [11] | Dezentje VO, Opdam FL, Gelderblom H, et al. CYP2D6 genotype-and endoxifen-guided tamoxifen dose escalation increases endoxifen serum concentrations without increasing side effects[J]. Breast Cancer Res Treat , 2015, 153 :583–590. DOI:10.1007/s10549-015-3562-5 |
| [12] | de Vries Schultink AH, Zwart W, Linn SC, et al. Effects of pharmacogenetics on the pharmacokinetics and pharmacody-namics of tamoxifen[J]. Clin Pharmacokinet , 2015, 54 :797–810. DOI:10.1007/s40262-015-0273-3 |
| [13] | Binkhorst L, Mathijssen RH, Jager A, et al. Individualization of tamoxifen therapy: much more than just CYP2D6 genotyp-ing[J]. Cancer Treat Rev , 2015, 41 :289–299. DOI:10.1016/j.ctrv.2015.01.002 |
| [14] | Ma Q, Lu AY. Pharmacogenetics, pharmacogenomics, and individualized medicine[J]. Pharmacol Rev , 2011, 63 :437–459. DOI:10.1124/pr.110.003533 |
| [15] | Johnson MD, Zuo H, Lee KH, et al. Pharmacological characterization of 4-hydroxy-N-desmethyl tamoxifen, a novel active metabolite of tamoxifen[J]. Breast Cancer Res Treat , 2004, 85 :151–159. DOI:10.1023/B:BREA.0000025406.31193.e8 |
| [16] | Stearns V, Johnson MD, Rae JM, et al. Active tamoxifen metabolite plasma concentrations after coadministration of tamoxifen and the selective serotonin reuptake inhibitor paroxetine[J]. J Natl Cancer Inst , 2003, 95 :1758–1764. DOI:10.1093/jnci/djg108 |
| [17] | Katzenellenbogen BS, Norman MJ, Eckert RL, et al. Bioac-tivities, estrogen receptor interactions, and plasminogen acti-vator-inducing activities of tamoxifen and hydroxy-tamoxifen isomers in MCF-7 human breast cancer cells[J]. Cancer Res , 1984, 44 :112–119. |
| [18] | Lim YC, Desta Z, Flockhart DA, et al. Endoxifen (4-hydroxy-N-desmethyl-tamoxifen) has anti-estrogenic effects in breast cancer cells with potency similar to 4-hydroxy-tamoxifen[J]. Cancer Chemother Pharmacol , 2005, 55 :471–478. DOI:10.1007/s00280-004-0926-7 |
| [19] | Mugundu GM, Sallans L, Guo Y, et al. Assessment of the impact of CYP3A polymorphisms on the formation of alpha-hydroxytamoxifen and N-desmethyltamoxifen in human liver microsomes[J]. Drug Metab Dispos , 2012, 40 :389–396. DOI:10.1124/dmd.111.039388 |
| [20] | Blevins-Primeau AS, Sun D, Chen G, et al. Functional significance of UDP-glucuronosyltransferase variants in the metabolism of active tamoxifen metabolites[J]. Cancer Res , 2009, 69 :1892–1900. DOI:10.1158/0008-5472.CAN-08-3708 |
| [21] | Squirewell EJ, Qin X, Duffel MW. Endoxifen and other metabolites of tamoxifen inhibit human hydroxysteroid sulfotransferase 2A1 (hSULT2A1)[J]. Drug Metab Dispos , 2014, 42 :1843–1850. DOI:10.1124/dmd.114.059709 |
| [22] | Konig J, Muller F, Fromm MF. Transporters and drug-drug interactions: important determinants of drug disposition and effects[J]. Pharmacol Rev , 2013, 65 :944–966. DOI:10.1124/pr.113.007518 |
| [23] | Eichelbaum M, Ingelman-Sundberg M, Evans WE. Pharma-cogenomics and individualized drug therapy[J]. Annu Rev Med , 2006, 57 :119–137. DOI:10.1146/annurev.med.56.082103.104724 |
| [24] | Griese EU, Zanger UM, Brudermanns U, et al. Assessment of the predictive power of genotypes for the in-vivo catalytic function of CYP2D6 in a German population[J]. Pharmaco-genetics , 1998, 8 :15–26. |
| [25] | Sachse C, Brockmoller J, Bauer S, et al. Cytochrome P450 2D6 variants in a Caucasian population: allele frequencies and phenotypic consequences[J]. Am J Hum Genet , 1997, 60 :284–295. |
| [26] | Sistonen J, Sajantila A, Lao O, et al. CYP2D6 worldwide genetic variation shows high frequency of altered activity variants and no continental structure[J]. Pharmacogenet Genomics , 2007, 17 :93–101. |
| [27] | Ji L, Pan S, Wu J, et al. Genetic polymorphisms of CYP2D6 in Chinese mainland[J]. Chin Med J (Engl) , 2002, 115 :1780–1784. |
| [28] | Zembutsu H. Pharmacogenomics toward personalized tamoxifen therapy for breast cancer[J]. Pharmacogenomics , 2015, 16 :287–296. DOI:10.2217/pgs.14.171 |
| [29] | Crews KR, Gaedigk A, Dunnenberger HM, et al. Clinical pharmacogenetics implementation consortium guidelines for cytochrome P450 2D6 genotype and codeine therapy: 2014 update[J]. Clin Pharmacol Ther , 2014, 95 :376–382. DOI:10.1038/clpt.2013.254 |
| [30] | Saladores P, Murdter T, Eccles D, et al. Tamoxifen metabo-lism predicts drug concentrations and outcome in premeno-pausal patients with early breast cancer[J]. Pharmacoge-nomics J , 2015, 15 :84–94. DOI:10.1038/tpj.2014.34 |
| [31] | Teh LK, Mohamed NI, Salleh MZ, et al. The risk of recurrence in breast cancer patients treated with tamoxifen: polymorphisms of CYP2D6 and ABCB1[J]. AAPS J , 2012, 14 :52–59. DOI:10.1208/s12248-011-9313-6 |
| [32] | Sukasem C, Sirachainan E, Chamnanphon M, et al. Impact of CYP2D6 polymorphisms on tamoxifen responses of women with breast cancer: a microarray-based study in Thailand[J]. Asian Pac J Cancer Prev , 2012, 13 :4549–4553. DOI:10.7314/APJCP.2012.13.9.4549 |
| [33] | Sirachainan E, Jaruhathai S, Trachu N, et al. CYP2D6 polymorphisms influence the efficacy of adjuvant tamoxifen in Thai breast cancer patients[J]. Pharmgenomics Pers Med , 2012, 5 :149–153. |
| [34] | Hertz DL, McLeod HL, Irvin WJ Jr. Tamoxifen and CYP2D6: a contradiction of data[J]. Oncologist , 2012, 17 :620–630. DOI:10.1634/theoncologist.2011-0418 |
| [35] | Kiyotani K, Mushiroda T, Imamura CK, et al. Significant effect of polymorphisms in CYP2D6 and ABCC2 on clinical outcomes of adjuvant tamoxifen therapy for breast cancer patients[J]. J Clin Oncol , 2010, 28 :1287–1293. DOI:10.1200/JCO.2009.25.7246 |
| [36] | Goetz MP, Kamal A, Ames MM. Tamoxifen pharmacoge-nomics: the role of CYP2D6 as a predictor of drug response[J]. Clin Pharmacol Ther , 2008, 83 :160–166. DOI:10.1038/sj.clpt.6100367 |
| [37] | Goetz MP, Suman VJ, Hoskin TL, et al. CYP2D6 metabo-lism and patient outcome in the Austrian breast and colorectal cancer study group trial (ABCSG) 8[J]. Clin Cancer Res , 2013, 19 :500–507. DOI:10.1158/1078-0432.CCR-12-2153 |
| [38] | Phan M, Venitz J. Summary Minutes of the Advisory Committee Pharmaceutical Science Clinical Pharmacology Subcommittee[M]. Silver Spring, MD: U.S. Food and Drug Administration, 2006 . |
| [39] | Hennig EE, Piatkowska M, Karczmarski J, et al. Limited predictive value of achieving beneficial plasma Z-endoxifen threshold level by CYP2D6 genotyping in tamoxifen-treated Polish women with breast cancer[J]. BMC Cancer , 2015 . DOI:10.1186/s12885-12015-11575-12884 |
| [40] | Kiyotani K, Mushiroda T, Tsunoda T, et al. A genome-wide association study identifies locus at 10q22 associated with clinical outcomes of adjuvant tamoxifen therapy for breast cancer patients in Japanese[J]. Hum Mol Genet , 2012, 21 :1665–1672. DOI:10.1093/hmg/ddr597 |
| [41] | Mwinyi J, Vokinger K, Jetter A, et al. Impact of variable CYP genotypes on breast cancer relapse in patients undergoing adjuvant tamoxifen therapy[J]. Cancer Chemother Pharmacol , 2014, 73 :1181–1188. DOI:10.1007/s00280-014-2453-5 |
| [42] | Madlensky L, Natarajan L, Tchu S, et al. Tamoxifen metabolite concentrations, CYP2D6 genotype, and breast cancer outcomes[J]. Clin Pharmacol Ther , 2011, 89 :718–725. DOI:10.1038/clpt.2011.32 |
| [43] | Murdter TE, Schroth W, Bacchus-Gerybadze L, et al. Activity levels of tamoxifen metabolites at the estrogen receptor and the impact of genetic polymorphisms of phase I and II enzymes on their concentration levels in plasma[J]. Clin Pharmacol Ther , 2011, 89 :708–717. DOI:10.1038/clpt.2011.27 |
| [44] | Teft WA, Gong IY, Dingle B, et al. CYP3A4 and seasonal variation in vitamin D status in addition to CYP2D6 contribute to therapeutic endoxifen level during tamoxifen therapy[J]. Breast Cancer Res Treat , 2013, 139 :95–105. DOI:10.1007/s10549-013-2511-4 |
| [45] | Lim JS, Chen XA, Singh O, et al. Impact of CYP2D6, CYP3A5, CYP2C9 and CYP2C19 polymorphisms on tamoxifen pharmacokinetics in Asian breast cancer patients[J]. Br J Clin Pharmacol , 2011, 71 :737–750. DOI:10.1111/j.1365-2125.2011.03905.x |
| [46] | Zafra-Ceres M, de Haro T, Farez-Vidal E, et al. Influence of CYP2D6 polymorphisms on serum levels of tamoxifen metabolites in Spanish women with breast cancer[J]. Int J Med Sci , 2013, 10 :932–937. DOI:10.7150/ijms.5708 |
| [47] | Fernandez-Santander A, Gaibar M, Novillo A, et al. Rela-tionship between genotypes Sult1a2 and CYP2D6 and tamoxifen metabolism in breast cancer patients[J]. PLoS One , 2013, 8 :e70183. DOI:10.1371/journal.pone.0070183 |
| [48] | Park IH, Ro J, Park S, et al. Lack of any association between functionally significant CYP2D6 polymorphisms and clinical outcomes in early breast cancer patients receiving adjuvant tamoxifen treatment[J]. Breast Cancer Res Treat , 2012, 131 :455–461. DOI:10.1007/s10549-011-1425-2 |
| [49] | Barginear MF, Jaremko M, Peter I, et al. Increasing tamoxifen dose in breast cancer patients based on CYP2D6 genotypes and endoxifen levels: effect on active metabolite isomers and the antiestrogenic activity score[J]. Clin Pharmacol Ther , 2011, 90 :605–611. DOI:10.1038/clpt.2011.153 |
| [50] | Stingl JC, Parmar S, Huber-Wechselberger A, et al. Impact of CYP2D6*4 genotype on progression free survival in tamoxifen breast cancer treatment[J]. Curr Med Res Opin , 2010, 26 :2535–2542. DOI:10.1185/03007995.2010.518304 |
| [51] | Lash TL, Cronin-Fenton D, Ahern TP, et al. CYP2D6 inhibition and breast cancer recurrence in a population-based study in Denmark[J]. J Natl Cancer Inst , 2011, 103 :489–500. DOI:10.1093/jnci/djr010 |
| [52] | Abraham JE, Maranian MJ, Driver KE, et al. CYP2D6 gene variants: association with breast cancer specific survival in a cohort of breast cancer patients from the United Kingdom treated with adjuvant tamoxifen[J]. Breast Cancer Res , 2010, 12 :R64. DOI:10.1186/bcr2629 |
| [53] | Park HS, Choi JY, Lee MJ, et al. Association between genetic polymorphisms of CYP2D6 and outcomes in breast cancer patients with tamoxifen treatment[J]. J Korean Med Sci , 2011, 26 :1007–1013. DOI:10.3346/jkms.2011.26.8.1007 |
| [54] | Chamnanphon M, Pechatanan K, Sirachainan E, et al. Association of CYP2D6 and CYP2C19 polymorphisms and disease-free survival of Thai post-menopausal breast cancer patients who received adjuvant tamoxifen[J]. Pharmgenomics Pers Med , 2013, 6 :37–48. |
| [55] | Rae JM, Drury S, Hayes DF, et al. CYP2D6 and UGT2B7 genotype and risk of recurrence in tamoxifen-treated breast cancer patients[J]. J Natl Cancer Inst , 2012, 104 :452–460. DOI:10.1093/jnci/djs126 |
| [56] | Regan MM, Leyland-Jones B, Bouzyk M, et al. CYP2D6 genotype and tamoxifen response in postmenopausal women with endocrine-responsive breast cancer: the breast interna-tional group 1-98 trial[J]. J Natl Cancer Inst , 2012, 104 :441–451. DOI:10.1093/jnci/djs125 |
| [57] | Zhang X, Pu Z, Ge J, et al. Association of CYP2D6*10, OATP1B1 A388G, and OATP1B1 T521C polymorphisms and overall survival of breast cancer patients after tamoxifen therapy[J]. Med Sci Monit , 2015, 21 :563–569. DOI:10.12659/MSM.893473 |
| [58] | Guengerich FP. Cytochromes P450, drugs, and diseases[J]. Mol Interv , 2003, 3 :194–204. DOI:10.1124/mi.3.4.194 |
| [59] | Wakeling AE, Slater SR. Estrogen-receptor binding and biologic activity of tamoxifen and its metabolites[J]. Cancer Treat Rep , 1980, 64 :741–744. |
| [60] | Johnson N, Walker K, Gibson LJ, et al. CYP3A variation, premenopausal estrone levels, and breast cancer risk[J]. J Natl Cancer Inst , 2012, 104 :657–669. DOI:10.1093/jnci/djs156 |
| [61] | Lee JS, Cheong HS, Kim LH, et al. Screening of genetic polymorphisms of CYP3A4 and CYP3A5 genes[J]. Korean J Physiol Pharmacol , 2013, 17 :479–484. DOI:10.4196/kjpp.2013.17.6.479 |
| [62] | Su HI, Sammel MD, Velders L, et al. Association of cyclophosphamide drug-metabolizing enzyme polymorphisms and chemotherapy-related ovarian failure in breast cancer survivors[J]. Fertil Steril , 2010, 94 :645–654. DOI:10.1016/j.fertnstert.2009.03.034 |
| [63] | Antunes MV, de Oliveira V, Raymundo S, et al. CYP3A4*22 is related to increased plasma levels of 4-hydroxytamoxifen and partially compensates for reduced CYP2D6 activation of tamoxifen[J]. Pharmacogenomics , 2015, 16 :601–617. DOI:10.2217/pgs.15.13 |
| [64] | ter Heine R, Binkhorst L, de Graan AJ, et al. Population pharmacokinetic modelling to assess the impact of CYP2D6 and CYP3A metabolic phenotypes on the pharmacokinetics of tamoxifen and endoxifen[J]. Br J Clin Pharmacol , 2014, 78 :572–586. DOI:10.1111/bcp.2014.78.issue-3 |
| [65] | Baxter SD, Teft WA, Choi YH, et al. Tamoxifen-associated hot flash severity is inversely correlated with endoxifen concentration and CYP3A4*22[J]. Breast Cancer Res Treat , 2014, 145 :419–428. DOI:10.1007/s10549-014-2963-1 |
| [66] | Tucker AN, Tkaczuk KA, Lewis LM, et al. Polymorphisms in cytochrome P4503A5 (CYP3A5) may be associated with race and tumor characteristics, but not metabolism and side effects of tamoxifen in breast cancer patients[J]. Cancer Lett , 2005, 217 :61–72. DOI:10.1016/j.canlet.2004.08.027 |
| [67] | Wegman P, Elingarami S, Carstensen J, et al. Genetic variants of CYP3A5, CYP2D6, SULT1A1, UGT2B15 and tamoxifen response in postmenopausal patients with breast cancer[J]. Breast Cancer Res , 2007, 9 :R7. DOI:10.1186/bcr1640 |
| [68] | Gjerde J, Geisler J, Lundgren S, et al. Associations between tamoxifen, estrogens, and FSH serum levels during steady state tamoxifen treatment of postmenopausal women with breast cancer[J]. BMC Cancer , 2010 . DOI:10.1186/1471-2407-1110-1313 |
| [69] | Yin SJ, Ni YB, Wang SM, et al. Differences in genotype and allele frequency distributions of polymorphic drug metabolizing enzymes CYP2C19 and CYP2D6 in mainland Chinese Mongo-lian, Hui and Han populations[J]. J Clin Pharm Ther , 2012, 37 :364–369. DOI:10.1111/jcp.2012.37.issue-3 |
| [70] | Rangel LB, Taraba JL, Frei CR, et al. Pharmacogenomic diversity of tamoxifen metabolites and estrogen receptor genes in Hispanics and non-Hispanic whites with breast cancer[J]. Breast Cancer Res Treat , 2014, 148 :571–580. DOI:10.1007/s10549-014-3191-4 |
| [71] | Schroth W, Antoniadou L, Fritz P, et al. Breast cancer treatment outcome with adjuvant tamoxifen relative to patient CYP2D6 and CYP2C19 genotypes[J]. J Clin Oncol , 2007, 25 :5187–5193. DOI:10.1200/JCO.2007.12.2705 |
| [72] | Moyer AM, Suman VJ, Weinshilboum RM, et al. SULT1A1, CYP2C19 and disease-free survival in early breast cancer patients receiving tamoxifen[J]. Pharmacogenomics , 2011, 12 :1535–1543. DOI:10.2217/pgs.11.97 |
| [73] | Greer AK, Dates CR, Starlard-Davenport A, et al. A potential role for human UDP-glucuronosyltransferase 1A4 promoter single nucleotide polymorphisms in the pharmacogenomics of tamoxifen and its derivatives[J]. Drug Metab Dispos , 2014, 42 :1392–1400. DOI:10.1124/dmd.114.058016 |
| [74] | Ahern TP, Christensen M, Cronin-Fenton DP, et al. Func-tional polymorphisms in UDP-glucuronosyl transferases and recurrence in tamoxifen-treated breast cancer survivors[J]. Cancer Epidemiol Biomarkers Prev , 2011, 20 :1937–1943. DOI:10.1158/1055-9965.EPI-11-0419 |
| [75] | Raftogianis RB, Wood TC, Otterness DM, et al. Phenol sulfotransferase pharmacogenetics in humans: association of common SULT1A1 alleles with TS PST phenotype[J]. Biochem Biophys Res Commun , 1997, 239 :298–304. DOI:10.1006/bbrc.1997.7466 |
| [76] | Cronin-Fenton DP, Damkier P, Lash TL. Metabolism and transport of tamoxifen in relation to its effectiveness: new perspectives on an ongoing controversy[J]. Future Oncol , 2014, 10 :107–122. DOI:10.2217/fon.13.168 |
| [77] | Teft WA, Mansell SE, Kim RB. Endoxifen, the active metabolite of tamoxifen, is a substrate of the efflux transporter P-glycoprotein (multidrug resistance 1)[J]. Drug Metab Dispos , 2011, 39 :558–562. DOI:10.1124/dmd.110.036160 |
| [78] | Iusuf D, Teunissen SF, Wagenaar E, et al. P-glycoprotein (ABCB1) transports the primary active tamoxifen metabolites endoxifen and 4-hydroxytamoxifen and restricts their brain penetration[J]. J Pharmacol Exp Ther , 2011, 337 :710–717. DOI:10.1124/jpet.110.178301 |
| [79] | Sensorn I, Sirachainan E, Chamnanphon M, et al. Association of CYP3A4/5, ABCB1 and ABCC2 polymorphisms and clinical outcomes of Thai breast cancer patients treated with tamoxifen[J]. Pharmgenomics Pers Med , 2013, 6 :93–98. |
| [80] | Argalacsova S, Slanar O, Vitek P, et al. Contribution of ABCB1 and CYP2D6 genotypes to the outcome of tamoxifen adjuvant treatment in premenopausal women with breast cancer[J]. Physiol Res , 2015, 64 :S539–8547. |
| [81] | Kauffmann HM, Keppler D, Gant TW, et al. Induction of hepatic mrp2 (cmrp/cmoat) gene expression in nonhuman primates treated with rifampicin or tamoxifen[J]. Arch Toxicol , 1998, 72 :763–768. DOI:10.1007/s002040050571 |
| [82] | Choi HK, Cho KB, Phuong NT, et al. SIRT1-mediated FoxO1 deacetylation is essential for multidrug resistance-associated protein 2 expression in tamoxifen-resistant breast cancer cells[J]. Mol Pharm , 2013, 10 :2517–2527. DOI:10.1021/mp400287p |
| [83] | Filipits M, Pohl G, Rudas M, et al. Clinical role of multidrug resistance protein 1 expression in chemotherapy resistance in early-stage breast cancer: the Austrian Breast and Colorectal Cancer Study Group[J]. J Clin Oncol , 2005, 23 :1161–1168. DOI:10.1200/JCO.2005.03.033 |
| [84] | Li W, Jia M, Qin X, et al. Harmful effect of ERβ on BCRP-mediated drug resistance and cell proliferation in ERα/PR-negative breast cancer[J]. FEBS J , 2013, 280 :6128–6140. DOI:10.1111/febs.12533 |
| [85] | Pu Z, Zhang X, Chen Q, et al. Establishment of an expression platform of OATP1B1 388GG and 521CC genetic polymorphism and the therapeutic effect of tamoxifen in MCF-7 cells[J]. Oncol Rep , 2015, 33 :2420–2428. |
| [86] | Province MA, Goetz MP, Brauch H, et al. CYP2D6 genotype and adjuvant tamoxifen: meta-analysis of heterogeneous study populations[J]. Clin Pharmacol Ther , 2014, 95 :216–227. DOI:10.1038/clpt.2013.186 |
 2016, Vol. 51
2016, Vol. 51


