核因子-κB (nuclear factor-kappa B,NF-κB) 是一类参与细胞黏附、分化、增殖和凋亡等多个细胞过 [1, 2, 3, 4, 5]。NF-κB家族由p50/NF-κB1、p52/NF-κB2、REL/ cREL、REL-A/p65和REL-B组成,以上蛋白质以同源或异源二聚体的形式组成具有功能的NF-κB[6]。不同的NF-κB蛋白二聚体具有不同的结合序列,识别不同的DNA靶标,从而调节不同基因的表 达。通常情况下,大部分的NF-κB二聚体存在于细胞质中,与包括NF-κB抑制蛋白α亚基 (NF-kappa-B inhibitor alpha,IκBα,由NFKBIA基因编码) 在内的三种抑制因子之一结合而保持无活性状态[7]。在炎症反应等产生的细胞因子刺激下,IκBα发生磷酸化及泛素化并迅速被蛋白酶体解离,解除了与IκBα结合状态的NF- κB得以进入细胞核内与特定DNA序列结合,从而发挥其对下游靶基因的转录调控作用[8]。研究[9]发现,NF-κB的结构元件p65能够竞争性地与RXR结合,影响PXR/RXR复合物的形成,减少PXR/RXR复合物与其相应基因调控序列的相互作用,从而抑制PXR对相关基因调控作用的发挥,影响受其调控的包括CYP3A4和MDR1等在内的基因表 达。环孢素A (CsA) 是CYP3A4、CYP3A5及P-糖蛋白 (P-glycoprotein,P-gp,由MDR1基因编码) 的底物[10, 11]。本实验室前期研究[12]发现,NFKBIA基因的两个SNP-2758G > A (rs696)、rs8904C > T与肾移植患者术后早期CsA血药浓度显著相关。rs8904与rs696都是存在于NFKBIA 3'非翻译区 (3' untranslated region,3'UTR) 上的位点,并且二者存在强连锁不平衡,然而截至到目前该两位点的具体生物学机制还不十分清楚。本研究拟构建含有以上2个位点的4种单倍型荧光素酶报告基因载体,并验证其功能,为确证上述2个位点发挥功能的机制提供依据。
材料与方法 试剂与仪器引物、DNA测序 (中国上海生工生物科技公司); LS174T细胞(中国科学院上海细胞库); JM110感受态细胞 (中国Biowit公司); pGEM®-T easy、pGL3-promoter载体质粒、DNA T4连接酶、双荧光素酶报告基因检测系统 (美国Promega公司); pRL-TK、Ex Taq酶 (日本Takara公司); 限制性内切酶XbaI、去磷酸化酶CIP (美国NEB公司); DNA凝胶回收试剂盒、质粒提取试剂盒 (中国TIANGEN公司); DMEM/高糖培养基 (美国Hyclone,Thermo Fisher Scientific公司); 胎牛血清 (美国GIBICO,Invitrogen公司); LipofectamineTM 2000转染试剂 (美国Invitrogen公司)。
NFKBIA基因3'UTR片段的获取根据GenBank (基因号: NM_020529) 确定NFKBIA基因3'UTR区序列 (503 bp),利用Premier5.0软件设计PCR引物,上游引物3'UTR PF序列和下游引物3'UTR PR序列中黑体下划线为XbaI酶切位点,之前的序列为保护碱基,见表 1。选取经过测序鉴定rs8904为CC型(该位点在3'UTR片段的第2位)、rs696为GA型(该位点在3'UTR片段的第126位) 且在3'UTR片段中无其他变异位点的中山大学附属第一医院肾移植患者DNA样本作为模板。患者为前期研究纳入的受试对象,在进行研究前已签署知情同意书。rs8904 T等位基因通过在3'UTR PF序列中引入突变 (MutPF中斜体加下划线位置) 获得 (表 1)。
| Table 1 The primer sequences of NFKBIA 3'UTR |
PCR反应体系25 μL,包括10× Ex Taq Buffer 2.5 μL、dNTP Mixture (各2.5 mmol·L−1) 2 μL、Forward Primer (10 μmol·L−1) 1 μL、Reverse Primer (10 μmol·L−1) 1 μL、Takara® Ex Taq (5 U·μL−1) 0.15 μL和全基因组DNA 3 μL。
PCR反应条件: 94 ℃预变性5 min; 94 ℃变性 30 s,60 ℃退火30 s,72 ℃延伸60 s,30个循环; 72 ℃延伸7 min。
双荧光素酶基因报告质粒及突变体的构建PCR反应产物在1% 琼脂糖凝胶上电泳,切胶回收后并且将其与pGEM®-T easy 载体连接,转化入JM110感受态细胞,培养后对质粒测序验证以保证目的片段序列正确。T-载体连接产物经Xba I酶切,pGL3-promoter载体经Xba I酶切、CIP酶去磷酸化,电泳分离目的片段切胶回收纯化后,在T4 DNA连接酶作用下进行连接,使目的片段插入线性化后的pGL3-promoter载体中,得到重组质粒。然后转化至JM110感受态细胞,涂在含有氨苄霉素的LB琼脂培养基上,于37 ℃过夜培养,挑取阳性单克隆,于含LB培养液5 mL的试 管中37 ℃摇床(240 r·min−1) 过夜培养。根据质粒提取试剂盒操作说明书提取目的重组质粒,经Xba I酶单酶切电泳验证后,将重组质粒送上海生工生物科技公司进行测序鉴定。将鉴定为目标序列且连接方向正确的重组双荧光素酶报告载体分别命名为pGL3- rs8904C/rs696G、pGL3-rs8904C/rs696A、pGL3-rs8904T/ rs696G和pGL3-rs8904T/rs696A。
双荧光素酶基因报告分析在37 ℃、5% CO2的条件下培养LS174T细胞,以60%~70% 的细胞密度接种于96孔板中,每孔总体积100 μL,待细胞生长至对数生长期时,取OPTI-MEM培养基25 μL稀释重组质粒和内参pRL-TK,OPTI-MEM培养基25 μL稀释LipofectamineTM 2000试剂,于5 min后将两者混合,轻轻摇匀,静置20 min形成转染复合物,将其加到细胞培养板的各孔中。其中,重组质粒转染为每孔100 ng,内参pRL-TK为每孔5 ng,每组设4个复孔。于6 h后更换含10% 胎牛血清的DMEM培养基。在转染24 h后裂解细胞,按双荧光素酶报告基因检测系统说明书操作,检测其荧光素酶的活性。
统计学处理采用GraphPad Prism5统计软件分析实验数据。数据以x±s表 示; 用t检验比较不同组间的报告基因表 达水平的差异。P < 0.05为差异有统计学意义。
结果 1 获取NFKBIA 3'UTR片段本研究设计了包含rs8904C等位基因或T等位 基因的NFKBIA 3'UTR全片段扩增引物,以rs8904为CC型、rs696为GA型的人全基因组DNA为模板,PCR扩增产物长度为503 bp,扩增的片段大小与预期相符。
2 构建pGL3-NFKBIA-3'UTR质粒 2.1 重组质粒酶切鉴定重组质粒经限制性内切酶Xba I酶切后,1% 琼脂糖凝胶电泳结果显示两条带,一条为线性pGL3-promoter载体 (约5 000 bp),一条与PCR扩增出的目的片段 (约500 bp) 大小接近。载体及插入片段显示在相应位置,如图 1。
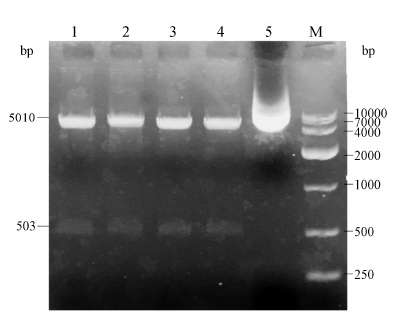 | Figure 1 Restriction enzyme analysis of pGL3-3'UTR recombinant plasmids. M: DL10 000 DNA marker; 1: pGL3-rs8904C/ rs696G digested with XbaI; 2: pGL3-rs8904C/rs696A digested with XbaI; 3: pGL3-rs8904T/rs696G digested with XbaI; 4: pGL3- rs8904T/rs696A digested with XbaI; 5: Recombinant plasmid without digestion |
经DNA测序,结果显示 4种NFKBIA 3'UTR目的片段 (由rs8904与rs696 构成的C/G、C/A、T/G、T/A 4种单倍型) 除2个目标SNP与实验设计相符,其余序列均与GenBank中NFKBIA 3'UTR序列一致 (图 2),且插入方向正确。至此,4种重组质粒pGL3-rs8904C/rs696G、pGL3- rs8904C/rs696A、pGL3-rs8904T/rs696G和pGL3- rs8904T/rs696A构建成功 (图 3)。
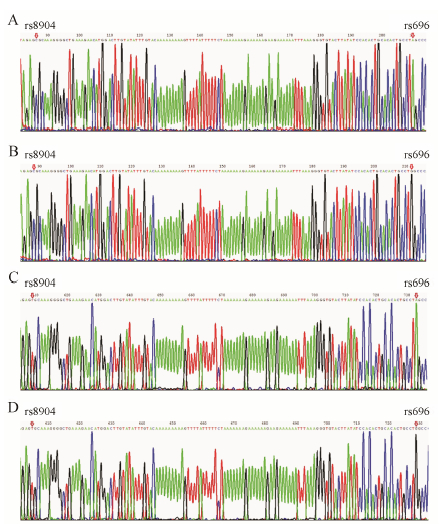 | Figure 2 Plasmid sequencing validation of two SNPs at rs8904 and rs696 mutant C to T and G to A respectively. A: Base C at rs8904 site and base A at rs696 site; B: Base C at rs8904 site and base G at rs696 site; C: Base T at rs8904 site and base A at rs696 site; D: Base T at rs8904 site and base G at rs696 site |
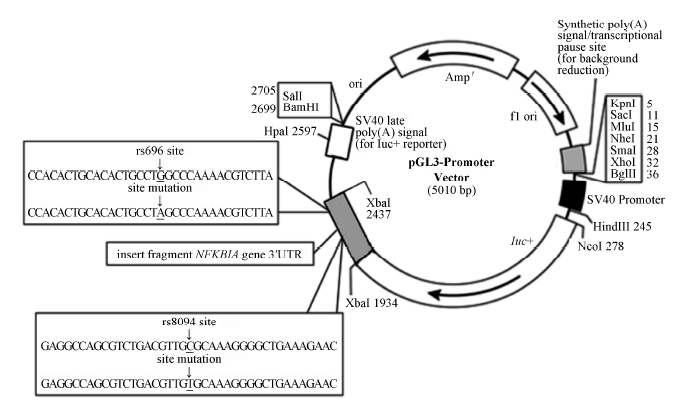 | Figure 3 Schematic diagram of recombinant plasmids |
将构建好的4种重组质粒及阴性对照pGL3-vector分别转染LS174T细胞。由双荧光素酶报告基因结果可知 (图 4),与转染了pGL3-vector组(阴性对照) 相比,4组转染重组双荧光素酶报告质粒的荧光素酶活性均显著下降 (P < 0.001)。对于rs696G > A,含等位基因A的两种重组质粒pGL3-rs8904C/rs696A和pGL3-rs8904T/rs696A的荧光素酶活性分别比含等位基因G的两种重组 质粒pGL3-rs8904C/rs696G和pGL3-rs8904T/rs696G下降约45.1% (P < 0.05) 和56.1% (P < 0.001)。对于rs8904C > T,含等位基因T的两种重组质粒pGL3- rs8904T/rs696G和pGL3-rs8904T/rs696A的荧光素 酶活性分别与含等位基因C的两种重组质粒pGL3- rs8904C/rs696G和pGL3-rs8904C/rs696A比较,其荧光素酶活性无显著性差异 (P > 0.05)。
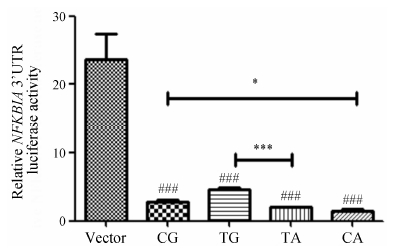 | Figure 4 The assays for LS174T cells which were transfected with luciferase reporter vectors containing different kinds of haplotypes. n = 4, x±s. P < 0.05, ***P < 0.001. ###P < 0.001 vs pGL3-vector group |
运用药物基因组学寻找相应生物标记物,在患者服药前预测其所需剂量,以期其血药浓度尽早处于治疗窗,提高疗效及减少不良反应的发生是近年来药物基因组学的研究热点之一[13]。位于基因编码区的基因变异一直是药物基因组学关注的焦点。但近年来,非翻译区在基因表 达调控中的重要作用逐渐被发现,比如3'UTR在mRNA末端PolyA的加工、控制mRNA的稳定性及降解速率、协助识别特殊密码子和调控mRNA的翻译等转录后的调控过程中发挥重要作用[14, 15]。具有功能性的3'UTR多态性也在人和动物体内发现并得到验证,位于3'UTR的基因多态性影响基因的表 达情况也已日渐受到关注[16, 17, 18, 19]。
目的基因片段体外克隆,并研究其功能的相关技术也日渐成熟,屡见报道[20, 21]。本实验室前期 研究发现位于NFKBIA基因3'UTR的rs8904C > T、rs696G > A位点显著影响肾移植患者术后的早期环抱素A血药浓度。其中,rs696位点的AA型携带者的CsA剂量校正谷浓度显著高于GA型 (P = 0.001) 及GG型携带者 (P < 0.001); rs8904 TT型携带者的CsA将剂量校正谷浓度显著高于CC型 (P = 0.003) 及CT型携带者 (P = 0.015)。因此,作者推测这两个位点发生突变可能会导致IκBα水平降低,从而导致其与NF-κB的结合量下降,使得NF-κB水平升高。增多的NF-κB进而影响PXR功能的发挥,导致PXR下游CYP3A4及CYP3A5水平降低,减弱对环孢素A的代谢,从而使得人体内环孢素A的血药浓度增高。而本研究发现rs696的G→A突变会导致荧光素酶活性水平降低,与之前推断该SNP可能会下调IκBα表 达水平一致。然而,本研究并未发现rs8904C > T对荧光素酶活性的影响,由于该位点与rs696G > A存在强连锁不平衡,推测该位点对CsA浓度的影响可能来源于rs696G > A的作用。本研究仅是通过构建报告基因的方法考察SNP对荧光素酶活性的影响,然而rs696G > A对IκBα与NF-κB的结合、PXR功能的发挥、CYP3A4及CYP3A5表 达水平的影响,以致对CsA代谢情况的影响的确切机制仍有待下一步的深入研究。
本研究还发现,与转染了pGL3-vector组 (阴性对照) 相比,转染重组双荧光素酶报告质粒的荧光素酶活性均显著下降 (P < 0.001),这与Li等[22]的发现一致,该研究发现NFKBIA 3'UTR中存在负调控基因 表 达的调控元件从而能显著下调荧光素酶报告基因的表 达。此外,3'UTR也是microRNA (miRNA) 的主要结合区域。miRNA可以通过与目标mRNA的3'UTR发生不完全结合,抑制该mRNA的翻译过程,或者导致目的基因降解和沉默,参与蛋白的转录后调控[23, 24]。已有研究发现,在miRNA靶基因结合位点上的基因变异如SNP,可以影响miRNA与靶基因mRNA的亲和力,改变该基因的降解程度,从而影响特定基因的表 达[25, 26]。因此,rs8904C > T和rs696G > A是否也会通过影响相关miRNA与NFKBIA 3'UTR的结合而影响其基因表 达仍有待下一步的验证。
本研究通过构建重组NFKBIA基因3'UTR野生型及突变型报告质粒,发现rs696G→A突变对荧光素酶活性有显著影响,这些重组质粒的构建也可为未来研究NFKBIA基因与miRNA相互作用奠定基础。
| [1] | Gilmore TD. Introduction to NF-κB: players, pathways, perspectives [J]. Oncogene, 2006, 25: 6680-6684. |
| [2] | Chen CD, Sawyers CL. NF-κB activates prostate-specific antigen expression and is upregulated in androgen-independent prostate cancer [J]. Mol Cell Biol, 2002, 22: 2862-2870. |
| [3] | Sen R, Baltimore D. Inducibility of κ immunoglobulin enhancer-bingding protein NF-κB by a postranslational mechanism [J]. Cell, 1986, 47: 921-928. |
| [4] | Baeuerle PA. IκB-NF-κB structures: at the interface of inflammation control [J]. Cell, 1998, 95: 729-731. |
| [5] | Baeuerle PA, Henkel T. Function and activation of NF-κB in the immune system [J]. Annu Rev Immunol, 1994, 12: 141-179. |
| [6] | Huxford T, Mishler D, Phelps CB, et al. Solvent exposed non-contacting amino acids play a critical role in NF-κB/IκBα complex formation [J]. J Mol Biol, 2002, 324: 587-597. |
| [7] | Dyson HJ, Komives EA. Role of disorder in IκB-NFκB interaction [J]. IUBMB Life, 2012, 64: 499-505. |
| [8] | Karin M, Ben-Neriah Y. Phosphorylation meets ubiquitination: the control of NF-κB activity [J]. Annu RevImmunol, 2000, 18: 621-663. |
| [9] | Gu X, Ke S, Liu D, et al. Role of NF-κB in regulation of PXR-mediated gene expression: a mechnaism for the suppression of cytochrome P-450 3A4 by proinflammatroy agents [J]. J Biol Chem, 2006, 281: 17882-17889. |
| [10] | Christians U, Strom T, Zhang YL, et al. Active drug transport of immunosuppressants: new insights for pharmacokinetics and pharmacodynamics [J]. Ther Drug Monit, 2006, 28: 39-44. |
| [11] | Jibodh RA, Lagas JS, Nuijen B, et al. Taxanes: old drugs, new oral formulations [J]. Eur J Pharmacol, 2013, 717: 40-46. |
| [12] | Zhang Y. Regulation of Polygenetic Network on Cyclosporine Concentrations in Chinese Renal Transplant Recipients (多基因网络调控下环孢素A在中国肾移植患者中的血药浓度研究) [D]. Guangzhou: Sun Yat-sen University, 2013. |
| [13] | Liu XM, Li JL, Wang HY, et al. Associations of SUMO4 polymorphisms with tacrolimus concentrations in Chinese renal transplant recipients [J]. Acta Pharm Sin (药学学报), 2015, 50: 180-184. |
| [14] | Wang L, Zhang FJ, Li ZY, et al. Cloning and sequence comparison of 3'-UTRs of antifreeze protein genes from Oodescelis chinensis and Tenebrio molitor [J]. Genomics Appl Biol (基因组学与应用生物学), 2015, 34: 692-699. |
| [15] | Zhang R, Dong DL, Yang BF. Basic research and application of microRNA-a novel target for regulating cardiac arrhythmias [J]. Acta Pharm Sin (药学学报), 2011, 46: 6-11. |
| [16] | Gervasini G, Garcia-Cerrada M, Coto E, et al. A 3'-UTR polymorphism in soluble epoxide hydrolase gene is associated with acute rejection in renal transplant recipients [J]. PLoS One, 2015, 10: e0133563. |
| [17] | Zambrano T, Hirata MH, Cerda Á, et al. Impact of 3'UTR genetic variants in PCSK9 and LDLR genes on plasma lipid traits and response to atorvastatin in Brazilian subjects: a pilot study [J]. Int J Clin Exp Med, 2015, 8: 5978-5988. |
| [18] | Sugimoto M, Gotoh Y, Kawahara T, et al. Molecular effects of polymorphism in the 3'UTR of Unc-5 homolog C associated with conception rate in holsteins [J]. PLoS One, 2015, 10: e0131283. |
| [19] | Mazumder B, Seshadri V, Fox PL. Translational control by the 3'-UTR: the ends specify the means [J]. Trends Biochem Sci, 2003, 28: 91-98. |
| [20] | Liang L, Guo QM, Zhang Z, et al. Cloning, prokaryotic expression, and functional identification of a sesquiterpene synthase gene (AsSS4) from Aquilaria sinensis [J]. Acta Pharm Sin (药学学报), 2014, 49: 1724-1729. |
| [21] | Jiang D, Rong QX, Yuan QJ, et al. Cloning and prokaryotic expression analysis of HDS from Salvia miltiorrhiza bge.f.alba [J]. Acta Pharm Sin (药学学报), 2014, 49: 1614-1620. |
| [22] | Li T. Preliminary Functional Studies and Identification of Interacting Protein for 3'UTR of NFBIA (NFKBIA 3'UTR功 能的初步研究及其相互作用蛋白的鉴定) [D]. Guangzhou: Southern Medical University, 2011. |
| [23] | Lee YS, Nakahara K, Pham JW, et al. Distinct roles for Drosophila Dicer-1 and Dicer-2 in the siRNA/miRNA silencing pathways [J]. Cell, 2004, 117: 69-81. |
| [24] | Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function [J]. Cell, 2004, 116: 281-297. |
| [25] | Clop A, Marcq F, Takeda H, et al. A mutation creating a potential illegitimate microRNA target site in the myostatin gene affects muscularity in sheep [J]. Nat Genet, 2006, 38: 813-818. |
| [26] | Mishra PJ, Humeniuk R, Mishra PJ, et al. A miR-24 microrna binding-site polymorphism in dihydrofolate reductase gene leads to methotrexate resistance [J]. Proc Natl Acad Sci U S A, 2007, 104: 13513-13518. |
 2016, Vol. 51
2016, Vol. 51


