2. 中国中医科学院中药资源中心, 道地药材国家重点实验室培育基地, 北京 100700
2. State Key laboratory of Dao-di Herbs, National Resources Center for Chinese Materia Medica, China Academy of Chinese Medical Sciences, Beijing 100700, China
金银花是临床常用的一种中药材,来源于忍冬科植物忍冬 (Lonicera japonica Thunb.) 的干燥花蕾或带初开的花,主要活性成分为黄酮类、有机酸、挥发油、三萜皂苷等[1, 2, 3],具有清热解毒、抑菌、抗病毒等作用[4, 5]。红白忍冬 (L. japonica var. chinensis (Wats.) Bak),别名红花忍冬、红金银花,为金银花的野生变种,其花色艳丽、香味浓郁,具有重要的药用及观赏价值[6],其与金银花在性状、成分上存在显著差异[5]。
为了进一步了解金银花活性成分变异的分子机制,本课题组在建立以活性成分为导向的金银花活性成分基因调控网络基础上,比较了金银花、红金银花活性成分生物合成关键酶基因的转录水平与基因结构变化。结果表明,尽管二者在转录水平上存在显著差异[7],但其氨基酸序列未发现变异。
传统遗传学认为基因结构的变异影响了基因表达,而表观遗传学则认为即使在DNA序列不变的情况下,基因表达也可产生可遗传的变化[8]。其中DNA甲基化是一种重要的表观遗传修饰方式,参与了众多的生物学过程: 如胚胎发育、细胞分化、基因的表达调控、基因组印记、X染色体失活等[9]。在植物的生长发育和环境应答过程中,DNA去甲基化可以激活一些特殊基团的功能并且对基因组后生状态进行重置,包括被动去甲基化和主动去甲基化[10]。DNA去甲基化机制主要包括: ① DNA葡糖基酶参与的碱基切除修复; ② 脱氨酶参与的碱基切除修复; ③ 核苷酸外切除修复机制; ④ 氧化去甲基化等[11]。
DNA的去甲基化水平取决于DNA去甲基化酶,其含有最保守的DNA糖苷酶结构域,包括DME、ROS1、DML2、DML3、DML4、DML5等6个亚家族[12]。目前关于DNA去甲基化酶的报道主要集中在拟南芥[13, 14, 15]、水稻[16, 17]等模式植物中,金银花类药用植物DNA去甲基化酶的理化性质及其表达模式的研究鲜见报道。本文运用生物信息学方法从转录组数据中筛选获得4个金银花DNA去甲基化酶基因,并分析了其核苷酸及编码蛋白的特性,比较了其在不同变种、不同产地间及不同组织部位的表达模式差异,为进一步解析金银花中DNA去甲基化酶功能奠定基础。
金银花样品采自于江苏、云南、山东、甘肃、北京等地,红金银花采自河北、云南、山东、甘肃、北京等地。所有样品均经鉴定后保存于 -80 ℃冰箱,凭证标本保存于中国中医科学院中药资源中心。实验材料详见表 1。
|
|
Table1 Samples used in this study (n = 3) |
从NCBI蛋白数据库中查询得到拟南芥DNA去甲基化酶序 列,并以其作为探针,通过Blast软件在金银花转录组数据库中搜寻同源序列,识别标准为e值≤1e-15,Score≥100,匹配数b = 1。
利用在线工具Protparam (http:// www.expasy.ch/tools/protparam.html) 预测基因编码蛋白的理化性质; 采用ExPASy PROSITE (http://www. expasy.ch/prosite) 和CDD (http://www.ncbi.mlm.nih. gov/Structure/cdd/wrpsb.cgi) 进行蛋白质结构域分析; 采用CFSSP (http://swissmodel.expasy.org/) 进行蛋白质二级结构分析; 使用SignalP4.0 Serve (http://www. cbs.dtu.dk/services/SignalP) 进行分泌蛋白预测; 利用WOLF PSORT (http://wolfpsort.org/) 进行蛋白定位信号预测; 利用PFAM (http://pfam.xfam.org) 进行蛋白保守域分析。利用Mega 5.0构建Neighbor-joining系统进化树,bootstrap值设为1000。
用Invitrogen公司的Trizol reagent提取样品总RNA,1% 琼脂糖凝胶电泳 确定RNA完整性,紫外分光光度仪测定总RNA的OD260和OD280值,选择OD260/OD280为1.8~2.0的总RNA进行反转录,反转录反应按照cDNA合成试剂盒 (TaKaRa公司) 说明书进行。
利用Primer Primer 5.0设计金银花中4个DNA去甲基化酶基因实时荧光定量引物,扩增产物长度在100~250 bp,送由生工生物工程 (上海) 有限公司合成,如表 2。反应体系按照SYBR Premix Ex Taq TM Kit (TaKaRa公司) 说明书配制,每个反应重复3次,设置阴性对照,在ABI 7500上进行,结束后进行熔解曲线分析。各基因表达量以内参基因LJ18S作为标准进行相对定量,相对定量方法采用2-△Ct法。
|
|
表2 Real-time PCR primers |
从NCBI蛋白数据库中查询得到拟南芥DNA去甲基化酶序列,并以其作为探针,通过Blast软件在金银花转录组数据库中搜寻同源序列,共获得4个金银花DNA去甲基化酶基因,分别命名为LJDME1、LJDME2、LJDME3和LJDME4。ORF Finder预测表明LJDME1全长为5 187 bp; LJDME2全长为4 174 bp; LJDME3全长为4 755 bp,LJDME4 ORF区不全,为922 bp。将所获得的4个金银花DNA去甲基化酶基因提交GenBank核酸序列库,GenBank号依次为: KP116104、KP116105、KP116106和KP116107。
利用Protparam对4个金银花DNA去甲基化酶进行蛋白质理化性质预测,结果表明,LJDME1编码 1 746个氨基酸,蛋白分子量为203.869 kD,理论PI
值为10.69; LJDME2编码1 493个氨基酸,蛋白分子量为161.539 kD,理论PI值为8.71; LJDME3编码 1 771个氨基酸,蛋白分子质量为197.750 kD,理论PI值为6.70; LJDME4编码455个氨基酸,蛋白分 子质量为48.479 kD,理论PI值为10.16。利用CDD数据库分析结构域,结果表明LJDME1、LJDME2、LJDME3、LJDME4均具有endonuclease III (ENDO3c) 特异识别区域。SinalP4.0Server软件分析表明LJDME1、LJDME2、LJDME3和LJDME4均为非分泌蛋白。WOLFPSORT预测表明LJDME1、LJDME2、LJDME3和LJDME4均定位在细胞核中。利用PFAM软件预测表明LJDME1、LJDME2、LJDME3和LJDME4均具有DNA去甲基化酶保守区域RRM-DME domain和Perm-CXXC domain,且RRM-DME domain中含有DNA葡糖基酶。虽然LJDME4 ORF不全,但其具有DNA去甲基化酶上述保守区域,故本文暂且不继续获得其全长,只对其基因及编码蛋白进行基本特性分析。除了LJDME4外,其余3个还具有最保守的DNA糖苷酶结构域: HhH-GPD domain (图 1)。

|
Figure1 The conservative regions of LJDME1,LJDME2 and LJDME3 |
利用CFSSP软件对金银花中4个DNA去甲基化酶蛋白进行二级结构预测(图 2),结果表明,LJDME1的α-螺旋 (H) 为76.1%、β-折叠 (E) 为58.6%、β-转角 (T) 为12.0%; LJDME2的α-螺旋(H) 为65.1%、β-折叠(E) 为70.8%、β-转角(T) 为10.7; LJDME3的α-螺旋 (H) 为67.8%、β-折叠 (E) 为34.6%、β-转角 (T) 为14.7。
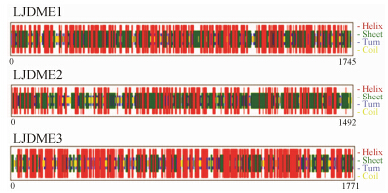
|
Figure2Predicts secondary structures of LJDME1,LJDME2 and LJDME3 amino acid sequence |
选取拟南芥DNA去甲基化酶和金银花中的LJDME1、LJDME2、LJDME3和LJDME4氨基酸序列,利用MEGA5.0 Neighbor-joining法构建DNA去甲基化酶系统进化树 (图 3)。刘秋香等[12]将高等植物中的DNA去甲基化酶分为6个亚家族: ROS1、DME、DML2、DML3、DML4和DML5,从图 3可以看出,金银花中的4个DNA去甲基化酶聚为一大类,且LJDME1、LJDME2聚为一类,LJDME3、LJDME4聚为一类,与拟南芥中的DME关系更为密切。

|
Figure3Phylogenetic analysis of LJDME1,LJDME2,LJDME3 and LJDME4 from Lonicera japonica and DNA demethylase from Arabidopsis thaliana using NJ method. AT: Arabidopsis thaliana |
为了进一步了解金银花中的DNA去甲基化酶基因的表达模式和表达水平,本文通过Real-time PCR分别分析了LJDME1、LJDME2、LJDME3和LJDME4基因在同一产地不同变种、不同产地相同种中的表达情况,以及比较其在花和叶中的表达差异情况。
金银花中4个DNA去甲基化酶基因在不同变种间的表达模式与聚类分析结果一致,即LJDME3、LJDME4基因在山东、云南中的表达量都是红金银 花>金银花,而在甘肃和北京中为金银花>红金银花; LJDME1、LJDME2基因在山东、甘肃、云南、北京、江苏中的表达量均为红金银花>金银花。
金银花中4个DNA去甲基化酶基因在不同产地间的表达模式与聚类分析结果也一致,但不同地区间表达差异不明显。一方面,在不同产地的金银花中LJDME1、LJDME2、LJDME3和LJDME4基因表达量由高到低均为: 江苏>甘肃>北京>云南>山东。另一方面,在不同产地中的红金银花表达量结果为: LJDME1由高到低为云南>甘肃>山东>河北>北京; LJDME2由高到低为云南>甘肃>山东>北京>河北; LJDME3和LJDME4表达模式一致,由高到低为山 东>云南>甘肃>北京>河北。
金银花中4个DNA去甲基化酶基因在花和叶中的表达量差异明显,由图 4可知,一方面,在5个不同产地的金银花中,LJDME2、LJDME3表达量均为 花蕾>叶子; LJDME4除了在山东中的表达情况为花蕾<叶子外,其余4个产地均为花蕾>叶子。另一方面,在5个不同产地的红金银花中,LJDME2、LJDME3、LJDME4表达量都是花蕾>叶子。而LJDME1在金银花及其变种的叶子中表达较少甚至不表达,因此本文中未列出。
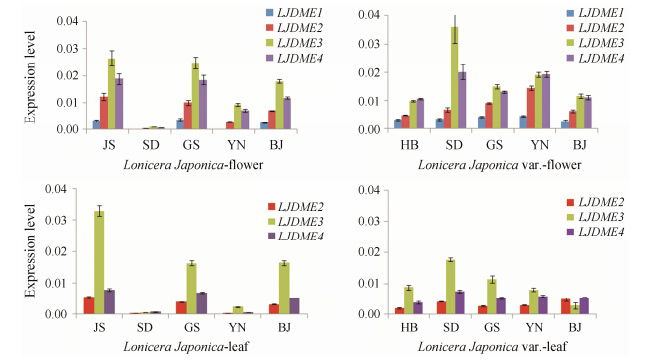
|
Figure4The expression levels of LJDME1,LJDME2,LJDME3 and LJDME4 genes in Lonicera japonica and their substitutes from different resources |
DNA去甲基化酶基因在表观遗传中具有重要的作用,本文共获得了4个金银花的DNA去甲基化酶基因,分别为LJDME1、LJDME2、LJDME3和LJDME4。通过生物信息学方法表明这4个基因编码的蛋白均具有DNA去甲基化酶的保守区域RRM- DME domain和Perm-CXXC domain,且RRM-DME domain中含有DNA葡糖基酶,推测它们的去甲基化机制可能是通过DNA糖基酶参与实现的。系统进化树分析表明,金银花中的4个DNA去甲基化酶聚为一大类,且LJDME1、LJDME2聚为一类,LJDME3、LJDME4聚为一类,与拟南芥中的DME关系更为密切,且与DME是双子叶植物所特有的DNA去甲基化酶,单子叶植物中不存在的结果相符[18]。基因表达模式及水平结果表明,金银花中4个DNA去甲基化酶基因种间差异表达非常明显,在山东、甘肃、云南、北京、江苏中的表达量均为红金银花>金银花,赵贤慧[6]研究结果表明红白忍冬比忍冬香味更浓; 本课题组前期研究结果表明红白忍冬的绿原酸含量显著高于忍冬[5],因此推测LJDME1、LJDME2基因可能参与调控金银花活性成分积累的过程。刘秋香等[12]研究结果表明水稻中的大多数DNA去甲基化酶基因在雌蕊、花序以及胚中的表达显著高于其他组织,证明DNA去甲基化酶基因具有组织表达特异性。本文结果表明金银花中4个DNA去甲基化酶基因在花蕾中的表达量明显高于叶子中的表达量,由此看出这4个金银花DNA去甲基化酶基因也具有组织表达特异性。
本文研究结果为进一步了解金银花类药用植物中DNA去甲基化酶的功能奠定基础,并为研究金银花活性成分生物合成与代谢调控的遗传变异提供依据。
| [1] | Chinese Pharmacopoeia Commission. The Pharmacopoeia of the People's Republic of China (中华人民共和国药典) [S]. 2010 ed. Part I. Beijing: China Medical Science Press, 2010. |
| [2] | Wang ZY, Jiang C, Chen M, et al. Cloning and bioinformatic analysis of FatB genes in Lonicera japonica Thunb and its substitutes [J]. Acta Pharm Sin (药学学报), 2012, 47: 1394- 1398. |
| [3] | Jiang C, Yuan Y, Liu GM, et al. EST-SSR identification of Lonicera japonica Thunb [J]. Acta Pharm Sin (药学学报), 2012, 47:803-810. |
| [4] | Wu XH, Tian ZY, Wang H. Research progress of Lonicera japonica [J]. Lishizhen Med Mater Med Res (时珍国医国药), 2005, 16: 1303-1304. |
| [5] | Yuan Y, Song L, Li M, et al. Genetic variation and metabolic pathway intricacy govern the active compound content and quality of the Chinese medicinal plant Lonicera japonica Thunb [J]. BMC Genomics, 2012, 13: 195. |
| [6] | Zhao XH. Studies on tissue culture of Lonicera japonica var. chinensis [J]. Northern Hort (北方园艺学), 2009, 9: 71-73. |
| [7] | Yuan Y, Wang ZY, Jiang C, et al. Exploiting genes and functional diversity of chlorogenic acid and luteolin biosyntheses in Lonicera japonica and their substitutes [J]. Gene, 2012, 534: 408-416. |
| [8] | Wolffe AP, Matzke MA. Epigenetics: regulation through repression [J]. Science, 1999, 286: 481-486. |
| [9] | Chen X, Wang ZC. Plant DNA methyltransferase [J]. Chem Life (生命的化学), 2009, 29: 534-538. |
| [10] | Zhang H, Zhu JK. Active DNA demethylation in plants and animals [J]. Cold spring Harb Symp Quant Biol, 2012, 77: 161-173. |
| [11] | Cao LG, Zhang Y, Lan XG, et al. Research progress of DNA methylation mechanism [J]. Prog Mod Biomed, 2012, 12: 160-162. |
| [12] | Liu QX, Xue QZ, Xu JH. Phylogenetic analysis of DNA demethylase genes in angiosperm [J]. Hereditas, 2014, 36: 276-285. |
| [13] | Lízal P, Relichová J. The effect of day length, vernalization and DNA demethylation on the flowering time in Arabidopsis thaliana [J]. Physiol Plant, 2001, 113: 121-127. |
| [14] | Penterman J, Zilberman D, Huh JH, et al. DNA demethylation in the Arabidopsis genome [J]. Proc Natl Acad Sci USA, 2007, 104: 6752-6757. |
| [15] | Choi Y, Harada JJ, Goldberg RB, et al. An invariant aspartic acid in the DNA glycosylase domain of DMETER is necessary for transcriptional activation of the imprinted MEDEA gene [J]. Proc Natl Acad Sci USA, 2004, 101: 7481-7486. |
| [16] | Ono A, Yamaguchi K, Fukada-Tanaka S, et al. A null mutation of ROS1a for DNA demethylation in rice is not transmittable to progeny [J]. Plant J, 2012, 71: 564-574. |
| [17] | Zemach A, Kim MY, Silva P, et al. Local DNA hypomethylation activates genes in rice endosperm [J]. Proc Natl Acad Sci USA, 2010, 107:18729-18734. |
| [18] | Choi Y, Gehring M, Johnson L, et al. DEMETER, a DNA glycosylase domain protein, is required for endosperm gene imprinting and seed viability in Arabidopsis [J]. Cell, 2002, 110: 33-42. |
 2015, Vol. 50
2015, Vol. 50


