2. 清华大学化学系生命有机磷化学及化学生物学教育部重点实验室, 北京 100084;
3. 辽宁中医药大学药学院, 辽宁 大连 116600
2. Key Laboratory of Bioorganic Phosphorus Chemistry and Chemical Biology, Ministry of Education, Department of Chemistry, Tsinghua University, Beijing 100084, China;
3. College of Pharmacy, Liaoning University of Traditional Chinese Medicine, Dalian 116600, China
网络药理学是一门对生物系统的网络进行分析并选取特定信号节点进行多靶点药物分子设计的新学科。通过生物学网络中节点的连接和关系来分析网络特性,进而阐明药物的作用机制,为分析药物的作用提供了崭新的角度[1, 2]。网络药理学的发展推动了对药物作用机制的重新认识,为药物发现和老药新用提供理论依据和技术支撑,因此网络药理学在药
物机制探讨[3, 4]、药物重新定位[5, 6]及新药筛选[7, 8]等方面得到了广泛的应用。
随着网络药理学研究的不断发展,其研究方法也呈现出多种多样、“百家争鸣”的特点,现在较常用的靶点预测的方法有基于配体特征的反向药效团匹配 (PharmMapper)、基于蛋白特征的反向分子对 接 (HTDocking) 等,此外本课题组前期研究建立了一种基于Kyoto encyclopedia of genes and genomes (KEGG) 等公共数据库的网络预测模型,即“药物副作用−通路−适应症药物”(side effect-pathway- indication drug,SEPID) 预测模型。PharmMapper方法应用范围广,预测速度快。然而,其仅考虑待测分子与数据库中分子的相似度来打分并不能客观公正地体现化合物与潜在靶点的亲和力,还要把数据库中分子间活性值差异大和因来源不同而导致同一个靶点的活性预测结果不统一的情况考虑在内。HTDocking最初是用来预测阿尔茨海默氏病领域相关的蛋白靶点,其数据库来源于蛋白质数据库,是与阿尔茨海默氏病相关的一个蛋白质数据库子集。该方法适用范围也较广,能够在线实时预测,速度也较快,能够为用户及时反馈预测结果。然而,本方法所依赖的打分函数的精确度仍需要进一步提高。本研究以罗格列酮复方为研究对象,采用3种网络药理学的研究方法 (Pharm Mapper、HTDocking及SEPID预测模型) 进行复方作用靶点的预测,并经过生物信息学分析,寻找罗格列酮复方的重要调节靶点和信号通路,比较不同网络药理学靶点预测方法的差异,探讨罗格列酮复方的作用机制。
材料与方法 药品罗格列酮复方由罗格列酮、阿米洛利和氢氯噻嗪配伍组成,3个药物的结构式如图 1,该复方主要用于治疗2型糖尿病,并可避免单用罗格列酮导致的水肿等副作用[9]。

|
Figure 1 Structures of the three drugs |
PharmMapper 在ChemBioDraw[10]软件中分别绘制3个药物分子的结构式,并将其以mol2的文件 格式导入PharmMapper数据库 (http://59.78.96.61/ pharmmapper/)。PharmMapper是一种基于配体特征的反向药效团匹配的靶点预测方法,通过已构建 好的包含很多药效团模型的数据库,用单个待测分子去反向搜索匹配,最后找到与搜索结构匹配较 好的靶点,PharmMapper数据库内嵌7 000多个药效团模型,它们关联1 627个化合物靶点信息[11]。PharmMapper能够在几分钟至十几分钟内完成靶点的预测,并根据匹配结果的相似度进行程序打分和排序。该方法为用户迅速反馈预测结果提供了可行 性[12]。
HTDocking HTDocking[13]数据库 (http://www. cbligand.org/AD/docking_search.php) 提供在线绘制结构式,是一种基于蛋白结构特征的反向分子对接的靶点预测方法,其将待测分子与靶点数据库中的一些化合物的靶点分子进行分子对接,从中筛选出结合效果最优的候选者,将其作为待测化合物的潜在靶点。HHTDocking程序将待测分子与数据库对接的亲和力值进一步转化为对接得分,最后根据对接得分的排名来预测潜在的靶点。
SEPID预测模型SEPID预测模型是基于side effect resource (SIDER)、genetic association database (GAD)、DrugBank和KEGG等数据库建立了特定药物副作用及其适应症治疗药物靶点的网络预测模型。该方法仅适合于对化合物已有报道或研究的靶点进行统计和预测,并对预测结果进行打分和随机扰动测试。本方法需要及时更新数据库信息,以便能更全面地包含化合物潜在的靶点。本文所选择的研究对象罗格列酮复方即是通过该方法预测得到的,其消除了单用罗格列酮的水肿副作用,已在动物模型上初步证实[9]。
3种网络药理学预测方法及靶点生物信息学分析中用到的数据库有SIDER、GAD、DrugBank、national center for biotechnology information (NCBI)、Molecule Annotation System 3.0 (MAS 3.0) 和KEGG等。这些数据库的网址以及主要功能如表 1所示。
|
|
Table 1 Database and its mainly function |
将3种预测方法得到的药物靶点信息分别在MAS 3.0生物分子功能注释系统中进行通路注释和分析,MAS 3.0系统可以分析差异 靶点所在的通路以及某个通路中靶点富集度的显著 性水平。通过统计分析,选择合适的计算方法计算通路显著性P值。最终选择有统计学意义的富集通路 (P < 0.05) 作为“药物−通路”网络构建的数据。
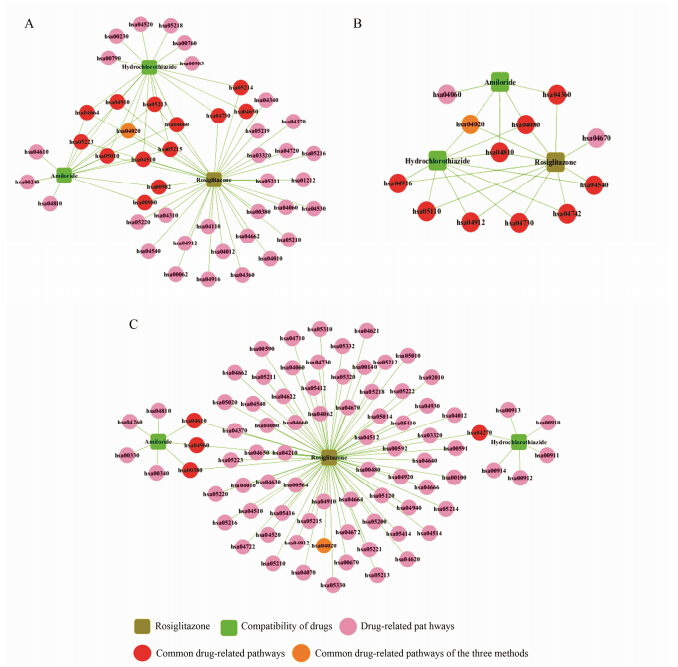
“药物-通路”网络的构建根据MAS 3.0通路注释得到的数据,采用Cytoscape软件[14]分别构建3种方法的“药物−通路”网络模型。在“药物−通路”& lt; /span>网络图中,包含两种节点 (node): 药物和通路,若某一通路为药物的相关作用通路,则以边 (edge) 相连。每种方法均建立“药物−通路”网络后,通过共有的作用通路构建包含3种预测方法所有信息的“药物−通路”网络 (图 2)。通过该网络可方便比较分析3种方法预测结果之间的关系。
结果 1 预测靶点信息为了比较PharmMapper和HTDocking预测方法之间的相关性并且尽可能多的包含潜在的靶点,均选择各自预测出的前30个靶点信息进行分析和讨论。以罗格列酮为例,表 2为PharmMapper预测得到的 罗格列酮靶点信息; 表 3为HTDocking预测得到的 罗格列酮靶点信息; SEPID预测模型的靶点是从DrugBan k和GAD数据库提取获得 (未详细列出)。需要特别注明的是,3种方法得到的药物预测靶点基本不相同,只有PharmMapper预测得到罗格列酮的靶点(peroxisome proliferator-activated receptor gamma) 及阿米洛利的靶点 (urokinase-type plasminogen activator) 已有研究证实 (与SEPID预测模型得到的结果一致),因此后续需要对药物靶点进行通路注释和分析,考察3种药物在调节的通路上是否具有更多潜在的联系。
|
|
Table 2 Information of rosiglitazone on drug targets of the method of PharmMapper |
|
|
Table 3 Information of rosiglitazone on drug targets of the method of HTDocking |
基于PharmMapper预测方法建立的“药物−通路”网络模型 (图 2A),共预测得到罗格列酮复方相关通路46条。在这些通路中,罗格列酮 (棕色节点) 与配伍药物 (绿色节点) 共同作用的通路有14条(图 2A红色和橙色节点),它们分别与疾病[15] (5条)、免疫系统[16] (3条)、代谢过程[17] (2条)、神经系统 (1条)、内分泌系统 (1条)、细胞过程 (1条) 和信号转导过程 (1条) 有关。由于罗格列酮在治疗2型糖尿病的同时,可能产生水肿的副作用,因此罗格列酮作用的37条通路中既包含药物的作用靶点 (即发挥有利的药效),也可能包含药物的副作用靶点。而配伍药物阿米洛利和氢氯噻嗪的主要药效作用是消除水肿,因此14条共同的通路可能与罗格列酮复方消除单用罗格列酮引起水肿等副作用的调节有关。
|
Figure 2 The “drug-pathway” network of the three forecast methods if a pathway is drug-related,connected by edge. A: PharmMapper,B: HTDocking,C: SEPID |
基于HTDocking预测方法建立的“药物−通路”网络模型 (图 2B),共预测得到罗格列酮复方相关通路12条。在这些通路中,罗格列酮得到的10条调节通路均是配伍药物的作用通路 (图 2B红色和橙色节点),这些通路不仅与信号传导[18](2条)、内分泌系统[19] (2条) 和细胞过程[20]有关 (2条),还与神经系统 (2条)、感觉系统 (1条) 和疾病 (1条) 等有关。这10条通路可能与罗格列酮复方消除单用罗格列酮引起水肿副作用的调节有关,其中是否有罗格列酮治疗2型糖尿病的作用通路,还需进一步探讨。
2.3 SEPID预测靶点的通路注释结果与分析基于SEPID预测方法建立的“药物−通路”网络模型 (图 2C),共预测得到罗格列酮复方相关通路82条。其中有18条通路是根据DrugBank数据库检索到的所有药物作用靶点经通路注释与分析得到 (作用的通路,P < 0.05),还有64条通路是根据GAD数据库检索到的罗格列酮水肿的基因经通路注释与分析得到 (副作用的通路,P < 0.05)。在以上通路中,罗格列酮与配伍药物共同作用的通路有4条 (图 2C红色节点)。这4条通路分别与氨基酸代谢[21]、免疫系统[22]、排泄系统[23]和循环系统[24]有关,这些通路可能是配伍药物消除罗格列酮副作用的主要调节通路。
讨论根据药物靶点通路注释结果与分析可以看出,在3种预测方法中,SEPID预测模型得到了82条 作用通路,PharmMapper预测得到了46条作用通路,HTDocking只得到12条作用通路。由此可知,SEPID 预测模型预测结果涉及更多的体内过程,HTDocking的预测结果涉及的体内调节通路相对较少。由于预测原理的差异,3种方法的预测结果差异较大,提示在实际研究中应该综合考虑多种预测方法的分析结果,不能单一倚重于某种方法。
PharmMapper预测方法得到的罗格列酮的靶点 (peroxisome proliferator-ac-tivated receptor gamma) 及阿米洛利的靶点 (urokinase-type plasminogen activator) 已有研究证实 (与SEPID预测模型得到的结果一致),而HTDocking预测方法却没有得到任何与研究证实相同的靶点。本研究结果表明,PharmMapper预测方法可能更适合用来分析药物的靶点信息,而HTDocking预测方法则可能偏重于疾病[13]方面的靶点预测。
从两种方法的预测方式来看,PharmMapper预测方法导入的是化合物的三维结构式而HTDocking预测方法是在线绘制二维结构式,因三维结构式存在构象差异等特点可能会比二维结构式预测更有效,从而导致PharmMapper预测方法得到更准确的结果。
SEPID预测模型与以上两种预测方法不同,它是从数据库出发检索靶点信息的,这些都是经过实验“筛选”后得到的有用信息,这种方法得到的数据真实性强,有利于进一步分析,但不利于新的作用靶点的发现。而前两种方法是单纯地从分子结构来预测靶点,这种方法预测量大,预测效率高,预测具有无偏性,但是未经过实验的初步筛选,包含无用的数据,需要再从预测结果中确定有效信息进行进一步验证。
通过对3种方法预测得到的靶点分析比较,发现钙信号转导通路 (hsa04020) 在每种方法的预测中都有出现 (并且在前两种预测方法中所有药物均与该通路相关),说明这条通路与罗格列酮复方的药效有着重要的关系。钙信号调控着细胞内许多重要的功 能,在许多病理状态下,细胞内钙离子水平的异常会引起细胞功能的改变,因此细胞内钙离子处于非常严格的调控之中。有文献[25, 26]报道钙通道的异常会引起各种糖尿病的发生,钙离子信号的异常会导致肺水肿[27],也有研究[28]表明钙通道阻滞剂过量时会导致门静脉水肿。此外钙离子转运障碍还与心肌功能异常有关[29],进而导致出现心力衰竭的症状[30, 31],而心力衰竭也是罗格列酮的副作用,同时配伍药物又能缓解此副作用。这进一步说明该通路与罗格列酮复方的作用关系密切。以上研究表明3种方法预测得到的共同通路是罗格列酮复方作用的重要调节通路。
综上所述,3种网络药理学的预测方法各有优缺点,在实际应用中应综合考虑各预测方法的优势和贡献,筛选最重要的靶点及通路,为后续实验研究提供有价值的线索。本研究通过对罗格列酮复方作用靶点及调节通路的预测分析发现,钙信号转导通路在3种方法中均可预测到,其可能是罗格列酮复方调节的主要通路,对后续该复方药效作用机制的深入探讨具有重要的指导意义。
| [1] | Hsin KY, Ghosh S, Kitano H. Combining machine learning systems and multiple docking simulation packages to improve docking prediction reliability for network pharmacology [J]. PLoS One, 2013, 8: e83922. |
| [2] | Tao W, Xu X, Wang X, et al. Network pharmacology-based prediction of the Radix Curcumae ingredients and potential targets of Chinese herbal italic formula for application to cardiovascular disease [J]. J Ethnopharmacol, 2013, 145: 1-10. |
| [3] | Azmi AS, Mohammad RM. Rectifying cancer drug discovery through network pharmacology [J]. Future Med Chem, 2014, 6: 529-539. |
| [4] | Cheng BF, Hou YY, Jiang M, et al. Anti-inflammatory mechanism of Qingfei Xiaoyan Wan studied with network pharmacology [J]. Acta Pharm Sin (药学学报), 2013, 48: 686-693. |
| [5] | Sanseau P, Agarwal P, Barnes MR, et al. Use of genome- wide association studies for drug repositioning [J]. Nat Biotechnol, 2012, 30: 317-320. |
| [6] | Wang W, Yang S, Zhang X, et al. Drug repositioning by integrating target information through a heterogeneous network model [J]. Bioinformatics, 2014, 30: 2923-2930. |
| [7] | Zhang ZQ, Chen XY, Duan YX, et al. A novel methodology that combined fast chromatography and antioxidant activities screening in Baimai prescription research [J]. J Am Chem Soc, 2013, 699: 349-353. |
| [8] | Liu QS, Zhang ZQ, Fang L, et al. Application of network pharmacology and high through-put technology on active compounds screening from traditional Chinese medicine [J]. China J Chin Mater Med (中国中药杂志), 2012, 37: 134-137. |
| [9] | Wang LL, Fan XM, Wang YM, et al. Rosiglitazone compound formula eliminates the side effects of rosiglitazone [J]. Cent South Pharm (中南药学), 2014, 12: 346-350. |
| [10] | Cousins KR. Computer review of ChemDraw Ultra 12.0 [J]. J Am Chem Soc, 2011, 133: 8388. |
| [11] | Liu XF, Ouyang S, Yu B, et al. PharmMapper server: a web server for potential drug target identification using pharmacophore mapping approach [J]. Nucleic Acids Res, 2010, 38: W609-W614. |
| [12] | Fang JS, Liu AL, Du GH. Research advance on drug target prediction based on Chemoinformatics [J]. Acta Pharm Sin (药学学报), 2014, 49: 1357-1364. |
| [13] | Liu H, Wang L, Lv M, et al. AlzPlatform: an Alzheimer's disease domain-specific chemogenomics knowledgebase for polypharmacology and target identification research [J]. J Chem Inf Model, 2014, 54: 1050-1060. |
| [14] | Zhang C, Wang J, Hanspers K, et al. NOA: a cytoscape plugin for network ontology analysis [J]. Bioinformatics, 2013, 29: 2066-2067. |
| [15] | Meng F, Dai E, Yu X, et al. Constructing and characterizing a bioactive small molecule and microRNA association network for Alzheimer's disease [J]. J R Soc Interface, 2013, 11: 20131057. |
| [16] | Russier M, Reynard S, Carnec X, et al. The exonuclease domain of lassa virus nucleoprotein is involved in antigen- presenting-cell-mediated NK cell responses [J]. J Virol, 2014, 88: 13811-13820. |
| [17] | von Grafenstein S, Fuchs JE, Huber MM, et al. Precursors for cytochrome P450 profiling breath tests from an in silico screening approach [J]. J Breath Res, 2014, 8: 046001. |
| [18] | Gunawardhana LP, Gibson PG, Simpson JL, et al. Characteristic DNA methylation profiles in peripheral blood monocytes are associated with inflammatory phenotypes of asthma [J]. Epigenetics, 2014, 9: 1302-1316. |
| [19] | Dang AK, Murtazina DA, Magee C, et al. GnRH evokes localized subplasmalemmal calcium signaling in gonadotropes [J]. Mol Endocrinol, 2014, 28: 2049-2059. |
| [20] | Takito J, Otsuka H, Yanagisawa N, et al. Regulation of osteoclast multinucleation by the actin cytoskeleton signaling network [J]. J Cell Physiol, 2015, 230: 395-405. |
| [21] | Voges R, Corsten S, Wiechert W, et al. Absolute quantification of corynebacterium glutamicum glycolytic and anaplerotic enzymes by QconCAT [J]. J Proteomics, 2015, 113: 366-377. |
| [22] | Dobó J, Schroeder V, Jenny L, et al. Multiple roles of complement MASP-1 at the interface of innate immune response and coagulation [J]. Mol Immunol, 2014, 61: 69- 78. |
| [23] | Gravez B, Tarjus A, Jimenez-Canino R, et al. The diuretic torasemide does not prevent aldosterone-mediated mineralocorticoid receptor activation in cardiomyocytes [J]. PLoS One, 2013, 8: e73737. |
| [24] | Otero C, Peñaloza JP, Rodas PI, et al. Temporal and spatial regulation of cAMP signaling in disease: role of cyclic nucleotide phosphodiesterases [J]. Fundam Clin Pharmacol, 2014, 28: 593-607. |
| [25] | Gutterman DD, Durand MJ. Vascular dysfunction in diabetes mellitus large conductance calcium-activated potassium channels as part of a subsarcolemmal signaling soiree [J]. Circ Res, 2014, 114: 588-590. |
| [26] | Obrosova IG. Diabetes and the peripheral nerve [J]. Biochim Biophys Acta, 2009, 1792: 931-940. |
| [27] | Hersi A, Armstrong PW, Choy JB, et al. Pulmonary edema postcardioversion: a potential calcium signaling problem [J]. Can J Cardiol, 2006, 22: 259-262. |
| [28] | Lu HC, Chen JD, How CK. Periportal edema after cardiac arrest due to calcium channel blocker overdose [J]. J Formos Med Assoc, 2014, 113: 266-267. |
| [29] | Shi YH, Zhu SW, Mao XZ, et al. Transcriptome profiling, molecular biological and physiological studies reveal a major role for ethylene in cotton fiber cell elongation [J]. Plant Cell, 2006, 18: 651-664. |
| [30] | Patel K, Fonarow GC, Ahmed M, et al. Calcium-channel blockers and outcomes in older patients with heart failure and preserved ejection fraction [J]. Circ Heart Failure, 2014, 7: 945-952. |
| [31] | Wilson K, Guggilam A, West TA, et al. Effects of a myofilament calcium sensitizer on left ventricular systolic and diastolic function in rats with volume overload heart failure [J]. Am J Physiol Heart Circ Physiol, 2014, 307: H1605-H1617. |
 2015, Vol. 50
2015, Vol. 50


