肝纤维化 (liver fibrosis) 是肝脏内的细胞外基质 (extracellular matrix,ECM),包括胶原 (特别是Ⅰ型胶原α1) 过度沉积的病理过程[1]。它是机体对急、慢性肝损伤的一种修复反应,大部分慢性肝病都伴随肝纤维化。肝星状细胞 (hepatic stellate cell,HSC) 的激活是肝纤维化的细胞学基础[2, 3],转化生长因子β1 (transforming growth factor beta 1,TGF-β1) 是目前已知最重要的致肝纤维化的细胞因子之一[2, 4,5]。肝纤维化的发展最终会导致肝硬化,而肝硬化在全球的发病率和死亡率中占很大比例,最终只能依靠肝移植来延续生命。研究表明,肝纤维化是可以逆转的[6, 7, 8, 9],因此研究如何控制肝纤维化这一可逆的病理过程,对于治疗肝纤维化及肝硬化十分重要。
近年来,肝纤维化的临床治疗取得一些新进展,但是目前仅有中国批准上市两种中药复方,除针对病因治疗以外,尚无其他治疗药物[10]。在药物筛选模型方面,抗丙型肝炎病毒 (HCV) 药物筛选系统研究较为成熟,已建立用于筛选抗肝脏脂肪变性及磷脂质病细胞模型,但这两种模型分别针对HCV及脂肪病变,并不适合所有抗肝纤维化药物的筛选。因此,建立新的药物筛选模型,寻找临床有效的抗肝纤维化药物,已成为新药研发的当务之急。
胶原的过度表达是肝纤维化产生的基本特征,同时也是造成器官器质性病变的重要原因[2, 4, 11]。近期Stefanovic等[12]建立了以Ⅰ型胶原5’SL与LARP6蛋白的相互作用为靶点的抗纤维化药物高通量筛选模型。Ⅰ型胶原5’SL与LARP6蛋白的相互作用对胶原产生有着重要作用,因此所筛选化合物能够抑制两者的相互作用,即能抑制胶原的产生,进而抑制纤维化。本研究也是以Ⅰ型胶原的表达水平为考察对象,但靶点是Ⅰ型胶原α1基因COL1A1的启动子活性,化合物能够抑制该启动子活性即能抑制胶原的产生,进而抑制肝纤维化。据此,作者构建了含COL1A1启动子的荧光素酶报告系统的高通量筛选模型,并对该细胞模型的可靠性进行了验证。
材料与方法 药品、试剂及材料分子克隆所需的胰蛋白胨 (tryptone) 及酵母提取物 (yeast extract) 购自Oxoid公司; 琼脂糖购自Biosharp公司; 限制性内切酶、T4 DNA连接酶及DNA Marker均购自Takara公司; 血液/组织/细胞基因组DNA提取试剂盒、普通琼脂糖凝胶DNA回收试剂盒及普通质粒小提试剂盒均购自天根生化科技有限公司; 细胞培养所需的DMEM培养基购自Gibco公司; 胎牛血清购自中国医学科学院血液研究所; 细胞培养瓶购自Nunc公司; 培养板购自Costar公司; 双抗混合物 (青霉素与链霉素) 购自Hyclone公司; 转染试剂lipofectamine 2000购自Invitrogen公司; G418购自Amerco公司; TGF-β1购自R&D公司; 人肝星状细胞LX2为本实验室培养、传代及冻存; 大肠杆菌E.coli DH5α感受态购自天 根生化科技有限公司; pGL4.17荧光素酶报告载体和海肾荧光素质粒Renilla为本实验室保存; PureYield Plasmid Midiprep System、Dual-Luciferase Reporter Assay System和Bright-Glo Luciferase Assay System购自Promega公司; NucleoSpin RNA Clean-up试剂盒购自MN公司; AffinityScript Multiple Temperature cDNA Synthesis Kit购自Agilent Technologies公司; FastStart Universal Probe Master (Rox) 购自Roche公司; Taqman引物和探针购自Applied Biosystem公司; 抗体β-actin购自Sigma-Aldrich公司; 抗体collagenⅠ购自Abcam公司; HRP标记的羊抗兔二抗和羊抗鼠二抗购自中杉金桥公司; Chemiliminescent HRP Substrate购自Millipore公司。
COL1A1启动子序列克隆及荧光素酶报告质粒的构建按基因组DNA提取试剂盒说明书提取LX2细胞基因组DNA,用PCR法扩增得到COL1A1启动子序列,引物序列为5'-AAGAGCTCGTGGGAAAGC CTGGATGG-3',5'-AAAGATCTTTTGGGACTTACTG TCTTCGT-3'; PCR反应条件为: 98 ℃变性10 s,59 ℃退火10 s,72 ℃延伸4 min,循环30次。将PCR产物和荧光素酶报告载体pGL4.17进行SacⅠ和BglⅡ双酶切,用T4 DNA连接酶连接这两个片段得到重组 质粒,最终经测序确认。将该质粒命名为pGL4.17- COL1A1P (图 1),并用去内毒素质粒提取试剂盒提取无内毒素的重组质粒备用。
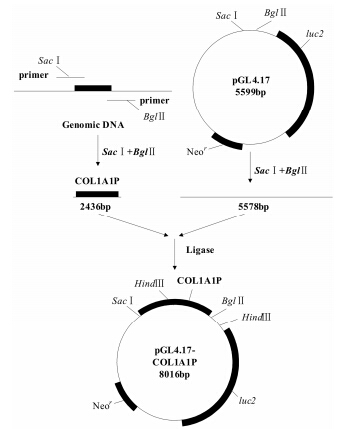
|
Figure 1 Construction of plasmid pGL4.17-COL1A1P |
在含10% 胎牛血清的DMEM 培养基,37 ℃,5% CO2条件下培养LX2细胞。传至48孔板,培养24 h,用转染试剂lipofectamine 2000推荐的方法瞬时转染6 h,其中质粒pGL4.17-COL1A1P转染量为100 ng/孔,Renilla为30 ng/孔。经TGF-β1 (2 ng·mL-1) 诱导24 h后,用双荧光素酶报告基因检测系统 (Dual-Luciferase Reporter Assay System) 检测荧光强度。
稳定转染pGL4.17-COL1A1P的单克隆细胞株的筛选在6孔板中培养LX2细胞达到接近90%~95% 汇合时,进行细胞转染。24 h后,加G418至终质量浓度为100 μg·mL-1,继续培养并及时换液。待 6孔板中的细胞长满后,铺单细胞到96孔板。待单细胞长成克隆 (约1~2周),加D-luciferin至终质量浓度60 μg·mL-1,用活体生物发光成像系统 (IVIS 200,Xenogen公司) 拍摄,选择信号最强的单克隆扩大培养,并命名为LX2-COL单克隆细胞。
荧光素酶报告基因检测系统筛选候选化合物LX2-COL单克隆细胞铺于48孔板,待细胞汇合度 约90%~95% 后无血清培养,24 h后加入TGF-β1 (2 ng·mL-1) 诱导,同时加入候选化合物。作用24 h后,用Bright-Glo Luciferase Assay System试剂盒检测启动子活性,检测方法按该说明书操作。
实时荧光定量RT-PCR在6孔板中培养LX2细胞,接近90%~95% 汇合时,换无血清培养基饥饿24 h后,加TGF-β1 (2 ng·mL-1) 诱导的同时给药,24 h后收集细胞。用Trizol (Invitrogen公司) 提取RNA,用NucleoSpin RNA Clean-up试剂盒纯化RNA,取2 μg/样品用AffinityScript Multiple Temperature cDNA Synthesis Kit逆转录得到cDNA,最后用ABI 7500 Fast实时荧光定量PCR仪进行检测。
Western blotting检测收集经过24 h处理的LX2细胞,用PBS冲洗2遍,加入RIPA裂解液,4 ℃裂解30 min,12 000×g、4 ℃离心15 min,收集上清液,按Bradford法测定蛋白含量后等量分装。SDS-PAGE凝胶电泳将不同分子量的蛋白质分离,然后用湿转 法将胶上的蛋白质分子转移到预处理后的PVDF膜上。在含5% 脱脂奶粉的PBST中轻轻晃动封闭1 h,然后将不同的蛋白条带用对应的一抗,4 ℃孵育过夜。PBST洗膜3次后,加入相应的二抗,室温孵育 1 h。再用PBST洗膜3次,加上HRP底物发光液,通过凝胶成像系统获取图片。
数据处理每组实验重复3次,组间比较采用t检验。
结果 1 双荧光素酶报告基因检测系统检测启动子活性萤火虫荧光素酶 (firefly luciferase) 的读数可以反应待测启动子的转录活性,海肾荧光素酶 (Renilla luciferase) 的读数作为内参,消除细胞数量和转染效率的差异。结果显示,COL1A1P的活性比对照组强15.98倍,表明TGF-β1能够明显增强COL1A1启动子活性。见图 2。
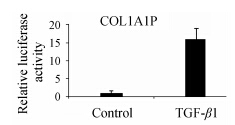
|
Figure 2 Detection of COL1A1 promoter activity in LX2 cells. Cells were transfected with pGL4.17-COL1A1P and Renilla for 6 h,and then treated with 2 ng·mL-1 TGF-β1 for 24 h. The activity of COL1A1 promoter was detected by Dual-Luciferase Reporter Assay System |
为了满足高通量筛选的要求,简化实验步骤,使每次筛选结果均一可靠,反应灵敏,应用单克隆细胞培养的方法筛选稳定转染pGL4.17-COL1A1P的LX2单克隆细胞株,即LX2-COL单克隆细胞株。利用活体生物发光成像系统筛选出总光通量最高的细胞株。结果显示,6号单克隆细胞株光通量最强 (图 3),将其扩大培养。
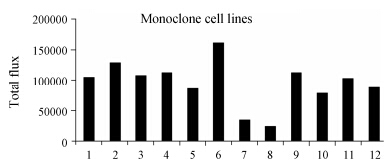
|
Figure 3 The screening of LX2-COL monoclone cell line. Different LX2-COL monoclone cell lines were treated with 2 ng·mL-1 TGF-β1 for 24 h. There after,60 μg·mL-1 D-luciferin was added and the total flux were detected by Xenogen IVIS System |
利用单荧光素酶报告基因检测系统,从候选化合物中筛选出抑制COL1A1启动子活性最强的化合物物。表没食子儿茶素没食子酸酯 (EGCG) 为阳性对照药,S1~S7是候选化合物。结果显示,加入化合物S7后,荧光强度最弱。与对照组相比,其活性抑制率为72.3%,强于EGCG活性抑制率 (55.35%),表明S7抑制COL1A1启动子活性最强。见图 4。
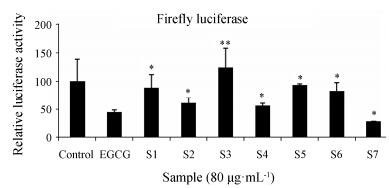
|
Figure 4 The activity detection of candidate drugs. LX2 monoclonal cells were treated with 2 ng·mL-1 TGF-β1,EGCG (positive control drug) and candidate drugs (S1-S7) for 24 h,and then firefly luciferase activity was detected by Bright-Glo Luciferase Assay System. n = 3,P < 0.05,**P < 0.01 vs EGCG group |
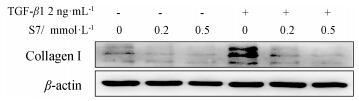
为了验证本细胞模型的可靠性,作者采用实时荧光定量RT-PCR和Western blotting的方法验证筛选出的化合物S7的抗肝纤维化效果。实时荧光定量RT-PCR的结果显示,S7能够使TGF-β1诱导升高的Ⅰ型胶原α1的mRNA水平显著降低,见图 5。此外,Western blotting的结果显示,S7能够逆转TGF-β1诱导升高的Ⅰ型胶原蛋白水平,见图 6。说明S7能够显著降低在肝纤维化产生过程中发挥重要作用的Ⅰ型胶原的mRNA及蛋白水平,证明该细胞筛选模型成功筛选出具有一定抗纤维化作用的化合物。

|
Figure 5 Effect of S7 on of collagenⅠ (α1) mRNA. LX2 was treated with various concentration of S7 (0.2 mmol∙L-1 and 0.5 mmol∙L-1),and 2 ng∙mL-1 TGF-β1 for 24 h,and then collagen Ⅰ mRNA was detected by real-time RT-PCR. n = 3. P < 0.05 vs TGF-β1 group |
|
Figure 6 Effect of S7 on protein expression of collagen Ⅰ. LX2-COL monoclonal cells were treated with various concentration of S7 (0.2 mmol∙L-1 and 0.5 mmol∙L-1),and 2 ng·mL-1 TGF-β1 for 24 h,and then collagen Ⅰ protein was detected by Western blotting |
Ⅰ型胶原α1是导致肝纤维化的一种重要胶原蛋白,在各种肝纤维化进程中均会高表达,且这种高表达表现在转录水平上,因此将化合物对胶原启动子活性的影响作为初步判断潜在药物抗肝纤维化作用的指标。HSC的激活是肝纤维化产生的细胞学基础,因此,采用人肝星状细胞LX2作为重组质粒的宿主细胞。基于TGF-β1可以通过TGFβ/ Smad通路促使COL1A1等肝纤维化关键基因的转录激活[13],利用TGF-β1诱导LX2的活化。将COL1A1启动子的基因片段和带有luciferase基因的报告载体重组得到荧光素酶报告质粒,借助luciferase的表达反映COL1A1启动子的活性强弱。建立稳定表达COL1A1启动子的单克隆细胞株,利用荧光素酶报告基因检测系统筛选候选化合物。并利用real-time RT-PCR和Western blotting的方法,初步证明筛选出的化合物对肝纤维化过程中collagenⅠ的表达有显著抑制作用,验证了该细胞筛选模型的可靠性。
该筛选模型是稳定表达COL1A1启动子活性的单克隆细胞株的细胞模型。首先,化合物在细胞中的活性及生物学功能更能接近于人的生理状态,经该模型筛选得到的化合物也具有很高的可靠性。其次,利用单克隆细胞株进行筛选,结果均一可靠、操作简便、高效、快速,能够满足高通量筛选的要求。此外,利用荧光素酶为报告基因,通过检测荧光素酶报告基因的表达强度反映COL1A1启动子的活性,效果直接,灵敏度高。最后,与Stefanovic等[12]建立的以Ⅰ型胶原5’SL与LARP6蛋白相互作用为靶点的筛选模型比较,本模型的靶点是COL1A1启动子的活性,所筛选的化合物只要能够影响COL1A1启动子表达的任何一个环节,便能影响collagenⅠ的表达,所以本模型应用范围更广。目前本研究已申请专利 (201410466788.5)。
总之,本细胞筛选模型能够实现对潜在的抗肝纤维化药物进行高通量筛选,为抗肝纤维化药物的筛选提供简单而可靠的方法。
| [1] | Kisseleva T, Brenner DA. Role of hepatic stellate cells in fibrogenesis and the reversal of fibrosis [J]. J Gastroenterol Hepatol, 2007, 22: S73-S78. |
| [2] | Guo J, Friedman SL. Hepatic fibrogenesis [J]. Semin Liver Dis, 2007, 27: 413-426. |
| [3] | Kisseleva T, Brenner DA. Hepatic stellate cells and the reversal of fibrosis [J]. J Gastroenterol Hepatol, 2006, 21: S84-S87. |
| [4] | Friedman SL. Hepatic fibrosis - overview [J]. Toxicology. 2008, 254: 120-129. |
| [5] | Shek FW, Benyon RC. How can transforming growth factor beta be targeted usefully to combat liver fibrosis? [J]. Eur J Gastroenterol Hepatol, 2004, 16: 123-126. |
| [6] | Traber PG, Chou H, Zomer E, et al. Regression of fibrosis and reversal of cirrhosis in rats by galectin inhibitors in thioacetamide-induced liver disease [J]. PLoS One, 2013, 8: e75361. |
| [7] | Fallowfield JA, Iredale JP. Reversal of liver fibrosis and cirrhosis - an emerging reality [J]. Scott Med J, 2004, 49: 3-6. |
| [8] | Giannone FA, Baldassarre M, Domenicali M, et al. Reversal of liver fibrosis by the antagonism of endocannabinoid CB1 receptor in a rat model of CCl(4)-induced advanced cirrhosis [J]. Lab Invest, 2012, 92: 384-395. |
| [9] | Fallowfield J, Hayes P. Pathogenesis and treatment of hepatic fibrosis: is cirrhosis reversible? [J]. Clin Med, 2011, 11: 179-183. |
| [10] | Zhao SS, Shao RG, He H. Potential targets for anti-liver fibrosis [J]. Acta Pharm Sin (药学学报), 2014, 49: 1365- 1371. |
| [11] | Bataller R, Brenner DA. Liver fibrosis [J]. J Clin Invest, 2005, 115: 209-218. |
| [12] | Stefanovic B, Stefanovic L. Screening for antifibrotic compounds using high throughput system based on fluorescence polarization [J]. Biology, 2014, 3: 281-294. |
| [13] | He H, Mennone A, Boyer JL, et al. Combination of retinoic acid and ursodeoxycholic acid attenuates liver injury in bile duct-ligated rats and human hepatic cells [J]. Hepatology, 2011, 53: 548-557. |
 2015, Vol. 50
2015, Vol. 50


