| 隐藏嗜酸菌Acidiphilium cryptum JF-5双组分信号转导系统 |
隐藏嗜酸菌(Acidiphilium cryptum)是一种能利用有机物、H2、单质硫或者还原Fe3+而获得能量,进行化能异养生长的嗜酸兼性异养细菌,为革兰氏阴性菌[1],该菌一般生长在酸性矿山废水、高温废水或一些重金属污染酸性矿山环境中.隐藏嗜酸菌为金属还原性细菌,能进行铁呼吸作用,使Fe3+还原成Fe2+,可使Cr6+还原成Cr3+以降低Cr的毒性[2-3].在生物浸出过程中,隐藏嗜酸菌可还原Fe3+并利用其它浸矿细菌合成的有机物,通过这种方式,消除这些有机物的抑制和毒害作用,可为浸矿细菌如嗜酸氧化亚铁硫杆菌(Acidithiobacillus ferrooxidans)提供Fe2+作为能源[4].有研究报道,通过对隐藏嗜酸菌和嗜酸氧化亚铁硫杆菌进行紫外诱变,诱变后混合菌浸矿,浸出效率大幅提高[5].此外,该菌还能合成聚-β-羟丁酸(Poly-β-hydroxybutyrate, PHB)[6-8].
隐藏嗜酸菌在生长繁殖过程中,需对多变的极端环境条件进行感知和反应.在原核生物中,通常这个功能是由双组分信号转导系统(Two-component signal transduction system, TCS)来完成的.双组分信号转导系统由组氨酸蛋白激酶(Histidine protein kinase, HPK)和反应调节蛋白(Response regulator protein, RR)组成[9],具有参与调控生物生长、毒力等多种重要生物学功能.通常,定位在细胞质膜上的HPK监视着环境的变化,而与之相对应的位于细胞质中的RR对环境信号的变化进行相应的响应.目前,国内外还未见有关隐藏嗜酸菌双组分信号转导系统的研究报道.本研究拟应用生物信息学手段,对隐藏嗜酸菌A. cryptumJF-5菌株双组分信号转导系统进行预测和结构分析,并对其功能进行预测,旨在为研究该菌在极端环境中适应性分子机制提供新的方向.
1 材料与方法 1.1 基因组序列来源从GenBank (ftp://ftp.ncbi.nlm.nih.gov/genomes/Bacteria/)下载隐藏嗜酸菌A. cryptum JF-5基因组信息(后缀名为.fna, .ffn, .ptt, .faa的4个文件),见表 1.
| 表1 隐藏嗜酸菌A. cryptum JF-5基因组信息 |
 |
| 点击放大 |
1.2 HKs和RRs预测
提交隐藏嗜酸菌A. cryptumJF-5的NC_009484.faa文件至Pfam蛋白序列数据库(http://pfam.sanger.ac.uk/)进行批量搜索(Cut-off选择Gathering threshold)[10],在返回的结果中用Pfam02518 (HATPase_c)和Pfam00072 (Response_reg)(HKs和RRs的保守结构域)搜索可能的HKs和RRs;然后通过NCBI的BLASTp程序(http://blast.ncbi.nlm.nih.gov/)进行在线确认:①催化结构域HATPase_c必须位于HKs的C-末端;② HATPase_c的上游具有二聚化或磷酸化结构域DHp;③ RRs的N-末端通常具有磷酸受体结构域REC;④ Response_reg结构域位于RRs的C-末端.基因间距小于300 bp且转录同向的HK-RR鉴定为HK-RR基因对(HK-RR gene pairs),同时拥有HK的磷酸转移酶结构域(phosphotransferase domain)和RR磷酰基接受域(phosphoryl-accepting domain)的HK-RR为杂合蛋白(HK-RR Fusion),单个的HK或RR基因鉴定为“Orphan”[11].
1.3 HKs和RRs功能域与进化分析HKs和RRs功能域分别用SMART分析(http://smart.embl-heidelberg.de/)[12]和BLASTp程序相似性比对分析(http://blast.ncbi.nlm.nih.gov/);采用MEGA5.1进行多重序列比对并建立系统发育树[13].
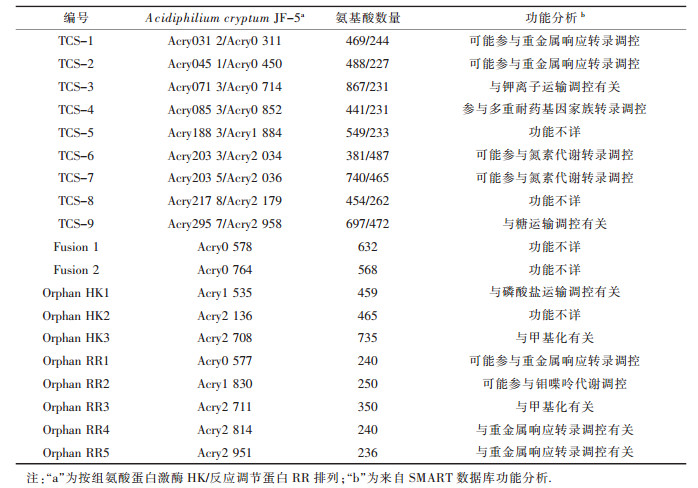
2 结果与分析 2.1 隐藏嗜酸菌双组份信号转导系统及功能分析利用保守结构域HATPase_c (Pfam02518)和Response_reg (Pfam00072)对A. cryptumJF-5菌株的TCSs进行了搜寻.结果表明,在A. cryptumJF-5菌株中,HKs和RRs都为14个,其中成对的TCS (HK-RR)为9对,杂合结构的TCS (Fusion)为2个,孤儿Orphan HKs和Orphan RRs分别为3和5个(见表 2和表 3).隐藏嗜酸菌TCSs的数量明显少于苏云金芽孢杆菌(成对的TCS在35~40对之间,孤儿HKs和RRs分别为15~16和6~8个,杂合结构的TCS为2~3个)[14],这可能与它们生长的环境复杂度差异密切相关,隐藏嗜酸菌生长在相对单一的环境中.通过分析发现这些双组分系统基因分散在基因组中,并不局限于某一区域,表明隐藏嗜酸菌A. cryptumJF-5菌株存在一个复杂的调控网络.
| 表2 隐藏嗜酸菌A. cryptumJF-5中双组分信号转导系统的数目/个 |
 |
| 点击放大 |
| 表3 隐藏嗜酸菌A. cryptumJF-5中双组分信号转导系统及功能分析 |
 |
| 点击放大 |
利用SMART数据库对TCS进行了功能分析,结果表明,TCS-1、TCS-2、Orphan RR1、Orphan RR4和Orphan RR5可能参与隐藏嗜酸菌对重金属响应转录调控,这可能有助于该菌生存于一些重金属污染酸性矿山环境中;TCS-4参与多重耐药基因家族转录调控;TCS-6和TCS-7可能参与氮素代谢转录调控;Orphan HK3和Orphan RR3与甲基化有关;还有部分功能仍然不清楚.这些分析结果也表明隐藏嗜酸菌A. cryptumJF-5菌株存在一个复杂的调控网络(见表 3).
2.2 隐藏嗜酸菌HKs结构域组成HKs是多结构域的同型二聚体蛋白,包括一个多变的N-末端和一个保守C-末端.N-末端为来自不同细胞位置(细胞外、细胞内或跨膜)的信号识别位点,大多数N-末端区域存在有1~20个跨膜螺旋(也有个例出现[15]);C-末端区域包含有将信号转导到相应RR蛋白的保守组氨酸残基,有典型的功能域如HATPase_c和His_kinase[10, 16].利用SMART数据库对隐藏嗜酸菌A. cryptumJF-5的HKs进行结构域分析,发现大多数HKs的N-末端具有接受信号的信号输入结构域(Input),如跨膜区、HAMP、PAS等结构域,见图 1.
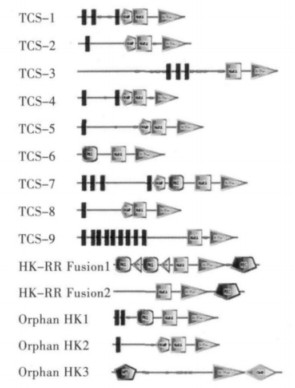 |
| 图 1 隐藏嗜酸菌Acidiphilium cryptum JF-5双组分信号转导系统中HKs结构域 |
2.3 隐藏嗜酸菌RRs结构域组成
RR蛋白大多数通常为转录因子调节相关基因转录[16],在N-末端具有一个保守的磷酸受体结构域(REC),在C-末端输出结构域(Output domain).大多数RRs中含有一个保守的天冬氨酸残基(Asp,D), 为接受磷酸化的位点[17].根据NCBI的BLASTp在线同源性比对分析,发现隐藏嗜酸菌A. cryptumJF-5的RRs可分为OmpR (6)、CopR (1)、NtrC (1)、AtoC (1)、PhoB (1)、unclassified (4)等亚家族(注:括号内为数量),隐藏嗜酸菌A. cryptumJF-5的RRs主要是OmpR亚家族,占总RRs的40 %以上.OmpR亚家族是目前已知的存在数目最多的一类应答调节蛋白,含有一个翼状螺旋-转角-螺旋的DNA结合基序[18].
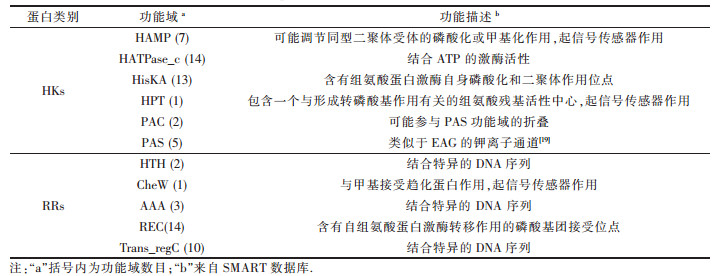
2.4 隐藏嗜酸菌双组分信号转导系统结构功能域分析基于SMART数据库,发现隐藏嗜酸菌A. cryptumJF-5的双组分调控系统基因中所存在的已知功能域(表 4),其中14个组氨酸蛋白激酶均存在结合ATP的激酶活性功能域(HATPase_c),而14个反应调节蛋白均存在接受磷酸基团的功能域(REC).此外,发现Orphan HK3(Acry2708)蛋白中存在有HPT功能域,该功能域包含一个与形成转磷酸基作用有关的组氨酸残基活性中心,而这种功能域一般只存在于真核生物中.
| 表4 隐藏嗜酸菌A. cryptumJF-5双组分调控系统基因中的已知功能域 |
 |
| 点击放大 |
2.5 隐藏嗜酸菌双组分信号转导系统基因进化分析
利用MEGA软件采用NJ法对隐藏嗜酸菌A. cryptumJF-5双组分调控系统的HKs和RRs构建进化树,见图 2.从进化关系上来看,在HKs蛋白进化中,TCS-1和TCS-2共同聚簇,Orphan HK3和TCS-9共同聚簇,TCS-4和Orphan HK2共同聚簇,TCS-3、TCS-5和Orphan HK1共同聚簇,杂合蛋白Fusion 1和Fusion 2共同聚簇(见图 2-HK);在RRs蛋白进化中,TCS-1和TCS-2共同聚簇,TCS-6、TCS-7和TCS-9共同聚簇,TCS-4、TCS-8和Orphan RR1共同聚簇,TCS-5和Orphan RR2共同聚簇(见图 2-RR),基因共同聚簇显示了高度同源性,说明这些处在同一分支上的共同聚簇基因可能具有相同的进化途径.
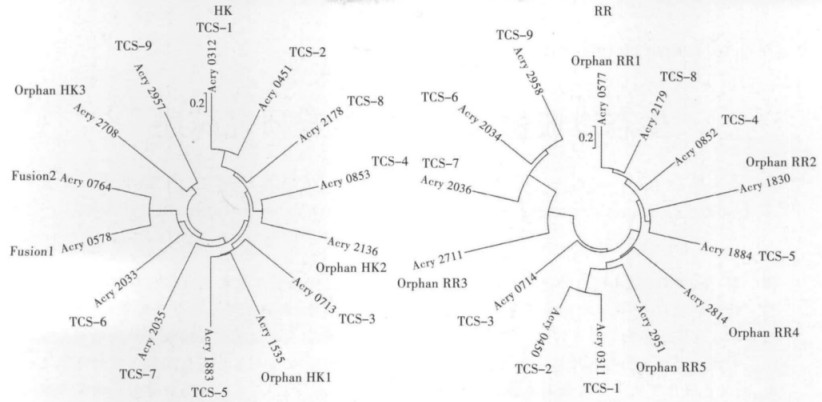 |
| 图 2 利用MEGA 5.1(NJ法)构建的隐藏嗜酸菌A. cryptumJF-5的HKs和RRs进化树 |
3 结论
本研究对隐藏嗜酸菌A. cryptumJF-5的TCSs进行了预测及相关功能分析,结果如下:
(1)在A. cryptumJF-5菌株TCS中,HKs和RRs都为14个,其中成对的TCS (HK-RR)为9对,杂合结构的TCS (Fusion)为2个,孤儿Orphan HKs和Orphan RRs分别为3个和5个.
(2)研究发现TCS-1、TCS-2、Orphan RR1、Orphan RR4和Orphan RR5可能参与隐藏嗜酸菌对重金属响应转录调控,这将有助于该菌生存在一些重金属污染酸性矿山环境中.
(3)A. cryptumJF-5菌株中大多数HKs的N-末端具有接受信号的信号输入结构域(Input),如跨膜区、HAMP、PAS等结构域.
(4)A. cryptumJF-5菌株的RRs可分为OmpR、CopR、NtrC、AtoC、PhoB、unclassified等亚家族,其中主要是OmpR亚家族,均占总RRs的40 %以上.
(5)A. cryptumJF-5菌株中的已知功能结构域主要包括HAMP、HATPase_c、HisKA、HPT、PAC、PAS、HTH、CheW、AAA、REC、Trans_regC等结构域.
(6)从进化关系上来看,发现A. cryptumJF-5菌株的部分TCS基因处在同一分支上的共同聚簇基因可能具有相同的进化途径.
在隐藏嗜酸菌中,TCS作为一种重要的调控机制,通过调控其多种生理生化过程,来适应外界环境的变化.深入研究隐藏嗜酸菌TCSs,将对阐明其适应多种极端环境因素的分子机制具有重大意义,但当前对其研究还十分有限.大多数TCSs在细胞中作用及它们激活基因,特别是RR调控的目标蛋白等尚不清楚,因此隐藏嗜酸菌TCS有待于研究者进一步深入研究和功能验证.
| [1] |
Kusel K, Dorsch T, Acker G, et al. Microbial reduction of Fe (Ⅲ) in acidic sediments: isolation of Acidiphilium cryptum JF-5 capable of coupling the reduction of Fe (Ⅲ) to the oxidation of glucose[J].
Appl Environ Microbiol, 1999, 65(8): 3633–3640. |
| [2] |
Bilgin A A, Silverstein J, Jenkins J D. Iron respiration by Acidiphilium cryptum at pH 5[J].
FEMS Microbiol Ecol, 2004, 49(1): 137–143. DOI: 10.1016/j.femsec.2003.08.018. |
| [3] |
Magnuson T S, Swenson M W, Paszczynski A J, et al. Proteogenomic and functional analysis of chromate reduction in Acidiphilium cryptum JF-5, an Fe (III)-respiring acidophile[J].
Biometals, 2010, 23(6): 1129–1138. DOI: 10.1007/s10534-010-9360-y. |
| [4] | 历丽.隐藏嗜酸菌对嗜酸氧化亚铁硫杆菌的As3+抗性基因表达与浸矿作用的影响研究[D].长沙:中南大学, 2010. http://cdmd.cnki.com.cn/Article/CDMD-10533-2010187753.htm |
| [5] | 杨宇, 张帅, 徐爱玲, 等. 隐藏嗜酸菌DX1-1和氧化亚铁硫杆菌CMS的紫外诱变育种及浸矿研究[J]. 中南大学学报:自然科学版, 2010, 41(2): 393–399. |
| [6] |
Xu A L, Xia J L, Liu K K, et al. Real-time PCR analysis of metabolic pathway of PHB in Acidiphilium cryptum DX1-1[J].
J Microbiol Biotechnol, 2010, 20(1): 71–77. |
| [7] | 徐爱玲, 张帅, 张燕飞, 等. 积累PHB菌种隐藏嗜酸菌DX1-1的诱变改良[J]. 微生物学通报, 2008, 35(10): 1516–1521. |
| [8] | 徐爱玲, 历丽, 张帅, 等. 隐藏嗜酸菌DX1-1产PHB条件的优化[J]. 武汉大学学报:理学版, 2008, 54(6): 707–712. |
| [9] |
West A H, Stock A M. Histidine kinases and response regulator proteins in two-component signaling systems[J].
Trends Biochem Sci, 2001, 26(6): 369–376. DOI: 10.1016/S0968-0004(01)01852-7. |
| [10] |
Finn R D, Mistry J, Tate J, et al. The Pfam protein families database[J].
Nucleic Acids Res, 2010, 38: 211–222. DOI: 10.1093/nar/gkp985. |
| [11] |
Mark de Been, Francke M, Moezelaar C R, et al. Comparative analysis of two-component signal transduction systems of Bacillus cereus, Bacillus thuringiensis and Bacillus anthracis[J].
Microbiology, 2006, 152: 3035–3048. DOI: 10.1099/mic.0.29137-0. |
| [12] |
Schultz J, Copley R R, Doerks T, et al. SMART: a web-based tool for the study of genetically mobile domains[J].
Nucleic Acids Res, 2000, 28(1): 231–234. DOI: 10.1093/nar/28.1.231. |
| [13] |
Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J].
Mol Biol Evol, 2011, 28(10): 2731–2739. DOI: 10.1093/molbev/msr121. |
| [14] | 张清仪, 王阶平, 程钢, 等. 苏云金芽孢杆菌双组份信号转导系统的生物信息学分析[J]. 微生物学通报, 2011, 38(9): 1385–1392. |
| [15] |
Mascher T, Helmann J D, Unden G. Stimulus perception in bacterial signal-transducing histidine kinases[J].
Microbiol Mol Biol Rev, 2006, 70(4): 910–938. DOI: 10.1128/MMBR.00020-06. |
| [16] |
Stock A M, Robinson V L, Goudreau P N. Two-component signal transduction[J].
Annu Rev Biochem, 2000, 69: 183–215. DOI: 10.1146/annurev.biochem.69.1.183. |
| [17] |
Pao G M, Saier M H, J r. Response regulators of bacterial signal transduction systems: selective domain shuffling during evolution[J].
J Mol Evol, 1995, 40(2): 136–154. DOI: 10.1007/BF00167109. |
| [18] |
Galperin M Y. Structural classification of bacterial response regulators: Diversity of output domains and domain combinations[J].
J Bacteriol, 2006, 188(12): 4169–4182. DOI: 10.1128/JB.01887-05. |
| [19] |
ZHU Lin I B, Taylor B L Dixon R. PAS domain S-boxes in Archaea, bacteria and sensors for oxygen and redox[J].
Trends Biochem Sci, 1997, 22(9): 331–333. DOI: 10.1016/S0968-0004(97)01110-9. |
 2013, Vol. 4
2013, Vol. 4

