猪圆环病毒(porcine circovirus,PCV)是一种环状、单股负链DNA病毒,属于圆环病毒科圆环病毒属[1]。2016年,Palinski等[2]在美国通过宏基因组分析,从患有PNDS的病死母猪和流产胎儿中首次发现PCV3。其后,美国、中国、巴西、意大利、韩国、泰国、西班牙、丹麦、德国、瑞典和波兰等许多国家都出现了大量关于检测出PCV3的报道,表明该病毒已快速在全球范围内传播[3-5]。2017年3月,广东省报道了我国首例PCV3感染病例,此后,在我国多个省份均有关于PCV3的报道[6-8]。除了在猪体中检测到PCV3外,在犬科动物和其他哺乳动物中也鉴定到PCV3的存在[9]。Cui等[10]提出PCV3起源于蝙蝠,鸟类是蝙蝠圆环病毒传播给猪的重要中间宿主,这种传播方式增加了跨物种传播的风险,影响到公共卫生安全。
PCV3的基因组总长为2 000 bp,包含三个主要开放阅读框(open reading frames,ORF),分别为ORF1、ORF2和ORF3。对不同国家PCV3分离株的基因序列分析显示,全基因组序列和衣壳蛋白(capsid protein,Cap蛋白)的核苷酸相似性分别为97.6%~99.8%和97.5%~100%,表明PCV3的序列高度保守[11]。进一步分析Cap蛋白的氨基酸序列可知,全球流行的PCV3毒株主要分为PCV3a、PCV3b和PCV3c三种亚型[5, 11-12],其中PCV3a的流行率正在不断上升[13]。PCV3基因组中ORF2基因编码的Cap蛋白N端与其它圆环病毒一样,富含高度碱性的精氨酸残基,是猪圆环病毒的主要结构蛋白,能引发特异性免疫反应。因此,Cap蛋白是研发检测方法的首要候选因子。当前检测PCV3常用的方法主要包括PCR与ELISA,进一步使用IHC、原位杂交以及病理观察等加以佐证。已报道文献中ELISA方法以间接ELISA方法为主,但是该方法特异性较差,容易发生交叉反应。
本研究将PCV3的Cap蛋白基因进行原核表达,成功制备得到1株具有良好阻断效果的2E6单克隆抗体。在此基础上,建立了一种阻断ELISA方法,该方法具有较高重复性和特异性,为PCV3检测提供了一种有效的技术手段。
1 材料与方法 1.1 材料SUMO-Cap重组质粒、PCV3-Cap真核质粒、PCV2-VLP以及PCV3杆状病毒液由本实验室制备与保存,猪圆环病毒3型(PCV3)、猪圆环病毒2型(PCV2)、猪伪狂犬病病毒(PRV)、猪繁殖与呼吸综合征病毒(PRRSV)、猪瘟病毒(CSFV)等阳性血清以及猪阴性对照血清由本实验室保存。BALB/c小鼠和ICR小鼠订购于上海西普尔-必凯实验动物有限公司,羊抗鼠IgG-HRP购自Cell Signaling Technology,TMB显色液购自上海碧云天生物技术有限公司。
1.2 SUMO-Cap重组蛋白的表达与纯化将SUMO-Cap重组质粒转化到Rosetta感受态细胞中,挑取单菌落摇菌至OD600 nm值为0.4~0.6后,加入终浓度为1 mmol·L-1的IPTG进行诱导。16 ℃,150 r·min-1摇菌24 h后,收集菌体超声破碎并进行SDS-PAGE鉴定,确定蛋白表达在沉淀后用尿素透析法纯化,并将纯化后的蛋白进行Western blot鉴定。鉴定成功后,测定蛋白浓度,保存于-80 ℃。
1.3 单克隆抗体的制备、纯化与鉴定用纯化后的SUMO-Cap重组蛋白100 μg·只-1免疫6~8周龄雌性BALB/c小鼠,3次免疫后再进行一次冲击免疫。取免疫后的小鼠脾细胞与骨髓瘤细胞SP2/0融合,进行3次亚克隆,采用间接ELISA方法进行筛选,获得阳性杂交瘤细胞株,将细胞扩大培养并制备腹水,将采集后的腹水送至南京钟鼎生物技术有限公司进行纯化与HRP酶标。用pCold-SUMO空载作为阴性对照,PCV2-VLP作为无关蛋白对照,进行Western blot鉴定。将PCV3-Cap真核质粒转染PK-15细胞,用制备的单抗作为一抗,进行间接免疫荧光(IFA)检测,以此来鉴定单抗的特异性。
1.4 阻断ELISA方法的建立与最佳反应条件的优化根据方阵滴定法,以2、1.5、1.0、0.5、0.25 μg·mL-1蛋白浓度100 μL·孔-1进行抗原包被;抗原包被条件分为37 ℃ 2 h、4 ℃ 12 h以及37 ℃ 2 h转至4 ℃ 12 h三种方法;PBST洗3次,封闭液选择1%BSA、2%BSA、5%脱脂乳、2%明胶,200 μL·孔-1在37 ℃下进行封闭,封闭时间设置为1、2和3 h;PBST洗3次,阳性血清和阴性血清按照1 ∶1、1 ∶5、1 ∶10、1 ∶50进行稀释,100 μL·孔-1加入后,在37 ℃下作用时间分为0.5、1、1.5、2、2.5 h;PBST洗3次,酶标单抗按照1 ∶2 000、1 ∶2 500、1 ∶3 000、1 ∶3 500进行稀释,100 μL·孔-1加入后,在37 ℃下作用时间分为0.5、1、1.5、2 h;PBST洗3次,避光加入100 μL·孔-1的TMB,在37 ℃下作用时间分为5、10、15、20、25 min;显色结束后,加入50 μL·孔-1终止液。酶标仪测定OD450值,阻断率(PI)=(阴性血清OD450 nm值-被检血清OD450 nm值)/阴性血清OD450 nm值×100%。
1.5 阻断ELISA方法临界值的确定用建立的阻断ELISA方法检测50份已知的PCV3阴性血清,计算PI的平均值(x)与标准差(s),然后以x +2 s和x +3 s的值确定临界值。
1.6 特异性试验按照已经优化好条件的阻断ELISA方法检测猪圆环病毒3型(PCV3)、猪圆环病毒2型(PCV2)、伪狂犬病病毒(PRV)、猪繁殖与呼吸综合征病毒(PRRSV)和猪瘟病毒(CSFV)阳性血清,重复3次取平均值后计算PI,以此评判该方法的特异性。
1.7 敏感性试验将3份PCV3阳性血清进行连续倍比稀释,用建立的阻断ELISA方法进行检测,以此评判该方法的敏感性。
1.8 重复性试验使用同一批次包被的酶标板检测3份PCV3阳性血清,重复3次,计算批内变异系数。包被3批不同批次的酶标板,检测3份阳性血清,重复3次取平均值,计算批间变异系数。综合上述结果,评判该方法的重复性。
1.9 符合率试验用本研究建立的阻断ELISA方法与IPMA方法相对比,检测150份猪血清样品,根据公式计算Kappa值。当0<Kappa≤0.4时,说明试验的一致性较差;当0.4<Kappa<0.75时,说明试验具有中度的一致性;当Kappa≥0.75时,说明试验具有高度的一致性。IPMA试验方法如下:将Sf9细胞铺于96孔板中,细胞汇合度为80%以上时,将PCV3杆状病毒液加入到细胞中培养48 h,弃掉培养基,每孔加入含1%H2O2的甲醇进行固定,15 min后弃掉固定液,PBST洗3次,5%脱脂乳200 μL·孔-1,4 ℃封闭过夜。弃掉封闭液,PBST洗3次,加入0.5%Triton X-100进行透膜,PBST洗3次,待检血清进行1 ∶100稀释,100 μL·孔-1加入到细胞板中,37 ℃作用1 h。PBST洗3次,用1 ∶5 000稀释HRP标记的羊抗猪酶标二抗,100 μL·孔-1加入到细胞板中,37 ℃作用45 min。PBST洗3次,100 μL·孔-1加入AEC显色液,37 ℃避光作用15 min,用超纯水洗3次,观察结果。显红色说明血清为阳性,不显色说明血清为阴性。
2 结果 2.1 目的蛋白的纯化与鉴定SDS-PAGE结果显示,SUMO-Cap重组蛋白主要存在于包涵体中,经包涵体纯化后可得到纯度较高的蛋白,相对分子质量大小约为40 ku(图 1A),与预期结果一致。Western blot结果表明,纯化后的重组蛋白可与PCV3猪源阳性血清发生特异性反应(图 1B)。
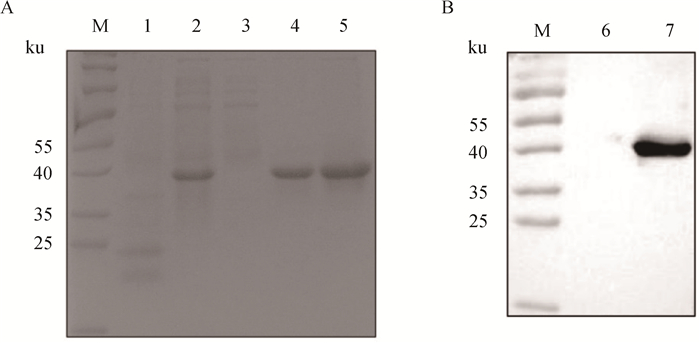
|
M. 180 ku蛋白相对分子质量标准; 1. 诱导后pCold-SUMO空载对照;2. 诱导后SUMO-Cap全菌对照;3. 超声破碎上清;4. 超声破碎沉淀;5. 纯化后SUMO-Cap重组蛋白;6. pCold-SUMO空载;7. 纯化后SUMO-Cap重组蛋白 M. 180 ku protein marker; 1. pCold-SUMO empty vector control after induction; 2. SUMO-Cap whole bacteria after induction; 3. Supernatant of sonication.; 4. Precipitation Supernatant of sonication; 5. Purified SUMO-Cap recombinant protein; 6. Production for pCold-SUMO empty vector; 7. Purified SUMO-Cap recombinant protein 图 1 SUMO-Cap重组蛋白的SDS-PAGE分析(A)及Western blot鉴定(B) Fig. 1 SDS-PAGE analysis (A) and Western blot identification (B) of SUMO-Cap recombinant protein |
多次亚克隆筛选后得到杂交瘤细胞株2E6,制备腹水使用ProteinA/G的方法纯化获得抗体(图 2A)。Western blot结果表明,得到的单克隆抗体可与SUMO-Cap重组蛋白发生特异性反应(图 2B)。IFA结果显示,2E6单克隆抗体与PCV3具有良好的反应性(图 2C)。
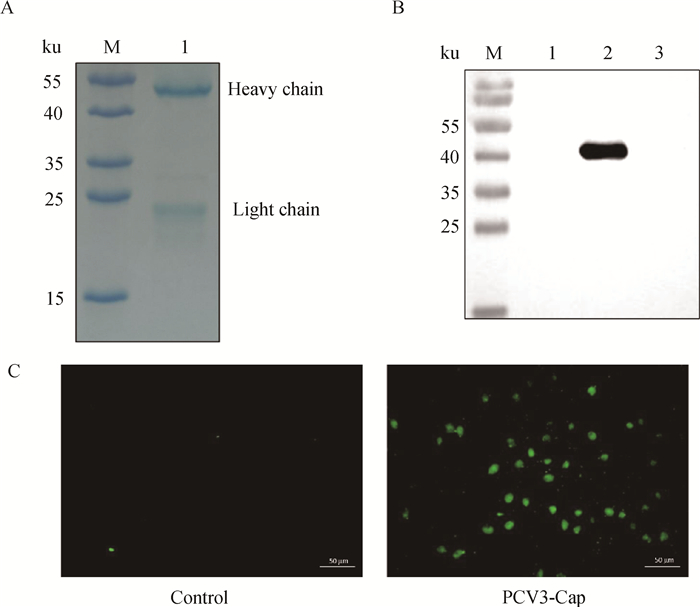
|
A.抗体纯化效果(M. 180 ku蛋白质相对分子质量标准;1. 纯化后的2E6单抗);B.抗体Western blot鉴定(M.180 ku蛋白质相对分子质量标准;1. pCold-SUMO空载;2. 纯化后SUMO-Cap重组蛋白;3. PCV2-VLP);C.2E6单抗IFA鉴定(400×) A.Purification of mAb (M. 180 ku protein marker; 1. Purified 2E6 mAb); B. Identification of mAb by Western blot (M.180 ku protein marker; 1. pCold-SUMO empty vector; 2. Purified SUMO-Cap recombinant protein; 3. PCV2-VLP); C. Identification of monoclonal antibody to 2E6 by IFA (400×) 图 2 2E6单克隆抗体的纯化效果(A)及特异性检验(B、C) Fig. 2 Purification effect (A) and specificity test (B, C) of 2E6 monoclonal antibody |
通过不断优化阻断ELISA的反应条件,确定了最佳条件如下:抗原包被浓度为0.5 μg·mL-1,包被条件为37 ℃ 2 h转至4 ℃ 12 h;最佳封闭剂为5%脱脂乳,封闭条件为37 ℃ 3 h;血清稀释度为1 ∶1,反应条件为37 ℃ 2.5 h;酶标单抗最佳稀释度为1 ∶3 000,反应条件为37 ℃ 1 h;TMB的最佳反应条件为37 ℃ 15 min。
2.4 临界值的确定按照优化后的阻断ELISA方法检测50份已知PCV3的阴性血清,如表 1所示,平均阻断率为14.81%,标准差为6.74%,x +2 s=28.30%,x +3 s=35.05%。综上可得:当PI≤28.30%时,判定结果为阴性;当PI≥35.05%时,判定结果为阳性;当28.30%<PI<35.05%时,判定为可疑,重复一次试验后如果结果仍为可疑,则判定为阳性。
|
|
表 1 阻断ELISA临界值的确定 Table 1 The Cut-off of the blocking ELISA |
使用本试验中建立好的阻断ELISA方法检测PCV3、PCV2、PRV、PRRSV和CSFV抗体阳性血清,结果如图 3所示,PCV3阳性血清的PI值为83.12%,其他病原的阳性血清PI值均小于28.30%,由此说明该方法的特异性较好。
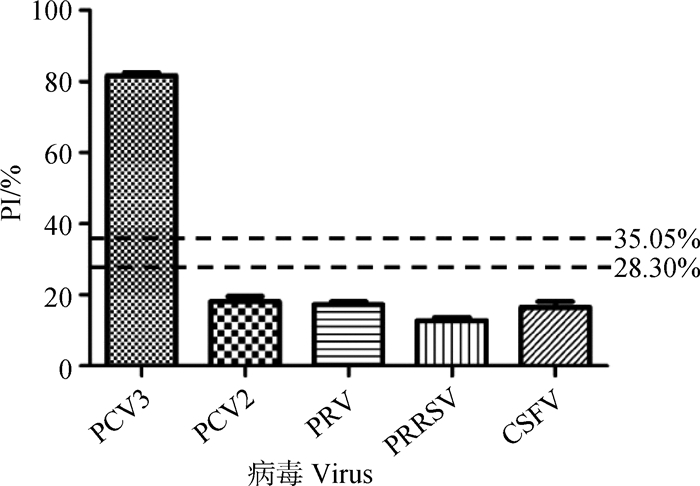
|
图 3 阻断ELISA特异性检验 Fig. 3 Results of the specificity test for blocking ELISA |
将3份PCV3阳性血清进行倍比稀释,按照已经优化好的阻断ELISA方法进行检测,结果如图 4所示,检测效价可达到1 ∶128。

|
图 4 阻断ELISA敏感性检验 Fig. 4 Results of the sensitivity test for blocking ELISA |
使用建立好的阻断ELISA方法检测3份PCV3阳性血清,进行批内和批间重复,结果如表 2、3所示,批内变异系数为0.46%~0.92%,批间变异系数为1.29%~4.11%,两者的变异系数均小于10%,说明该方法具有良好的重复性。
|
|
表 2 阻断ELISA的批内重复试验结果 Table 2 The result of intra-repeatability test by blocking ELISA |
|
|
表 3 阻断ELISA的批间重复试验结果 Table 3 The result of inter-repeatability by blocking ELISA |
2.8.1 IPMA法检测PCV3阳性血清 将PCV3杆状病毒液感染Sf9细胞48 h后,待检临床猪血清作为一抗,二抗使用HRP标记的羊抗猪酶标二抗,用AEC法显色。如图 5所示,不显色说明血清为阴性,显红色说明血清为阳性。

|
图 5 IPMA法检测PCV3血清 Fig. 5 Detection of PCV3 serum by IPMA method |
2.8.2 比对试验 使用本研究中优化后的阻断ELISA方法,与本实验室前期建立的IPMA方法同时检测150份猪血清样品,根据公式计算Kappa值,结果如表 4所示,与IPMA方法比对,计算得到的Kappa值为0.9。该阻断ELISA方法与IPMA方法比对后的Kappa值大于0.75,说明该方法具有良好的符合率。
|
|
表 4 比对试验结果 Table 4 Comparison test results |
当猪体感染PCV3后,据文献报道主要损伤的是呼吸与消化系统[7, 14]。与此同时,会引起肠道微生物群的动态变化,破坏了菌群的平衡,其次伴随着渗出性皮炎等症状[15-16]。PCV3存在垂直传播,会穿过胎盘屏障并且在胎儿的组织内复制,产生较高的病毒载量,甚至导致怀孕母猪出现死胎与木乃伊胎[17-20]。PCV3持续性病毒血症的出现,增加了再次感染的可能性,虽然病毒可能在猪体内长时间存在,但却不会表现出明显的临床症状[11, 21-23]。不同阶段的猪均可以检出PCV3,在木乃伊胎儿中的比例最高,该病毒可能是导致繁殖失败的原因之一[24-25]。研究表明,PCV3可感染多种动物,不只局限于猪[9]。由于该病毒存在众多的中间宿主,此特性也为其在种间传播提供了可能,将对养猪业的健康发展造成了巨大的威胁。
在临床检测中,PCV3会出现病毒血症,部分猪体在咽拭子与肛门拭子中也可检出PCV3阳性[26]。关于PCV3病毒载量的测定,淋巴结中的病毒载量明显高于肺和其他组织[4]。该病毒具有广泛的组织嗜性,使用免疫组织化学法(IHC),可在感染PCV3猪体的肾、肺以及淋巴结中观察到病原的位置[27-28]。当前检测PCV3常用的方法有PCR、ELISA、IHC、原位杂交以及组织病理学观察等[29-30]。
目前,尚无有效预防PCV3感染的相关疫苗以及治疗药物,这为防范PCV3的感染增加了难度[31]。加强猪场的日常管理与环境卫生是预防PCV3的主要手段,做到尽早发现并快速阻止病毒的进一步扩散[32]。因此,实时监测临床中PCV3的流行情况,持续分析相关分子流行病学动态变化是至关重要的。本研究中的重组蛋白有大规模可获得性且操作简便等优点,将重组蛋白免疫BALB/c小鼠制备获得了一株分泌阻断效果良好抗体的杂交瘤细胞株2E6,以此制备腹水得到的单克隆抗体具有构象表位,可识别具有空间结构的PCV3病毒粒子,为后期对PCV3的深入研究提供了有利的条件。此外,市面上并没有特异性较好的商品化阻断ELISA抗体检测试剂盒,本研究中建立的方法特异性较高,与PCV2、PRV、PRRSV、CSFV抗体阳性血清均不发生反应;具有可重复性,批内变异系数为0.46%~ 0.92%,批间变异系数为1.29%~4.11%,两者的变异系数均小于10%;该方法与IPMA方法比对后的Kappa值均大于0.75,说明该方法具有良好的符合率;能够快速检测出PCV3抗体的存在,可应用于猪场的日常管理中。与此同时,应该尽快开发出针对性强的疫苗,不断提高对PCV3的防控,减少养殖过程中引发的经济损失。
4 结论本研究成功筛选到1株具有良好阻断效果的猪圆环病毒3型Cap蛋白单克隆抗体,并在此基础上建立了一种具有较高重复性和特异性的阻断ELISA检测方法,为临床PCV3流行病学调查提供了技术支撑。
| [1] |
TODD D. Circoviruses: immunosuppressive threats to avian species: a review[J]. Avian Pathol, 2000, 29(5): 373-394. DOI:10.1080/030794500750047126 |
| [2] |
PALINSKI R, PIÑEYRO P, SHANG P C, et al. A novel porcine circovirus distantly related to known circoviruses is associated with porcine dermatitis and nephropathy syndrome and reproductive failure[J]. J Virol, 2016, 91(1): e01879-16. |
| [3] |
KWON T, YOO S J, PARK C K, et al. Prevalence of novel porcine circovirus 3 in Korean pig populations[J]. Vet Microbiol, 2017, 207: 178-180. DOI:10.1016/j.vetmic.2017.06.013 |
| [4] |
KEDKOVID R, WOONWONG Y, ARUNORAT J, et al. Porcine circovirus type 3 (PCV3) infection in grower pigs from a Thai farm suffering from porcine respiratory disease complex (PRDC)[J]. Vet Microbiol, 2018, 215: 71-76. DOI:10.1016/j.vetmic.2018.01.004 |
| [5] |
LI G R, WANG H J, WANG S L, et al. Insights into the genetic and host adaptability of emerging porcine circovirus 3[J]. Virulence, 2018, 9(1): 1301-1313. DOI:10.1080/21505594.2018.1492863 |
| [6] |
SHEN H, LIU X, ZHANG P, et al. Genome characterization of a porcine circovirus type 3 in South China[J]. Transbound Emerg Dis, 2018, 65(1): 264-266. DOI:10.1111/tbed.12639 |
| [7] |
QI S S, SU M J, GUO D H, et al. Molecular detection and phylogenetic analysis of porcine circovirus type 3 in 21 Provinces of China during 2015—2017[J]. Transbound Emerg Dis, 2019, 66(2): 1004-1015. DOI:10.1111/tbed.13125 |
| [8] |
吕其壮, 覃婷, 龚紫凤, 等. 猪圆环病毒3型流行病学研究进展[J]. 中国预防兽医学报, 2021, 43(5): 567-571. LÜ Q Z, QIN T, GONG Z F, et al. Epidemiological research progress on porcine circovirus type 3[J]. Chinese Journal of Preventive Veterinary Medicine, 2021, 43(5): 567-571. (in Chinese) |
| [9] |
SUN W C, WANG W, XIN J L, et al. An epidemiological investigation of porcine circovirus 3 infection in dogs in the Guangxi Province from 2015 to 2017, China[J]. Virus Res, 2019, 270: 197663. DOI:10.1016/j.virusres.2019.197663 |
| [10] |
CUI Y Q, HOU L, PAN Y, et al. Reconstruction of the evolutionary origin, phylodynamics, and phylogeography of the porcine circovirus type 3[J]. Front Microbiol, 2022, 13: 898212. DOI:10.3389/fmicb.2022.898212 |
| [11] |
GE M, REN J, XIE Y L, et al. Prevalence and genetic analysis of porcine circovirus 3 in China from 2019 to 2020[J]. Front Vet Sci, 2021, 8: 773912. DOI:10.3389/fvets.2021.773912 |
| [12] |
CHUNG H C, NGUYEN V G, PARK Y H, et al. Genotyping of PCV3 based on reassembled viral gene sequences[J]. Vet Med Sci, 2021, 7(2): 474-482. DOI:10.1002/vms3.374 |
| [13] |
HA Z, LI J F, XIE C Z, et al. Prevalence, pathogenesis, and evolution of porcine circovirus type 3 in China from 2016 to 2019[J]. Vet Microbiol, 2020, 247: 108756. DOI:10.1016/j.vetmic.2020.108756 |
| [14] |
ZHANG F F, YUAN W F, LI Z Q, et al. Porcine circovirus type 3 in pig farms experiencing diarrhea in Jiangxi, China: prevalence, genome sequence and pathogenicity[J]. Animals (Basel), 2020, 10(12): 2324. |
| [15] |
HOU L, WANG J, ZHANG W, et al. Dynamic alterations of gut microbiota in porcine circovirus type 3-infected piglets[J]. Front Microbiol, 2020, 11: 1360. DOI:10.3389/fmicb.2020.01360 |
| [16] |
JIANG H J, WANG D, WANG J, et al. Induction of porcine dermatitis and nephropathy syndrome in piglets by infection with porcine circovirus type 3[J]. J Virol, 2019, 93(4): e02045-18. |
| [17] |
YANG Z, MARTHALER D G, ROVIRA A. Frequency of porcine circovirus 3 detection and histologic lesions in clinical samples from swine in the United States[J]. J Vet Diagn Invest, 2022, 34(4): 602-611. DOI:10.1177/10406387221099538 |
| [18] |
RUIZ A, SAPORITI V, HUERTA E, et al. Exploratory study of the frequency of detection and tissue distribution of porcine circovirus 3 (PCV-3) in pig fetuses at different gestational ages[J]. Pathogens, 2022, 11(2): 118. DOI:10.3390/pathogens11020118 |
| [19] |
何庆, 李周勉, 王欢, 等. 猪流产胎儿中猪圆环病毒3型的检测及其遗传演化分析[J]. 畜牧兽医学报, 2022, 53(5): 1527-1535. HE Q, LI Z M, WANG H, et al. Detection and genetic evolution of porcine circovirus type 3 in swine aborted fetus[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(5): 1527-1535. (in Chinese) |
| [20] |
KROEGER M, TEMEEYASEN G, PIÑEYRO P E. Five years of porcine circovirus 3:What have we learned about the clinical disease, immune pathogenesis, and diagnosis[J]. Virus Res, 2022, 314: 198764. DOI:10.1016/j.virusres.2022.198764 |
| [21] |
SIRISEREEWAN C, THANAWONGNUWECH R, KEDKOVID R. Current understanding of the pathogenesis of porcine circovirus 3[J]. Pathogens, 2022, 11(1): 64. DOI:10.3390/pathogens11010064 |
| [22] |
TEMEEYASEN G, LIERMAN S, ARRUDA B L, et al. Pathogenicity and immune response against porcine circovirus type 3 infection in caesarean-derived, colostrum-deprived pigs[J]. J Gen Virol, 2021, 102(11): 001502. |
| [23] |
MORA-DÍAZ J, PIÑEYRO P, SHEN H G, et al. Isolation of PCV3 from perinatal and reproductive cases of PCV3-associated disease and in vivo characterization of PCV3 replication in CD/CD growing pigs[J]. Viruses, 2020, 12(2): 219. DOI:10.3390/v12020219 |
| [24] |
SUKMAK M, THANANTONG N, POOLPERM P, et al. The retrospective identification and molecular epidemiology of porcine circovirus type 3 (PCV3) in swine in Thailand from 2006 to 2017[J]. Transbound Emerg Dis, 2019, 66(1): 611-616. DOI:10.1111/tbed.13057 |
| [25] |
DAL SANTO A C, CEZARIO K C, BENNEMANN P E, et al. Full-genome sequences of porcine circovirus 3 (PCV3) and high prevalence in mummified fetuses from commercial farms in Brazil[J]. Microb Pathog, 2020, 141: 104027. DOI:10.1016/j.micpath.2020.104027 |
| [26] |
WOŹNIAK A, MIŁEK D, STADEJEK T. Wide range of the prevalence and viral loads of porcine circovirus type 3 (PCV3) in different clinical materials from 21 polish pig farms[J]. Pathogens, 2020, 9(5): 411. DOI:10.3390/pathogens9050411 |
| [27] |
LIU Y Y, ZHANG S, SONG X H, et al. The prevalence of novel porcine circovirus type 3 isolates in pig farms in China[J]. Transbound Emerg Dis, 2019, 66(5): 2143-2151. DOI:10.1111/tbed.13266 |
| [28] |
TAN C Y, LIN C N, OOI P T. What do we know about porcine circovirus 3 (PCV3) diagnosis so far?: A review[J]. Transbound Emerg Dis, 2021, 68(6): 2915-2935. DOI:10.1111/tbed.14185 |
| [29] |
OH T, CHAE C. First isolation and genetic characterization of porcine circovirus type 3 using primary porcine kidney cells[J]. Vet Microbiol, 2020, 241: 108576. DOI:10.1016/j.vetmic.2020.108576 |
| [30] |
OUYANG T, NIU G Y, LIU X H, et al. Recent progress on porcine circovirus type 3[J]. Infect Genet Evol, 2019, 73: 227-233. DOI:10.1016/j.meegid.2019.05.009 |
| [31] |
JIANG Z X, WU J J, JIANG M, et al. A novel technique for constructing infectious cloning of type 3 porcine circovirus[J]. Front Microbiol, 2020, 11: 1067. DOI:10.3389/fmicb.2020.01067 |
| [32] |
NIU G Y, CHEN S, LI X, et al. Advances in crosstalk between porcine circoviruses and host[J]. Viruses, 2022, 14(7): 1419. DOI:10.3390/v14071419 |
(编辑 白永平)



