2. 西南民族大学青藏高原研究院, 成都 610207
2. Institute of Tibetan Plateau Research, Southwest Minzu University, Chengdu 610207, China
山羊肉具有肉质细腻,低脂高蛋白等特点,受到众多国民的喜爱[1]。肌内脂肪(IMF)为肌肉纤维之间的脂肪组织,是肉产品生产中一个重要的经济性状,其也被称为大理石花纹,适当的IMF含量可以增加肉产品的嫩度、多汁性和风味,从而改善肉产品的口感和品质[2-4]。IMF沉积由脂肪合成与分解代谢共同决定[5-6],其中脂肪合成的机制尚不明确,因此,需要对IMF沉积过程中脂肪合成的分子调控机制进行研究,为改善山羊肉品质提供理论参考依据。
蛋白酶体是存在于真核细胞中溶酶体外的蛋白水解体系,是依赖ATP的蛋白水解酶复合物[7],Xu等[8]研究发现蛋白酶体受到抑制后骨髓瘤细胞中脂质代谢发生改变。蛋白酶体包括两种形式:20S蛋白酶体和26S蛋白酶体[9]。有研究报道,26S蛋白酶体能直接或间接调控脂质代谢过程[10]。26S蛋白酶体非ATPase调节亚基9(proteasome 26S subunit, non-ATPase 9,PSMD9),是26S蛋白酶体的调节因子,定位于染色体12q24.31~q24.32上[11-12]。Parker等[13]通过脂质组学和蛋白组学研究发现PSMD9参与脂质代谢的调控。在疾病上研究发现,PSMD9与肥胖、Ⅱ型糖尿病、血脂异常和心血管等疾病相关[14-18]。PSMD9可通过调节胰岛素基因转录,促进脂肪合成,从而导致高胆固醇血症[19-20]。此外,PSMD9可作为酰基甘油的驱动因子调节肝酰基甘油代谢[21]。在小鼠上的研究发现,干扰PSMD9基因能够减少肝脏脂肪生成[13]。这些研究结果表明,PSMD9对机体脂质代谢具有重要作用,然而,目前对PSMD9的研究主要集中在人类疾病和小鼠等模式动物上,关于PSMD9基因在山羊肌内前体脂肪细胞脂质沉积中的作用尚未见报道。
因此,本试验以简州大耳羊为研究对象,利用PCR技术克隆山羊PSMD9基因序列,测定其在不同分化时期及不同组织中的表达水平,并进行生物信息学分析。通过过表达和干扰PSMD9基因进一步探究PSMD9对肌内脂肪沉积的调控作用,为提高山羊IMF含量改善羊肉品质提供理论基础。
1 材料与方法 1.1 试验材料从四川省简阳大哥大牧业有限公司随机挑选12只健康1周岁左右的简州大耳羊公羊,早上空腹,颈动脉放血致死,然后在无菌条件下采集山羊心脏、肝脏、脾脏、肾脏、肺、背最长肌、股二头肌、臂三头肌、小肠和皮下脂肪组织样品,用配好的DEPC水清洗洁净后快速分割,随后立即用锡纸包裹并存放于液氮罐中用于提取组织总RNA。本试验所用的山羊肌内前体脂肪细胞来自本实验室前期冻存的1日龄羔羊的前体脂肪细胞。
主要试剂:Gel Extraction Kit购自凯杰生物工程(深圳)有限公司(德国),gDNAse、Trizol、反转录试剂盒、限制性内切酶Hind Ⅲ与BamH I、pMD19-T购自宝生物工程(大连)有限公司,琼脂糖凝胶DNA回收试剂盒购自北京天恩泽生物技术公司(中国),QuantiFastTMSYBR®Green PCR Kit、QuantiTect Reverse Transcription Kit、DEME/F-12、opti-MEM购自赛默飞世尔科技公司(美国),DH5α感受态细胞、高纯度质粒小提中量试剂盒、2×Taq PCR Master Mix、质粒小提试剂盒购自天根生化科技(北京)有限公司,宿主菌大肠杆菌E.coli DH5α购自北京擎科生物技术有限公司,氨苄购自北京孚博生物科技有限公司(德国),甘油三酯试剂盒购自北京普利莱基因技术有限公司,I型胶原酶购自西格玛奥德里奇(上海)贸易有限公司,胚胎牛血清购自北京全式金生物技术股份有限公司,无菌PBS、胰蛋白酶购自武汉博士德生物工程有限公司。
1.2 试验方法1.2.1 总RNA的提取及cDNA合成 Trizol法提取山羊各组织的总RNA,用紫外分光光度计NanoDrop-ND-1000(Agilent)测定RNA样品的浓度与OD值,所测数值均在1.8~2.0之间。根据反转录试剂盒(HiScript Ⅲ All-in one RT SuperMix Perfect for qPCR)说明书,将总RNA反转录为cDNA,具体操作如下:在RNase-free离心管中配制如下混合液:模板RNA 1 μg,Enzyme Mix 1 μL,5×All-in-one qRT SuperMix 4 μL,RNase-free ddH2O补充到总体积20 μL,使其充分混合,在PCR仪中进行反转录,反应程序为:50 ℃ 15 min,85 ℃ 5 s,-20 ℃保存备用。
1.2.2 目的基因克隆 根据GenBank公布的山羊PSMD9的预测序列(XM_005691404.3)在Primer Premier 5.0软件上设计引物(F: GCTACAGTTTCCTCGGGCGTCC; R: ACCACCTAGT-TCAGGCGTCCAG),预测长度564 bp。
以山羊的肝脏组织为模板,进行PCR反应,PCR扩增体系如下:1×T3 Super PCR Mix 12.5 μL,上、下游引物各1.0 μL,肝脏cDNA 1.0 μL,ddH2O 9.5 μL。反应条件为98 ℃ 3 min;98 ℃ 30 s,62.2 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 7 min。PCR结束之后,进行琼脂糖凝胶电泳检测,根据TIANGEN公司生产的琼脂糖凝胶电泳胶回收试剂盒说明书对克隆获得的目的片段进行回收。将回收片段连接到pMD-19 T载体,连接体转化至DH5α感受态细胞,挑取单一呈圆形的阳性菌落,菌液PCR鉴定单一正确的目的基因条带后,送至北京擎科生物科技有限公司测序。
1.2.3 生物信息学分析 对克隆得到的山羊PSMD9基因序列进行生物信息学分析,具体生物信息学分析内容及分析工具见表 1。
|
|
表 1 序列分析及其工具 Table 1 Sequence analysis and analysis tools |
1.2.4 山羊PSMD9基因组织表达差异分析和时序表达谱构建 采用Primer Premier 5.0软件设计目的基因的荧光特异性引物(表 2),以UXT作为内参基因矫正基因的相对表达水平,从12只公羊中随机选取3只公羊组织样,利用RT-qPCR技术检测PSMD9基因在简州大耳羊各组织中的mRNA表达水平和山羊肌内前体脂肪细胞诱导分化过程中0~8 d每间隔2 d的表达水平。RT-qPCR反应体系:cDNA稀释液1 μL,上、下游引物各0.2 μL,荧光染料(Taq Pro Universal SYBR qPCR Master Mix)5 μL,ddH2O 3.6 μL。RT-qPCR程序:95 ℃预变性3 min;95 ℃变性10 s,60 ℃退火30 s,72 ℃延伸30 s,共40个循环。
|
|
表 2 实时荧光定量PCR(RT-qPCR)引物信息 Table 2 Primers information for real-time quantitative PCR (RT-qPCR) |
1.2.5 山羊PSMD9过表达载体构建和干扰序列合成 根据山羊PSMD9基因CDS区763 bp的序列,利用BioXM2.6找出适宜的酶切位点(Hind Ⅲ、BamH I),并设计亚克隆引物(F:CCCAAGCT-TGCCACCATGGATTACAAGGATGACGACGA-TAAGTCGGAGAAGGCGGGG;R: CGCGGATC-CTCATCTTTGCAATGGAATAATGTTGCA)。利用Hind Ⅲ和BamH I对pcDNA3.1载体和亚克隆获得完整的山羊PSMD9基因CDS区序列进行双酶切,双酶切体系为亚克隆产物10 μL,Hind Ⅲ1 μL,BamH I 1 μL,10*K Buffer 2.5 μL,ddH2O 5.5 μL。37 ℃水浴过夜,利用通用型DNA纯化回收试剂盒(Universal DNA Purification kit)进行纯化,T4连接酶连接纯化产物,随后转化至DH5α感受态细胞,接种,挑取阳性菌落及测序,最后使用限制性内切酶Hind Ⅲ和BamH I对重组的pcDNA3.1-PSMD9质粒进行酶切后电泳,即双酶切鉴定。山羊siRNA序列(S:GCGACAUUCAGGAACUGAUTT;A:AUCAGUUCCUGAAUGU-CGCTT)和Negative control(S: UUCUUCGAAC-GUGUCACGUTT; A: ACGUGACACGUUCGG-AGAATT)序列由上海吉玛公司合成。siRNA经12 000 r·min-1离心10 min,并溶解于62.5 μL DEPC水。
1.2.6 山羊肌内前体脂肪细胞培养及转染 37 ℃水浴解冻实验室冻存的山羊肌内前体脂肪细胞,将细胞接种含1%双抗及10 mL·L-1胎牛血清的DEME/F-12培养基的培养瓶中进行培养,待F3代细胞生长至80%时,将细胞接种至6孔板中,每孔细胞生长至80%时转染。山羊PSMD9过表达组设置pcDNA 3.1-PSMD9组和pcDNA3.1阴性对照组。按转染试剂的说明书配制预混液,即pcDNA 3.1-PSMD9质粒1 μg,转染试剂4 μL及90 μL的无血清培养基,轻微振荡混匀后,室温静置15 min。将静置好的预混液均匀滴加到饥饿4 h细胞中,振荡混匀并置于培养箱中培养6 h后,使用50 μmol·L-1油酸诱导细胞分化,48 h后利用Trizol法收集6孔板的细胞,提取细胞RNA后反转录,将cDNA置于-20 ℃冰箱保存。山羊PSMD9干扰组设置siPSMD9和Negative control(阴性对照组),转染方法如下:siPSMD9和Negative control试剂6 μL,转染试剂4 μL及90 μL无血清培养基配制好混合液,将混合液添加至饥饿的细胞中,振荡混匀并置于培养箱中培养6 h后,使用50 μmol·L-1油酸诱导细胞分化,48 h后利用Trizol法收集6孔板的细胞,提取细胞RNA后反转录,将cDNA置于-20 ℃冰箱保存。
1.2.7 油红O染色和甘油三酯测定 用于染色的细胞接种于6孔板,10 mL·L-1甲醛固定诱导48 h的细胞30 min,在固定细胞前后各用PBS缓冲液清洗,油红O染色:利用适量油红O工作液染色30 min,PBS洗去油红O工作液,使用显微镜观察脂滴情况并拍照[22]。按照甘油三酯测定试剂盒的说明书进行操作,具体操作如下:每孔加入200 mL裂解液,吹打收集细胞于离心管中,混匀后室温静置10 min。取适量上清液70 ℃加热10 min,室温2 000 r·min-1离心5 min,上清液即可用于甘油三酯测定。
1.2.8 数据统计分析 组织表达差异分析、时序表达分析、过表达和干扰效率分析均以UXT为内参基因[23],采用2-ΔΔCt法对RT-qPCR数据进行计算,其中△△Ct=[(Ctgene-CtUXT)]试验组-[(Ctgene-CtUXT)]对照组。本研究中所有的结果数据用“平均值±标准误(Mean±SEM)”表示,显著性检验使用Graphpad prism 8.0软件中One-way ANOVA(and nonparametric or mixed)分析,P < 0.05表示差异显著,P < 0.01表示差异极显著;利用Graphpad prism 8.0绘图。每个待测样本设3个生物学重复和3个技术重复。
2 结果 2.1 山羊PSMD9基因克隆与生物信息学分析以简州大耳羊肝脏组织RNA反转录得到的cDNA为模板,采用RT-PCR扩增获得山羊PSMD9基因核苷酸序列763 bp(图 1),其中5′UTR 67 bp,CDS区564 bp,3′UTR 132 bp,编码187个氨基酸残基,起始密码子为ATG,终止密码子为TGA。
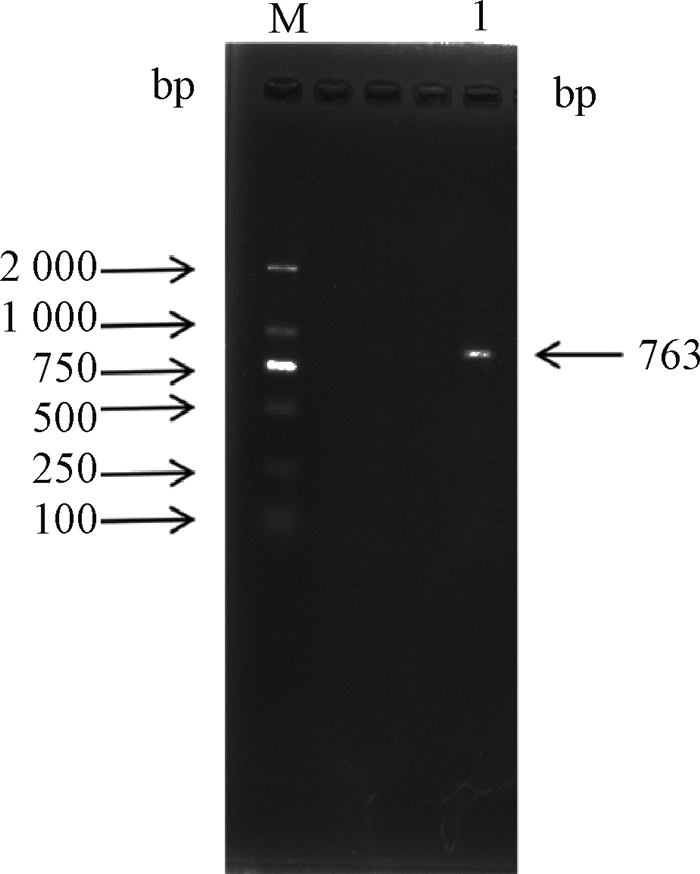
|
M. DNA相对分子质量标准;1. PSMD9基因PCR扩增产物 M. DL2000 DNA marker; 1. PCR products of PSMD9 gene 图 1 PSMD9基因目的片段的扩增 Fig. 1 Amplification of target fragment of PSMD9 gene |
利用BLAST对不同物种PSMD9基因的同源性进行比较,发现简州大耳羊与绵羊(XP_004017391.1)、牛NP_001029613.1)、野猪(XP_003132922.1)、犬(XP_025329610.1)、人(NP_002804.2)、马(XP_001496522.2)、水牛(XP_006047226.1)、大熊猫(XP_034528225.1)、骆驼(XP_006178110.1)的相似性分别为82.84%、81.86%、76.21%、74.76%、70.04%、74.63%、81.37%、75.24%、76.47%(图 2A)。采用MEGA5软件将各物种的氨基酸序列构建了系统进化树(图 2B)。
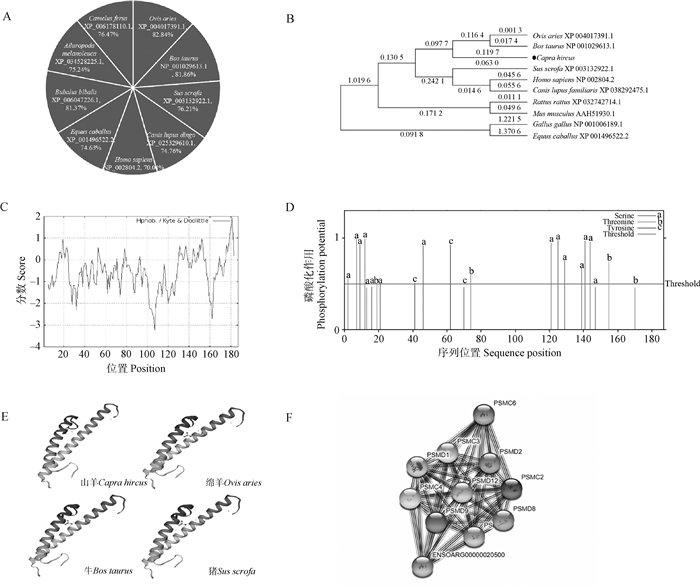
|
A. PSMD9氨基酸相似性分析;B. PSMD9系统进化树分析;C. PSMD9亲、疏水性分析;D. PSMD9磷酸化位点分析;E. 不同物种PSMD9蛋白的三级结构预测;F. 山羊PSMD9蛋白相互作用蛋白预测分析 A. PSMD9 amino acid similarity analysis; B. PSMD9 phylogenetic tree analysis; C. Hydrophilicity and hydrophobicity analysis of PSMD9; D. Analysis of PSMD9 phosphorylation site prediction; E. Prediction of tertiary structure of PSMD9 proteins from different species; F. Prediction of PSMD9 protein-interacting proteins in goats 图 2 山羊PSMD9基本理化性质及序列分析 Fig. 2 Basic physicochemical properties and sequence analysis of goat PSMD9 |
理化性质分析发现,PSMD9蛋白分子式为C881H1450N274O280S7,分子量为20 585.29 u,理论等电点(theoretical PI)为7.77,该蛋白体外半衰期在哺乳动物网织红细胞离体状态下为30 h,其蛋白不稳定系数为57.37。带负电荷的残基总数(Asp+Glu)为25,带正电荷的残基总数(Arg+Lys)为26,原子总数为2 892,脂肪系数为84.01,总平均亲水系数为-0.598。
PSMD9蛋白在176~183区有较强的疏水性,在25~52、55~70、80~92、94~129区有较强的亲水性(图 2C),整个蛋白质中亲水性最大值是3.222(第107位氨基酸),最小值是-1.933(第181位氨基酸),推测出PSMD9为亲水性蛋白。磷酸位点分析显示,PSMD9具有36个磷酸位点(图 2D)。信号肽的预测显示,发现山羊PSMD9不存在信号肽。跨膜结构分析发现PSMD9蛋白无跨膜结构。
二级结构预测显示,山羊PSMD9蛋白主要含α螺旋31.03%,无规则卷曲44.92%,β折叠5.88%。三级结构预测显示,山羊、绵羊、牛、和猪的PSMD9三级结构相似(图 2E)。利用STRING交互式数据库进行蛋白相互作用分析(图 2F),分析得出PSMD9蛋白可能与PSMC6、PSMC3、PSMC2、PSMD1、PSMD2、PSMD8和PSMD12等蛋白存在相互作用。
2.2 山羊PSMD9基因组织表达差异分析和时序表达谱构建以UXT为内参基因,以PSMD9在心脏中的表达水平做归一化处理。RT-qPCR检测结果显示,PSMD9在山羊不同组织中均有表达,在肝脏中表达量最高,脾脏中表达量最低(图 3A)。山羊PSMD9基因在诱导分化的第0~8天肌内脂肪细胞中均存在表达,诱导分化第8天时表达水平最高(图 3B),且显著高于诱导分化之前表达水平(P < 0.05)。
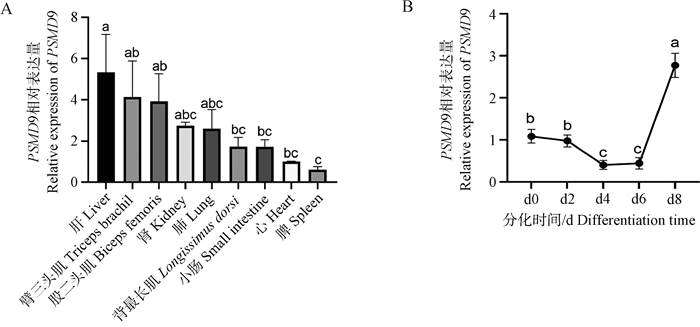
|
A. 山羊PSMD9基因组织表达谱;B. 山羊PSMD9基因在山羊肌内脂肪细胞分化过程中的相对表达水平。不同小写字母表示差异显著(P < 0.05);相同小写字母表示差异不显著(P>0.05) A. Tissue expression profile of PSMD9 gene in goats; B. Relative expression level of goat PSMD9 gene during the differentiation of goat intramuscular adipocytes. Different lowercase letters indicate significant difference(P < 0.05); The same letters indicate insignificant difference(P>0.05) 图 3 山羊PSMD9基因组织及肌内脂肪细胞不同分化阶段的相对表达水平 Fig. 3 Relative expression levels of PSMD9 gene in goat tissues and intramuscular adipocytes at different differentiation stages |
pcDNA3.1-PSMD9转染山羊肌内前体脂肪细胞并诱导48 h后,收集细胞,利用RT-qPCR检测PSMD9过表达效率,结果显示,过表达PSMD9基因后其相对表达量极显著高于对照组,表达效率约为对照组的192倍(P < 0.01);干扰PSMD9后其相对表达量显著低于对照组(P < 0.05),干扰效率为72%(图 4A)。甘油三酯测定结果显示,pcDNA3.1-PSMD9组甘油三酯含量显著高于pcDNA3.1组(P < 0.05);干扰PSMD9基因后,siPSMD9组甘油三酯含量低于Negative control组(图 4B)。油红O染色结果显示pcDNA3.1-PSMD9组较pcDNA3.1组脂滴增加;siPSMD9组较Negative control组脂滴减少(图 4C)。
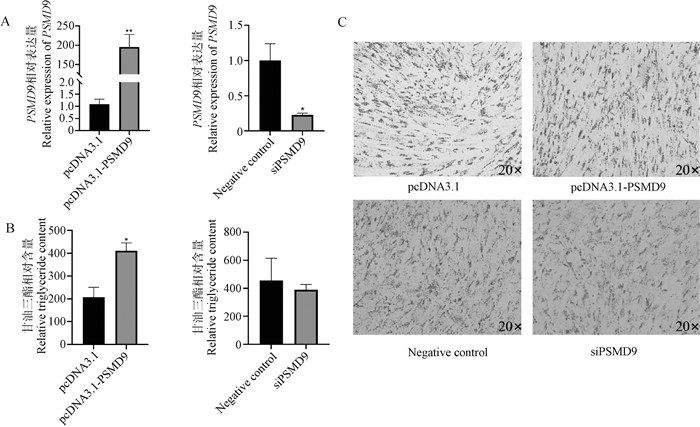
|
A. PSMD9过表达和干扰效率检测;B. 以GPO-Trinder酶法检测甘油三酯含量;C. 油红O染色。*.P < 0.05, **.P < 0.01,下同 A. PSMD9 overexpression and interference efficiency detection; B. Triglyceride content by GPO-Trinder enzyme reaction; C. Oil Red O staining.*.P < 0.05, **.P < 0.01, the same as below 图 4 PSMD9基因对山羊肌内前体脂肪细胞的影响 Fig. 4 Effect of PSMD9 gene on intramuscular precursor adipocytes of goat |
干扰山羊PSMD9后,通过RT-qPCR检测发现,二酰甘油酰基转移酶1(DGAT1)和脂肪酸合成酶(FASN)显著下调(P < 0.05),过氧化物酶体增殖物激活受体γ(PPARγ)和乙酰辅酶A羧化酶(ACC)极显著下调(P < 0.01),而二酰甘油酰基转移酶2(DGAT2)和硬脂酰辅酶A去饱和酶1(SCD1)无显著变化。过表达山羊PSMD9后,DGAT2极显著上调(P < 0.01),FASN和ACC显著上调(P < 0.05),而DGAT1、PPARγ和SCD1无显著变化(图 5)。
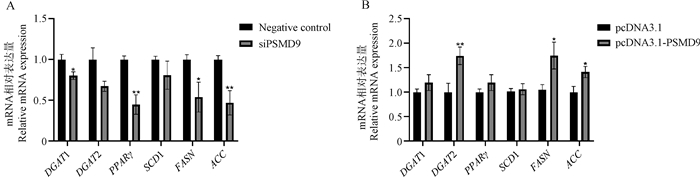
|
A.干扰PSMD9基因对脂代谢相关基因表达的影响;B.过表达PSMD9基因对脂代谢相关基因表达的影响 A. Effect of interfering with PSMD9 gene on the expression of genes related to lipid metabolism; B. Effects of overexpression of PSMD9 gene on the expression of lipid metabolism-related genes 图 5 PSMD9对脂代谢相关基因表达的影响 Fig. 5 Effect of PSMD9 on the expression of genes related to lipid metabolism |
脂肪生成过程是一个非常复杂的生物学过程,可分为两个主要阶段:转化(从间充质干细胞到前体脂肪细胞)和终末分化(从前体脂肪细胞到脂肪细胞)[24-25]。脂肪细胞的生长是由脂肪细胞数量的积累以及体积的变化导致的,而成熟的脂肪细胞不具备分裂和分化的能力,因此脂肪细胞数量上的积累是因为前体脂肪细胞的增殖分化,脂肪细胞体积的增大主要是因为胞内甘油三酯升高[26]。PSMD9基因在山羊肌内前体脂肪细胞脂质沉积中的作用是未知的,为了探索PSDM9基因在山羊肌内前体脂肪细胞中对的脂质沉积的作用,本试验克隆了PSMD9基因,得到了CDS区全序列,通过生物信息学发现PSMD9基因具有36个磷酸化位点修饰,这将有助于蛋白的翻译后修饰及细胞信号转导[27]。将山羊PSMD9氨基酸序列与其他物种比对发现, 山羊与绵羊、牛的相似性较高,说明其在不同物种中的保守性较高,这与Hopper等[12]的研究结果一致。系统进化树结果表明, 山羊PSMD9与绵羊、牛具有较高的亲缘性;对三级结构预测发现, 不同物种间的结构相似,因此推测PSMD9基因在不同物种间可能发挥着相似的作用。
脂肪细胞的分化对肌内脂肪沉积具有重要作用,为进一步探讨PSMD9基因对山羊肌内脂肪沉积的作用,本研究利用RT-qPCR检测了PSMD9基因在不同组织中的表达和山羊前体脂肪细胞分化过程中的表达。结果表明, PSMD9基因在山羊肝脏表达量最高,在肾和小肠等组织中均有表达,这与Uhlen等[28]的研究结果PSMD9在肾脏组织中具有较高表达相一致。肝脏是脂质代谢比较旺盛的组织[29],脂肪酸作为肠道的主要产物[30],本试验结果在肝脏和小肠中的高表达也体现了PSMD9基因对调控脂质代谢具有重要作用。通过分析PSMD9基因的时序表达,结果发现PSMD9基因在山羊肌内前体脂肪细胞分化的第0~8天均有表达,且在诱导分化的第8天达到最高水平,显著高于诱导分化之前表达水平(P < 0.05),由此推测,PSMD9基因在不同分化时期可能发挥不同的作用。
为了阐明PSMD9基因对山羊肌内前体脂肪细胞脂质沉积的影响,本试验利用过表达和干扰手段进行研究,结果显示,过表达PSMD9基因后促进山羊肌内前体脂肪细胞的脂滴积累和甘油三酯合成,干扰PSMD9基因后山羊肌内前体脂肪细胞脂滴聚集减少、甘油三酯合成减少,这与Parker等[13]在小鼠中的研究结果一致,上调PSMD9基因可促进脂质增加。PPARs是研究脂质代谢的重要候选基因,它属于II型核受体超家族,是一类能够被天然或人工合成的配体和前列腺素代谢物等激活,在动物体内发挥重要调控作用的核转录因子。PPARs家族包括3种亚型,PPARα、PPARβ和PPARγ。三者在体内的分布差别较大而功能也不尽相同。研究表明,PPARγ的表达足以维持脂肪细胞分化,且是脂肪细胞分化和脂质生成必不可少的转录因子[31-32]。DGAT1和DGAT2是甘油三酯合成所需的限速酶,是外源脂肪酸形成甘油三酯的关键酶,且研究证明过表达DGAT1和DGAT2基因可促进脂质沉积[33-36]。PSDM9的表达能够显著上调DGAT2的表达,而siPSMD9处理显著降低了DGAT1和PPARγ的表达,这些数据表明,PSMD9能够促进脂质沉积。FASN由多个亚基组成,是一种多功能复合酶,也是脂肪酸合成过程中的限速酶,其活性与动物体内脂肪酸的合成和体脂的沉积具有密切关系[37]。ACC在脂肪从头生成中限制速率,并调节脂肪酸氧化,ACC可以抑制肝脏丙二酰辅酶A的产生,减少脂肪酸的合成和增加肝脏脂肪酸的氧化[38-39]。PSMD9的表达显著增加了FASN和ACC的表达,而siPSMD9处理显著降低了FASN和ACC的表达,这些数据表明,PSMD9似乎能促进脂肪酸从头合成。综合以上结果,推测山羊PSMD9基因可能通过促进脂肪合成代谢途径从而促进脂质沉积。但阐明山羊PSMD9基因调控肌内脂肪细胞脂质沉积的分子机制还需要进一步研究。
4 结论本研究克隆得到了山羊PSMD9基因核苷酸序列763 bp,编码187个氨基酸残基;其在肝脏组织中表达量最高,脾脏组织中表达量最低;山羊PSMD9基因随着肌内脂肪细胞的分化而逐渐降低,在第4天表达量最低,之后表达量逐渐升高;PSMD9基因表达能够显著促进山羊前体脂肪细胞脂质沉积。这些结果将为进一步阐明PSMD9在山羊肌内脂肪沉积过程中的作用机制提供理论基础。
| [1] |
KUMAR P, SHARMA N, SHARMA S, et al. In-vitro meat: a promising solution for sustainability of meat sector[J]. J Anim Sci Technol, 2021, 63(4): 693-724. DOI:10.5187/jast.2021.e85 |
| [2] |
BAIK M, KANG H J, PARK S J, et al. TRIENNIAL GROWTH AND DEVELOPMENT SYMPOSIUM: Molecular mechanisms related to bovine intramuscular fat deposition in the longissimus muscle[J]. J Anim Sci, 2017, 95(5): 2284-2303. |
| [3] |
LIU K Q, YU W S, WEI W, et al. Melatonin reduces intramuscular fat deposition by promoting lipolysis and increasing mitochondrial function[J]. J Lipid Res, 2019, 60(4): 767-782. DOI:10.1194/jlr.M087619 |
| [4] |
NISHIMURA T, HATTORI A, TAKAHASHI K. Structural changes in intramuscular connective tissue during the fattening of Japanese black cattle: effect of marbling on beef tenderization[J]. J Anim Sci, 1999, 77(1): 93-104. DOI:10.2527/1999.77193x |
| [5] |
BAIK M, KANG H J, PARK S J, et al. TRIENNIAL GROWTH AND DEVELOPMENT SYMPOSIUM: Molecular mechanisms related to bovine intramuscular fat deposition in the longissimus muscle[J]. J Anim Sci, 2017, 95(5): 2284-2303. |
| [6] |
JEONG J, KWON E G, IM S K, et al. Expression of fat deposition and fat removal genes is associated with intramuscular fat content in longissimus dorsi muscle of Korean cattle steers[J]. J Anim Sci, 2012, 90(6): 2044-2053. DOI:10.2527/jas.2011-4753 |
| [7] |
倪晓光, 赵平. 泛素-蛋白酶体途径的组成和功能[J]. 生理科学进展, 2006, 37(3): 255-258. NI X G, ZHAO P. The components and functions of the ubiquitin-proteasome pathway[J]. Progress in Physiological Sciences, 2006, 37(3): 255-258. (in Chinese) |
| [8] |
XU G J, HUANG S, PENG J, et al. Targeting lipid metabolism in multiple myeloma cells: rational development of a synergistic strategy with proteasome inhibitors[J]. Br J Pharmacol, 2021, 178(23): 4741-4757. DOI:10.1111/bph.15653 |
| [9] |
贾彩凤. REGγ调控脂质代谢的研究[D]. 上海: 华东师范大学, 2014. JIA C F. The essential role of REGγ in regulating lipid metabolism[D]. Shanghai: East China Normal University, 2014. (in Chinese) |
| [10] |
王威. Rpn和Rac及脂质识别蛋白ML对褐飞虱发育、生殖的影响[D]. 杭州: 浙江大学, 2021. WANG W. Effects of Rpn, Rac and lipid recognition protein ML on development and reproduction of brown planthopper (Nilaparvata lugens)[D]. Hangzhou: Zhejiang University, 2021. (in Chinese) |
| [11] |
WATANABE T K, SAITO A, SUZUKI M, et al. cDNA cloning and characterization of a human proteasomal modulator subunit, p27(PSMD9)[J]. Genomics, 1998, 50(2): 241-250. DOI:10.1006/geno.1998.5301 |
| [12] |
HOPPER J L, BEGUM N, SMITH L, et al. The role of PSMD9 in human disease: future clinical and therapeutic implications[J]. AIMS Mol Sci, 2015, 2(4): 476-484. DOI:10.3934/molsci.2015.4.476 |
| [13] |
PARKER B L, CALKIN A C, SELDIN M M, et al. An integrative systems genetic analysis of mammalian lipid metabolism[J]. Nature, 2019, 567(7747): 187-193. DOI:10.1038/s41586-019-0984-y |
| [14] |
GRAGNOLI C. PSMD9 gene in the NIDDM2 locus is linked to type 2 diabetes in Italians[J]. J Cell Physiol, 2010, 222(2): 265-267. DOI:10.1002/jcp.21954 |
| [15] |
GRAGNOLI C. Overweight condition and waist circumference and a candidate gene within the 12q24 locus[J]. Cardiovasc Diabetol, 2013, 12: 2. DOI:10.1186/1475-2840-12-2 |
| [16] |
GRAGNOLI C, CRONSELL J. PSMD9 gene variants within NIDDM2 may rarely contribute to type 2 diabetes[J]. J Cell Physiol, 2007, 212(3): 568-571. DOI:10.1002/jcp.21127 |
| [17] |
WILSON S G, ADAM G, LANGDOWN M, et al. Linkage and potential association of obesity-related phenotypes with two genes on chromosome 12q24 in a female dizygous twin cohort[J]. Eur J Hum Genet, 2006, 14(3): 340-348. DOI:10.1038/sj.ejhg.5201551 |
| [18] |
ABERG K, DAI F, SUN G Y, et al. A genome-wide linkage scan identifies multiple chromosomal regions influencing serum lipid levels in the population on the Samoan islands[J]. J Lipid Res, 2008, 49(10): 2169-2178. DOI:10.1194/jlr.M800194-JLR200 |
| [19] |
THOMAS M K, YAO K M, TENSER M S, et al. Bridge-1, a novel PDZ-domain coactivator of E2A-mediated regulation of insulin gene transcription[J]. Mol Cell Biol, 1999, 19(12): 8492-8504. DOI:10.1128/MCB.19.12.8492 |
| [20] |
GRAGNOLI C. Hypercholesterolemia and a candidate gene within the 12q24 locus[J]. Cardiovasc Diabetol, 2011, 10(1): 38. DOI:10.1186/1475-2840-10-38 |
| [21] |
KEATING M F, VALLIM T Q, PARKER B L, et al. PSMD9 identified as a novel regulator of hepatic acylglycerol metabolism using an integrated systems-biology approach[J]. Obes Res Clin Pract, 2019, 13(3): 272. |
| [22] |
林森, 林亚秋, 朱江江, 等. Wnt10b对山羊前体脂肪细胞分化相关基因表达的影响[J]. 畜牧兽医学报, 2018, 49(4): 685-692. LIN S, LIN Y Q, ZHU J J, et al. Effect of Wnt10b on the expression of precursor adipocytes differentiation related genes in goat[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(4): 685-692. (in Chinese) |
| [23] |
许晴, 林森, 朱江江, 等. 山羊肌内前体脂肪细胞诱导分化过程中内参基因的表达稳定性分析[J]. 畜牧兽医学报, 2018, 49(5): 907-918. XU Q, LIN S, ZHU J J, et al. The expression stability analysis of reference genes in the process of goat intramuscular preadipocytes differentiation in goat[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(5): 907-918. (in Chinese) |
| [24] |
OTTO T C, LANE M D. Adipose development: from stem cell to adipocyte[J]. Crit Rev Biochem Mol Biol, 2005, 40(4): 229-242. DOI:10.1080/10409230591008189 |
| [25] |
杨阳. Zfp217参与转录调控和转录后m6A修饰调控脂肪细胞生成的机制[D]. 武汉: 华中农业大学, 2019. YANG Y. Mechanism of Zfp217 regulating adipogenesis through transcriptional regulation and M6A posttranscriptional modification[D]. Wuhan: Huazhong Agricultural University, 2019. (in Chinese) |
| [26] |
宋兴亚, 彭巍, 刘贤, 等. m6A甲基化修饰及其影响动物脂肪生成的分子机制研究进展[J]. 中国畜牧杂志, 2022, 58(10): 53-58. SONG X Y, PENG W, LIU X, et al. The research progress in methylation modification of m6A and its molecular mechanism affecting the adipogenesis of animals[J]. Chinese Journal of Animal Science, 2022, 58(10): 53-58. (in Chinese) |
| [27] |
ARDITO F, GIULIANI M, PERRONE D, et al. The crucial role of protein phosphorylation in cell signaling and its use as targeted therapy (Review)[J]. Int J Mol Med, 2017, 40(2): 271-280. DOI:10.3892/ijmm.2017.3036 |
| [28] |
UHLEN M, OKSVOLD P, FAGERBERG L, et al. Towards a knowledge-based Human Protein Atlas[J]. Nat Biotechnol, 2010, 28(12): 1248-1250. DOI:10.1038/nbt1210-1248 |
| [29] |
CALNAN D R, BRUNET A. The FoxO code[J]. Oncogene, 2008, 27(16): 2276-2288. DOI:10.1038/onc.2008.21 |
| [30] |
黄馨仪, 郭飞, 曾祥昌, 等. 短链脂肪酸作为信号分子在肠道炎症中的研究进展[J]. 中国临床药理学与治疗学, 2019, 24(11): 1293-1299. HUANG X Y, GUO F, ZENG X C, et al. Short-chain fatty acids as signal molecules in intestinal inflammation[J]. Chinese Journal of Clinical Pharmacology and Therapeutics, 2019, 24(11): 1293-1299. (in Chinese) |
| [31] |
SIERSBÆK R, NIELSEN R, MANDRUP S. Transcriptional networks and chromatin remodeling controlling adipogenesis[J]. Trends Endocrinol Metab, 2012, 23(2): 56-64. DOI:10.1016/j.tem.2011.10.001 |
| [32] |
刘珂含, 王永, 李艳艳, 等. SRSF10对山羊肌内前体脂肪细胞分化的影响[J]. 畜牧兽医学报, 2022, 53(6): 1768-1778. LIU K H, WANG Y, LI Y Y, et al. Effects of SRSF10 on the differentiation of intramuscular preadipocytes in goats[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(6): 1768-1778. (in Chinese) |
| [33] |
YANG C H, LI Q, LIN Y Q, et al. Diacylglycerol acyltransferase 2 promotes the adipogenesis of intramuscular preadipocytes in goat[J]. Anim Biotechnol, 2022, 24: 1-8. |
| [34] |
POL A, GROSS S P, PARTON R G. Biogenesis of the multifunctional lipid droplet: lipids, proteins, and sites[J]. J Cell Biol, 2014, 204(5): 635-646. DOI:10.1083/jcb.201311051 |
| [35] |
杨昌恒, 李琪, 黄维, 等. 山羊DGAT1基因的克隆及对前体脂肪细胞脂质沉积的调控作用研究[J]. 畜牧兽医学报, 2022, 53(1): 76-87. YANG C H, LI Q, HUANG W, et al. Cloning of goat DGAT1 gene and its regulation on lipid deposition in preadipocytes[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(1): 76-87. (in Chinese) |
| [36] |
LI S, WU T, LU Y X, et al. Obesity promotes gastric cancer metastasis via diacylglycerol acyltransferase 2-dependent lipid droplets accumulation and redox homeostasis[J]. Redox Biol, 2020, 36: 101596. DOI:10.1016/j.redox.2020.101596 |
| [37] |
王芳, 马红, 张冬杰, 等. 猪FASN基因功能性单核苷酸多态性的生物信息学分析[J]. 黑龙江畜牧兽医, 2023(11): 62-66, 137. WANG F, MA H, ZHANG D J, et al. Bioinformatics analysis of functional SNPs in pig FASN genes[J]. Heilongjiang Animal Science and Veterinary Medicine, 2023(11): 62-66, 137. (in Chinese) |
| [38] |
XU H F, LUO J, ZHAO W S, et al. Overexpression of SREBP1 (sterol regulatory element binding protein 1) promotes de novo fatty acid synthesis and triacylglycerol accumulation in goat mammary epithelial cells[J]. J Dairy Sci, 2016, 99(1): 783-795. DOI:10.3168/jds.2015-9736 |
| [39] |
DANDAN M, HAN J L, MANN S, et al. Acetyl-CoA carboxylase inhibitor increases LDL-apoB production rate in NASH with cirrhosis: prevention by fenofibrate[J]. J Lipid Res, 2023, 64(3): 100339. |
(编辑 郭云雁)



