我国是世界上地方鸡种资源最丰富的国家之一,《国家畜禽遗传资源品种名录(2021版)》显示,我国现有地方鸡品种115个。地方鸡种大多具有外貌特征多样、适应性强、肉质风味独特、蛋品质优良等特点[1-3],符合我国传统消费习惯,为培育地方特色肉鸡和蛋鸡新品种提供了丰富的育种素材。然而,大量品种的精准鉴定和保种方法仍然有较大的提升空间。利用特征性分子标记对地方品种和商业化品系进行精准标识,对推动畜禽种质资源保护和商业化利用具有重要意义。
单核苷酸多态性(single nucleotide polymorphism, SNP)位点作为第三代遗传标记与其他分子标记相比具有数量多、分布广泛等优越性[4],已有技术可对其进行快速和规模化筛查,进而实现基因分型[5]。随着二代基因组测序成本降低,全基因组重测序成为种质资源研究[6]、群体进化[7]、基因组育种[8]等研究的常规技术方法,可以挖掘到大量目标性状相关的SNP标记[9-10]。然而,针对家禽具有地方品种和专门化品系众多的特点,需要筛选数量较少的SNP集合进行品种/品系区分,建立简便快速的鉴定方法,辅助种质资源保护和鉴定工作。
目前,中低密度SNP标记集合的检测方法主要包括SNP固相芯片、基于靶向SNP标记集合检测的液相芯片、基于质谱原理的SNP标记集合检测方法等[11-14]。中国农业科学院北京畜牧兽医研究所已研发55K SNP芯片“京芯一号”[15],中国农业大学研发50K SNP芯片“凤芯一号”[16],江苏省家禽科学研究所研发23K液相芯片“酉芯一号”,山东省农业科学院家禽研究所研发11K液相芯片“鲁芯一号”等,主要服务于经济性状功能基因和分子标记挖掘[17-19]、基因组育种工作[20-21]和种质资源鉴定[22]。
群体分化指数(fixation index, Fst)是检测群体受到自然或人工选择基因组变异的常用方法,是群体间分化程度的衡量指标,可筛选受到选择压力影响的SNP标记[23-26]。连锁不平衡(linkage disequilibrium, LD)分析常应用于独立SNP提取[27-29],快速型白羽肉鸡父系LD衰变距离390 kb, 京星黄鸡专门化品系LD衰变距离129 kb,不同品系LD差异较大,可通过各品种/品系的LD分析对SNP标记进行缩减[16, 30]。
综上,本研究通过群体分化指数分析和连锁不平衡分析获得少量SNP标记,可以将目标品种与其它代表性品种区分,从而建立目标品种特征性SNP标记集合,为挖掘鸡品种/品系特征性SNP标记集合提供快速有效的方案。
1 材料与方法 1.1 试验数据试验数据选取来源于中国农业科学院北京畜牧兽医研究所的北京油鸡群体(BJY AC (n=59))、BJY E (n=40)、BJY F (n=40))、京星黄鸡选育系D2系(JXH.D2 (n=49))、京星黄鸡选育系H系(JXH.H (n=59))、茶花鸡(CH (n=30))、大围山微型鸡(DWS (n=24))、武定鸡(WD (n=21)、藏鸡(ZJ (n=10))、大骨鸡(DG (n=7))和瓢鸡(P(n=21))重测序数据; 来源于佛山高明区新广农牧有限公司的快速型白羽肉鸡(B (n=60))重测序数据; 来源于山东农业科学院家禽研究所的7个山东地方鸡品种A、B、BRG、BRM、D、L和S (SD A (n=10)、SD B (n=10)、SD BRG (n=10)、SD BRM (n=10)、SD D (n=20)、SD L (n=80)、SD S (n=20))重测序数据。试验数据共涉及19个品种/品系共580个个体。
1.2 数据质控基于10x以上的全基因组重测数据,利用PLINK(V 1.90)[31]软件对SNP进行标准的质量控制,删除缺失率>0.1的个体、删除缺失率>0.1的SNP以及删除次等位基因频率<0.05的个体(--mind 0.1 --geno 0.1 --maf 0.05),并将19个品种品系测序数据合并为一个37.14 G数据量的vcf文件。保留1~28号染色体上的位点,共保留16 927 197个SNPs标记位点用于后续分析。
1.3 主成分分析和系统进化树使用GCTA 64(V 1.93.2)[32]软件构建亲缘关系矩阵(--make-grm)后计算PCA,计算每个主成分解释百分比,选择前2个主成分,用RStudio(V1.1.463)绘制主成分分析(principal component analysis,PCA)平面图。
随机提取19个品种/品系各10个个体,大骨鸡7个个体,共187个个体,利用PLINK(V1.90)软件对SNP频率构建遗传距离矩阵(--distance-matrix)。通过MEGA(V 7.0.26)[33]软件,采用领接法(Neighbour Joining, NJ)绘制进化树。
1.4 群体分化指数分析以1个品种/品系作为目标品种/品系,利用VCFTools(V0.1.13)[34]软件计算采用1对N的方式进行Fst分析,以1 kb为窗口大小、1 kb为步长计算SNP单点Fst值(--fst-window-size 1 --fst-window-step 1)。
1.5 连锁不平衡分析利用PLINK(V1.90)软件提取群体分化指数分析结果中MEAN_FAST≥0.65 SNP位点形成SNP标记集合,对SNP标记位点进行LD分析(--blocks no-pheno-req)。提取全部非LD SNP标记位点以及每个LD中1个SNP标记位点,作为独立SNP标记位点。
2 结果 2.1 遗传结构分析对19个品种/品系质控后16 927 197个SNPs进行PCA(图 1a)和NJ进化树(图 1b)分析,结果表明北京油鸡、快速型白羽肉鸡品系、京星黄鸡H系和京星黄鸡D2系与其它品种遗传距离较远,分层明显。茶花鸡和大围山微型鸡聚成一支,武定鸡和瓢鸡聚成一支,山东地方品种/品系聚在一起。

|
a.多品种主成分分析;b. NJ进化树结果。B.快速型白羽肉鸡;BJY AC、BJY E、BJY F.北京油鸡;CH.茶花鸡;DG.大骨鸡;DWS.大围山微型鸡;JXH.D2.京星黄鸡选育系D2系;JXH.H.京星黄鸡选育系H系;P.瓢鸡;SD A、SD B、SD BRG、SD BRM、SD D、SD L、SD S.山东地方鸡品种品系A、B、BRG、BRM、D、L、S;WD.武定鸡;ZJ.藏鸡 a. Results of multi-variety principal component analysis; b. Results of neighbour joining tree. B. The fast growing white feather broiler; BJY AC, BJY E, BJY F. Beijing yellow chicken; CH. Chahua chicken; DG. Dagu chicken; DWS. Daweishan mini chicken; JXH.D2. Jingxing yellow chicken selective line D2; JXH.H. Jingxing yellow chicken selective line H; P. Piao chicken; SD A, SD B, SD BRG, SD BRM, SD D, SD L, SD S. Shandong local chicken breed A, B, BRG, BRM, D, L, S; WD. Wuding chicken; ZJ. Tibetan chicken 图 1 多品种主成分分析和NJ进化树结果 Fig. 1 Results of multi-variety principal component analysis and neighbour joining tree |
根据遗传结构分析结果挑选独立于其它群体的北京油鸡、快速型白羽肉鸡、京星黄鸡H系和京星黄鸡D2系,分别通过单位点Fst分析和LD分析筛选特征性SNP标记集合。以快速型白羽肉鸡为目标品种进行单位点Fst分析(图 2a),结果表明,与其他品种/品系显著差异的SNP标记主要位于1、5、18和28号染色体上。提取MEAN_FAST≥0.80共346个SNPs标记进行PCA分析,可将快速型白羽肉鸡与其它群体分开(图 2b)。对346个SNPs标记进行LD分析,提取所有不连锁的SNPs和每个LD中1个SNP标记,共114个SNPs标记进行群体PCA分析,结果表明114个SNPs标记可将快速型白羽肉鸡与其它品种/品系分开(图 2c)。
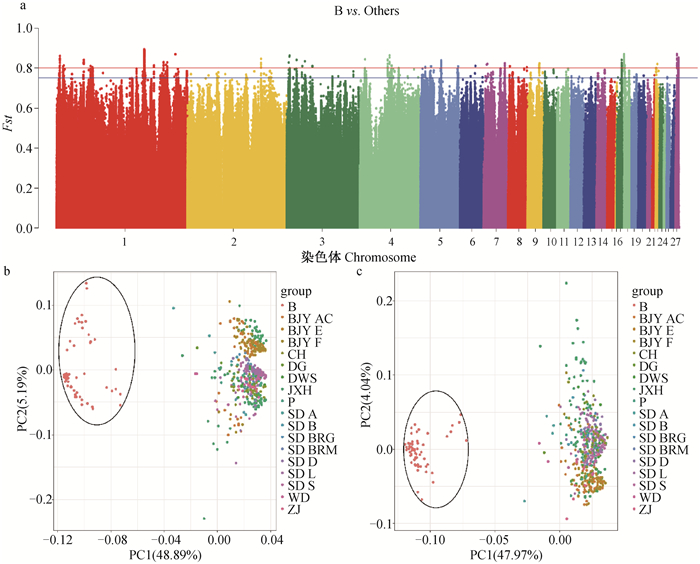
|
a.快速型白羽肉鸡vs.其它品种/品系群体分化指数分析结果;b.选择性清除分析筛选MEAN_FAST≥0.80 SNP标记主成分分析结果;c.连锁不平衡分析筛选的SNP标记主成分分析结果 a. The result of Fst between the fast growing white feather broiler with other breed and strains; b. The result of PCA analysis that selected the MEAN_FAST≥0.80 SNP markers; c. The result of PCA analysis that selected the SNP markers by LD analysis 图 2 快速型白羽肉鸡vs.其它品种品系Fst和PCA结果 Fig. 2 The result of Fst and PCA between the fast growing white feather broiler with other breeds and strains |
以京星黄鸡选育系H系为目标品系进行单位点Fst分析(图 3a),结果表明,与其他品种显著差异的SNP标记主要位于1、2、3和4号染色体上。提取MEAN_FAST≥0.76共356个SNPs标记进行PCA分析,可将京星黄鸡选育系H系与其它群体分开(图 3b)。对356个SNPs标记进行LD分析,提取所有不连锁的SNPs和每个LD中1个SNP标记,共220个SNPs标记进行群体PCA分析,结果表明220个SNPs标记可将京星黄鸡选育系H系与其它品种分开(图 3c)。
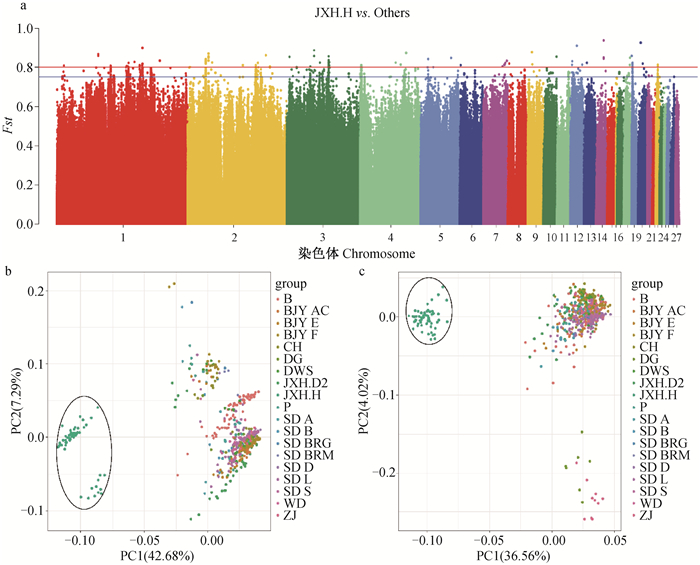
|
a.京星黄鸡选育系H系vs.其它品种/品系群体分化指数分析结果;b.选择性清除分析筛选MEAN_FAST≥0.76 SNP标记主成分分析结果;c.连锁不平衡分析筛选的SNP标记主成分分析结果 a. The result of Fst between Jingxing yellow chicken selective line H with other breeds and strains; b. The result of PCA analysis that selected the MEAN_FAST≥0.76 SNP markers; c. The result of PCA analysis that selected the SNP markers by LD analysis 图 3 京星黄鸡选育系H系vs.其它品种品系Fst和PCA结果 Fig. 3 The result of Fst and PCA between Jingxing yellow chicken selective line H with other breeds and strains |
以京星黄鸡选育系D2系为目标品系进行单位点Fst分析(图 4a),结果表明,与其他品种/品系显著差异的SNP标记主要位于1、2、4、7、14和23号染色体上。提取MEAN_FAST≥0.76共321个SNPs标记进行PCA分析,可将京星黄鸡选育系D2与其它群体分开(图 4b)。对321个SNPs标记进行LD分析,提取所有不连锁的SNPs和每个LD中1个SNP标记,共226个SNPs标记进行群体PCA分析,结果表明226个SNPs标记可将京星黄鸡选育系D2系与其它品种/品系分开(图 4c)。
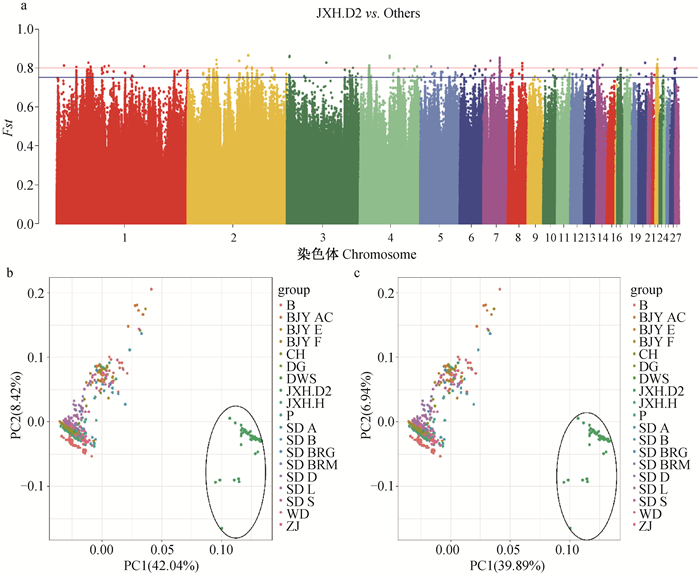
|
a.京星黄鸡选育系D2系vs.其它品种/品系群体分化指数分析结果;b.选择性清除分析筛选MEAN_FAST≥0.76 SNP标记主成分分析结果;c.连锁不平衡分析筛选的SNP标记主成分分析结果 a. The result of Fst between Jingxing yellow chicken selective line D2 with other breeds and strains; b. The result of PCA analysis that selected the MEAN_FAST≥0.76 SNP markers; c. The result of PCA analysis that selected the SNP markers by LD analysis 图 4 京星黄鸡选育系D2 vs.其它品种品系Fst和PCA结果 Fig. 4 The result of Fst and PCA between Jingxing yellow chicken selective line D2 with other breeds and strains |
根据遗传结构分析结果挑选聚集在一个分支的武定鸡和瓢鸡,分别通过单位点Fst分析和LD分析筛选特征性SNP标记集合。以武定鸡为目标品种进行单位点Fst分析(图 5a),结果表明,与其他品种显著差异的SNP标记主要位于1、2、4、5和15号染色体上。提取MEAN_FAST≥0.70共368个SNPs标记进行PCA分析,可将武定鸡与其它群体分开(图 5b)。对368个SNPs标记进行LD分析,提取所有不连锁的SNPs和每个LD中1个SNP标记,共204个SNPs标记进行群体PCA分析,结果表明204个SNPs标记可将武定鸡与其它品种分开(图 5c)。

|
a.武定鸡vs.其它品种/品系群体分化指数分析结果;b.选择性清除分析筛选MEAN_FAST≥0.70 SNP标记主成分分析结果;c.连锁不平衡分析筛选的SNP标记主成分分析结果 a. The result of Fst between Wuding chicken with other breeds and strains; b. The result of PCA analysis that selected the MEAN_FAST≥0.70 SNP markers; c. The result of PCA analysis that selected the SNP markers by LD analysis 图 5 武定鸡vs.其它品种品系Fst和PCA结果 Fig. 5 The result of Fst and PCA between Wuding chicken with other breeds and strains |
以瓢鸡为目标品种进行单位点Fst分析(图 6a),结果表明,与其他品种显著差异的SNP标记主要位于1、2和4号染色体上。提取MEAN_FAST≥0.65共1 178个SNPs标记进行PCA分析,可将瓢鸡与其它群体分开(图 6b)。对1 178个SNPs标记进行LD分析,提取所有不连锁的SNPs和每个LD中1个SNP标记,共178个SNPs标记进行群体PCA分析,结果表明178个SNPs标记可将瓢鸡与其它品种分开(图 6c)。

|
a.瓢鸡vs.其它品种/品系群体分化指数分析结果;b.选择性清除分析筛选MEAN_FAST≥0.65 SNP标记主成分分析结果;c.连锁不平衡分析筛选的SNP标记主成分分析结果 a. The result of Fst between Piao chicken with other breeds and strains; b. The result of PCA analysis that selected the MEAN_FAST≥0.65 SNP markers; c. The result of PCA analysis that selected the SNP markers by LD analysis 图 6 瓢鸡vs.其它品种品系Fst和PCA结果 Fig. 6 The result of Fst and PCA between Piao chicken with other breeds and strains |
目前SNP标记集可以基于单倍型分析[35]、连锁不平衡分析[36]、全基因组关联分析[37]等方法获得。Judge等[38]基于有系谱和重测序数据的大群体以及中等密度SNP芯片的大群体, 利用Delta统计、Fst统计、结合Delta统计和成对Fst值的索引进行计算等方法识别信息量最大的SNPs,通过300个以上SNPs标记精准量化生物样本中安格斯牛和赫里福德牛的比例。Seo等[39]基于鸡600K SNP芯片,GWAS分析后,对病例组和对照组进行LD修剪,得到96个SNPs标记可以将目标鸡与其它鸡群分开。这些基于系谱与GWAS分析的研究可进行少量群体特征性SNP集合的挖掘,但是均需要较大群体的表型信息与测序,而本研究方法仅需要试验群体代表性个体的重测序数据,一般30个左右个体可代表一个品种的遗传多样性,数量较少的个体可作为背景,在试验材料和数据准备上更为简便。
Bertolini等[40]基于奶牛大群体Bovine SNP50 v1 BeadChip芯片,利用基于Delta、Fst、PCA-chrom和PCA-whole等技术,通过品种分配和随机森林筛选出96个SNPs组成的SNP-set可以将品种区分开。Schiavo等[41]基于猪大群体PorcineSNP60 BeadChip芯片,保留LD分析中所有r2<0.3的SNP和1个LD中任一SNP,然后使用Delta、Fixation指数、主成分分析统计和两种随机森林分类方法筛选到96个SNPs标记位点可进行目标品种鉴定。Cho等[42]基于鸡大群体600K SNP芯片,通过GWAS和LD分析筛选,得到初步的SNP标记集合,然后通过随机森林(RF)和AdaBoost(AB)两种机器学习算法,筛选到Yeonsan Ogye鸡群的38(RF)和43(AB)个共81个最佳SNPs标记集合,在品种区分上显示了100%的准确性。Kumar等[43]基于小等位基因频率连锁不平衡的方法,鉴定到591个品种特异性SNPs组成的集合,适用于鉴别牛的亲缘关系的分配。Gao等[44]基于24个猪品种的62 822个SNPs基因型文件,通过LD、PCA、随机森林及相应的包外误差估计(OOB)和MDA筛选方法获得1 000个SNPs可将目标品种区分。利用随机森林等一系列方法可筛选出较少的品种特征性SNP标记,是未来发展的重点,但需要较强的方法学作为基础。而本方法仅通过一次单点群体分化指数分析和连锁不平衡分析,即可挖掘到114~226个SNPs标记将目标品种与其它代表性品种区分开,更为快捷。
4 结论本研究应用19个鸡品种/品系全基因重测序数据进行1对N的单位点群体分化指数分析,以MEAN_FAST≥0.65为筛选标准,对筛选得到的SNP标记进行连锁不平衡分析,在多个品种中确定了114~226个不同染色体上SNPs标记可以将目标品种与其它代表性品种区分开来,从而建立目标品种特征性SNP标记集合。该SNP标记集合筛选方法是实现低成本和快速品种鉴定的基础。
| [1] |
张小波. 地方鸡品种特性及规模化高效养殖技术[J]. 中国畜牧业, 2022(20): 62-63. ZHANG X B. Local chicken breed characteristics and scale up efficient breeding technology[J]. China Animal Industry, 2022(20): 62-63. (in Chinese) |
| [2] |
陈力力, 张爱琼, 王可人. 乌蒙凤鸡的品种特性与饲养管理概述[J]. 吉林畜牧兽医, 2022, 43(7): 61-62. CHEN L L, ZHANG A Q, WANG K R. Overview of breed characteristics and feeding management of Wumeng crested chicken[J]. Jilin Animal Husbandry and Veterinary Medicine, 2022, 43(7): 61-62. (in Chinese) |
| [3] |
霍献强, 杨祝良, 邹乐勤, 等. 广西6个地方鸡肉品质测定及差异性分析[J]. 家畜生态学报, 2022, 43(5): 47-51. HUO X Q, YANG Z L, ZOU L Q, et al. Meat quality determination and difference analysis of six local chicken breeds in Guangxi[J]. Acta Ecologae Animalis Domastici, 2022, 43(5): 47-51. (in Chinese) |
| [4] |
唐一通, 肖娜, 李智山, 等. 单核苷酸多态性检测方法研究进展[J]. 现代生物医学进展, 2013, 13(27): 5393-5397. TANG Y T, XIAO N, LI Z S, et al. Progress in detection methods of single nucleotide polymorphisms[J]. Progress in Modern Biomedicine, 2013, 13(27): 5393-5397. DOI:10.13241/j.cnki.pmb.2013.27.022 (in Chinese) |
| [5] |
平措占堆. 奶牛精液品质性状的分子标记技术研究进展[J]. 中国奶牛, 2018(1): 15-19. PINGCUO Z D. The research progress of molecular markers for dairy semen quality[J]. China Dairy Cattle, 2018(1): 15-19. DOI:10.19305/j.cnki.11-3009/s.2018.01.005 (in Chinese) |
| [6] |
ZHANG W, LI X J, JIANG Y, et al. Genetic architecture and selection of Anhui autochthonous pig population revealed by whole genome resequencing[J]. Front Genet, 2022, 13: 1022261. DOI:10.3389/fgene.2022.1022261 |
| [7] |
DESTA Z A, ORTIZ R. Genomic selection: genome-wide prediction in plant improvement[J]. Trends Plant Sci, 2014, 19(9): 592-601. DOI:10.1016/j.tplants.2014.05.006 |
| [8] |
宋志芳, 芦春莲, 曹洪战. 全基因组重测序及其在动物育种的研究进展[J]. 畜牧与兽医, 2017, 49(11): 145-148. SONG Z F, LU C L, CAO H Z. Research progress on Whole genome resequencing in animal breeding[J]. Animal Husbandry & Veterinary Medicine, 2017, 49(11): 145-148. (in Chinese) |
| [9] |
YANG Z L, ZOU L Q, SUN T T, et al. Genome-wide association study using whole-genome sequencing identifies a genomic region on chromosome 6 associated with comb traits in Nandan-Yao chicken[J]. Front Genet, 2021, 12: 682501. DOI:10.3389/fgene.2021.682501 |
| [10] |
ZHANG Z R, QIU M H, DU H R, et al. Whole genome re-sequencing identifies unique adaption of single nucleotide polymorphism, insertion/deletion and structure variation related to hypoxia in Tibetan chickens[J]. Gene Expr Patterns, 2021, 40: 119181. DOI:10.1016/j.gep.2021.119181 |
| [11] |
KWOK P Y, CHEN X N. Detection of single nucleotide polymorphisms[J]. Curr Issues Mol Biol, 2003, 5(2): 43-60. |
| [12] |
汪琳, 赖平安, 张鹤晓, 等. 液相芯片——高通量检测技术的新亮点[J]. 检验检疫科学, 2005, 15(6): 55-56. WANG L, LAI P A, ZHANG H X, et al. Liquichip-a new highlight of high throughput detection technology[J]. Quality Safety Inspection and Testing, 2005, 15(6): 55-56. DOI:10.3969/j.issn.1674-5354.2005.06.020 (in Chinese) |
| [13] |
岳龙涛, 高雪芹, 樊景凤. 固相生物芯片表面处理及化学修饰的研究进展[J]. 中国生物制品学杂志, 2008, 21(8): 727-731. YUE L T, GAO X Q, FAN J F. Advances in surface treatment and chemical modification of solid-phase biochips[J]. Chinese Journal of Biologicals, 2008, 21(8): 727-731. DOI:10.3969/j.issn.1004-5503.2008.08.025 (in Chinese) |
| [14] |
朱兰, 王鹏, 欧阳依娜, 等. 基于SNP分子标记的云上黑山羊3个核心群遗传多样性分析[J]. 中国畜牧杂志, 2022, 58(10): 136-143. ZHU L, WANG P, OYANG Y N, et al. Genetic diversity analysis of 3 Core Yunshang black goat populations based on SNP markers[J]. Chinese Journal of Animal Science, 2022, 58(10): 136-143. (in Chinese) |
| [15] |
LIU R R, XING S Y, WANG J, et al. A new chicken 55K SNP genotyping array[J]. BMC Genomics, 2019, 20(1): 410. DOI:10.1186/s12864-019-5736-8 |
| [16] |
LIU Z, SUN C J, YAN Y Y, et al. Design and evaluation of a custom 50K Infinium SNP array for egg-type chickens[J]. Poult Sci, 2021, 100(5): 101044. DOI:10.1016/j.psj.2021.101044 |
| [17] |
LI W, ZHENG M Q, ZHAO G P, et al. Identification of QTL regions and candidate genes for growth and feed efficiency in broilers[J]. Genet Sel Evol, 2021, 53(1): 13. DOI:10.1186/s12711-021-00608-3 |
| [18] |
杨欣婷, 郑麦青, 谭晓冬, 等. 快大型黄羽肉鸡肉品质性状的遗传参数估计和关键基因挖掘[J]. 畜牧兽医学报, 2021, 52(9): 2416-2428. YANG X T, ZHENG M Q, TAN X D, et al. Genetic parameters estimation and key genes identification for meat quality traits of fast-growing yellow-feather meat-type chickens[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(9): 2416-2428. (in Chinese) |
| [19] |
LIU L, CUI H X, XING S Y, et al. Effect of divergent selection for intramuscular fat content on muscle lipid metabolism in chickens[J]. Animals (Basel), 2020, 10(1): 4. |
| [20] |
HE Z X, LI S, LI W, et al. Comparison of genomic prediction methods for residual feed intake in broilers[J]. Anim Genet, 2022, 53(3): 466-469. |
| [21] |
HUANG S W, HE Y T, YE S P, et al. Genome-wide association study on chicken carcass traits using sequence data imputed from SNP array[J]. J Appl Genet, 2018, 59(3): 335-344. |
| [22] |
KAWAGUCHI F, NAKAMURA M, KOBAYASHI E, et al. Comprehensive assessment of genetic diversity, structure, and relationship in four Japanese cattle breeds by Illumina 50 K SNP array analysis[J]. Anim Sci J, 2022, 93(1): e13770. |
| [23] |
曾滔, 赵福平, 王光凯, 等. 基于群体分化指数Fst的绵羊全基因组选择信号检测[J]. 畜牧兽医学报, 2013, 44(12): 1891-1899. ZENG T, ZHAO F P, WANG G K, et al. Genome-wide detection of selection signatures in sheep populations with use of population differentiation index Fst[J]. Acta Veterinaria et Zootechnica Sinica, 2013, 44(12): 1891-1899. (in Chinese) |
| [24] |
WANG Q, LI D, GUO A, et al. Whole-genome resequencing of Dulong Chicken reveal signatures of selection[J]. Br Poult Sci, 2020, 61(6): 624-631. |
| [25] |
YANG W J, LIU Z, ZHAO Q Q, et al. Population genetic structure and selection signature analysis of beijing black pig[J]. Front Genet, 2022, 13: 860669. |
| [26] |
XIA X T, ZHANG S J, ZHANG H J, et al. Assessing genomic diversity and signatures of selection in Jiaxian Red cattle using whole-genome sequencing data[J]. BMC Genomics, 2021, 22(1): 43. |
| [27] |
尼桂琰, 张哲, 姜力, 等. 利用全基因组连锁不平衡估计中国荷斯坦牛有效群体大小[J]. 遗传, 2012, 34(1): 50-58. NI G Y, ZHANG Z, JIANG L, et al. Chinese Holstein Cattle effective population size estimated from whole genome linkage disequilibrium[J]. Hereditas (Beijing), 2012, 34(1): 50-58. (in Chinese) |
| [28] |
张鹏, 李金泉. 利用连锁不平衡法发掘作物种质资源中优异基因的研究进展[J]. 中国农学通报, 2009, 25(8): 34-37. ZHANG P, LI J Q. Reviews of the advancement on mining exotic genes in crop germplasm resources by linkage disequilibrium method[J]. Chinese Agricultural Science Bulletin, 2009, 25(8): 34-37. (in Chinese) |
| [29] |
谢水华, 李加琪, 张豪, 等. 利用连锁不平衡进行畜禽QTL定位的研究进展[J]. 中国畜牧兽医, 2007, 34(6): 36-39. XIE S H, LI J Q, ZHANG H, et al. Research progress of livestock QTL localization by linkage disequilibrium[J]. China Animal Husbandry & Veterinary Medicine, 2007, 34(6): 36-39. (in Chinese) |
| [30] |
TAN X D, LIU R R, LI W, et al. Assessment the effect of genomic selection and detection of selective signature in broilers[J]. Poult Sci, 2022, 101(6): 101856. |
| [31] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. |
| [32] |
YANG J, LEE S H, GODDARD M E, et al. Genome-wide complex trait analysis (GCTA): methods, data analyses, and interpretations[M]//GONDRO C, VAN DER WERF J, HAYES B. Genome-Wide Association Studies and Genomic Prediction. Totowa: Humana Press, 2013: 215-236.
|
| [33] |
KUMAR S, STECHER G, LI M, et al. MEGA X: molecular evolutionary genetics analysis across computing platforms[J]. Mol Biol Evol, 2018, 35(6): 1547-1549. |
| [34] |
DANECEK P, AUTON A, ABECASIS G, et al. The variant call format and VCFtools[J]. Bioinformatics, 2011, 27(15): 2156-2158. |
| [35] |
ZHANG K, DENG M H, CHEN T, et al. A dynamic programming algorithm for haplotype block partitioning[J]. Proc Natl Acad Sci U S A, 2002, 99(11): 7335-7339. |
| [36] |
BADKE Y M, BATES R O, ERNST C W, et al. Methods of tagSNP selection and other variables affecting imputation accuracy in swine[J]. BMC Genet, 2013, 14: 8. |
| [37] |
LIU Y X, CHEN H J, HEINE J, et al. A genome-wide association study of mammographic texture variation[J]. Breast Cancer Res, 2022, 24(1): 76. |
| [38] |
JUDGE M M, KELLEHER M M, KEARNEY J F, et al. Ultra-low-density genotype panels for breed assignment of Angus and Hereford cattle[J]. Animal, 2017, 11(6): 938-947. |
| [39] |
SEO D, CHO S, MANJULA P, et al. Identification of target chicken populations by machine learning models using the minimum number of SNPs[J]. Animals (Basel), 2021, 11(1): 241. |
| [40] |
BERTOLINI F, GALIMBERTI G, SCHIAVO G, et al. Preselection statistics and Random Forest classification identify population informative single nucleotide polymorphisms in cosmopolitan and autochthonous cattle breeds[J]. Animal, 2018, 12(1): 12-19. |
| [41] |
SCHIAVO G, BERTOLINI F, GALIMBERTI G, et al. A machine learning approach for the identification of population-informative markers from high-throughput genotyping data: application to several pig breeds[J]. Animal, 2020, 14(2): 223-232. |
| [42] |
CHO E, CHO S, KIM M, et al. Single nucleotide polymorphism marker combinations for classifying Yeonsan Ogye chicken using a machine learning approach[J]. J Anim Sci Technol, 2022, 64(5): 830-841. |
| [43] |
KUMAR H, PANIGRAHI M, CHHOTARAY S, et al. Comparative analysis of five different methods to design a breed-specific SNP panel for cattle[J]. Anim Biotechnol, 2021, 32(1): 130-136. |
| [44] |
GAO J, SUN L W, ZHANG S S, et al. Screening discriminating SNPs for Chinese indigenous pig breeds identification using a random forests algorithm[J]. Genes (Basel), 2022, 13(12): 2207. |
(编辑 郭云雁)



