2. 天津市宁河原种猪场有限责任公司, 天津 301504;
3. 中国农业科学院农业基因组研究所, 深圳 518120
2. Tianjin Ninghe Breeding Pig Farm Co., Ltd., Tianjin 301504, China;
3. Agricultural Genomics Institute at Shenzhen, Chinese Academy of Agricultural Sciences, Shenzhen 518120, China
富含半胱氨酸的清道夫受体超家族(scavenger receptor cysteine-rich superfamily)是一类结构相关的蛋白质超家族,包括多种跨膜蛋白,并且都具有富含半胱氨酸的清道夫受体(scavenger receptor cysteine-rich, SRCR)结构域[1]。清道夫受体超家族根据序列和结构相似性,可分为10多个类型[2],CD163蛋白属于SRCR家族B型成员[3],包含9个SRCR结构域,每个结构域含有8个半胱氨酸残基,仅在单核细胞和巨噬细胞中表达。它参与机体免疫生理过程,已被证实是猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus, PRRSV)的必需受体,在动物抗病育种中有重要的应用价值。
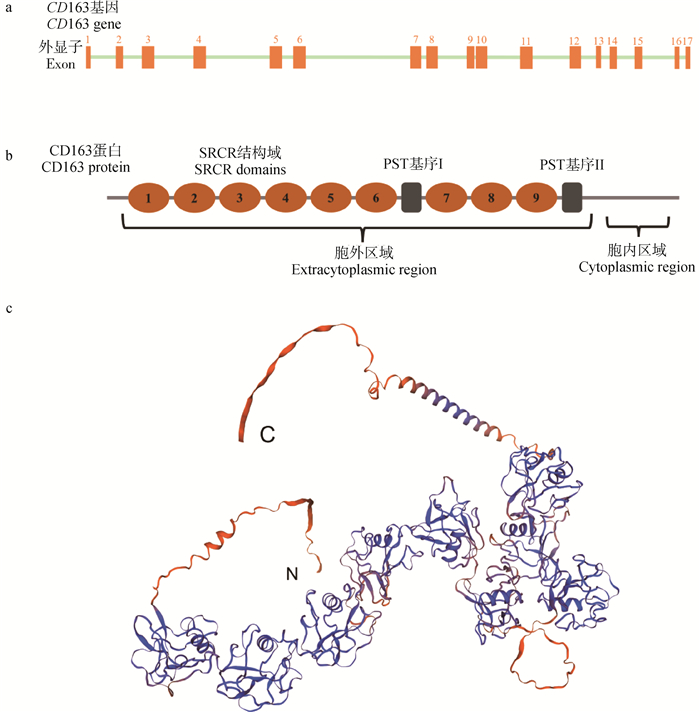
1.2 CD163基因结构CD163蛋白的全称为分化抗原163(cluster of differentiation 163, CD163)。因其分子量大小为130 ku因此也称为M130或MM130蛋白。猪的CD163基因位于5号染色体上,目前在GenBank数据库中共有5个转录本,分别为NM_213976.1;XM_021091121.1;XM_021091120.1;XM_021091123.1;XM_021091122.1。其中,转录本NM_213976.1的编码区(coding domain sequence, CDS)全长3 333 bp,含有17个外显子和16个内含子(图 1a),编码1 110个氨基酸(图 1b)。起始密码子位于外显子1上,信号肽由外显子1和外显子2编码。9个SRCR结构域分别由外显子3、外显子4、外显子5、外显子6、外显子7、外显子8、外显子10、外显子11和外显子12编码。外显子9和外显子13编码两个富含脯氨酸-丝氨酸-苏氨酸(proline-serine-threonine, PST)的连接基序,外显子14、外显子15和外显子16编码胞内区部分,终止密码子位于外显子16上(图 1)。
|
a.猪CD163基因结构示意图;b.猪CD163蛋白二级结构示意图;c.通过序列同源性预测的猪CD163蛋白三维结构示意图,N代表蛋白N端,C代表蛋白C端 a. Diagram of porcine CD163 gene structure; b. Diagram of porcine CD163 protein domains; c. The predicted three-dimensional structure diagram of porcine CD163 based on sequence homology, N and C represent the N-terminal and C-terminal ends of CD163 protein, respectively 图 1 猪CD163基因和蛋白结构 Fig. 1 Gene and protein structure of porcine CD163 |
CD163是一种Ⅰ型膜蛋白,由胞外区、跨膜区和胞内区3部分组成(图 1b)。具体包括:N端信号肽、连续的6个SRCR结构域、PST连接基序、连续的3个SRCR结构域、第二个PST连接基序、跨膜区、细胞质尾区等部分[4]。CD163蛋白的每个SRCR结构域都由100~110个氨基酸残基组成,是由以α-螺旋为核心的5~6个β折叠构成的(图 1c)。
CD163蛋白存在可变剪切,会生成含有不同长度的细胞质尾区的CD163蛋白异构体[5-6]。短尾形式的CD163蛋白细胞质尾区有49个氨基酸,而长尾形式的细胞质尾区分别有84和89个氨基酸[4]。除细胞质尾区异构体外,在人类细胞中还发现了SRCR5和SRCR6结构域中间插入33个氨基酸的CD163蛋白异构体[4]。位于细胞膜上的CD163蛋白可通过蛋白水解形成包含有9个SRCR的可溶性CD163蛋白(soluble CD163, sCD163),sCD163蛋白的生理功能尚不完全明确,但其在体液中的浓度与多种疾病显著相关[7-8]。
2 CD163蛋白的功能CD163蛋白几乎仅在单核细胞以及由单核细胞分化的细胞中表达,例如巨噬细胞,其表达量随单核细胞的活化或成熟而增加,而且与机体的免疫应答紧密相关[9-10]。目前的研究显示,CD163蛋白具有重要的生物学功能,一方面CD163蛋白可以作为受体介导病毒、细菌等多种病原体的感染;另一方面,CD163蛋白还可以影响红细胞的成熟分化,清除机体游离的血红蛋白(hemoglobin, Hb),影响铁离子的循环[11];还与炎症和免疫调节密切相关,可以刺激免疫反应,也可以抑制免疫反应,并参与动脉粥样硬化[8, 12]、糖尿病[13]、癌症等[14-15]疾病的发生发展;可溶性CD163分子还可以作为某些疾病的生物标记物[7],具有重要的生理功能。
2.1 CD163蛋白是介导PRRSV感染的必需受体2.1.1 猪繁殖与呼吸综合征简介 猪繁殖与呼吸综合征(porcine reproductive and respiratory syndrome, PRRS),俗称蓝耳病,是由PRRSV引发的高致病性和高致死性的猪烈性传染性疫病,给全球养猪业带来重大经济损失。受PRRSV感染的猪可能出现食欲不振、发烧、嗜睡和呼吸困难等症状[16],但主要导致母猪的流产和死胎,仔猪的呼吸道疾病和哺乳仔猪的大量死亡。在自然宿主中,PRRSV仅能感染单核细胞/巨噬细胞谱系的细胞,如猪肺泡巨噬细胞(pulmonary alveolar macrophages, PAMs)[17]。
2.1.2 CD163在PRRSV感染中的作用 PRRSV对宿主的选择性取决于宿主细胞的表面受体。目前已经有多个受体被证明参与PRRSV感染(表 1)。这些受体包括CD163[16, 18-21]、CD169[22-24]、硫酸乙酰肝素(heparan sulfate, HS)[25]、波形蛋白(vimentin)[26]、非肌肉肌球蛋白重链9(myosin heavy chain 9, MYH9)[27-28]、CD209[29]和Siglec-10[30]等。目前已通过基因编辑动物以及活体攻毒试验证明CD163蛋白是PRRSV感染的必需受体[16, 18-21, 31-35]。
|
|
表 1 PRRSV细胞受体及其作用 Table 1 Summary of PRRSV receptors |
早期在多个物种的体外细胞试验中发现CD163蛋白是PRRSV感染的重要受体。多种不感染PRRSV的细胞系,如BHK-21(仓鼠肾细胞系)[39]、PK-15(猪肾细胞系)[40]等,过表达CD163后即可感染PRRSV。Li等[41]在小鼠肺泡巨噬细胞衍生细胞和鼠腹膜巨噬细胞样细胞中过表达猪CD163蛋白后,可支持PRRSV感染和复制,而且被PRRSV感染后,小鼠肺泡巨噬细胞衍生细胞的抗炎细胞因子和促炎细胞因子表达模式与PAM细胞相似。Wang等[42]的研究发现CD163表达水平对PRRSV传染性至关重要,增加CD163的丰度会增强细胞对PRRSV的易感性,高效的PRRSV感染需要高水平的组成型CD163表达。Xu等[43]扩增了来自猪PAM细胞的CD163基因的编码区域,并使用慢病毒表达系统将其整合到永生化单克隆PAM细胞系(monoclonal PAM, mPAM)中,发现稳定表达CD163蛋白的mPAM细胞系能够被高致病性PRRSV毒株JXA1或经典PRRSV毒株SD1感染,并产生高滴度的PRRSV。这些研究初步建立了CD163蛋白与PRRSV感染的正相关性,使得CD163成为了最受关注的PRRSV受体之一。
另一方面,大量研究也发现干扰或者敲除CD163,或者阻断CD163蛋白与PRRSV的结合则可以使原本能够感染PRRSV的细胞对PRRSV产生抗性。如Li等[44]研究表明ssc-miR-124a通过抑制CD163的表达来实现其抗病毒作用,过表达ssc-miR-124a可显著抑制PRRSV在PAM中的复制。Yu等[45]使用CRISPR/Cas9基因编辑工具,在MARC-145细胞中精准删除CD163蛋白的SRCR5结构域后,可使MARC-145细胞完全抵抗PRRSV的感染。Xia等[46]和Chen等[47]分别使用腺病毒载体过表达可溶性的CD169和CD163蛋白,发现可以在细胞和个体水平抵抗PRRSV感染。此外,Xu等[48]在体外细胞试验中发现,使用CD163单克隆抗体可以有效阻断PRRSV对PAM和MARC-145细胞的感染。这些结果进一步证实了CD163是PRRSV的核心受体的假说[49],但是仍然不清楚CD163蛋白是否是PRRSV感染的必需受体。
Whitworth等[21]利用CRISPR/Cas9基因编辑技术制备了CD163基因敲除猪,并进行了PRRSV攻毒试验,发现缺失CD163蛋白的猪可以完全抵抗PRRSV感染。该研究首次在活体水平证明CD163蛋白是PRRSV的必需受体,为猪的抗PRRS育种奠定了基础。在该研究中,对CD163基因敲除猪接种Ⅱ型PRRSV NVSL 97-7895毒株后,敲除猪完全没有出现呼吸困难、发烧等典型的PRRS临床症状;肺部组织结构完好,完全没有水肿和炎症反应;也没有出现病毒血症和抗体反应[21]。后续试验还证明CD163基因敲除的母猪能够保护胎儿免受PRRSV的母体感染[20](表 2)。我国研究人员Yang等[19]获得了CD163基因敲除杜洛克猪,并使用高致病性PRRSV毒株(HP-PRRSV TP)对其进行攻毒,发现CD163基因敲除猪能够完全抵抗高致病性PRRSV毒株的感染。Xu等[18]使用另一种高致病性PRRSV毒株(HP-PRRSV WUH3)对CD163-pAPN双基因敲除猪进行攻毒,同样发现CD163基因敲除猪能够完全抵抗高致病性PRRSV毒株的感染。这些研究结果证实了猪CD163蛋白是PRRSV感染不可或缺的受体,不表达CD163蛋白的基因编辑猪可以完全抵抗Ⅰ型、Ⅱ型以及高致病性等多种PRRSV,进一步证明了CD163基因在猪抗病育种中的价值。
|
|
表 2 CD163基因编辑猪研究 Table 2 Summary of CD163 gene editing pigs |
2.1.3 猪CD163基因的精准编辑 尽管已经有多项研究表明CD163基因敲除猪可以完全抵抗PRRSV感染,但是由于CD163蛋白在机体某些代谢产物的清除以及免疫调控等方面具有重要的作用,完全敲除CD163基因可能会影响猪的某些生理功能,进而产生非预期效应。较为理想的方式是对CD163蛋白进行精准修饰,使其不能介导PRRSV感染,同时又保持生理功能。要实现这一目标,首先要找到CD163蛋白与PRRSV互作的关键位点。
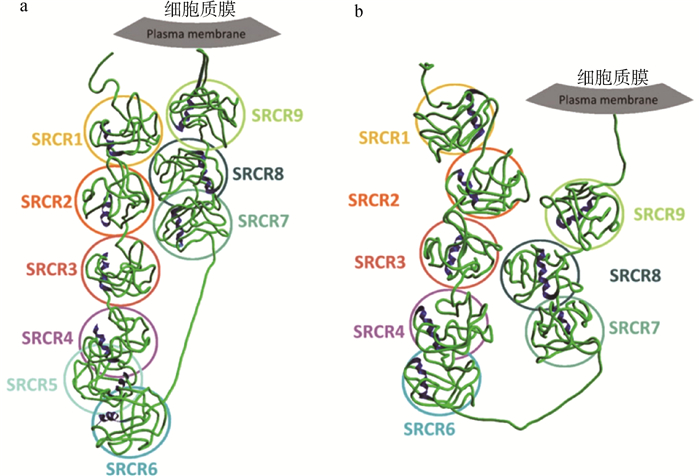
Van Gorp等[3]通过对CD163蛋白重要结构域的依次替换,发现CD163的第5个SRCR结构域在PRRSV感染过程中是必需的,而N端第1至4个SRCR结构域以及胞内尾区不是PRRSV感染所必需的。基于Van Gorp等[3]的发现,Burkard等[16]通过CRISPR/Cas9技术精确删除了CD163蛋白的SRCR5结构域(图 2),并制备了缺失该结构域的猪。他们的研究发现, SRCR5结构域缺失对猪的体重和血细胞数量无影响,对PAM细胞和外周血单核细胞(peripheral blood monocytes, PBMCs)的细胞特异性表面标志物无影响,并且PAM细胞仍然具有清除血红蛋白-触珠蛋白复合物(haemoglobin-haptoglobin,Hb-Hp)的生物活性,证明SRCR5缺失的CD163蛋白在PAM细胞中能够正确表达、折叠并定位在细胞表面发挥其生物学功能。PRRSV攻毒试验还表明, 缺失SRCR5结构域的PAM细胞能够完全抵抗Ⅰ型和Ⅱ型PRRSV感染[16]。后续研究还发现,SRCR5结构域缺失也不影响血浆中sCD163蛋白浓度,并在活体水平证明,缺失SRCR5结构域的基因编辑猪同样能够完全抵抗Ⅰ型PRRSV感染[34]。Wang等[31]通过CRISPR/Cas9和体细胞核移植技术获得了CD163蛋白SRCR5结构域缺失的基因编辑猪,并使用高致病性PRRSV毒株(HP-PRRSV JXA1)对其进行攻毒试验。结果发现,CD163蛋白SRCR5结构域缺失猪能够抵抗高致病性PRRSV,仅在攻毒试验后期表现出轻微的临床症状,同时SRCR5结构域的缺失不影响猪血浆中触珠蛋白(haptoglobin, Hp)的浓度[31]。这些研究结果都证实了CD163蛋白的SRCR5区域是介导PRRSV感染宿主的关键结合区域。
|
a.CD163蛋白结构示意图;b.缺失SRCR5结构域的CD163蛋白示意图 a. Diagram of CD163 protein; b. Diagram of CD163 protein without SRCR5 domain 图 2 CD163以及缺失SRCR5结构域的CD163蛋白结构预测图[34] Fig. 2 Predicted protein structure of CD163 and CD163 without SRCR5 domain[34] |
Guo等[32]进一步缩小了对CD163基因的编辑范围,只删除了SRCR5结构域中的41个氨基酸(481-521位氨基酸),并使用高致病性PRRSV毒株(HP-PRRSV JXA1和MY毒株)进行攻毒,发现基因编辑猪无高烧等PRRS相关临床症状,无病毒血症和抗体反应,能够完全抵抗高致病性PRRSV毒株的感染。Xu等[53]构建了CD163基因SRCR5结构域缺失40个氨基酸(523-562位氨基酸)的永生化PAM细胞系,发现其能完全抵抗Ⅱ型高致病性PRRSV毒株的感染(WUH3毒株)。Guo等[32]和Xu等[53]的研究为确定CD163蛋白介导PRRSV感染的必需区域提供了参考,表明对CD163蛋白进行精准修饰也可以实现对PRRSV的完全抵抗。Ma等[55]基于对猴CD163 SRCR5、人CD163L1 SRCR8以及猪CD163 SRCR5的晶体结构的比较和突变研究,推测猪CD163 SRCR5结构域长环中第534位与561位的带电氨基酸残基在体外对Ⅱ型PRRSV毒株感染起关键作用,这一研究为CD163蛋白精准修饰提供了重要的候选靶点。基于该发现,Xu等[53]创制了CD163蛋白第561位精氨酸精准替换为丙氨酸的CD163-R561A基因编辑猪,并分离该猪的PAM细胞进行PRRSV攻毒试验,发现CD163蛋白R561A精准替换可以显著抑制PRRSV的感染,但不能完全抵抗PRRSV。这一研究表明猪CD163蛋白第561位氨基酸对PRRSV感染有显著影响,但并不是PRRSV感染的必要条件。同时,这一研究也是首次通过单个氨基酸的精准替换研究CD163蛋白与PRRSV互作的关键位点,为精准基因编辑抗病育种提供了重要的研究思路。
以上研究为定位CD163蛋白与PRRSV互作的关键位点提供了重要参考。但是,需要注意的是,SRCR5结构域可能不是PRRSV感染宿主细胞的充分条件,例如Van Gorp等[3]发现非易感细胞仅表达CD163蛋白SRCR5结构域不能介导PRRSV的感染,但表达SRCR5到SRCR9结构域则能感染。说明SRCR5结构域不足以使PRRSV感染,还需要其他结构域的辅助。因此,还需要进一步深入解析CD163蛋白与PRRSV互作的关键位点。
除了将CD163蛋白与PRRSV互作的关键位点删除外,还可以将猪CD163蛋白的SRCR5结构域替换为同源片段,例如Wells等[35]利用人CD163L1蛋白的SRCR8结构域替换猪CD163蛋白SRCR5结构域,发现替换后可以抵抗Ⅰ型PRRSV毒株感染,但是却不抵抗Ⅱ型PRRSV毒株的感染。Chen等[33]同样获得了人CD163L1蛋白的SRCR8结构域替换猪CD163蛋白SRCR5结构域的基因编辑猪,体内和体外攻毒试验表明,虽然同源替换后的CD163突变体不能完全抵抗HP-PRRSV毒株,但是可以通过抑制病毒脱壳和基因组释放来强烈抑制HP-PRRSV的复制。
2.2 CD163蛋白与ASFV除了介导PRRSV的感染外,有研究发现CD163蛋白可能也参与非洲猪瘟病毒(African swine fever virus, ASFV)的吸附和内化。ASFV是一种猪的烈性传染性疾病,可感染各日龄阶段的猪,发病率和致死率可高达100%,给世界各国生猪养殖业造成了重大的经济损失[56],尚无有效的治疗药物或疫苗。与PRRSV类似,ASFV也主要感染猪的单核细胞/巨噬细胞及其衍生细胞,并且成熟后期的细胞优先被感染。这种对细胞的选择性说明ASFV的感染可能需要特异性的受体。Gao等[57]发现ASFV感染过表达CD163和Siglec-1的PK15和3D4-21细胞系以及稳定表达CD163和Siglec-1的PK15S1-CD163细胞系,但单独过表达CD163或者Siglec-1的PK15和3D4-21细胞系则不具有感染性,表明CD163和Siglec-1是ASFV感染宿主细胞的关键受体,在ASFV感染过程中起协同作用。还有研究发现表达CD163蛋白的单核细胞比不表达CD163的单核细胞更容易感染AFSV,使用CD163特异性抗体可以抑制ASFV感染,这些都表明CD163可能参与了ASFV感染[58]。但是CD163蛋白并不是ASFV的必需受体,例如在不感染ASFV的细胞中过表达CD163质粒后,该细胞仍然不能感染ASFV[59]。因此可以推测,其他巨噬细胞相关受体也可能参与ASFV感染过程[60]。
2.3 CD163作为血红蛋白-触珠蛋白复合物受体清除游离的血红蛋白细胞外游离的Hb是一种潜在的毒性物质,因为其中的血红素基团发生化学反应后会释放大量的自由基,进而可能对多种组织,特别是对肾造成严重的氧化损伤[4]。另外,游离的Hb还会与体内的一氧化氮(NO)发生反应,降低NO的生物利用度,阻碍体内NO的稳态[4]。为了避免这种损伤,哺乳动物进化出了游离Hb的高容量清除系统。其中一种就是由CD163和Hp介导的。
当发生生理性或病理性溶血时,从红细胞中释放的游离Hb会立即被Hp捕获以形成具有很高亲和力的Hb-Hp复合物[61]。Hp的结合对Hb毒性具有抑制作用,并防止Hb的过氧化修饰[62]。单核细胞或者巨噬细胞表面的CD163蛋白的SRCR3结构域在相应Ca2+和pH条件的调控下,特异性的结合Hb-Hp复合物,然后将其内吞到巨噬细胞中[63],之后CD163迅速脱离复合体,重新转移到巨噬细胞表面,而Hb-Hp复合物则在溶酶体中被水解。释放的血红素通过胞质溶胶中的血红素加氧酶1(heme oxygenase-1, HO-1)转化为毒性较低的化合物[64]。这种Hp-CD163依赖性的Hb清除机制被认为主要在肝和脾巨噬细胞中起作用,可以有效地防止血红素介导的血管系统以及肾损伤[65-66]。同时,CD163与Hb-Hp复合物的结合还会激活抗炎症因子的释放,如白介素-10(interleukin-10, IL-10),Fe2+,一氧化碳(CO)和胆红素[4, 67]。
此外,CD163蛋白与Hb也有较低的亲和力,在不存在Hp的情况下,CD163蛋白也可以与Hb结合并将其内化。Schaer等[68]的研究表明,在游离Hb的浓度较低时,Hp与Hb形成高亲和力的Hb-Hp复合物,可极大地促进CD163对游离Hb的清理。但是在游离Hb浓度较高(≥100 μg·mL-1),超过Hp的结合能力时,Hb-Hp复合物反而会与游离的Hb竞争结合CD163蛋白。
2.4 CD163作为成红细胞黏附受体促进红细胞生长除了能够清理游离的Hb外,CD163还对红细胞的生长发育具有重要作用[11]。骨髓、脾以及胎儿肝中的红细胞岛是红细胞生成的功能单位[11],这是一个多细胞结构,由处于不同分化阶段的红细胞及其包围在中央的巨噬细胞组成。CD163蛋白的SRCR2结构域中的13个氨基酸基序能够直接与成红细胞结合,因此CD163蛋白可以作为红细胞岛中的成红细胞黏附受体发挥作用,介导红细胞和巨噬细胞之间的接触,不仅将铁运输到成红细胞以合成血红蛋白,而且还分泌许多细胞因子从而支持成红细胞的生存和分化[69]。因此,巨噬细胞上的CD163可能具有双重功能,即同时介导Hb的清除和促进红细胞的生成,形成了铁元素的循环代谢机制。
2.5 CD163蛋白的其他功能CD163也是一种肿瘤坏死因子样凋亡微弱诱导剂(tumor necrosis factor like weak inducer of apoptosis, TWEAK)的受体蛋白[70]。TWEAK是一种通过非死亡结构域依赖机制诱导细胞凋亡的信号分子,参与细胞凋亡、细胞增殖等多种生理过程,它还介导血管生成和炎症[71]。CD163以可溶性结合的形式作用于TWEAK,是TWEAK的受体之一[71]。
CD163特异性地在单核细胞和巨噬细胞中高表达,而巨噬细胞及其祖细胞单核细胞是免疫系统中的关键角色。它们维持体内平衡,在各种病理状态如感染、炎症、动脉粥样硬化和癌症中发挥重要作用。成熟的巨噬细胞是识别和清除潜在病原体的第一道防线,它们能够吞噬、降解自身和外来物质、建立细胞间相互作用和产生炎症介质。抗炎介质糖皮质激素、IL-10和白介素-6(interleukin-6, IL-6)等能够强烈诱导CD163蛋白的表达,形成独特的“替代激活的巨噬细胞”细胞群,它们与伤口愈合、血管生成和保护宿主免受压倒性的炎症反应有关[72],因此CD163的上调表达是炎症中巨噬细胞向替代激活表型转变的标志之一。此外,CD163还同时与抗炎、促炎细胞因子的产生有关。例如CD163与Hb-Hp复合物的结合会激活CO和胆红素的释放[4, 67],这两者都具有很强的抗氧化和抗炎作用[73]。而CD163与CD163特异性单克隆抗体的交联能够诱导NO,白细胞介素-1β(IL-1β),IL-6和肿瘤坏死因子-α(TNF-α)的分泌[74]。IL-1β和TNF-α是促炎细胞因子,而NO和IL-6同时发挥促炎和抗炎作用。CD163还显示出与HMGB1-Hp复合物相互作用并以HO-1依赖性方式调节炎症反应[75]。因此,CD163在免疫调节方面同时发挥着抗炎和促炎两种作用,能够刺激和抑制免疫反应。另外,在临床上,血液或体液中的sCD163常作为某些疾病的生物标记物[7, 76]。
3 小结与展望综上所述,CD163有着广泛的功能,参与了不同生命现象的发生,有着重要的生理作用。目前已有大量证据表明CD163是PRRSV的必需受体,并在病毒脱衣壳以及病毒基因组释放过程中发挥作用[33]。CD163基因敲除猪可以完全抵抗PRRSV的感染[19, 21],本团队前期研究表明CD163和pAPN双基因敲除猪能够抵抗3种病毒,而且具有正常的生产性能[18]。对CD163基因进行精准编辑可以在完全抵抗PRRSV的感染的同时,不影响CD163基因的正常生理功能[31-32, 34]。这些发现为解决猪繁殖与呼吸综合征的危害提供了一种新思路。国际著名猪育种公司Genus已经开展了基因编辑抗猪繁殖与呼吸综合征新品种的培育工作[77]。
然而,对于CD163的研究还远远没有结束,仍有很多关于CD163的未知信息需要探讨。例如CD163蛋白在清除Hb-Hp复合物的同时会激活信号级联反应,产生抗炎分子,因此可以推测,在CD163敲除的情况下,出血性疾病的发生应当会增强炎症反应。但是当CD163敲除猪感染ASFV并且出现出血性症状后,CD163敲除猪和野生型猪之间的临床结果和组织病理学却并没有显著差异[60]。因此,CD163在炎症和免疫反应中的具体作用还需要进一步深入研究。
| [1] |
TABAN Q, MUMTAZ P T, MASOODI K Z, et al. Scavenger receptors in host defense: From functional aspects to mode of action[J]. Cell Commun Signal, 2022, 20(1): 2. DOI:10.1186/s12964-021-00812-0 |
| [2] |
CHENG C, ZHENG E L, YU B W, et al. Recognition of lipoproteins by scavenger receptor class A members[J]. J Biol Chem, 2021, 297(2): 100948. DOI:10.1016/j.jbc.2021.100948 |
| [3] |
VAN GORP H, VAN BREEDAM W, VAN DOORSSELAERE J, et al. Identification of the CD163 protein domains involved in infection of the porcine reproductive and respiratory syndrome virus[J]. J Virol, 2010, 84(6): 3101-3105. DOI:10.1128/JVI.02093-09 |
| [4] |
NIELSEN M J, M∅LLER H J, MOESTRUP S K. Hemoglobin and heme scavenger receptors[J]. Antioxid Redox Signal, 2010, 12(2): 261-273. DOI:10.1089/ars.2009.2792 |
| [5] |
NIELSEN M J, MADSEN M, MøLLER H J, et al. The macrophage scavenger receptor CD163:Endocytic properties of cytoplasmic tail variants[J]. J Leukoc Biol, 2006, 79(4): 837-845. DOI:10.1189/jlb.1005602 |
| [6] |
STOIAN A M M, ROWLAND R R R, BRANDARIZ-NUÑEZ A. Identification of CD163 regions that are required for porcine reproductive and respiratory syndrome virus (PRRSV) infection but not for binding to viral envelope glycoproteins[J]. Virology, 2022, 574: 71-83. DOI:10.1016/j.virol.2022.07.012 |
| [7] |
HUANG Y J, LIN C H, YANG H Y, et al. Urine soluble CD163 is a promising biomarker for the diagnosis and evaluation of lupus nephritis[J]. Front Immunol, 2022, 13: 935700. DOI:10.3389/fimmu.2022.935700 |
| [8] |
DAVID C, DIVARD G, ABBAS R, et al. Soluble CD163 is a biomarker for accelerated atherosclerosis in systemic lupus erythematosus patients at apparent low risk for cardiovascular disease[J]. Scand J Rheumatol, 2020, 49(1): 33-37. DOI:10.1080/03009742.2019.1614213 |
| [9] |
BUECHLER C, RITTER M, ORSÓ E, et al. Regulation of scavenger receptor CD163 expression in human monocytes and macrophages by pro- and antiinflammatory stimuli[J]. J Leukoc Biol, 2000, 67(1): 97-103. DOI:10.1002/jlb.67.1.97 |
| [10] |
VAN DEN HEUVEL M M, TENSEN C P, VAN AS J H, et al. Regulation of CD163 on human macrophages: Cross-linking of CD163 induces signaling and activation[J]. J Leukoc Biol, 1999, 66(5): 858-866. DOI:10.1002/jlb.66.5.858 |
| [11] |
BORGES M D, SESTI-COSTA R. Macrophages: Key players in erythrocyte turnover[J]. Hematol Transfus Cell Ther, 2022, 44(4): 574-581. DOI:10.1016/j.htct.2022.07.002 |
| [12] |
GUTIÉRREZ-MUÑOZ C, MÉNDEZ-BARBERO N, SVENDSEN P, et al. CD163 deficiency increases foam cell formation and plaque progression in atherosclerotic mice[J]. FASEB J, 2020, 34(11): 14960-14976. DOI:10.1096/fj.202000177R |
| [13] |
SIWAN E, TWIGG S M, MIN D Q. Alterations of CD163 expression in the complications of diabetes: A systematic review[J]. J Diabetes Complicat, 2022, 36(4): 108150. DOI:10.1016/j.jdiacomp.2022.108150 |
| [14] |
成红军, 潘志昂, 祝成楼, 等. CD163在常见肿瘤中的临床意义及研究进展[J]. 临床荟萃, 2022, 37(7): 653-657. CHENG H J, PAN Z A, ZHU C L, et al. Clinical significance and research progress of CD163 in common tumors[J]. Clinical Focus, 2022, 37(7): 653-657. (in Chinese) |
| [15] |
TROIANO G, CAPONIO V C A, ADIPIETRO I, et al. Prognostic significance of CD68+ and CD163+ tumor associated macrophages in head and neck squamous cell carcinoma: A systematic review and meta-analysis[J]. Oral Oncol, 2019, 93: 66-75. DOI:10.1016/j.oraloncology.2019.04.019 |
| [16] |
BURKARD C, LILLICO S G, REID E, et al. Precision engineering for PRRSV resistance in pigs: Macrophages from genome edited pigs lacking CD163 SRCR5 domain are fully resistant to both PRRSV genotypes while maintaining biological function[J]. PLoS Pathog, 2017, 13(2): e1006206. DOI:10.1371/journal.ppat.1006206 |
| [17] |
DUAN X, NAUWYNCK H J, PENSAERT M B. Effects of origin and state of differentiation and activation of monocytes/macrophages on their susceptibility to porcine reproductive and respiratory syndrome virus (PRRSV)[J]. Arch Virol, 1997, 142(12): 2483-2497. DOI:10.1007/s007050050256 |
| [18] |
XU K, ZHOU Y R, MU Y L, et al. CD163 and pAPN double-knockout pigs are resistant to PRRSV and TGEV and exhibit decreased susceptibility to PDCoV while maintaining normal production performance[J]. Elife, 2020, 9: e57132. DOI:10.7554/eLife.57132 |
| [19] |
YANG H Q, ZHANG J, ZHANG X W, et al. CD163 knockout pigs are fully resistant to highly pathogenic porcine reproductive and respiratory syndrome virus[J]. Antiviral Res, 2018, 151: 63-70. DOI:10.1016/j.antiviral.2018.01.004 |
| [20] |
PRATHER R S, WELLS K D, WHITWORTH K M, et al. Knockout of maternal CD163 protects fetuses from infection with porcine reproductive and respiratory syndrome virus (PRRSV)[J]. Sci Rep, 2017, 7(1): 13371. DOI:10.1038/s41598-017-13794-2 |
| [21] |
WHITWORTH K M, ROWLAND R R R, EWEN C L, et al. Gene-edited pigs are protected from porcine reproductive and respiratory syndrome virus[J]. Nat Biotechnol, 2016, 34(1): 20-22. DOI:10.1038/nbt.3434 |
| [22] |
VANDERHEIJDEN N, DELPUTTE P L, FAVOREEL H W, et al. Involvement of sialoadhesin in entry of porcine reproductive and respiratory syndrome virus into porcine alveolar macrophages[J]. J Virol, 2003, 77(15): 8207-8215. DOI:10.1128/JVI.77.15.8207-8215.2003 |
| [23] |
VAN GORP H, VAN BREEDAM W, DELPUTTE P L, et al. The porcine reproductive and respiratory syndrome virus requires trafficking through CD163-positive early endosomes, but not late endosomes, for productive infection[J]. Arch Virol, 2009, 154(12): 1939-1943. DOI:10.1007/s00705-009-0527-1 |
| [24] |
YUSTE M, FERNÁNDEZ-CABALLERO T, PRIETO C, et al. Splenic CD163+ macrophages as targets of porcine reproductive and respiratory virus: Role of siglecs[J]. Vet Microbiol, 2017, 198: 72-80. DOI:10.1016/j.vetmic.2016.12.004 |
| [25] |
DELPUTTE P L, VANDERHEIJDEN N, NAUWYNCK H J, et al. Involvement of the matrix protein in attachment of porcine reproductive and respiratory syndrome virus to a heparinlike receptor on porcine alveolar macrophages[J]. J Virol, 2002, 76(9): 4312-4320. DOI:10.1128/JVI.76.9.4312-4320.2002 |
| [26] |
KIM J K, FAHAD A M, SHANMUKHAPPA K, et al. Defining the cellular target(s) of porcine reproductive and respiratory syndrome virus blocking monoclonal antibody 7G10[J]. J Virol, 2006, 80(2): 689-696. DOI:10.1128/JVI.80.2.689-696.2006 |
| [27] |
LI L L, XUE B Y, SUN W Y, et al. Recombinant MYH9 protein C-terminal domain blocks porcine reproductive and respiratory syndrome virus internalization by direct interaction with viral glycoprotein 5[J]. Antiviral Res, 2018, 156: 10-20. DOI:10.1016/j.antiviral.2018.06.001 |
| [28] |
GAO J M, XIAO S Q, XIAO Y H, et al. MYH9 is an essential factor for porcine reproductive and respiratory syndrome virus infection[J]. Sci Rep, 2016, 6(1): 25120. DOI:10.1038/srep25120 |
| [29] |
HUANG Y W, DRYMAN B A, LI W, et al. Porcine DC-SIGN: Molecular cloning, gene structure, tissue distribution and binding characteristics[J]. Dev Comp Immunol, 2009, 33(4): 464-480. DOI:10.1016/j.dci.2008.09.010 |
| [30] |
XIE J X, CHRISTIAENS I, YANG B, et al. Molecular cloning of porcine siglec-3, siglec-5 and siglec-10, and identification of siglec-10 as an alternative receptor for porcine reproductive and respiratory syndrome virus (PRRSV)[J]. J Gen Virol, 2017, 98(8): 2030-2042. DOI:10.1099/jgv.0.000859 |
| [31] |
WANG H T, SHEN L C, CHEN J Y, et al. Deletion of CD163 exon 7 confers resistance to highly pathogenic porcine reproductive and respiratory viruses on pigs[J]. Int J Biol Sci, 2019, 15(9): 1993-2005. DOI:10.7150/ijbs.34269 |
| [32] |
GUO C H, WANG M, ZHU Z B, et al. Highly efficient generation of pigs harboring a partial deletion of the CD163 SRCR5 domain, which are fully resistant to porcine reproductive and respiratory syndrome virus 2 infection[J]. Front Immunol, 2019, 10: 1846. DOI:10.3389/fimmu.2019.01846 |
| [33] |
CHEN J Y, WANG H T, BAI J H, et al. Generation of pigs resistant to highly pathogenic-porcine reproductive and respiratory syndrome virus through gene editing of CD163[J]. Int J Biol Sci, 2019, 15(2): 481-492. DOI:10.7150/ijbs.25862 |
| [34] |
BURKARD C, OPRIESSNIG T, MILEHAM A J, et al. Pigs lacking the scavenger receptor cysteine-rich domain 5 of CD163 are resistant to porcine reproductive and respiratory syndrome virus 1 infection[J]. J Virol, 2018, 92(16): e00415-18. |
| [35] |
WELLS K D, BARDOT R, WHITWORTH K M, et al. Replacement of porcine CD163 scavenger receptor cysteine-rich domain 5 with a CD163-like homolog confers resistance of pigs to genotype 1 but not genotype 2 porcine reproductive and respiratory syndrome virus[J]. J Virol, 2017, 91(2): e01521-16. |
| [36] |
WU J J, PENG X W, ZHOU A, et al. Mir-506 inhibits PRRSV replication in MARC-145 cells via CD151[J]. Mol Cell Biochem, 2014, 394(1): 275-281. |
| [37] |
SHANMUKHAPPA K, KIM J K, KAPIL S. Role of CD151, a tetraspanin, in porcine reproductive and respiratory syndrome virus infection[J]. Virol J, 2007, 4(1): 62. DOI:10.1186/1743-422X-4-62 |
| [38] |
HOU G P, XUE B Y, LI L L, et al. Direct interaction between CD163 N-terminal domain and MYH9 c-terminal domain contributes to porcine reproductive and respiratory syndrome virus internalization by permissive cells[J]. Front Microbiol, 2019, 10: 1815. DOI:10.3389/fmicb.2019.01815 |
| [39] |
CALVERT J G, SLADE D E, SHIELDS S L, et al. CD163 expression confers susceptibility to porcine reproductive and respiratory syndrome viruses[J]. J Virol, 2007, 81(14): 7371-7379. DOI:10.1128/JVI.00513-07 |
| [40] |
WANG X P, WEI R F, LI Q Y, et al. PK-15 cells transfected with porcine CD163 by PiggyBac transposon system are susceptible to porcine reproductive and respiratory syndrome virus[J]. J Virol Methods, 2013, 193(2): 383-390. DOI:10.1016/j.jviromet.2013.06.035 |
| [41] |
LI L L, WU C Y, HOU G P, et al. Generation of murine macrophage-derived cell lines expressing porcine CD163 that support porcine reproductive and respiratory syndrome virus infection[J]. BMC Biotechnol, 2017, 17(1): 77. DOI:10.1186/s12896-017-0399-5 |
| [42] |
WANG T Y, LIU Y G, LI L, et al. Porcine alveolar macrophage CD163 abundance is a pivotal switch for porcine reproductive and respiratory syndrome virus infection[J]. Oncotarget, 2018, 9(15): 12174-12185. DOI:10.18632/oncotarget.24040 |
| [43] |
XU Y L, WU S P, LI Y G, et al. A porcine alveolar macrophage cell line stably expressing CD163 demonstrates virus replication and cytokine secretion characteristics similar to primary alveolar macrophages following PRRSV infection[J]. Vet Microbiol, 2020, 244: 108690. DOI:10.1016/j.vetmic.2020.108690 |
| [44] |
LI N, HUANG K, CHEN Y J, et al. MicroRNA ssc-miR-124a exhibits antiviral activity against porcine reproductive and respiratory syndrome virus via suppression of host genes CD163[J]. Vet Microbiol, 2021, 261: 109216. DOI:10.1016/j.vetmic.2021.109216 |
| [45] |
YU P, WEI R P, DONG W J, et al. CD163ΔSRCR5 MARC-145 cells resist PRRSV-2 infection via inhibiting virus uncoating, which requires the interaction of CD163 with Calpain 1[J]. Front Microbiol, 2020, 10: 3115. DOI:10.3389/fmicb.2019.03115 |
| [46] |
XIA W L, WU Z, GUO C M, et al. Recombinant adenovirus-delivered soluble CD163 and sialoadhesin receptors protected pigs from porcine reproductive and respiratory syndrome virus infection[J]. Vet Microbiol, 2018, 219: 1-7. DOI:10.1016/j.vetmic.2018.04.006 |
| [47] |
CHEN Y, GUO R, HE S, et al. Additive inhibition of porcine reproductive and respiratory syndrome virus infection with the soluble sialoadhesin and CD163 receptors[J]. Virus Res, 2014, 179: 85-92. DOI:10.1016/j.virusres.2013.11.008 |
| [48] |
XU H L, LIU Z H, ZHENG S Y, et al. CD163 antibodies inhibit PRRSV infection via receptor blocking and transcription suppression[J]. Vaccines (Basel), 2020, 8(4): 592. DOI:10.3390/vaccines8040592 |
| [49] |
刘铃, 王丹丹, 崔凯, 等. 猪繁殖与呼吸综合征抗病育种研究进展[J]. 畜牧兽医学报, 2023, 54(2): 434-442. LIU L, WANG D D, CUI K, et al. Advances of disease-resistant breeding on porcine reproductive and respiratory syndrome[J]. Acta Veterinaria et Zootechnica Sinica, 2023, 54(2): 434-442. (in Chinese) |
| [50] |
魏迎辉, 刘志国, 徐奎, 等. CD163双等位基因编辑猪的制备及传代[J]. 中国农业科学, 2018, 51(4): 770-777. WEI Y H, LIU Z G, XU K, et al. Generation and propagation of cluster of differentiation 163 biallelic gene editing pigs[J]. Scientia Agricultura Sinica, 2018, 51(4): 770-777. (in Chinese) |
| [51] |
韩晓松, 高杨, 刘海龙, 等. 利用CRISPR/Cas9技术制备CD163基因SRCR5序列敲除猪[J]. 农业生物技术学报, 2020, 28(9): 1535-1542. HAN X S, GAO Y, LIU H L, et al. Generation of CD163 gene SRCR5 deleted pig (Sus scrofa) via CRISPR/Cas9[J]. Journal of Agricultural Biotechnology, 2020, 28(9): 1535-1542. (in Chinese) |
| [52] |
赵为民, 王慧利, 曹少先, 等. 猪CD163基因的单碱基编辑研究[J]. 畜牧兽医学报, 2022, 53(4): 1041-1050. ZHAO W M, WANG H L, CAO S X, et al. The study of base editing of porcine CD163 gene[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(4): 1041-1050. (in Chinese) |
| [53] |
XU K, ZHOU Y R, SHANG H T, et al. Pig macrophages with site-specific edited CD163 decrease the susceptibility to infection with porcine reproductive and respiratory syndrome virus[J/OL]. J Int Agricul, 2022. (2022-11-29). https://doi.org/10.1016/j.jia.2022.11.010.
|
| [54] |
张健, 吴珍芳, 杨化强. CD163基因敲除大白猪的抗蓝耳病性能和主要生产性能研究[J]. 华南农业大学学报, 2023, 44(3): 333-339. ZHANG J, WU Z F, YANG H Q. Resistance to blue ear disease and production performance assessment of CD163 gene-edited Large White pigs[J]. Journal of South China Agricultural University, 2023, 44(3): 333-339. (in Chinese) |
| [55] |
MA H F, LI R, JIANG L G, et al. Structural comparison of CD163 SRCR5 from different species sheds some light on its involvement in porcine reproductive and respiratory syndrome virus-2 infection in vitro[J]. Vet Res, 2021, 52(1): 97. DOI:10.1186/s13567-021-00969-z |
| [56] |
赵旭阳, 靳家鑫, 路闻龙, 等. 非洲猪瘟病毒免疫逃逸分子机制研究进展[J]. 畜牧兽医学报, 2022, 53(7): 2074-2082. ZHAO X Y, JIN J X, LU W L, et al. Advances in the molecular mechanism of immune escape of African swine fever virus[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(7): 2074-2082. (in Chinese) |
| [57] |
GAO Q, YANG Y L, LUO Y Z, et al. Adaptation of African swine fever virus to porcine kidney cells stably expressing CD163 and Siglec1[J]. Front Immunol, 2022, 13: 1015224. DOI:10.3389/fimmu.2022.1015224 |
| [58] |
SÁNCHEZ E G, PÉREZ-NÚÑEZ D, REVILLA Y. Mechanisms of entry and endosomal pathway of African swine fever virus[J]. Vaccines (Basel), 2017, 5(4): 42. DOI:10.3390/vaccines5040042 |
| [59] |
LITHGOW P, TAKAMATSU H, WERLING D, et al. Correlation of cell surface marker expression with African swine fever virus infection[J]. Vet Microbiol, 2014, 168(2-4): 413-419. DOI:10.1016/j.vetmic.2013.12.001 |
| [60] |
李玲, 夏应菊, 宋新宇, 等. 非洲猪瘟病毒细胞嗜性的研究进展[J]. 中国兽医科学, 2023, 53(4): 514-519. LI L, XIA Y J, SONG X Y, et al. Research progress on cytotoxicity of African swine fever virus[J]. Chinese Veterinary Science, 2023, 53(4): 514-519. DOI:10.16656/j.issn.1673-4696.2023.0069 (in Chinese) |
| [61] |
HWANG P K, GREER J. Interaction between hemoglobin subunits in the hemoglobin. haptoglobin complex[J]. J Biol Chem, 1980, 255(7): 3038-3041. DOI:10.1016/S0021-9258(19)85848-9 |
| [62] |
BUEHLER P W, ABRAHAM B, VALLELIAN F, et al. Haptoglobin preserves the CD163 hemoglobin scavenger pathway by shielding hemoglobin from peroxidative modification[J]. Blood, 2009, 113(11): 2578-2586. DOI:10.1182/blood-2008-08-174466 |
| [63] |
MADSEN M, MØLLER H J, NIELSEN M J, et al. Molecular characterization of the haptoglobin·Hemoglobin receptor CD163:Ligand binding properties of the scavenger receptor cysteine-rich domain region[J]. J Biol Chem, 2004, 279(49): 51561-51567. DOI:10.1074/jbc.M409629200 |
| [64] |
BELCHER J D, BECKMAN J D, BALLA G, et al. Heme degradation and vascular injury[J]. Antioxid Redox Signal, 2010, 12(2): 233-248. DOI:10.1089/ars.2009.2822 |
| [65] |
BELCHER J D, MAHASETH H, WELCH T E, et al. Heme oxygenase-1 is a modulator of inflammation and vaso-occlusion in transgenic sickle mice[J]. J Clin Invest, 2006, 116(3): 808-816. DOI:10.1172/JCI26857 |
| [66] |
LIM Y K, JENNER A, ALI A B, et al. Haptoglobin reduces renal oxidative DNA and tissue damage during phenylhydrazine-induced hemolysis[J]. Kidney Int, 2000, 58(3): 1033-1044. DOI:10.1046/j.1523-1755.2000.00261.x |
| [67] |
PHILIPPIDIS P, MASON J C, EVANS B J, et al. Hemoglobin scavenger receptor CD163 mediates interleukin-10 release and heme oxygenase-1 synthesis: Antiinflammatory monocyte-macrophage responses in vitro, in resolving skin blisters in vivo, and after cardiopulmonary bypass surgery[J]. Circ Res, 2004, 94(1): 119-126. DOI:10.1161/01.RES.0000109414.78907.F9 |
| [68] |
SCHAER D J, SCHAER C A, BUEHLER P W, et al. CD163 is the macrophage scavenger receptor for native and chemically modified hemoglobins in the absence of haptoglobin[J]. Blood, 2006, 107(1): 373-380. DOI:10.1182/blood-2005-03-1014 |
| [69] |
FABRIEK B O, POLFLIET M M J, VLOET R P M, et al. The macrophage CD163 surface glycoprotein is an erythroblast adhesion receptor[J]. Blood, 2007, 109(12): 5223-5229. DOI:10.1182/blood-2006-08-036467 |
| [70] |
MORENO J A, MUÑOZ-GARCÍA B, MARTÍN-VENTURA J L, et al. The CD163-expressing macrophages recognize and internalize TWEAK: Potential consequences in atherosclerosis[J]. Atherosclerosis, 2009, 207(1): 103-110. DOI:10.1016/j.atherosclerosis.2009.04.033 |
| [71] |
RATAJCZAK W, ATKINSON S D, KELLY C. The TWEAK/Fn14/CD163 axis-implications for metabolic disease[J]. Rev Endocr Metab Disord, 2022, 23(3): 449-462. DOI:10.1007/s11154-021-09688-4 |
| [72] |
ETZERODT A, MOESTRUP S K. CD163 and inflammation: Biological, diagnostic, and therapeutic aspects[J]. Antioxid Redox Signal, 2013, 18(17): 2352-2363. DOI:10.1089/ars.2012.4834 |
| [73] |
OTTERBEIN L E, BACH F H, ALAM J, et al. Carbon monoxide has anti-inflammatory effects involving the mitogen-activated protein kinase pathway[J]. Nat Med, 2000, 6(4): 422-428. DOI:10.1038/74680 |
| [74] |
POLFLIET M M J, FABRIEK B O, DANIËLS W P, et al. The rat macrophage scavenger receptor CD163:Expression, regulation and role in inflammatory mediator production[J]. Immunobiology, 2006, 211(6-8): 419-425. DOI:10.1016/j.imbio.2006.05.015 |
| [75] |
YANG H, WANG H C, LEVINE Y A, et al. Identification of CD163 as an antiinflammatory receptor for HMGB1-haptoglobin complexes[J]. JCI Insight, 2016, 1(7): e85375. |
| [76] |
侯亚芝, 张晋欣, 陈小飞. 可溶性血红蛋白清道夫受体163在不同疾病中的研究进展[J]. 中西医结合心脑血管病杂志, 2022, 20(18): 3341-3348. HOU Y Z, ZHANG J X, CHEN X F. Research progress of soluble hemoglobin scavenger receptor 163 in different diseases[J]. Chinese Journal of Integrative Medicine on Cardio-Cerebrovascular Disease, 2022, 20(18): 3341-3348. (in Chinese) |
| [77] |
刘志国, 王冰源, 牟玉莲, 等. 分子编写育种——动物育种的发展方向[J]. 中国农业科学, 2018, 51(12): 2398-2409. LIU Z G, WANG B Y, MU Y L, et al. Breeding by molecular writing (BMW): The future development of animal breeding[J]. Scientia Agricultura Sinica, 2018, 51(12): 2398-2409. (in Chinese) |
(编辑 郭云雁)



