主要组织相容性复合体(major histocompatibility complex,MHC)是一组编码主要组织相容性抗原的基因群总称,最早在小鼠体内被发现[1-2]。MHC作为组织细胞的遗传标记分子,与动物的抗病性、抗原识别、递呈和免疫调节密切相关[3-5]。此外,MHC编码的高度多态的蛋白对器官移植成功与否具有关键作用[6-9]。猪MHC又称猪白细胞抗原(swine leukocyte antigen,SLA),位于猪第7号染色体并横跨着丝点,是包含猪基因组中许多免疫相关基因的紧密连锁区域[10-11]。SLA基因包含高度多态的SLA Ⅰ类、SLA Ⅱ类和相对保守的SLA Ⅲ类基因,高度多态的SLA-1基因是经典的SLA Ⅰ类基因,目前IPD-MHC(Immuno Polymorphism Database-MHC)数据库共收录了89个SLA-1基因(Release 3.9.0.1 07/2022 build 209)。SLA-1基因编码的SLA Ⅰ类分子通过内源性抗原递呈,将胞内的抗原肽递呈到细胞表面,经CD8+T细胞识别并引起细胞免疫,对于机体的健康以及对疾病的抗病性具有重要作用。SLA-1包含8个外显子,第1外显子编码前导多肽链,第2~4外显子分别编码α1~α3胞外域,第5外显子编码跨膜结构域,第6~8外显子编码胞内域,其中,第2和第3外显子编码的α1和α2胞外域多态性最高,二者形成的抗原肽结合槽(peptide-binding groove,PBG)决定了与之结合的T淋巴细胞表位的特异性[12]。有研究利用SLA-1分子体外构建SLA Ⅰ类复合体研究与病毒T细胞表位结合的特异性,发现不同SLA-1分子结合T细胞表位的能力不同[13],这就提示携带不同SLA-1等位基因的猪对疫病的抵抗力不同。
猪是世界范围内广泛培育的重要经济动物,是人们日常生活主要的动物蛋白来源,而且由于猪与人类在基因组、血液生理指标、解剖学结构和成分等方面高度相似,使其逐渐成为一种重要的实验动物,应用于临床器官移植、动物疾病模型构建和病原致病机制研究等领域[14-17]。荣昌猪是中国西南地区的本土猪种,由于其肉质好和抗逆性强成为中国主要的优良猪种之一,目前,已在各领域表现出重要的应用价值,如用于建立人类医学动物模型,MITF-M基因突变形成的白化耳聋荣昌猪可以作为一种人类遗传性神经性听力缺陷的大型哺乳动物模型,在人工耳蜗应用以及研究中国耳聋遗传方面具有十分重要的意义[18-20]。同时荣昌猪具有较强的抗病性,已用于建立猪病原感染模型并研究自身免疫机制,为重大疫病防控提供支撑,同时也用于筛选独特的免疫相关分子,为抗病育种奠定基础。2019年, 中国农业科学院哈尔滨兽医研究所从重庆市畜牧科学院生物工程研究所引入27头荣昌猪,屏障环境饲养繁育,主要用于培育标准化的实验猪种群,同时开展了疫病净化、疾病模型构建及应用研究。本研究就该荣昌猪种群SLA-1基因特征进行分析,旨在为标准化实验荣昌猪的培育和推广应用奠定基础,也为异种移植研究提供遗传学参考。
1 材料与方法 1.1 试验材料通过前腔静脉采集中国农业科学院哈尔滨兽医研究所保存的27头荣昌猪(: 6,: 21)的抗凝血5 mL,置于EDTA抗凝管,用于分离外周血淋巴细胞,27头荣昌猪的编号如表 1所示。
|
|
表 1 荣昌猪SLA-1等位基因 Table 1 SLA-1 allele of Rongchang pig |
猪外周血淋巴细胞分离液购自天津市灏洋生物制品科技有限公司,Simply总RNA提取试剂盒购自Biosharp公司,DNA胶回收试剂盒购自OMEGA公司,One step PCR试剂盒、感受态细胞大肠杆菌(Escherichia coli)DH5α和载体pMD18-T vector均购自宝生物工程(大连)有限公司。
1.3 外周血淋巴细胞的分离和RNA的提取根据猪外周血淋巴细胞分离液说明书,将猪外周血淋巴细胞分离,-80 ℃保存。根据Simply总RNA提取试剂盒说明书提取外周血淋巴细胞总RNA,利用超微量分光光度计(NanoPhotometer,德国)测量总RNA浓度,-80 ℃保存。
1.4 引物设计与合成参考GenBank中大白猪H01单倍型序列(登录号:AJ251829),利用Primer Premier 5软件设计引物扩增SLA-1基因,扩增片段约为1 537 bp。SLA1-F:5′-CTCAGCTTCTCCCCAGACCCCGA-GGCTGAGGATC-3′,SLA1-R:5′-GGATTCTGGAAGGTTCTCAATCCTTCCATTTATTTCCTC-3′。引物由吉林省库美生物科技有限公司合成。
1.5 SLA-1的扩增和测序利用One Step PCR试剂盒以及合成的引物对目的基因进行扩增。RT-PCR反应体系50 μL:PrimeScript 1 Step Enzyme Mix 2 μL,2×1 step Buffer 25 μL,上、下游引物(10 pmol·μL-1)各1.5 μL,RNA模板2 μL(约300 ng·μL-1),RNase Free H2O 18 μL。PCR反应条件:50 ℃反转录30 min;94 ℃预变性2 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,35次循环;72 ℃延伸8 min。利用1%琼脂糖凝胶电泳对扩增产物进行鉴定,由吉林省库美生物科技有限公司直接测序。对于杂合子,利用胶回收试剂盒纯化目的片段,与pMD-18 T载体在16 ℃条件下连接2 h,然后转化至大肠杆菌DH5α感受态细胞中,并利用含Amp抗性的LB平板进行筛选。鉴定阳性单克隆菌落后,送至吉林省库美生物科技有限公司测序。
1.6 序列比对及分析测序获得的原始序列数据通过Chromas Version 2.6.6进行人工编辑,校对电泳峰图与碱基对应关系。利用MEGA 7.0软件对直接测序和克隆测序结果进行比对,根据不同猪个体中SLA-1基因序列差异确定等位基因序列。当同一个体克隆测序中至少3个单克隆的序列相同且该序列在不同个体的直接或克隆测序中多次出现,判定为一个等位基因,即一个等位基因的判定必须至少出现在2个不同的个体或来自2个独立的PCR反应。利用DnaSP 5.10.01软件进行等位基因多态性分析,利用DNASTAR Lasergene软件进行同源性分析,以人白细胞抗原(human leukocyte antigen,HLA)频率最高的HLA-A*02:01等位基因(GenBank登录号:FN806801)为参照[21],利用MEGA 7.0采用邻接法(Neighbor-Joining,N-J)构建系统进化树。所有序列提交GenBank获得登录号。将新等位基因提交国际动物遗传学学会(The International Society of Animal Genetics,ISAG)SLA命名委员会获得官方命名。SLA等位基因的命名采用HLA命名系统,等位基因前两位数字用于指定具有相似DNA序列的等位基因组(“组”是基于系统发育分析和DNA序列基序的识别);第3和第4位数字用于指定具有不同蛋白质序列的等位基因;第5和第6位数字用于指定因同义核苷酸替换而不同的等位基因;以冒号(: )作为区域分隔符。
2 结果 2.1 RT-PCR扩增与克隆RT-PCR扩增获得1 537 bp左右的目的条带(图 1),清晰且单一,表明已成功扩增SLA-1基因。将27头猪的扩增产物全部进行直接测序,分析测序结果,发现4头猪测序结果为单峰(猪编号为3、9、13和27),且序列相同,表明是纯合子个体,对其中1头(3号猪)进行了克隆。其它23头猪均为套峰,表明为杂合子个体,将具有相同杂合套峰的个体进行分类后,分别对10个个体(猪编号为3、5、10、15、16、17、18、19、23和24)进行了克隆。所有克隆鉴定阳性单克隆菌落并测序(图 2)。
|
M. DL 2000 DNA分子标记;1. PCR产物(猪编号2);2.PCR产物(猪编号3);3. PCR产物(猪编号5);4. PCR产物(猪编号10);5. PCR产物(猪编号14);6. PCR产物(猪编号15);7. PCR产物(猪编号17);8. PCR产物(猪编号18) M. DL 2000 DNA marker; 1. PCR product of No. 2; 2.PCR product of No. 3; 3. PCR product of No. 5; 4. PCR product of No. 10; 5. PCR product of No. 14; 6. PCR product of No. 15; 7. PCR product of No. 17; 8. PCR product of No. 18 图 1 SLA-1基因部分扩增结果 Fig. 1 Partial amplification results of SLA-1 |
|
M.DL 2000 DNA分子标记;1~4.编号10的单克隆鉴定部分结果;5~8.编号14的单克隆鉴定结果 M. DL 2000 DNA marker; 1-4. Partial results of no.10 monoclonal identification; 5-8. Partial results of no.14 monoclonal identification 图 2 部分阳性单克隆菌落PCR扩增结果 Fig. 2 Partial results of monoclonal identification |
分析27头荣昌猪SLA-1基因直接测序和克隆测序结果,根据等位基因的判定原则共鉴定出11个等位基因(表 1),将其提交GenBank数据库和IPD-MHC数据库,分别获得登录号和ISAG SLA命名委员会的官方命名(表 2)。与IPD-MHC数据库中已提交的89个SLA-1等位基因序列相比较,获得2个已知等位基因,SLA-1*11:03和SLA-1*15:03,分别存在于韩国地方猪种和中国融水小型猪、皖南花猪中。此外,发现9个新等位基因,命名为SLA-1*02:04、02:05、10:03、16:05、16:06、18:03、23:05、24:01和25:01。27头荣昌猪均至少包含1个SLA-1新等位基因,且10头荣昌猪中至少包含3个SLA-1等位基因,编号10的个体中含有4个不同的SLA-1等位基因,表明SLA-1是双拷贝基因。在该群体中,对被鉴定出在SLA-1基因座上具有两个以上等位基因的个体进行等位基因之间的两两组合分析,对在不同基因型个体中出现次数超过3次以上的组合确定为同一条染色体上的双拷贝等位基因,在编号分别为1、6、10、18、24、25的个体中不仅鉴定出SLA-1*23:05、SLA-1*11:03等位基因而且还有其他等位基因存在,因此在该群体中SLA-1*23:05与SLA-1*11:03,SLA-1*02:04与SLA-1*18:03之间存在连锁关系。连锁的等位基因SLA-1*23:05和SLA-1*11:03以及SLA-1*02:04和SLA-1*18:03分别用双拷贝等位基因A和B表示,等位基因SLA-1*02:05、SLA-1*10:03、SLA-1*15:03、SLA-1*16:05、SLA-1*16:06、SLA-1*24:01、SLA-1*25:01分别用等位基因C、D、E、F、G、H、I表示,该群体在SLA-1基因座上共获得11种基因型(表 1)。群体的等位基因频率显示,频率最高的是等位基因H(SLA-1*24:01),频率为44.23%,其次是双拷贝等位基因A(SLA-1*23:05,SLA-1*11:03)和等位基因E(SLA-1*15:03),频率分别为19.23%、17.31%,H(SLA-1*24:01)是该荣昌猪群体中最流行的等位基因(图 3)。
|
|
表 2 荣昌猪SLA-1等位基因提交信息 Table 2 Submission information for SLA-1 allele in Rongchang pig |
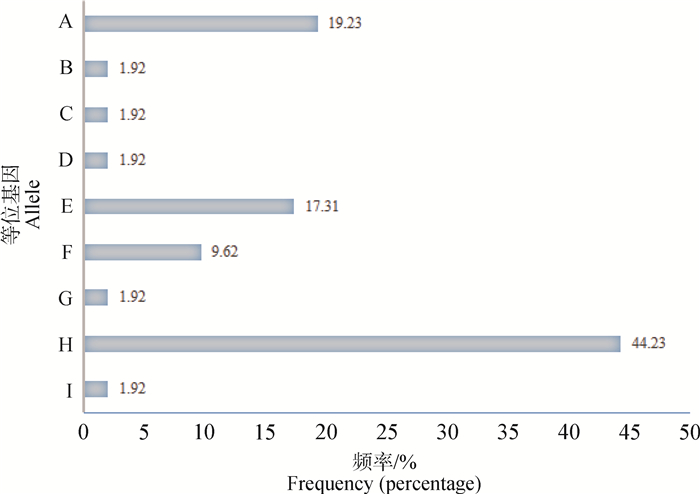
|
图 3 SLA-1等位基因及频率分布 Fig. 3 The distribution of SLA-1alleles and frequency |
11个SLA-1等位基因编码区(coding sequences,CDS)DNA多态性分析显示(图 4),碱基变异(base variation,S)主要集中在225~325 bp和500~575 bp两个区域,核苷酸多样度(nucleotide diversity,Pi)的变化趋势与碱基变异数的分布趋势基本保持一致,而且两者的峰值均处于第2和第3外显子区域内,表示该区域是SLA-1基因的高频变异位点区。在CDS区内,整体的Pi为0.044 9,单倍型多样度(haplotype diversity,Hd)为1,平均核苷酸差异数(average number of nucleotide differences,K)为48.782,G+C含量为64.9%,在所编码的361个氨基酸中,产生了75个氨基酸变异位点(amino acid variable sites,AV);在1~1 086 bp的核苷酸位点中,涵盖了127个核苷酸多态位点,其中, 包含简约信息位点85个,单一多态位点42个,此外,核苷酸多态位点包含18个同义单核苷酸多态性位点和109个非同义单核苷酸多态性位点。第2和第3外显子区域的多态性规律与整个CDS区变异相似(表 3)。
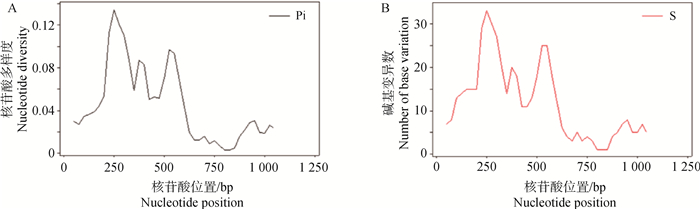
|
图 4 SLA-1等位基因碱基变异位置分布(A)和核苷酸多样度变化趋势(B) Fig. 4 The distribution of base variation positions (A) and nucleotide diversity (B) of SLA-1 allele sequence |
|
|
表 3 SLA-1等位基因序列多态性分析 Table 3 Polymorphism analysis of SLA-1 allele sequences |
由于SLA提呈不同抗原,因此第2和第3外显子编码的α1和α2结构域构成的PBG多态性较高,对荣昌猪SLA-1编码蛋白的α1和α2结构域的氨基酸序列变异性进行分析,结果显示,有14个氨基酸位点的Wu-Kabat指数高于6.0,其中,第74个氨基酸指数最高,达到13.8(图 5A)。第2外显子区域的氨基酸变异程度高于第3外显子,且变异程度最高的位点位于第2外显子区域。此外,由于SLA-1*04:01分子所编码的蛋白结构已经解析[22],共存在33个关键氨基酸组成PBG的A~F 6个口袋,因此将获得的荣昌猪SLA-1的α1和α2结构域氨基酸序列与SLA-1*04:01进行比对分析,结果显示,182个氨基酸位点中,121个位点完全保守,而33个关键氨基酸位点中,11个氨基酸位点完全保守,分别为Leu5(L)、Tyr7(Y)、Met45(M)、Tyr59(Y)、Leu81(L)、Tyr84(Y)、Tyr123(Y)、Lys146(K)、Tyr159(Y)、Leu160(L)、Tyr171(Y),其余22个位点均发生高频率突变(图 5B)。比对HLA-A*02:01、HLA-A*11:01与荣昌猪SLA-1等位基因氨基酸序列,结果显示,在与β2-微球蛋白(β2-microglobulin,β2 m)结合的19个氨基酸位点中,有10个氨基酸在人HLA-A与荣昌猪SLA-1等位基因中保持一致,分别是Tyr7(Y)、Phe8(F)、Val25(V)、Tyr27(Y)、Phe33(F)、Phe36(F)、His93(H)、Leu110(L)、Gly112(G)和Gln115(Q)。另外,比较人和荣昌猪CD8分子与MHC结合的7个关键氨基酸位点(223~229)[23],只有225(T/S)和228(T/M)两个位点存在差异,其它位点高度保守(表 4)。二级结构分析发现,荣昌猪不同SLA-1等位基因显示出SLA-1基因的典型二级结构,α1和α2区主要为α-螺旋和β-折叠,α1区具有1个α-螺旋和4个β-折叠,α2区具有4个α-螺旋和4个β-折叠(图 5B)。
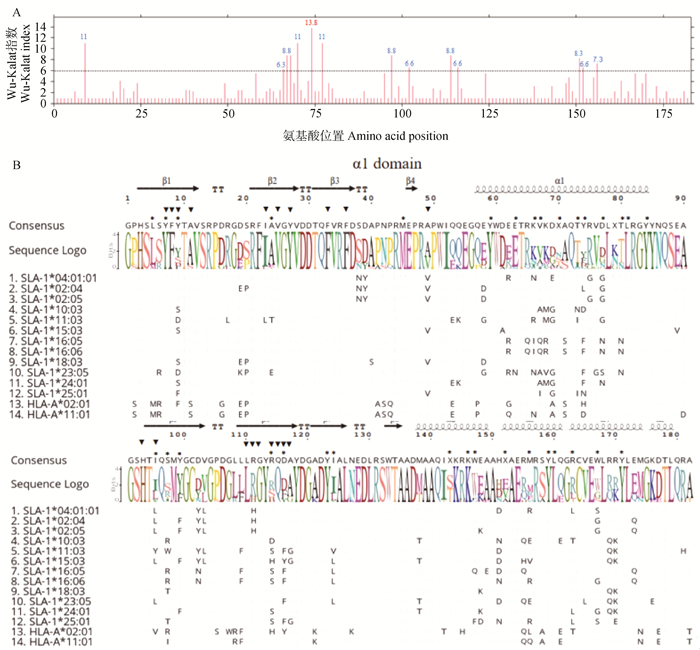
|
▼.人白细胞抗原(HLA)与β2-微球蛋白(β2m)结合位点,*.SLA-1*04:01等位基因抗原肽结合槽关键位点 ▼. Amino acid position binding human leukocyte antigen (HLA) and β2-microglobulin (β2m), *. Key amino acid position of PBG located in SLA-1*04:01 allele 图 5 荣昌猪SLA-1等位基因第2和第3外显子区域氨基酸变异性(A)和氨基酸序列比对(B) Fig. 5 The amino acid variability (A) and comparison (B) of exon 2 and exon 3 of SLA-1 alleles in Rongchang pig |
|
|
表 4 人和荣昌猪CD8分子与MHC结合的关键氨基酸氨基酸位点比较 Table 4 Comparison of key amino acid position of human and Rongchang pig CD8 molecules combine with MHC |
荣昌猪不同SLA-1等位基因的核苷酸相似性为93.6%~99.4%,氨基酸相似性为86.5%~98.3%,其中,SLA-1*02:04和SLA-1*02:05同源性最高,核苷酸和氨基酸相似性分别为99.4%和98.3%,二者存在7个核苷酸变异和6个氨基酸变异。荣昌猪与IPD-MHC数据库中已提交的69个具有CDS全长序列的SLA-1等位基因相似性分析显示,核苷酸序列和氨基酸序列分别为92.6%~99.9%和85.9%~99.4%。荣昌猪SLA-1等位基因和人群中流行的HLA-A*02:01和HLA-A*11:01等位基因的核苷酸相似性为83.3%~86.8%,氨基酸相似性为70.3%~76.9%,其中,与HLA-A*02:01相似性最高的等位基因是SLA-1*10:03(核苷酸85.0%,氨基酸74.4%),与HLA-A*11:01相似性较高的等位基因是SLA-1*18:03(核苷酸86.8%,氨基酸76.9%)(表 5)。
|
|
表 5 荣昌猪SLA-1和IPD-MHC数据库中提交的SLA-1以及人HLA-A流行等位基因的相似性比较 Table 5 Comparison of nucleotide and amino acid homology among SLA-1 alleles of Rongchang pig, SLA-1 alleles submitted in IPD-MHC database and frequent human HLA-A alleles |
从IPD-MHC数据库中选取与荣昌猪SLA-1等位基因核苷酸相似性较高(93.3%~99.9%)的31个具有CDS全长的SLA-1等位基因,与荣昌猪获得的11个SLA-1等位基因序列比对,以人群中HLA-A频率最高的HLA-A*02:01和HLA-A*11:01等位基因为外群对照,利用邻接法构建系统进化树(图 6)。结果显示,荣昌猪中9个新等位基因分布在不同聚类分支中,根据系统进化树,ISAG SLA命名委员会对其进行了命名。SLA-1*02:04和SLA-1*02:05聚在02等位基因组分支中,SLA-1*02:05与SLA-1*02:02具有3个核苷酸差异,SLA-1*02:04与SLA-1*02:01具有4个核苷酸差异,与多种猪品种和猪细胞系亲缘关系较近,如:辛克莱小型猪、NIH小型猪和小型猪肾细胞系等。SLA-1*10:03等位基因与汉福德、大花白以及融水小型猪中获得的SLA-1*10:01等位基因聚于同一分支,与SLA-1*10:01具有5个核苷酸差异。SLA-1*16:05和SLA-1*16:06遗传距离较近,与SLA-1*16:03聚为一支,属于16等位基因组,特别是SLA-1*16:05与SLA-1*16:03仅有一个核苷酸差异,两个等位基因均与亚洲野猪的亲缘关系较高。SLA-1*18:03与SLA-1*18:01聚于同一分支。SLA-1*23:05独立聚为一支,表明有特定的肽结合基序,而且与SLA-1*11:10和SLA-1*23:01聚为一大类分支,表明与巴马小型猪亲缘关系较近。SLA-1*24:01与亚洲野猪、巴马小型猪中获得的SLA-1*19:01聚于一大支,表明荣昌猪和其亲缘关系较近。SLA-1*25:01独立分为一支,表明也具有独特的肽结合基序。此外,已知的SLA-1*11:03和SLA-1*15:03等位基因在韩国地方猪、中国融水小型猪和皖南花猪也存在,表明这些品种猪可能有相同血缘关系的祖先。
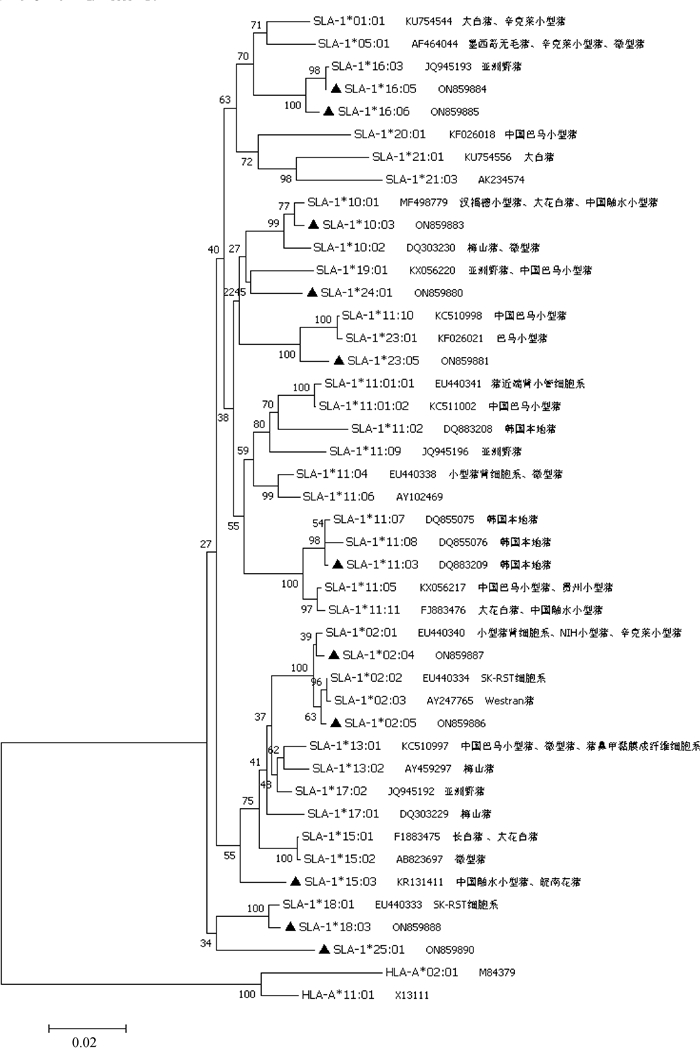
|
▲.荣昌猪SLA-1等位基因 ▲. SLA-1 alleles of Rongchang pig 图 6 荣昌猪SLA-1等位基因系统发育树 Fig. 6 Phylogenetic tree of SLA-1 allele from Rongchang pig |
实验猪在畜牧兽医生产实践中发挥重要作用,被广泛用于研究病原致病机制、T细胞表位识别和疫苗评估等,在这些研究中SLA对抗原的识别具有严格的MHC限制性,从而引起个体差异[24-27]。因此鉴定SLA基因可减少实验动物遗传背景差异对各项研究结果的影响。经典的SLA Ⅰ类基因SLA-1编码的Ⅰ类分子主要参与内源性抗原递呈,识别抗原并诱导细胞免疫应答。SLA-1基因具有高度多态性,目前IPD-MHC数据库共收录了89个SLA-1等位基因(Release 3.9.0.1 07/2022 build 209),存在于不同的猪品种或猪细胞系。荣昌猪是我国优良的地方猪品种,已作为实验动物在动物医学模型、药物评价模型和异种器官供体等研究中广泛应用,但目前尚未对荣昌猪的SLA-1基因进行系统鉴定,本研究在荣昌猪SLA-1基因座上共获得11个等位基因,其中,9个为新等位基因,2个为已知等位基因,在现有群体数量有限的情况下SLA-1基因座表现出较高的多态性。
荣昌猪SLA-1基因也发现了双拷贝现象。在之前的研究中,有学者发现SLA-1基因座双拷贝现象在韩国本地猪、SNU小型猪、NIH小型猪、巴克夏猪、约克夏猪、杜洛克猪、长白猪、梅山猪和巴马小型猪等众多猪品种中均存在,且部分双拷贝现象在某些SLA单倍型中持续存在[28-31]。一般认为,基因多样性或杂合度的增加有利于机体抵御不良环境,但无论是不同SLA基因座的等位基因杂合还是SLA-1基因座双拷贝杂合,在病原体和SLA等位基因之间的关系都是较为复杂的。首先,在同一个SLA基因座存在多个等位基因可能会增加其对不同抗原的结合能力,从而提高生存概率;其次,如果在群体中不存在对特定病原敏感的特定等位基因,即便杂合度很高,该种群对于病原的抵抗力依然较弱,而存在特定等位基因的纯合子群体却能够获得高的抵抗力,这可以从杂合子优势假说以及稀有等位基因优势假说得到解释[32-33]。
SLA-1基因的第2和第3外显子所编码的抗原肽结合槽相较于其它区域具有非常高的多态性,这种现象在不同猪品种中都存在[34-36]。本研究对荣昌猪SLA-1等位基因的DNA多态性进行了分析,从核苷酸多态性位点数量占比和非同义核苷酸多态性位点数量的比例发现,第2和第3外显子区域都表现出绝对的优势。氨基酸变异位点数量以及氨基酸变异系数和位置与核苷酸多态性具有相似性,同样证明SLA-1基因的高度多态区域集中于第2和第3外显子。
SLA Ⅰ类分子主要通过递呈病原的抗原肽给CD8+T淋巴细胞识别[37],继而诱导细胞杀伤效应清除感染性细胞。抗原肽是通过结合SLA Ⅰ类分子PBG中的6个口袋(A~F),然后与β2 m形成复合物递呈在细胞表面供T细胞识别[38-39]。因此不同等位基因和递呈抗原肽的差异会对PBG的立体结构造成影响,利用已解析的SLA-1*04:01的三维结构,对荣昌猪SLA-1等位基因6个口袋的关键氨基酸进行比对,发现有较高的差异性,提示获得的等位基因可能展现出与SLA-1*04:01不同的三维结构。
猪已被选定是最佳的异种移植供体,但猪-人异种器官/细胞移植主要障碍是人体对植入外源器官的免疫反应,其中SLA是与同种和异种免疫识别密切相关的一个重要分子标记基因,能被各种人类免疫细胞亚群直接识别[40]。因此异种移植成功与否的前提在于猪SLA与人HLA之间的适配性所导致的急性或慢性免疫排斥反应能否得到解决。比较荣昌猪SLA-1与β2 m结合以及人和荣昌猪CD8分子与MHC结合的关键氨基酸位点,发现氨基酸位点保守性较高。将获得的SLA-1等位基因与人群体中频率较高的等位基因进行同源性分析,显示SLA-1*10:03和SLA-1*18:03相似性最高,预示着携带这两个等位基因的猪可能和人之间存在较高的适配性。
目前,IPD-MHC数据库已收录了世界各类猪种以及猪细胞系的SLA-1等位基因,选取与荣昌猪相似性较高的SLA-1等位基因建立了系统进化树,发现荣昌猪种群中获得的SLA-1*15:03和SLA-1*11:03也分别存在于中国融水小型猪、皖南花猪以及韩国本地猪中,这些猪种起初都源自亚洲。获得的新SLA-1等位基因少部分与各类猪源细胞系亲缘性较高,大多数均与亚洲猪种亲缘性较高,因此可推测荣昌猪可能和这些猪种存在血缘相同的祖先。
本研究获得的荣昌猪SLA-1等位基因遗传信息仅针对于饲养于屏障设施中这个小型封闭群,并不能代表全国整个荣昌猪品种的群体遗传信息,但是对标准化实验荣昌猪的培育和推广应用必不可少。此外在荣昌猪上获得的SLA-1基因也可用于深入研究病原的抗原递呈机制,筛选猪重要病原的SLA限制性T细胞表位,对开发安全有效的表位肽疫苗意义重大。
4 结论本研究通过对荣昌猪SLA-1基因CDS区域进行PCR扩增测序,成功获得了11个SLA-1等位基因,其中,9个为新发现等位基因,SLA-1*24:01是该荣昌猪群体中最流行的等位基因。SLA-1基因多态性主要集中在第2和第3外显子区域,PBG中的33个关键氨基酸位点有11个位点完全保守;与β2 m结合的19个关键氨基酸位点有10个位点在人与荣昌猪之间保持一致,CD8分子与MHC结合的关键氨基酸位点高度保守,只有225(Thr/Ser)和228(Thr/Met)位点不同。同源性以及系统进化树表明,SLA-1*10:03和SLA-1*18:03分别与人HLA-A*02:01和HLA-A*11:01等位基因的同源性最高,荣昌猪与亚洲野猪、巴马小型猪和融水小型猪等亚洲猪种具有较近的亲缘关系。本研究结果为荣昌猪SLA遗传背景的研究以及抗原递呈过程中SLA与不同抗原肽的相互作用关系奠定基础。
| [1] |
马丽颖, 岳秉飞. 主要组织相容性复合体(MHC)研究进展[J]. 中国比较医学杂志, 2005, 15(3): 182-185. MA L Y, YUE B F. Advances in research on major histocompatibility complex[J]. Chinese Journal of Comparative Medicine, 2005, 15(3): 182-185. (in Chinese) |
| [2] |
刘子展. 贵州小型猪SLA经典Ⅰ类和Ⅱ类基因克隆和生物信息学分析及PCR-SSP分型方法的建立[D]. 合肥: 安徽农业大学, 2012. LIU Z Z. Cloning and bioinformatics analysis of swine leukocyte antigens class Ⅰ and class Ⅱ genes in Guizhou minipigs and establishment of PCR-SSP typing method[D]. Hefei: Anhui Agricultural University, 2012. (in Chinese) |
| [3] |
YAMAGUCHI T, DIJKSTRA J M. Major histocompatibility complex (MHC) genes and disease resistance in fish[J]. Cells, 2019, 8(4): 378. DOI:10.3390/cells8040378 |
| [4] |
CARRINGTON M, BONTROP R E. Effects of MHC class Ⅰ on HIV/SIV disease in primates[J]. AIDS, 2002, 16: S105-S114. DOI:10.1097/00002030-200216004-00015 |
| [5] |
ROCK K L, REITS E, NEEFJES J. Present Yourself! By MHC class Ⅰ and MHC class Ⅱ molecules[J]. Trends Immunol, 2016, 37(11): 724-737. DOI:10.1016/j.it.2016.08.010 |
| [6] |
SEETHARAM A, TIRIVEEDHI V, MOHANAKUMAR T. Alloimmunity and autoimmunity in chronic rejection[J]. Curr Opin Organ Transplant, 2010, 15(4): 531-536. DOI:10.1097/MOT.0b013e32833b31f4 |
| [7] |
AFZALI B, LOMBARDI G, LECHLER R I. Pathways of major histocompatibility complex allorecognition[J]. Curr Opin Organ Transplant, 2008, 13(4): 438-444. DOI:10.1097/MOT.0b013e328309ee31 |
| [8] |
WEISS R A. Xenotransplantation[J]. BMJ, 1998, 317(7163): 931-934. DOI:10.1136/bmj.317.7163.931 |
| [9] |
COOPER D K C, GASTON R, ECKHOFF D, et al. Xenotransplantation-the current status and prospects[J]. Br Med Bull, 2018, 125(1): 5-14. DOI:10.1093/bmb/ldx043 |
| [10] |
CELIS-GIRALDO C T, BOHÓRQUEZ M D, CAMARGO M, et al. A comparative analysis of SLA-DRB1 genetic diversity in Colombian (creoles and commercial line) and worldwide swine populations[J]. Sci Rep, 2021, 11(1): 4340. DOI:10.1038/s41598-021-83637-8 |
| [11] |
权金强, 江新杰, 李昌文, 等. 实验用SPF大白猪和长白猪SLA-1基因特征分析[J]. 农业生物技术学报, 2017, 25(5): 770-780. QUAN J Q, JIANG X J, LI C W, et al. Characterization analysis of SLA-1 gene from SPF yorkshire (Sus scrofa) and landrace[J]. Journal of Agricultural Biotechnology, 2017, 25(5): 770-780. (in Chinese) |
| [12] |
HAMMER S E, HO C S, ANDO A, et al. Importance of the major histocompatibility complex (Swine leukocyte antigen) in swine health and biomedical research[J]. Annu Rev Anim Biosci, 2020, 8: 171-198. DOI:10.1146/annurev-animal-020518-115014 |
| [13] |
GAO C X, HE X W, QUAN J Q, et al. Specificity characterization of SLA class Ⅰ molecules binding to swine-origin viral cytotoxic T lymphocyte epitope peptides in vitro[J]. Front Microbiol, 2017, 8: 2524. DOI:10.3389/fmicb.2017.02524 |
| [14] |
LUNNEY J K. Advances in swine biomedical model genomics[J]. Int J Biol Sci, 2007, 3(3): 179-184. |
| [15] |
SWINDLE M M, MAKIN A, HERRON A J, et al. Swine as models in biomedical research and toxicology testing[J]. Vet Pathol, 2012, 49(2): 344-356. DOI:10.1177/0300985811402846 |
| [16] |
GEISLER K, KVNZEL J, GRUNDTNER P, et al. The perfused swine uterus model: long-term perfusion[J]. Reprod Biol Endocrinol, 2012, 10: 110. DOI:10.1186/1477-7827-10-110 |
| [17] |
RAMIREZ-MEDINA E, VUONO E, RAI A, et al. Deletion of E184L, a putative DIVA target from the pandemic strain of african swine fever virus, produces a reduction in virulence and protection against virulent challenge[J]. J Virol, 2022, 96(1): e0141921. DOI:10.1128/JVI.01419-21 |
| [18] |
CHEN W, YI H J, ZHANG L, et al. Establishing the standard method of cochlear implant in Rongchang pig[J]. Acta Oto-Laryngol, 2017, 137(5): 503-510. DOI:10.1080/00016489.2016.1267406 |
| [19] |
任丽丽. 白化荣昌猪耳聋的分子病理机制研究[D]. 北京: 中国人民解放军医学院, 2013. REN L L. Probing the molecular pathological mechanism underlying the deafness in albino Rongchang swine[D]. Beijing: Chinese PLA General Hospital & Medical School, 2013. (in Chinese) |
| [20] |
陈伟, 陈磊, 杨仕明. 荣昌猪遗传性听力缺陷家系的发掘与应用[J]. 中华耳科学杂志, 2016, 14(1): 10-14. CHEN W, CHEN L, YANG S M. Excavation and application of the Rongchang pig hereditary pedigree connected to hearing defects[J]. Chinese Journal of Otology, 2016, 14(1): 10-14. (in Chinese) |
| [21] |
周福波. 汉族白癜风患者HLA-A*0201限制性CTL表位的筛查与鉴定[D]. 西安: 第四军医大学, 2014. ZHOU F B. Screening and identification of HLA-A*0201-restricted CTL epitope in Chinese vitiligo patients[D]. Xi'an: The Fourth Military Medical University, 2014. (in Chinese) |
| [22] |
ZHANG N Z, QI J X, FENG S J, et al. Crystal structure of swine major histocompatibility complex class Ⅰ SLA-1*0401 and identification of 2009 pandemic swine-origin influenza A H1N1 virus cytotoxic T lymphocyte epitope peptides[J]. J Virol, 2011, 85(22): 11709-11724. DOI:10.1128/JVI.05040-11 |
| [23] |
SALTER R D, BENJAMIN R J, WESLEY P K, et al. A binding site for the T-cell co-receptor CD8 on the α3 domain of HLA-A2[J]. Nature, 1990, 345(6270): 41-46. DOI:10.1038/345041a0 |
| [24] |
GAO F S, ZHAI X X, JIANG P, et al. Identification of two novel foot-and-mouth disease virus cytotoxic T lymphocyte epitopes that can bind six SLA-I proteins[J]. Gene, 2018, 653: 91-101. DOI:10.1016/j.gene.2018.02.025 |
| [25] |
FAN S H, WU Y N, WANG S, et al. Structural and biochemical analyses of swine major histocompatibility complex class Ⅰ complexes and prediction of the epitope map of important influenza a virus strains[J]. J Virol, 2016, 90(15): 6625-6641. DOI:10.1128/JVI.00119-16 |
| [26] |
PAN X C, ZHANG N Z, WEI X H, et al. Illumination of PRRSV cytotoxic T lymphocyte epitopes by the three-dimensional structure and peptidome of swine lymphocyte antigen class Ⅰ (SLA-I)[J]. Front Immunol, 2020, 10: 2995. DOI:10.3389/fimmu.2019.02995 |
| [27] |
李婧娜, 疏泽, 吴俊超, 等. 基于重测序的民猪与大白猪SLA Ⅰ类基因多态性及抗病潜能差异分析[J]. 畜牧兽医学报, 2021, 52(7): 1820-1830. LI J N, SHU Z, WU J C, et al. Analysis of SLA class Ⅰ gene polymorphism and potential disease resistance difference between Min pigs and Large White pigs based on resequencing[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(7): 1820-1830. (in Chinese) |
| [28] |
LUNNEY J K, HO C S, WYSOCKI M, et al. Molecular genetics of the swine major histocompatibility complex, the SLA complex[J]. Dev Comp Immunol, 2009, 33(3): 362-374. DOI:10.1016/j.dci.2008.07.002 |
| [29] |
GAO C X, JIANG Q, GUO D C, et al. Characterization of swine leukocyte antigen (SLA) polymorphism by sequence-based and PCR-SSP methods in Chinese Bama miniature pigs[J]. Dev Comp Immunol, 2014, 45(1): 87-96. DOI:10.1016/j.dci.2014.02.006 |
| [30] |
GAO C X, QUAN J Q, JIANG X J, et al. Swine leukocyte antigen diversity in Canadian specific pathogen-free Yorkshire and landrace pigs[J]. Front Immunol, 2017, 8: 282. |
| [31] |
SØRENSEN M R, ILSØE M, STRUBE M L, et al. Sequence-based genotyping of expressed swine leukocyte antigen class Ⅰ alleles by next-generation sequencing reveal novel swine leukocyte antigen class Ⅰ haplotypes and alleles in Belgian, Danish, and Kenyan fattening pigs and Göttingen minipigs[J]. Front Immunol, 2017, 8: 701. DOI:10.3389/fimmu.2017.00701 |
| [32] |
SOMMER S. The importance of immune gene variability (MHC) in evolutionary ecology and conservation[J]. Front Zool, 2005, 2: 16. DOI:10.1186/1742-9994-2-16 |
| [33] |
SPURGIN L G, RICHARDSON D S. How pathogens drive genetic diversity: MHC, mechanisms and misunderstandings[J]. Proc Biol Sci, 2010, 277(1684): 979-88. |
| [34] |
MACHUKA E M, MUIGAI A W T, AMIMO J O, et al. Comparative analysis of SLA-1, SLA-2, and DQB1 genetic diversity in locally-adapted Kenyan pigs and their wild relatives, warthogs[J]. Vet Sci, 2021, 8(9): 180. DOI:10.3390/vetsci8090180 |
| [35] |
GAO C X, XIN C, WANG X Y, et al. Molecular genetic characterization and haplotype diversity of swine leukocyte antigen in Chinese Rongshui miniature pigs[J]. Mol Immunol, 2019, 112: 215-222. DOI:10.1016/j.molimm.2019.05.009 |
| [36] |
FAN S H, WANG Y L, WANG S, et al. Polymorphism and peptide-binding specificities of porcine major histocompatibility complex (MHC) class Ⅰ molecules[J]. Mol Immunol, 2018, 93: 236-245. DOI:10.1016/j.molimm.2017.06.024 |
| [37] |
FAN S, WANG Y, WANG X, et al. Analysis of the affinity of influenza A virus protein epitopes for swine MHC I by a modified in vitro refolding method indicated cross-reactivity between swine and human MHC I specificities[J]. Immunogenetics, 2018, 70(10): 671-680. DOI:10.1007/s00251-018-1070-6 |
| [38] |
MADDEN D R. The three-dimensional structure of peptide-MHC complexes[J]. Annu Rev Immunol, 1995, 13: 587-622. DOI:10.1146/annurev.iy.13.040195.003103 |
| [39] |
YUE C, XIANG W Z, HUANG X W, et al. Mooring stone-like Arg114 pulls diverse bulged peptides: first insight into African swine fever virus-derived T cell epitopes presented by swine major histocompatibility complex class Ⅰ[J]. J Virol, 2022, 96(4): e0137821. DOI:10.1128/jvi.01378-21 |
| [40] |
COWAN P J, TECTOR A J. The resurgence of xenotransplantation[J]. Am J Transplant, 2017, 17(10): 2531-2536. DOI:10.1111/ajt.14311 |
(编辑 白永平)



