2. 广西大学动物科学技术学院, 南宁 530004
2. College of Animal Science and Technology, Guangxi University, Nanning 530004, China
禽传染性支气管炎(infectious bronchitis, IB)可引起养禽业严重的经济损失,禽传染性支气管炎病毒(infectious bronchitis virus, IBV)属于套式病毒目冠状病毒科Gamma-冠状病毒属的一员[1]。IBV为单股正链RNA病毒,全长约27.6 kb,编码4种结构蛋白:刺突(S)蛋白、小膜(E)蛋白、膜(M)蛋白和核衣壳(N)蛋白[2]。其中,S蛋白被蛋白酶切割成S1和S2两个亚蛋白[1]。由于S1蛋白含有决定血清型、病毒优势抗原位点等关键区域,大多数科研工作者均以S1蛋白作为靶对象进行研究[3-5]。然而,IBV的其它结构蛋白也具有重要的作用[6]。研究发现,N蛋白亦含有决定血清型的关键位点,其氨基酸的变异有可能导致IBV毒株血清型的改变[7]。此外,N蛋白是唯一位于膜内的蛋白,主要参与病毒RNA的合成、转录和翻译,在细胞免疫中起重要作用[8]。因此,为了更准确深入了解IBV毒株的遗传信息和分子变异机制,很有必要选择病毒相对保守的N基因作为研究对象进行分析。
基因重组是IBV甚至其它动物冠状病毒进化和免疫逃避的重要手段[9]。研究发现,IBV由于其RNA病毒的特性,病毒在转录复制的过程中缺乏矫正机制,极容易发生基因的变异和重组[8, 10]。目前,国内外很多研究只针对S1基因发生的重组进行分析,而IBV其它结构基因特别是N基因是否发生重组亦未可知。前期研究表明,国内IBV分离株已形成了有别于其它国家和地区独特的重组热点区域[2]。因此,基于N基因的重要性,很有必要进行国内所有IBV分离株N结构基因的重组分析,但目前国内相关研究还非常有限。
中国是世界上主要的养禽大国,禽的地方品种丰富、饲养模式多样,不同的免疫程序、不同日龄群体混养、放养模式等无疑会加大IB防控的难度[11]。由于IBV频繁的碱基突变与基因重组容易导致新基因毒株的不断产生,并且不同时间、不同地区的IBV流行特点也不相同,因此持续的流行病学分析对IB防控具有重要意义[10]。目前,国内学者进行IBV流行病学分析一般具有地域限制,分离株数量、分离株跨度时间以及分析手段均有限。此外,至今国内关于IBV的时空动态及传播路径的研究很有限,非常有必要对全国范围内所有的IBV毒株进行长期、深入、系统的流行病学分析。
鉴于此,本研究筛选具有完整毒株分离信息的1985—2022年国内所有IBV分离株(217株)N结构基因序列,分别进行系统进化树、基因重组、毒株溯源、谱系地理及种群动态历史等分析,旨在全面了解国内IBV N结构基因的遗传进化、起源及时空传播动态,为全国乃至世界范围内的IBV防控提供更有针对性的策略,也可以为其它动物冠状病毒甚至新型冠状病毒(SARS-CoV-2)的防控提供参考。
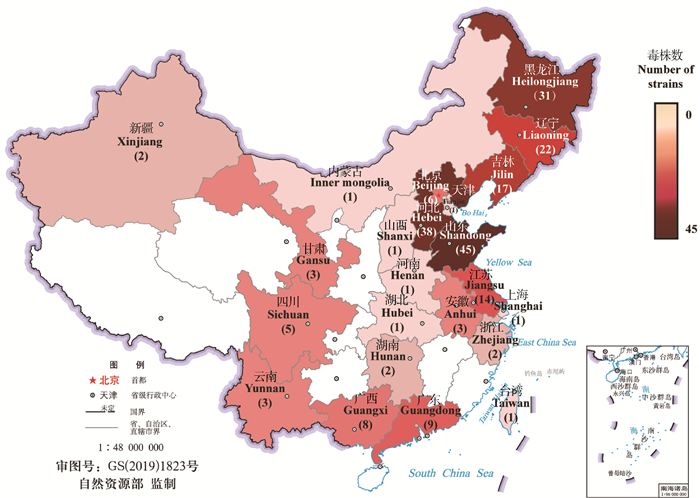
1 材料和方法 1.1 中国分离株N基因序列来源及背景基于GenBank数据库(https://www.ncbi.nlm.nih.gov)选取截至2022年9月30日上传的所有国内IBV分离株的全基因组序列,剔除分离年份或分离地点(省份)不明确的毒株,共获得217株IBV全基因组序列。国家地图为标准地图服务系统(http://bzdt.ch.mnr.gov.cn/)的标准地图,将217株国内IBV分离株在地图上展示其具体分离信息(图 1)。分析所用的IBV N结构基因序列均来源于全基因的截取(IBV分离株的GenBank登录号、分离年份、分离地点等详细信息见图 2)。32株IBV参考株包括常用疫苗株、国际上主要血清型的毒株以及国内代表毒株[2]。
|
每一个省份或直辖市(自治区)显示毒株数,从毒株最多(45,深红色)至毒株最少(0,白色) Each province or autonomous city (zone) shows the number of isolates, from most cases (45, dark red) to no case (0, white) 图 1 217株IBV分离株在中国的分布图 Fig. 1 Distribution of 217 IBV isolates in China |
|
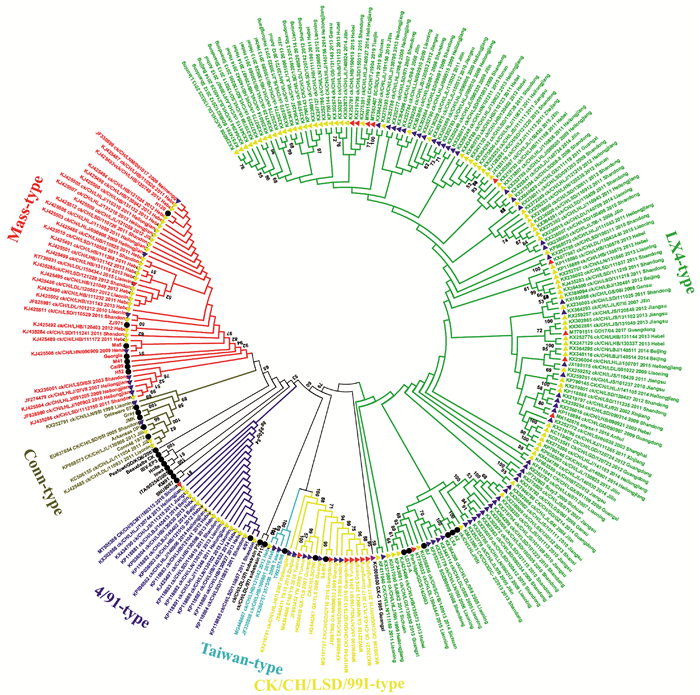
每种基因型IBV以不同颜色标记。32株IBV参考株以●标记。1985—1998年的1株IBV分离株以 标记。1999—2010年的69株IBV分离株以 标记。1999—2010年的69株IBV分离株以 标记。2011—2014年的129株IBV分离株以 标记。2011—2014年的129株IBV分离株以 标记。2015—2022年18株IBV分离株以 标记。2015—2022年18株IBV分离株以 标记
Each type of IBV was highlighted in different color. The 32 IBV reference strains were marked with filled ●. One IBV strain isolated during 1985-1998 was marked with filled 标记
Each type of IBV was highlighted in different color. The 32 IBV reference strains were marked with filled ●. One IBV strain isolated during 1985-1998 was marked with filled  . Sixty-nine IBV strains isolated during 1999-2010 were marked with filled . Sixty-nine IBV strains isolated during 1999-2010 were marked with filled  . One hundred and twenty-nine IBV strains isolated during 2011-2014 were marked with filled . One hundred and twenty-nine IBV strains isolated during 2011-2014 were marked with filled  and eighteen IBV strains isolated during 2015-2022 were marked with filled and eighteen IBV strains isolated during 2015-2022 were marked with filled  图 2 217株IBV分离株与32株IBV参考株的N基因遗传进化树分析
Fig. 2
Phylogenetic trees of the N gene between 217 IBV isolates and 32 IBV reference strains
图 2 217株IBV分离株与32株IBV参考株的N基因遗传进化树分析
Fig. 2
Phylogenetic trees of the N gene between 217 IBV isolates and 32 IBV reference strains
|
将217株国内IBV分离株和32株参考株的N结构基因序列应用在线生物软件Multiple Alignment with Fast Fourier Transformation (MAFFT),网址:https://mafft.cbrc.-jp/alignment/server/进行比对[12]。应用软件DNAStar 7.1进行核苷酸序列相似性分析,应用软件MEGA 6.0的邻接法(Neighbor-Joining, NJ)进行系统进化树构建,设置bootstrap值为1 000[13]。
1.3 IBV N基因选择压力分析应用在线生物软件Datamonkey (http://www.datamonkey.org/)的单一祖先计数方法Single-likelihood ancestor counting (SLAC)、固定效应似然模型Fixed effects likelihood (FEL)以及混合效应进化模型方法Mixed effects model of evolution (MEME)进行选择压力分析[6]。3种方法均检测到的氨基酸位点同为阳性或阴性证明有效。当dN(非同义置换率)/dS(同义置换率)>1时,为正向选择,反之则为净化选择。
1.4 IBV N基因重组分析应用重组分析软件Recombination Detection Program (RDP 4.95)对N结构基因进行重组分析。RDP 4.95软件的7种检测方法:RDP、GENECONV、Bootscan、MaxChi、Chimera、SiScan及3Seq,至少4种检测方法可检测到重组断点且P值<10-6证明检测结果可信[6]。应用另一重组分析软件SimPlot 3.5.1进行进一步验证,两种重组软件分析结果相一致证明重组分析结果准确无误[6]。
1.5 IBV N基因分子进化速率分析将经过核苷酸序列比对后的217株IBV N结构基因序列,应用软件jModel Test 2.1.7进行基于88种模型的最佳模型筛选[14],筛选的最佳模型结果:GTR+G+I。应用软件TempEst v1.5.1对217株IBV毒株的N结构基因序列进行R值相关系数矫正,筛选序列信息处于合理区间的数据进行进一步分析[15]。应用BEAST v1.10.4版本进行贝叶斯树构建,选择严格分子钟(strict clock)模型,采用基于蒙特卡洛马尔科夫链(Markov Chain Monte Carlo, MCMC)统计算法对N结构基因进行运算,以运算链的最终有效样本量(effective sample size, ESS)>200为标准进行调整,运算代次为2亿代次[2, 16]。参数设置完毕后,运行BEAST v1.10.4软件生成.xml文件,应用Tracer v1.7.1进行各项参数分析,并确认N结构基因的分子进化速率值[17]。
1.6 IBV N基因进化起源及种群动态分析应用TreeStar v1.10.4将上述运算结果生成Trees文件,剔除10%的抽样老化树枝,运行并生成最大分支置信树(maximum clade credibility tree, MCC Tree),通过FigTree v1.4.3软件查看MCC树,进行最近共同祖先(the most recent common ancestor, tMRCA)分析[15]。应用BEAST v1.10.4软件进行N结构基因的贝叶斯天际线图(Bayesian skyline plots, BSP)分析,应用Tracer v1.7.1软件绘制贝叶斯天际线图[17]。
1.7 IBV N基因贝叶斯谱系地理学分析应用BEAST v1.10.4软件的贝叶斯随机搜索变量选择(Bayesian Stochastic Search Variable Selection, BSSVS)模型对N结构基因的MCC树每个节点的可能地域进行评估[16]。将Tree Annotator软件生成的文件导入SpreaD3 v0.9.7软件,采用贝叶斯因子(Bayes Factor, BF)以及后验概率值(posterior probability, PP)对生成的传播路径进行验证,当BF>6且PP>5时证明可信[2, 18]。在SpreaD3 v0.9.7软件中导出文件,将分析结果在地图上展示,以此模拟中国IBV的时空传播动态。
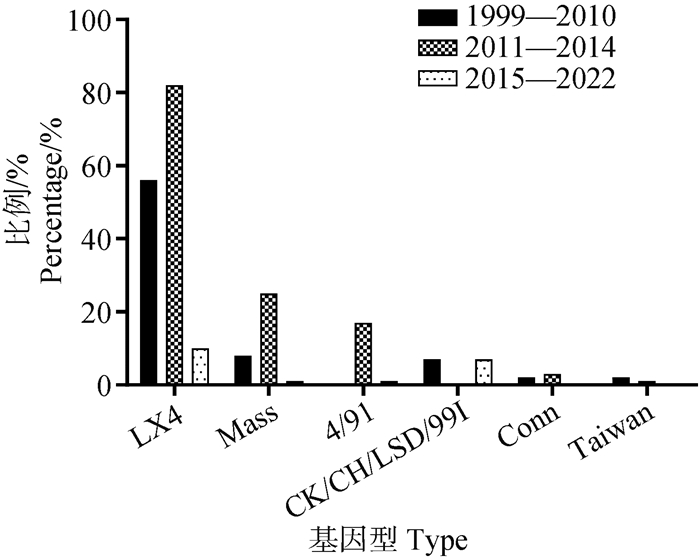
2 结果 2.1 IBV N基因序列的相似性分析和系统进化树构建相似性分析结果显示,217株国内IBV分离株N结构基因的核苷酸序列与参考株相似性为84.8%~100%。217株IBV分离株N基因与常用疫苗株H120、4/91、LDT3-A的核苷酸相似性分别为84.8%~100%、84.7%~100%、84.9%~100%;与上述疫苗株比较,N基因存在广泛的氨基酸突变。系统进化树分析结果显示,国内217株IBV分离株(除GX-C外)分属LX4 (65.44%, 142/217)、Mass (15.67%, 34/217)、4/91 (8.29%, 18/217)、CK/CH/LSC/99I (6.45%, 14/217)、Conn (2.31%, 5/217)和Taiwan (1.38%, 3/217)共6种基因型(图 2)。其中,LX4为优势基因型,Mass以及4/91分别为第二和第三大基因型(图 3)。
|
1985—1998年只有1株IBV毒株(GX-C)分离到,且该毒株与其它基因型毒株同源性均很低,属于单独一群 There was only one IBV strain (GX-C) isolated during 1985-1998, which showed considerable low homology with the above other genotypes and belonged to a separate group 图 3 IBV分离株N基因在不同时间段基因型的占比 Fig. 3 The percentages of different genotypes based on the N gene of IBV isolates in different years |
选择压力分析结果显示,217株IBV N结构基因的dN/dS值:0.159 (dN/dS<1),表明N基因处于净化选择压力,承受外界免疫压力下整体处于抑制状态以自身进化为主;但存在11个正向选择位点(表 1)。
|
|
表 1 217株IBV分离株N结构蛋白的选择压力 Table 1 The selection profiles of N protein of 217 IBV isolates |
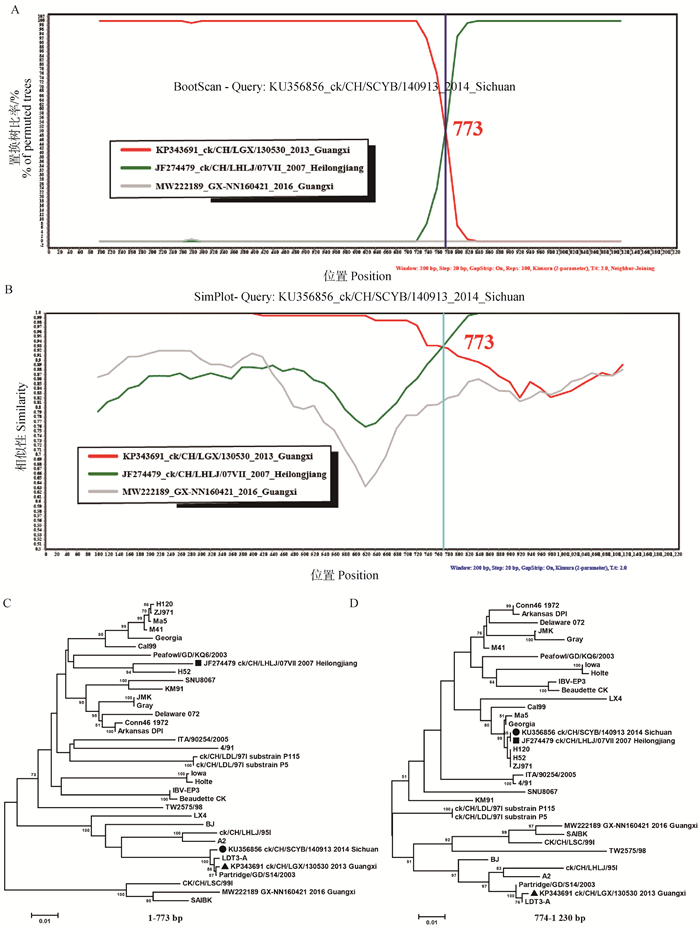
应用RDP 4.95和Simplot 3.5.1两种重组分析软件对国内217株IBV N基因进行重组分析,两种重组分析软件的结果相一致证明结果可信。分析结果显示,2014年, 在四川省分离的LX4型IBV分离株ck/CH/SCYB/140913 (GenBank登录号:KU356856)发生了重组。该毒株N基因来源于2013年LX4型广西分离株ck/CH/LGX/130530(主亲本)与2007年Mass型黑龙江分离株ck/CH/LHLJ/07VII(次亲本)之间的重组(图 4)。其中,RDP 4.95软件分析计算的RDP、GENECONV、Bootscan、MaxChi、Chimaera、3Seq的P值分别达到1.844×10-19、1.150×10-16、1.857×10-19、2.560×10-12、2.545×10-12和1.248×10-34。该分离株ck/CH/SCYB/140913的重组区域为N基因的C-端部分(773—1 230 bp),重组区域和Mass型黑龙江分离株ck/CH/LHLJ/07VII相似性达到99.8%;其它区域的N基因序列与主亲本LX4型广西分离株ck/CH/LGX/130530的相似性则达到99.9%。
|
A. BootScan分析;B. SimPlot相似性分析;C. 重组区域构建的进化树;D. 非重组区域构建的进化树;●为重组毒株,▲为主亲本毒株,■为次亲本毒株 A. BootScan analysis; B. SimPlot similarity analysis; C. Phylogenetic tree for recombinant regions; D. Phylogenetic tree for non-recombinant regions. ●. Potential recombinant, ▲. Potential major parent, ■. Potential minor parent 图 4 1株四川IBV分离株N基因的重组分析 Fig. 4 Recombination analysis of one Sichuan IBV isolates in N gene |
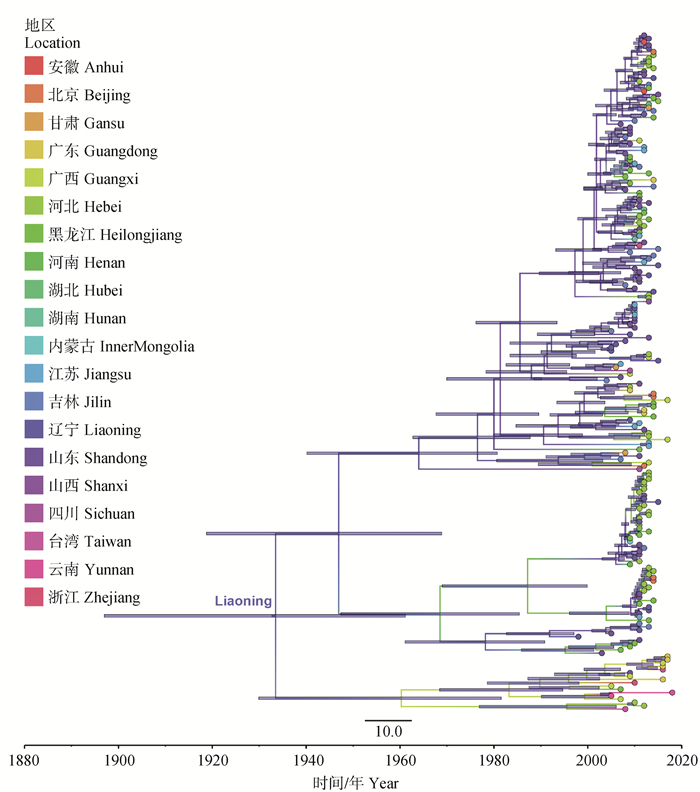
分子进化速率分析结果显示,IBV毒株N基因的分子进化速率为2.00×10-3 (95%置信区间:1.50×10-3~2.55×10-3)。最大分支置信树分析结果显示,国内IBV分离株最可能起源(tMRCA)时间为1 931.19年(95%置信区间:1 896.99~1 961.16);最可能起源地为辽宁省(图 5)。
|
图 5 IBV分离株的分子钟系统进化树 Fig. 5 The maximum clade credibility tree of IBV isolates |
居群动力学重构描述了IBV的5个流行生长期(图 6):20世纪50年代以前,IBV毒株的遗传多样性较为平稳,未发现明显的基因变异(缓慢增长期);在1950—2000年毒株的遗传多样性上升趋势明显且在2000年达到高峰(快速增长期);2000—2006年毒株的遗传多样性趋于稳定(平稳期);2006—2008年毒株的遗传多样性突然急剧下降(急速下降期);到了2008年末毒株的遗传多样性再次呈现上升趋势并趋于平稳(恒定增长期)。提示在1950—2006年IBV感染宿主的居群数量显著增多并呈现指数级增长,从而导致IBV遗传多样性大幅提高;但在2006—2008年IBV遗传变异频率突然下降,在2008年以后,开始处于稳定的遗传变异水平。

|
图 6 IBV分离株居群动力学分析结果 Fig. 6 Population genetic dynamics of IBV isolates |
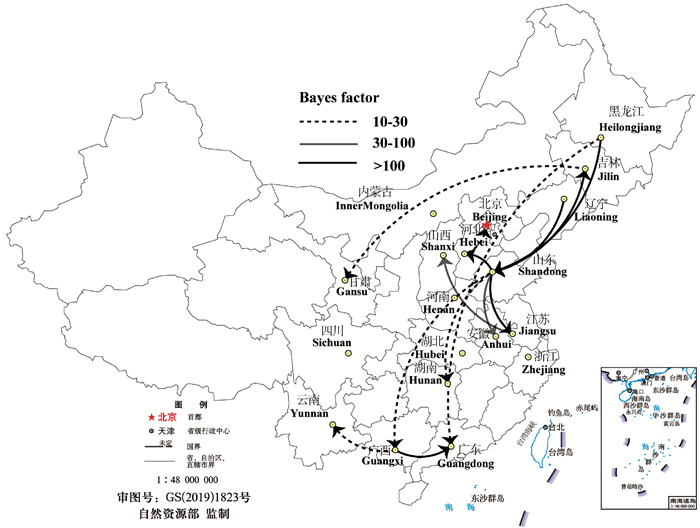
基于国内IBV分离株N结构基因组序列应用spreaD3 v0.9.7软件重塑IBV时空传播路径。通过贝叶斯因子(BF>6)对传播路径进行检验,图 7及表 2分析结果显示,中国IBV传播路径总体趋势为北方传播到南方。共形成5条主要的传播路径:第一条传播路径为东北地区的辽宁、黑龙江传播到山东(BF>100);第二条传播路径为华东地区的山东传播到吉林、河北和江苏(BF>100);第三条传播路径为山东传播到安徽、江苏传播到山西(BF>30);第四条传播路径为华南地区的广西传播到广东(BF>100)、广西传播到云南(BF>10);第五条传播路径属于长距离传播,从东北地区的黑龙江传播到华南地区的广东、山东传播到广西、湖南以及吉林传播到甘肃(BF>10)。第六条传播路径属于短距离传播,从河北传播到北京(BF>10)。此外,山东传播到河北、山东传播到吉林以及山东传播到江苏的贝叶斯因子值均超过10 000,推测山东省最可能成为IBV在中国各省区传播过程的毒株来源库。随后,以山东为中心快速散播到其它省份,由此导致了IBV在中国的流行(图 7)。
|
图 7 基于贝叶斯因子(BF)检验的IBV分离株区域传播分析图 Fig. 7 Map showing the regional transmission analysis for IBV isolates based on Bayes factor (BF) test |
|
|
表 2 217株IBV分离株N基因的贝叶斯因子检验 Table 2 Bayes Factor test for N gene of 217 IBV isolates among geographical regions |
目前,中国已成为世界主要的家禽生产和消费大国[11]。尽管Mass型(H120、H52、Ma5、28/86、M41和W93)、4/91型(NNA)、LDT3-A、QXL87等疫苗已在养殖场广泛使用,但IB问题依然困扰养殖场[2-4, 6, 10]。为了全面深入了解我国IBV N基因的遗传变异趋势以及时空传播动态,本研究对1985—2022年全国共217株IBV N结构基因序列进行了跨度37年的流行病学分析。
系统进化树分析结果显示,IBV N结构基因分为6个基因型。其中,LX4型、Mass型、4/91型分列优势基因型的第1~3位。Valastro等[19]基于IBV S1基因的系统进化树分析结果,将国际上IBV分为6大类共32个谱系(基因型)。前期研究表明,基于S1基因分析结果国内IBV呈现LX4型(GI-19,约57%)占据优势地位,Mass型(GI-1,约17%)次之,4/91型(GI-13,约11%)占据第3位,共分为7个谱系(基因型)的情况[2]。本研究结果与前期研究基于S1基因序列的分析结果相近而不完全相同。因此,基于N基因的系统进化树分析亦可真实反映IBV的基因型流行情况。此外,本研究结果显示IBV的N基因相对保守且处于净化选择压力下,但亦存在若干正向选择位点。正向选择位点对RNA病毒的结合活性至关重要[20]。N基因的研究不仅有利于深入了解N蛋白的相关功能,而且有助于重建冠状病毒的进化路径[21]。因此,本研究选择相对保守但具有重要生物学意义的N基因进行相关生物信息学分析。
IBV容易发生基因重组,不仅可造成新变异株的出现而且可引起抗原位点的漂移和变换,导致免疫失败的发生[9, 22-23]。重组分析结果显示,1株2014年分离的四川IBV分离株ck/CH/SCYB/140913在N基因发生了重组(次亲本为Mass型毒株)。前期研究结果显示,该毒株在非结构蛋白区域和S基因亦发生了基因重组,且次亲本亦为Mass型毒株[2]。因此,Mass型毒株(众多商品活毒疫苗株)参与基因重组的现象需引起人们足够的重视。Kuo等[20-21]基于台湾分离株N基因分析发现,台湾分离株N基因亦出现基因重组现象,重组不仅驱动了IBV种群进化导致新毒株种群的出现,而且增强了毒株的适应能力。因此,该四川分离株频繁的基因重组现象是否会导致其致病性的改变和免疫逃避现象的发生同样值得我们关注。
目前,基于贝叶斯理论的分析技术已在SARS-CoV-2等冠状病毒的溯源及时空传播方面应用广泛[24],但该技术在家禽的疾病防控及监测中暂未得到广泛的应用。贝叶斯最大分支置信树分析结果显示,国内IBV最可能起源于20世纪30年代初的辽宁省。因此,本研究与前期研究基于全基因组序列和S1基因序列的分析结果相比呈现溯源地相同、溯源时间相近而不完全一致的结果[2]。提示不同的结构基因片段可能具有不同的遗传进化史。
研究表明,禽流感病毒的传播路径与家禽的贸易路线高度吻合[25]。有趣的是,本研究发现IBV的传播路径也与家禽的贸易路线高度吻合。基于IBV的传播路径分析显示,国内IBV的传播表现出地理传播的连续性。研究表明,禽流感病毒的谱系传播与各省份的家禽贸易量以及候鸟迁徙密度呈正相关;与公路运输的距离呈负相关[25]。本研究结果显示,国内IBV的传播路径与活禽交易网络呈正相关,贸易越密集、病毒输入与输出越频繁、贝叶斯因子值越大、分析的传播路径越可信。因此,可以得出以下结论:国内IBV的传播主要是基于活禽的贸易和运输进行传播;而长距离传播路径(黑龙江传播到广东、山东传播到广西以及吉林传播到甘肃等)极有可能是通过候鸟的迁徙进行传播。结合前期研究[2]、本研究的贝叶斯分析结果和中国实际情况,推测国内IBV未来相当长一段时间内LX4型IBV将依然占据优势基因型地位,但其占比会逐渐缩小,而其它基因型(如Taiwan型等)毒株占比将逐渐增加;此外,随着频繁的贸易往来LX4型IBV已呈现浪潮式向中东、欧洲以及非洲传播并将极有可能成为上述地区的优势基因型。本研究运用的贝叶斯谱系地理学分析方法有助于科研工作者准确了解国内IBV的时空传播动态,对IBV的防控具有重要的意义。当然,本研究也有一定的局限性,不同省份、不同时间段的IBV分离株数量并不均匀有可能限制了时空分析结果的准确性。
研究表明,病毒遗传变异的发生、维持与病毒种群动态大小的变化息息相关[26]。种群动态分析结果表明,20世纪50年代以前, 国内IBV遗传多样性较为平稳,但1950年以后遗传多样性显著上升,猜测可能与新中国成立后国民经济缓慢恢复,鸡群养殖量逐年上升有关。但在2006年以后病毒的遗传多样性急剧下降,可能与2005年在青海湖的候鸟暴发H5N1禽流感导致家禽养殖业遭受毁灭性打击,家禽养殖量急剧萎缩进而导致IBV感染宿主的居群数量逐渐减少有关[27]。有趣的是,2008年后IBV的种群动态出现显著上升。病毒种群动态的变化与疫苗的免疫与否息息相关[28-29]。因此,2008年IBV种群动态的上升,推测与现有疫苗不能有效防控在2008年后即成为优势基因型的LX4型IBV感染有关。2018年后政府相关部门批准了LX4型商品弱毒疫苗株QXL87的上市,但该疫苗的免疫对鸡群的保护效果如何暂未可知,本研究团队将长期持续地进行IBV流行病学监测。
4 结论基于N基因的分析结果表明国内IBV处于LX4型占优、多基因型并存的现状。基因重组分析结果显示,1株IBV毒株的N基因出现基因重组现象。国内IBV最可能起源于20世纪30年代初期的辽宁省。IBV毒株在国内存在多条传播途径且山东省最可能成为国内IBV的毒株来源库。本研究结果有助于人们深入了解国内IBV流行毒株N基因的演化和时空传播动态,也为我国IBV流行毒株的防控提供了更有针对性的策略。
| [1] |
CAVANAGH D. Coronavirus avian infectious bronchitis virus[J]. Vet Res, 2007, 38(2): 281-297. DOI:10.1051/vetres:2006055 |
| [2] |
FAN W S, CHEN J M, ZHANG Y, et al. Phylogenetic and spatiotemporal analyses of the complete genome sequences of avian coronavirus infectious bronchitis virus in China during 1985-2020:revealing coexistence of multiple transmission chains and the origin of LX4-type virus[J]. Front Microbiol, 2022, 13: 693196. DOI:10.3389/fmicb.2022.693196 |
| [3] |
LIAN J M, WANG Z X, XU Z Y, et al. Distribution and molecular characterization of avian infectious bronchitis virus in southern China[J]. Poult Sci, 2021, 100(7): 101169. DOI:10.1016/j.psj.2021.101169 |
| [4] |
YAN W J, QIU R B, WANG F Y, et al. Genetic and pathogenic characterization of a novel recombinant avian infectious bronchitis virus derived from GI-1, GI-13, GI-28, and GI-19 strains in Southwestern China[J]. Poult Sci, 2021, 100(7): 101210. DOI:10.1016/j.psj.2021.101210 |
| [5] |
VILLALOBOS-AGVERO R A, LEÓN B, ZAMORA-SANABRIA R, et al. Molecular characterization of the S1 gene in GI-17 and GI-13 type isolates of avian infectious bronchitis virus (IBV) in Costa Rica, from 2016 to 2019[J]. VirusDisease, 2022, 33(1): 84-95. DOI:10.1007/s13337-022-00762-2 |
| [6] |
FAN W S, TANG N, DONG Z H, et al. Genetic analysis of avian coronavirus infectious bronchitis virus in Yellow chickens in Southern China over the past decade: revealing the changes of genetic diversity, dominant genotypes, and selection pressure[J]. Viruses, 2019, 11(10): 898. DOI:10.3390/v11100898 |
| [7] |
FINGER P F, PEPE M S, DUMMER L A, et al. Combined use of ELISA and Western blot with recombinant N protein is a powerful tool for the immunodiagnosis of avian infectious bronchitis[J]. Virol J, 2018, 15(1): 189. DOI:10.1186/s12985-018-1096-2 |
| [8] |
范文胜, 唐宁, 董志华, 等. 一株鸭源传染性支气管炎病毒的分离鉴定及结构蛋白基因和血清型分析[J]. 微生物学报, 2019, 59(3): 523-532. FAN W S, TANG N, DONG Z H, et al. Identification and analysis of structural protein genes and serotype of an infectious bronchitis virus in ducks[J]. Acta Microbiologica Sinica, 2019, 59(3): 523-532. (in Chinese) |
| [9] |
THOR S W, HILT D A, KISSINGER J C, et al. Recombination in avian gamma-coronavirus infectious bronchitis virus[J]. Viruses, 2011, 3(9): 1777-1799. DOI:10.3390/v3091777 |
| [10] |
黄梦姣, 张芸, 薛春宜, 等. 应对日益严峻的挑战: 中国禽传染性支气管炎研究[J]. 微生物学通报, 2019, 46(7): 1837-1849. HUANG M J, ZHANG Y, XUE C Y, et al. To meet the growing challenge: research of avian infectious bronchitis in China[J]. Microbiology China, 2019, 46(7): 1837-1849. (in Chinese) |
| [11] |
韦平. 当前优质鸡产业面临的挑战与发展机遇——论优质鸡产业发展九大议题[J]. 中国家禽, 2019, 41(11): 1-6. WEI P. Challenge and development opportunity of quality chicken industry in China: a review about 9 issues on the topic[J]. China Poultry, 2019, 41(11): 1-6. (in Chinese) |
| [12] |
KATOH K, STANDLEY D M. MAFFT multiple sequence alignment software version 7:improvements in performance and usability[J]. Mol Biol Evol, 2013, 30(4): 772-780. DOI:10.1093/molbev/mst010 |
| [13] |
TAMURA K, STECHER G, PETERSON D, et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [14] |
DARRIBA D, TABOADA G L, DOALLO R, et al. jModelTest 2:more models, new heuristics and parallel computing[J]. Nat Methods, 2012, 9(8): 772. |
| [15] |
DENG Q M, SHI M Y, LI Q H, et al. Analysis of the evolution and transmission dynamics of the field MDV in China during the years 1995-2020, indicating the emergence of a unique cluster with the molecular characteristics of vv+ MDV that has become endemic in southern China[J]. Transbound Emerg Dis, 2021, 68(6): 3574-3587. DOI:10.1111/tbed.13965 |
| [16] |
SUCHARD M A, LEMEY P, BAELE G, et al. Bayesian phylogenetic and phylodynamic data integration using BEAST 1.10[J]. Virus Evol, 2018, 4(1): vey016. |
| [17] |
RAMBAUT A, DRUMMOND A J, XIE D, et al. Posterior summarization in Bayesian phylogenetics using tracer 1.7[J]. Syst Biol, 2018, 67(5): 901-904. DOI:10.1093/sysbio/syy032 |
| [18] |
BIELEJEC F, BAELE G, VRANCKEN B, et al. SpreaD3:interactive visualization of spatiotemporal history and trait evolutionary processes[J]. Mol Biol Evol, 2016, 33(8): 2167-2169. DOI:10.1093/molbev/msw082 |
| [19] |
VALASTRO V, HOLMES E C, BRITTON P, et al. S1 gene-based phylogeny of infectious bronchitis virus: an attempt to harmonize virus classification[J]. Infect Genet Evol, 2016, 39: 349-364. DOI:10.1016/j.meegid.2016.02.015 |
| [20] |
KUO S M, WANG C H, HOU M H, et al. Evolution of infectious bronchitis virus in Taiwan: characterisation of RNA recombination in the nucleocapsid gene[J]. Vet Microbiol, 2010, 144(3-4): 293-302. DOI:10.1016/j.vetmic.2010.02.027 |
| [21] |
KUO S M, KAO H W, HOU M H, et al. Evolution of infectious bronchitis virus in Taiwan: positively selected sites in the nucleocapsid protein and their effects on RNA-binding activity[J]. Vet Microbiol, 2013, 162(2-4): 408-418. DOI:10.1016/j.vetmic.2012.10.020 |
| [22] |
吴翠兰, 何怡宁, 李和鸣, 等. 一株重组鸡传染性支气管炎病毒的分离及结构蛋白基因变异和血清型分析[J]. 畜牧兽医学报, 2015, 46(5): 815-823. WU C L, HE Y N, LI H M, et al. Isolation and analysis of structural protein genes variation and serotype of a recombinant strain of chicken infectious bronchitis virus[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(5): 815-823. (in Chinese) |
| [23] |
盛晓丹, 徐怀英, 刘霞, 等. 传染性支气管炎病毒S1和N基因遗传变异频率的分析[J]. 畜牧兽医学报, 2015, 46(6): 989-997. SHENG X D, XU H Y, LIU X, et al. Genetic variation frequency of S1 and N gene of infectious bronchitis virus[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(6): 989-997. (in Chinese) |
| [24] |
NABIL B, SABRINA B, ABDELHAKIM B. Transmission route and introduction of pandemic SARS-CoV-2 between China, Italy, and Spain[J]. J Med Virol, 2021, 93(1): 564-568. DOI:10.1002/jmv.26333 |
| [25] |
YANG Q Q, ZHAO X, LEMEY P, et al. Assessing the role of live poultry trade in community-structured transmission of avian influenza in China[J]. Proc Natl Acad Sci U S A, 2020, 117(11): 5949-5954. DOI:10.1073/pnas.1906954117 |
| [26] |
GAO F L, LIU X W, DU Z G, et al. Bayesianphylodynamic analysis reveals the dispersal patterns of tobacco mosaic virus in China[J]. Virology, 2019, 528: 110-117. |
| [27] |
CHEN H, SMITH G J D, ZHANG S Y, et al. Avian flu: H5N1 virus outbreak in migratory waterfowl[J]. Nature, 2005, 436(7048): 191-192. |
| [28] |
FRANZO G, NAYLOR C J, LUPINI C, et al. Continued use of IBV 793B vaccine needs reassessment after its withdrawal led to the genotype's disappearance[J]. Vaccine, 2014, 32(50): 6765-6767. |
| [29] |
FRANZO G, TUCCIARONE C M, BLANCO A, et al. Effect of different vaccination strategies on IBV QX population dynamics and clinical outbreaks[J]. Vaccine, 2016, 34(46): 5670-5676. |
(编辑 白永平)



