2. 农业部动物疫病病原生物学重点实验室东北科学观测实验站, 哈尔滨 150030
2. Northeast Scientific Inspection Observation Station, Key Laboratory of Animal Pathogen Biology of Ministry of Agriculture, Harbin 150030, China
猪流行性腹泻(porcine epidemic diarrhea, PED)是由猪流行性腹泻病毒(porcine epidemic diarrhea virus, PEDV)感染机体引起的一种高度接触性肠道传染病,主要临床症状为水样腹泻、呕吐和脱水等,对不同年龄阶段的猪都具有易感性[1-3]。自2010年以来,PEDV变异株疫情开始在我国国内和世界各地暴发,导致7日龄内的哺乳仔猪发生感染,死亡率为100%,严重影响养猪业的健康发展[4-5]。PEDV是一种单股正链RNA病毒,主要编码4种结构蛋白和16种非结构蛋白[6]。PEDV N蛋白能与病毒RNA相结合,形成螺旋核糖糖蛋白,为病毒RNA提供结构基础,和基因组密切关联,在病毒的复制、转录和组装过程中起着重要的作用[7-8]。
最近几年,重组酶聚合酶扩增(recombinase polymerase amplification, RPA)技术的问世,受到了大家广泛的关注,该项技术消除了常规RT-PCR反应过程中需要精准热循环的限制,它利用某些特定的蛋白和酶,实现对DNA双链的解旋以及DNA片段的特异性扩增的过程,从而达到检测特定病原的目的[9-11]。由于该技术的这些特性,使其在扩增反应中无需较高温度的变性、退火及延伸等步骤,因此使核酸扩增变的更加方便和快捷[12],与其他常规PCR技术相比,它可以在20~45 ℃的温度范围内进行反应,而且反应速度较快,通常情况下,从开始到完成只需15 ~25 min[13-15]。RPA技术可以利用琼脂糖凝胶电泳[16]、侧流层析试纸条[17]等对结果进行检测。在侧流层析试纸条上分别设有一条检测线和一条质控线,在检测线上包被生物素抗体,质控线上包被抗金标抗体。在对样品进行检测的过程中,利用胶体金标记的抗FAM抗体与扩增子上的FAM相结合,形成抗原抗体复合物,在毛细效应的作用下,复合物通过层析膜蔓延致检测线时,检测线处的生物素抗体与扩增子上的生物素发生特异性的结合,形成复合物,使检测线显色。综上所述,RPA技术在反应速度和反应条件方面,相比较一些常规PCR技术具有较大的优势,更适用于对某些特定的病原微生物进行快速检测[18-19],不需要昂贵的温度控制仪器辅助反应的发生,有效地减少了检测成本,更适合在实验条件欠佳的基层单位或边远山区推广。因此,本研究利用RPA扩增技术快速、准确、灵敏的优点,结合琼脂糖凝胶电泳以及侧流层析试纸条,建立了一种新的检测猪流行性腹泻病毒的方法。
1 材料与方法 1.1 细胞、材料及病料猪流行性腹泻病毒(PEDV,CV777株)、猪丁型冠状病毒(PDCoV,CH-HLJ-20株)、猪传染性胃肠炎病毒(TGEV,TH-98株)、猪轮状病毒(PoRV,JL-94株)和猪圆环病毒2型(PCV2,PCV2d株)均由本实验室提供并保存。OMEGA产物纯化试剂盒购于Omega公司;Trizol试剂盒以及质粒小提试剂盒购于天根生化科技有限公司;TwistAmp Basic购于英国TwistDx公司;Milenia HybriDetect试纸条为Milenia Biotec公司产品。pMD-19 T载体和E. coli DH5α感受态细胞购于大连宝生物工程有限公司。猪粪便样品共56份,分别采集自宾县、肇州和通辽地区的养殖场。
1.2 引物的设计与筛选采用GenBank中上发布的PEDV N基因序列作为模板,利用DNAMAN软件对其进行序列的比对,根据序列比对结果,查找出其中相对保守的基因序列,然后按照产品操作说明书上的要求,设计多对特异性引物(见表 1)进行筛选,以获得最佳的引物序列,引物均由库美生物有限公司合成。在引物的设计中,首先需要明确扩增片段的长度会影响反应扩增的效率,所以设计的扩增的片段长度需小于500 bp;同时也应当考虑到Basic RPA引物比传统的RT-PCR引物更长,注意避免引物序列中连续出现4个以上的G或C,避免引物的3′端的是以A结尾;LF-RPA引物设计要求下游引物的5′端采用生物素进行标记,探针的5′端由羧基荧光素(FAM)进行标记,3′端采用阻断聚合酶扩增的基团(C3 Spacer)进行修饰,并使用四氢呋喃脱碱基位点(THF)对探针的内部修饰。在引物设计完成后,还需要在NCBI中利用BLAST工具验证其特异性。
|
|
表 1 RPA引物序列 Table 1 RPA primer sequences |
按照TRNzol Universal总RNA提取试剂操作说明要求,进行病毒RNA提取。取500 μL细胞悬液离心加入1 mL Trizol,室温静置5 min,加入200 μL氯仿,剧烈震荡30 s,室温静置10 min,4 ℃,12 000 r·min-1离心15 min吸取350 μL上层于新的Ep管中,加入等量的异丙醇,充分混匀,室温下静置10 min;在4 ℃、12 000 r·min-1的条件下离心10 min,弃去上清液,每管加入75%乙醇1 mL;4 ℃,12 000 r·min-1离心1 min,弃上清,使用移液器吸干净残留液体,室温晾置3 min,每管加入RNase Free水80 μL混匀,得到病毒RNA。取23 μL RNA加入2 μL Oligo(dT)18,75 ℃孵育10 min,冰浴5 min后,然后依次加入8 μL RT Buffer、4 μL dNTP Mix、2 μL RRI、2 μL RTACE。42 ℃作用2 h,72 ℃水浴10 min,冰浴5 min。获得病毒cDNA。
1.4 阳性标准品的制备将上述获得的病毒cDNA作为模板,以PEDV-F/R(见表 2)为引物进行PCR扩增。通过琼脂糖凝胶电泳展示扩增结果,并在紫外灯照射下将带有目的片段的凝胶切下,使用OMEGA公司凝胶回收试剂盒对目的片段进行回收处理。回收纯化后的产物,克隆至pMD-19 T载体中,然后将其转化到E. coli DH5α感受态细胞中,将培养好的感受态菌液用涂布棒均匀涂于含有氨苄青霉素的LB固体培养基中。倒置于37 ℃恒温培养箱,培养10 h左右,挑选出的阳性菌落,进行扩大培养,使用质粒小提试剂盒提取质粒。并通过测序鉴定对其进行序列分析,证实目的片段成功克隆到pMD-19 T载体中,命名为pMD-19 T-PEDV/DH5α。
|
|
表 2 阳性标准品扩增引物 Table 2 Positive standard amplification primers |
使用分光光度计测量得到质粒的浓度为119 ng·μL-1,测序结果显示目的片段碱基数为1 045 bp,利用拷贝浓度计算公式:质粒拷贝浓度(copies·μL-1)=6.02×1023×浓度(ng·μL-1)×10-9/(重组载体碱基数×660),经计算,得出质粒的初始拷贝浓度为3.16×1010 copies·μL-1,对其进行10倍梯度稀释,使重组质粒pMD-19 T-PEDV/DH5α的拷贝浓度在1×109~1×101 copies·μL-1之间进行后续的试验研究。
1.6 RT-PCR以及RT-qPCR反应体系的建立1.6.1 RT-PCR反应体系的建立 以提取的病毒cDNA为样品模板,加入上下游引物(表 3)各1 μL,2×Es Taq MasterMix(Dye)12.5 μL,去离子水(ddH2O)9.5 μL,模板1 μL,将上述扩增体系混合后放入PCR仪中进行反应。RT-PCR扩增程序如下:预变性94 ℃ 3 min;94 ℃变性30 s,55 ℃退火30 s;72 ℃延伸1 min,30个循环;最后,72 ℃终止7 min。
|
|
表 3 猪流行性腹泻病毒检测引物 Table 3 Primer sequences for detection of Porcine epidemic diarrhea virus |
1.6.2 RT-qPCR反应体系的建立 同样以病毒cDNA为模板加入2 μL,上下游引物(表 3)各0.5 μL,FastStartUniversal SYBR Geen Master(ROX)10 μL,去离子水(ddH2O)7 μL,共计20 μL,在避光条件下混匀后进行如下扩增程序:95 ℃预变性10 min;95 ℃变性15 s,60 ℃退火30 s,72 ℃延伸30 s,共40个循环,在反应结束后记录各样品所对应的Ct值。
1.7 RPA反应体系和反应条件的建立以构建的阳性质粒为模板,使用所设计的引物进行RPA扩增反应。向含有冻干酶粉的0.2 mL TwistAmp Basic反应管中加入Rehydration Buffer 29.5 μL,上下游引物各2.4 μL(10 mol·μL-1),去离子水12.2 μL,模板质粒1 μL,将该扩增体系充分混匀,最后再加入醋酸镁溶液2.5 μL(280 mol·μL-1)催动反应快速发生。在加入醋酸镁溶液后,立即将反应管置于38 ℃水浴锅内计时30 min,等待RPA反应结束后,Basic RPA方法需先使用OMEGA公司的产物纯化试剂盒,对RPA扩增产物进行纯化处理,将纯化后的RPA产物添加至15 g·L-1琼脂糖凝胶中进行电泳,观察试验结果;LF-RPA方法则是在Basic RPA方法的基础上,将引物更换为针对LF-RPA所设计的引物,并加入0.6 μL探针、1 μL NFO酶、5 μL酶切Buffer后,同样需要加入醋酸镁溶液启动扩增反应,在反应结束后将20 μL反应产物与80 μL的稀释缓冲液进行混匀,将商品化抗FAM和Biotin标记的LFD试纸条插入混合均匀的体系中进行检测,根据检测结果确定反应体系的最佳条件。
1.8 RPA反应温度的优化为确定RPA反应的最适温度,将拷贝浓度为108 copies·μL-1的阳性标准品作为模板进行RPA扩增。根据上述反应体系,取8个TwistAmp Basic反应管,分别将其放置在15、20、25、30、36、39、42、45 ℃条件下进行反应,在反应开始30 min后取出反应管,置于冰上终止反应。并采用琼脂糖凝胶电泳以及侧流层析试纸条两种结果检测方法,对反应结果进行判定,以确定两种检测方法的最佳反应温度。
1.9 RPA反应时间的优化同样以拷贝浓度为108 copies·μL-1的阳性标准品为模板进行RPA扩增,优化其反应的最佳时间。根据对反应温度的优化,将在30 ℃条件下进行试验,将混合好的反应体系置于水浴锅内,在反应开始后的5、10、15、20、25、30、35、40 min取出反应管并立即置于冰上终止反应。同样使用两种结果检测方法,对反应结果进行判定,以确定两种检测方法的最佳反应时间。
1.10 敏感性试验将稀释好的108 ~101 copies·μL-1阳性质粒作为模板,根据上述建立好的RPA和RT-PCR反应体系和反应程序进行扩增反应,并设置阴性对照,通过将两种本研究所建立的检测方法与传统RT-PCR扩增方法相比较,来判定检测方法的敏感性。
1.11 特异性试验将实验室保存的猪丁型冠状病毒、猪传染性胃肠炎病毒、猪轮状病毒进行RNA提取,再反转录成cDNA,将猪圆环病毒2型直接进行病毒DNA提取,以4种病毒的cDNA为模板,按照上述的体系进行RPA扩增反应,同时设置阴性对照,再使用琼脂糖凝胶电泳和侧流层析试纸条对反应结果进行判定,检测其反应的特异性。
1.12 重复性试验以3种不同浓度的阳性标准质粒作为模板,在相同的体系、温度和时间条件下,重复进行3组试验。在每次的试验中,观察不同浓度的阳性标准品,经RPA反应扩增后所展示的结果;在3组试验中,观察每组试验中相同浓度的阳性标准品的RPA反应扩增结果,同时设置阴性对照并对检测结果进行分析。
1.13 临床样品的符合率检测根据本研究建立的方法,对猪场采集的56份猪粪便样品进行了检测。使用拭子蘸取少量粪便,浸泡于含有1 mL PBS的Ep管中,4 ℃放置2 h,12 000 r·min-1离心5 min,取500 μL上清,之后利用TRNzol Universal总RNA提取试剂提取病毒RNA,然后反转录成cDNA,进行RPA方法检测,将检测结果与常规RT-PCR和RT-qPCR进行对照,检测两种方法的结果符合率。
2 结果 2.1 目的基因的克隆以提取的病毒cDNA为模板,PEDV-F/R为引物进行PCR扩增。PCR产物经琼脂糖凝胶电泳后,在1 045 bp处显示出特异性的条带扩增,扩增结果与设计的目的片段相符(见图 1)。
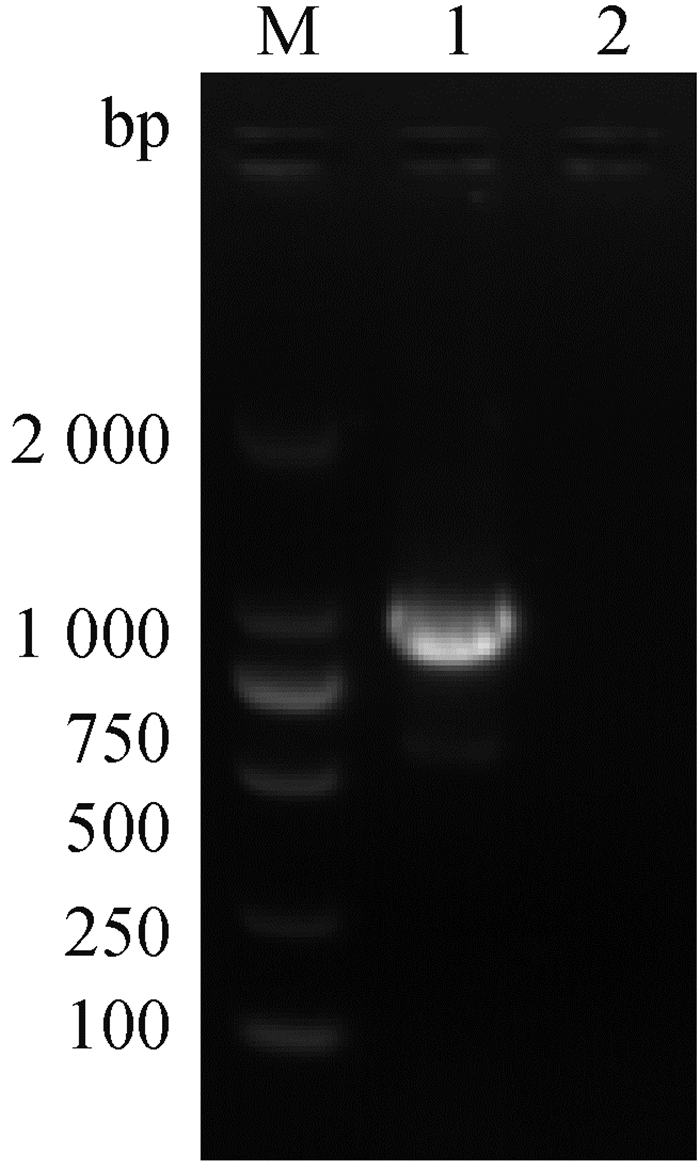
|
M. DNA相对分子质量标准;1. PEDV目的片段的扩增结果;2. 阴性对照 M. DNA marker; 1. Amplification node of target fragment from PEDV; 2. Negative control 图 1 目的片段的克隆 Fig. 1 Cloning of target fragments |
通过对GenBank中所提供的PEDV N基因(LT906 620.1、DQ355 221.1、KU664 503.1、KM887 144.1、KR610991、KJ646 625.1、MN486 588.1、JX560761、FJ473 390.1、KP890336)序列进行比对,各序列之间的一致性为98.45%,根据其保守基因序列设计引物。以3.16×108 copies·μL-1的阳性质粒为模板,对设计的引物进行筛选,结果如图 2A所示,Basic-PEDV-F1/R1的扩增条带较为单一和清晰,LFD-PEDV-F1/R1以及PEDV-PROBE1的扩增效果优于LFD-PEDV-F2/R2以及PEDV-PROBE2(图 2B),因此选用Basic-PEDV-F1/R1、LFD-PEDV-F1/R1进行后续试验。
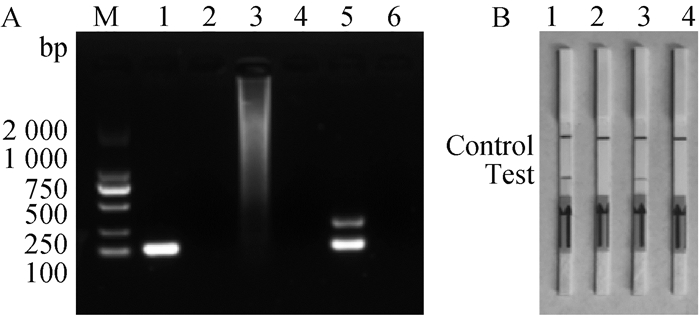
|
A. Basic RPA引物的筛选;B. LF-RPA引物的筛选;M. DNA相对分子质量标准;1~2. 第1组引物扩增结果及阴性对照;3~4. 第2组引物扩增结果及阴性对照;5~6. 第3组引物扩增结果及阴性对照 A. Screening of Basic RPA primers; B. Screening of LF-RPA primers; M. DNA marker; 1-2. Group 1 primer amplification results and negative control; 3-4. Group 2 primer amplification results and negative control; 5-6. Group 2 primer amplification results and negative control 图 2 RPA引物筛选结果 Fig. 2 Results of RPA primer screening |
以质粒拷贝浓度为3.16×1010 ~3.16×101 copies·μL-1的阳性标准品为模板,利用上述的RT-qPCR反应体系以及扩增程序,进行实时荧光定量PCR,将结果的Ct值设置为X轴、lg质粒拷贝数设置为Y轴构建标准曲线,其标准方程:y=-0.270 2x+11.351,相关系数R2为0.996 8(图 3)。结果表明,Ct值与质粒拷贝数之间呈良好的线性关系,可以看出,当Ct值低于30时,可记录样品为阳性;Ct值超过32或无有效值时,样品均检测结果为阴性。
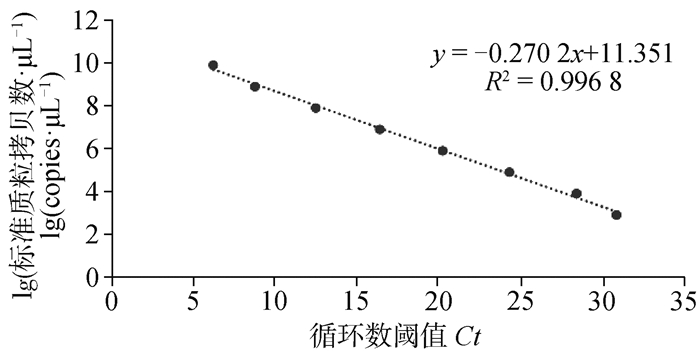
|
图 3 PEDV实时荧光定量PCR标准曲线 Fig. 3 Standard curve of PEDV real-time fluorescent quantitative PCR |
在使用琼脂糖凝胶电泳检测RPA反应时,在15 ℃的反应条件下,就可以观察到特异性条带的扩增,在15~42 ℃的温度范围内可较为清晰地看到特异性片段的扩增产量,随着反应温度的升高随之增加的现象,当温度继续升高到45 ℃时,出现条带亮度减弱的现象。在使用侧流层析试纸条的检测方法中,当反应温度到达30 ℃时,试纸条已经观察不到扩增产量随温度增加的情况,结合两种检测方法的结果可以看出,当反应温度范围在30~42 ℃时RPA反应的扩增效果最佳(图 4)。据此,本方法选用30 ℃的作为最佳反应温度。
|
A. Basic RPA反应温度的优化;B. LF-RPA反应温度的优化;M. DNA相对分子质量标准;N. 阴性对照;1. 15 ℃;2. 20 ℃;3. 25 ℃;4. 30 ℃;5. 36 ℃;6. 39 ℃;7. 42 ℃;8. 45 ℃ A. Optimization of reaction temperature of Basic RPA; B. Optimization of reaction temperature of LF-RPA; M. DNA marker; N. Negative control; 1. 15 ℃; 2. 20 ℃; 3. 25 ℃; 4. 30 ℃; 5. 36 ℃; 6. 39 ℃; 7. 42 ℃; 8. 45 ℃ 图 4 RPA反应温度的优化 Fig. 4 Optimization of reaction temperature of RPA |
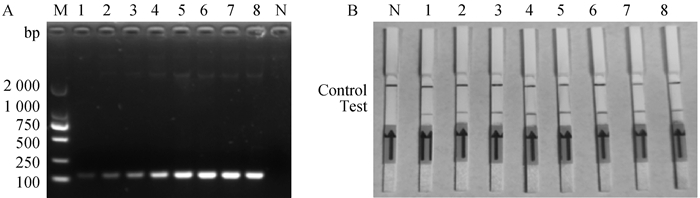
在琼脂糖凝胶的结果显示中,在30 ℃的反应条件下,反应开始的5 min内已经有目的片段开始扩增的现象,当反应时间到达20 min时,可以观察到清晰明亮的条带产生,表明已产生了大量的扩增产物(图 5)。由此可知,当RPA反应时间在超过5 min后,便可实现对样品的检测。在LF-RPA的检测结果中,与Basic RPA检测的结果有类似的情况,反应时间在20~40 min时Basic RPA和LF-RPA的检测结果无明显的差异。所以,本方法选用的最佳反应时间为20 min。
|
A. Basic RPA反应时间的优化;B. LF-RPA反应时间的优化;M. DNA相对分子质量标准;N. 阴性对照;1. 5 min;2. 10 min;3. 15 min;4. 20 min;5. 25 min;6. 30 min;7. 35 min;8. 40 min A. Optimization of reaction time of Basic RPA; B. Optimization of reaction time of LF-RPA; M. DNA marker; N. Negative control; 1. 5 min; 2. 10 min; 3. 15 min; 4. 20 min; 5. 25 min; 6. 30 min; 7. 35 min; 8. 40 min 图 5 RPA反应时间的优化 Fig. 5 Optimization of the RPA reaction time |
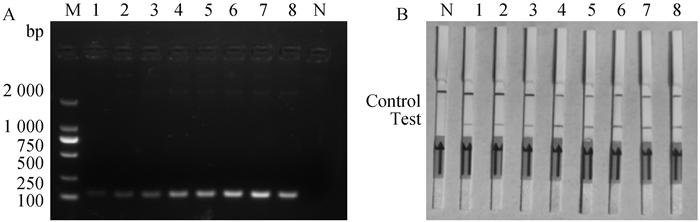
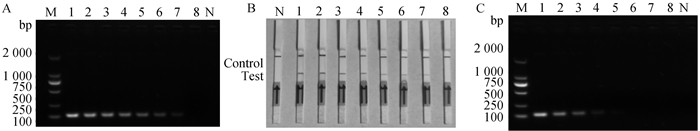
2.6.1 RPA反应的敏感性 将108 ~101 copies·μL-1的阳性质粒作为模板,在30 ℃水浴锅中进行20 min的RPA扩增反应,分别采用Basic RPA和LF-RPA两种检测方法对模板样品进行检测。从图 6A、B中可以看出,当标准品的拷贝浓度为3.16×102 copies·μL-1时两种检测方法均能检测到目的片段的扩增,而对照组无扩增现象,展现出RPA扩增技术良好的敏感性。
|
A. Basic RPA的灵敏度试验;B. LF-RPA的灵敏度试验;C. RT-PCR的灵敏度试验;M. DNA相对分子质量标准;N. 阴性对照;1~8. 3.16×108 ~3.16×101 copies·μL-1 A. Sensitivity tests of Basic RPA; B. Sensitivity tests of LF-RPA; C. Sensitivity tests of RT-PCR; M. DNA marker; N. Negative control; 1-8. 3.16×108-3.16×101 copies·μL-1 图 6 敏感性试验 Fig. 6 Sensitivity tests |
2.6.2 RT-PCR反应的敏感性 以同样的阳性标准品为模板,使用与RPA反应相同的引物进行RT-PCR的扩增,在凝胶成像系统上观察RT-PCR的扩增结果(图 6C)。从图中可以看出,当标准品的拷贝浓度为3.16×105 copies·μL-1时可以观察到较为暗淡的扩增条带产生,当以拷贝浓度更低的样品为模板时,没有出现目的片段的扩增现象,同时对照组也无条带扩增,表明该检测方法的可以检出的最低检测限度为3.16×105 copies·μL-1。
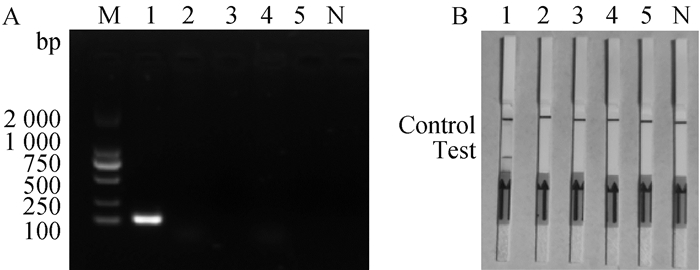
2.7 特异性试验为验证本方法中RPA反应的特异性,以猪流行性腹泻病毒、猪丁型冠状病毒、猪传染性胃肠炎病毒、猪轮状病毒和猪圆环病毒的cDNA为模板,水作为阴性对照进行RPA扩增反应。结果显示(图 7),在其余4种非PEDV的病毒样品以及阴性对照泳道中,均未出现目的片段扩增的现象,在侧流层析试纸条的检测试验中表现出相同的结果。两种检测方法均对猪流行性腹泻病毒展现出了良好的特异性扩增效果,展现出了RPA扩增技术在以病毒cDNA为模板的检测中,仍具有良好的特异性。
|
A. Basic RPA的特异性试验;B. LF-RPA的特异性试验;M. DNA相对分子质量标准;N. 阴性对照;1. 猪流行性腹泻病毒;2. 猪丁型冠状病毒;3. 猪传染性胃肠炎病毒;4. 猪轮状病毒;5. 猪圆环病毒2型 A. Specificity tests of Basic RPA; B. Specificity tests of LF-RPA; M. DNA marker; N. Negative control; 1. PEDV; 2. PDCoV; 3. TGEV; 4. PoRV; 5. PCV2 图 7 特异性试验 Fig. 7 Specificity tests |
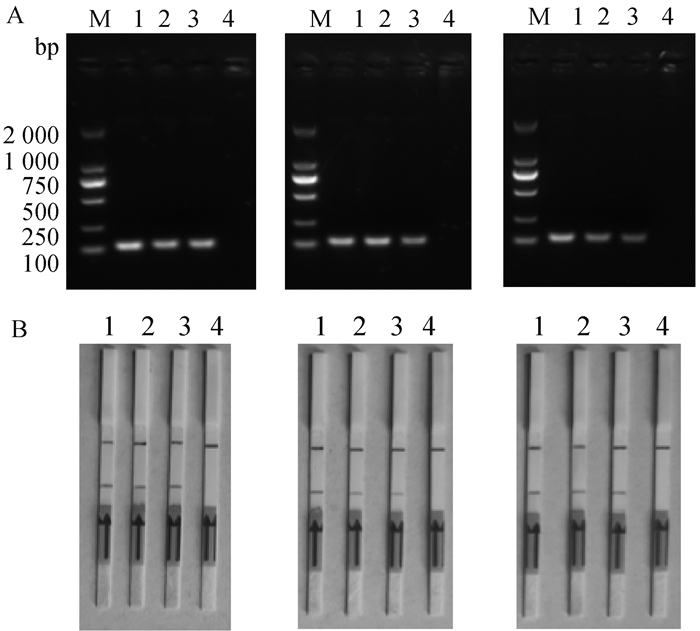
以3.16×107、3.16×106、3.16×105 copies·μL-1这三个不同拷贝浓度的阳性标准质粒作为模板,在30 ℃的水浴锅内反应20 min,相同的反应条件下重复进行3次RPA扩增试验,以Basic RPA和LF-RPA两种检测方法对样品进行检测。结果显示,3种不同拷贝浓度的质粒,在同一次试验中展现出对浓度的敏感性;在重复的3次试验中,相同拷贝浓度的质粒RPA扩增显示无明显差异,且阴性对照无扩增条带产生(图 8)。这样的结果表明了本研究所建立的RPA检测方法具有良好的批间重复性和批内重复性。
|
A. Basic RPA的重复性试验;B. LF-RPA的重复性试验;M. DNA相对分子质量标准;1~3. 3.16×107~3.16×105 copies·μL-1;4. 阴性对照 A. Assay reproducibility tests of Basic RPA; B. Assay reproducibility tests of LF-RPA; M. DNA marker; 1-3. 3.16×107-3.16×105 copies·μL-1; 4. Negative control 图 8 重复性试验 Fig. 8 Assay reproducibility tests |
使用Basic RPA、LF-RPA对56份临床粪便样品进行检测,两种方法的符合率见表 4。结果显示,以RT-PCR和RT-qPCR检测结果为最终判定标准,Basic RPA、LF-RPA与RT-PCR和RT-qPCR结果符合率分别为89.29%、91.07%、85.71%、87.50%。其中Basic RPA与RT-qPCR的符合率最高,LF-RPA与RT-PCR为符合率最低。
|
|
表 4 Basic RPA、LF-RPA与RT-PCR、qPCR检测结果的符合率 Table 4 The coincidence rates of Basic RPA, LF-RPA and RT-PCR, qPCR results |
当前,猪流行性腹泻的流行仍严重危害着中国和全球的生猪产业,目前已有的商品化疫苗并不能彻底阻止其传播[20-21],因此及早诊断对于该病的预防和治疗显得尤为重要,所以开发一种快速、高灵敏度的PEDV诊断技术是非常必要的。随着分子生物学技术的不断发展,已有多种方法应用于PEDV的检测,其中主要包括常规RT-PCR[22]、实时荧光定量PCR[23]和间接免疫荧光[24]。然而,上述这些技术都对仪器和设备的要求较高,不能实现对临床粪便样品中PEDV快速、直观的检测。目前,在已有的一些研究报道中,RPA技术在人类和动植物的传染病检测以及食品卫生安全等领域得到了广泛应用[25-26],甚至在案件现场中也可以利用RPA技术对体液进行检测[27],然而,还没有相关的研究报告将该技术应用与PEDV的检测。综上所述,本研究以RPA技术为基础,建立一种新的快速检测方法来检测猪群中的PEDV感染,该技术以琼脂糖凝胶电泳和侧流层析试纸条作为结果判定的方法,并对RPA技术在PEDV检测的应用进行了评估和比较,从而为进一步开展PEDV的临床快速诊断研究提供了理论依据。
在本研究中,作者参照了英国Twist Dx公司的产品使用说明书,以相对保守的PEDV N基因作为靶基因,与其他基因相比,该基因更适合于PEDV的病原检测,更易于提高检测的敏感性和特异性[28-29],对RPA反应进行了多组引物的设计。RPA技术不同于PCR技术,没有经过变性、退火等步骤可将引物之间的错配降低,因此,对于RPA扩增技术要格外注意对引物的筛选,以免产生错误的检测结果。但是到目前为止,未开发出针对RPA技术的引物设计软件,在引物的筛选过程中更多的是依靠试验去验证;其次RPA技术扩增片段的长度会影响其扩增的效率[31],较长的扩增片段会导致反应时间增长,不能体现RPA技术快速检测的优点,因此RPA方法多用在样品检测方面而不适用对片段进行克隆。在试验中发现,RPA具有相对较高的反应灵敏度,最低检测限度达到102 copies·μL-1,低于常规RT-PCR的检测限度。本试验中RT-PCR所检测的敏感性相较于Ding等[31]较低,可能的原因是所用的引物是根据RPA所设计导致在RT-PCR的过程中效果不佳导致,但还需要进一步的试验验证。此外,将本研究设计的引物与常见的其他腹泻病毒进行RPA扩增反应,结果显示无特异性条带产生,验证了该方法具有良好的特异性。从本研究中可以看出,在RPA反应开始5 min后就能显示出特异性扩增条带,在20~40 min时扩增效果最好,且没有显著的差别,整个反应只需30 min即可完成检测,这就说明了RPA技术在检测时间上的优越性;RPA反应的扩增条带在30~42 ℃时最亮,说明在这一温度区间扩增效果最好,且无显著差别。在温度超过42 ℃后,扩增条带呈减弱的趋势,造成这样的原因可能是较高的温度降低了反应中酶的活性。所以,将最佳反应温度确定为30 ℃,最佳反应时间确定为20 min,这些结果与蔡应奎等[32]对猪嵴病毒以及Wang等[33]对猪塞内卡病毒的检测中表现出类似的现象。
在实际应用中发现,采用Basic RPA及LF-RPA技术对检测猪粪便样品进行检测,与RT-PCR和RT-qPCR相比,具有更高的符合率,且检出率均高于常规RT-PCR的检测方法,由此可见本试验建立的Basic RPA和LF-RPA方法具有高敏感性、特异性的特点。虽然本研究中的样本数量并不多,但是这些资料仍然可以为今后这两种新方法在临床实际应用中提供初步的参考意见。总之,本研究开发了一种以RPA扩增技术为基础的高灵敏性、高特异性且低成本的PEDV快速检测技术,此方法只需简单的加热,不需要精确的温度控制设备,即使在设备不佳的实验室或现场也可以对PEDV感染进行诊断分析。
4 结论本研究在探索与优化RPA反应条件的基础上,成功建立了PEDV Basic RPA和LF-RPA的检测方法,且运用两种检测方法对猪粪便临床样品进行了检测和分析,与RT-PCR和RT-qPCR方法比较,结果显示本研究建立的两种检测方法具有较高的结果符合率,为今后PEDV的临床诊断提供了更多的选择以及更有效的技术支持。
| [1] |
KIM O, CHAE C. Comparison of reverse transcription polymerase chain reaction, immunohistochemistry, and in situ hybridization for the detection of porcine epidemic diarrhea virus in pigs[J]. Can J Vet Res, 2002, 66(2): 112-116. |
| [2] |
JUNG K, SAIF L J, WANG Q H. Porcine epidemic diarrhea virus (PEDV): an update on etiology, transmission, pathogenesis, and prevention and control[J]. Virus Res, 2020, 286: 198045. DOI:10.1016/j.virusres.2020.198045 |
| [3] |
SONG D, PARK B. Porcine epidemic diarrhoea virus: a comprehensive review of molecular epidemiology, diagnosis, and vaccines[J]. Virus Genes, 2012, 44(2): 167-175. DOI:10.1007/s11262-012-0713-1 |
| [4] |
万颖, 周改静, 麻园, 等. 猪流行性腹泻病毒N蛋白阻断ELISA抗体检测方法的建立及初步应用[J]. 畜牧兽医学报, 2022, 53(4): 1173-1181. WAN Y, ZHOU G J, MA Y, et al. Development and application of N-protein blocking ELISA for detecting porcine epidemic diarrhea virus antibodies[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(4): 1173-1181. (in Chinese) |
| [5] |
WANG F Q, WANG M, ZHANG L, et al. Generation and functional analysis of single chain variable fragments (scFvs) targeting the nucleocapsid protein of Porcine epidemic diarrhea virus[J]. Appl Microbiol Biotechnol, 2022, 106(3): 995-1009. DOI:10.1007/s00253-021-11722-z |
| [6] |
DONG S J, KONG N, ZHANG Y, et al. TARDBP inhibits porcine epidemic diarrhea virus replication through degrading viral nucleocapsid protein and activating type Ⅰ interferon signaling[J]. J Virol, 2022, 96(10): e0007022. DOI:10.1128/jvi.00070-22 |
| [7] |
冯力, 孙东波, 陈建飞, 等. 猪流行性腹泻病毒N蛋白单克隆抗体的制备与鉴定[J]. 中国预防兽医学报, 2008, 30(3): 190-193. FENG L, SUN D B, CHEN J F, et al. Preparation and characterization of the monoclonal antibodies against nucleocapsid protein of PEDV[J]. Chinese Journal of Preventive Veterinary Medicine, 2008, 30(3): 190-193. (in Chinese) |
| [8] |
QIN W Z, KONG N, ZHANG Y, et al. Nuclear ribonucleoprotein RALY targets virus nucleocapsid protein and induces autophagy to restrict porcine epidemic diarrhea virus replication[J]. J Biol Chem, 2022, 298(8): 102190. DOI:10.1016/j.jbc.2022.102190 |
| [9] |
LOBATO I M, O'SULLIVAN C K. Recombinase polymerase amplification: basics, applications and recent advances[J]. TrAC Trends Analyt Chem, 2018, 98: 19-35. DOI:10.1016/j.trac.2017.10.015 |
| [10] |
DAHER R K, STEWART G, BOISSINOT M, et al. Recombinase polymerase amplification for diagnostic applications[J]. Clin Chem, 2016, 62(7): 947-958. DOI:10.1373/clinchem.2015.245829 |
| [11] |
MOTA D S, GUIMARÃES J M, GANDARILLA A M D, et al. Recombinase polymerase amplification in the molecular diagnosis of microbiological targets and its applications[J]. Can J Microbiol, 2022, 68(6): 383-402. DOI:10.1139/cjm-2021-0329 |
| [12] |
吴耀东, 徐民俊, 郑文斌, 等. 重组酶聚合酶扩增技术及其在动物病原快速检测中的应用[J]. 中国兽医学报, 2016, 36(10): 1797-1802. WU Y D, XU M J, ZHENG W B, et al. Development of recombinant polymerase amplification technology and its applications in quick diagnosis of animal pathogen[J]. Chinese Journal of Veterinary Science, 2016, 36(10): 1797-1802. DOI:10.16303/j.cnki.1005-4545.2016.10.29 (in Chinese) |
| [13] |
TOMAR S, LAVICKOVA B, GUIDUCCI C. Recombinase polymerase amplification in minimally buffered conditions[J]. Biosens Bioelectron, 2022, 198: 113802. DOI:10.1016/j.bios.2021.113802 |
| [14] |
MUNAWAR M A. Critical insight into recombinase polymerase amplification technology[J]. Expert Rev Mol Diagn, 2022, 22(7): 725-737. DOI:10.1080/14737159.2022.2109964 |
| [15] |
PIEPENBURG O, WILLIAMS C H, STEMPLE D L, et al. DNA detection using recombination proteins[J]. PLoS Biol, 2006, 4(7): e204. DOI:10.1371/journal.pbio.0040204 |
| [16] |
ZHAO L W, WANG J C, SUN X X, et al. Development and evaluation of the rapid and sensitive RPA assays for specific detection of Salmonella spp. in food samples[J]. Front Cell Infect Microbiol, 2021, 11: 631921. DOI:10.3389/fcimb.2021.631921 |
| [17] |
LI J, ZHONG Q, SHANG M Y, et al. Preliminary evaluation of rapid visual identification of Burkholderia pseudomallei using a newly developed lateral flow strip-based recombinase polymerase amplification (LF-RPA) system[J]. Front Cell Infect Microbiol, 2022, 11: 804737. DOI:10.3389/fcimb.2021.804737 |
| [18] |
罗雪琮, 安梦楠, 吴元华, 等. 重组酶聚合酶扩增技术在植物病毒检测中的应用[J]. 生物技术通报, 2022, 38(2): 269-280. LUO X C, AN M N, WU Y H, et al. Applications of recombinase polymerase amplification in plant virus detection[J]. Biotechnology Bulletin, 2022, 38(2): 269-280. (in Chinese) |
| [19] |
WANG F, WANG Y, LIU X, et al. Rapid, simple, and highly specific detection of Streptococcus pneumonia with visualized recombinase polymerase amplification[J]. Front Cell Infect Microbiol, 2022, 12: 878881. DOI:10.3389/fcimb.2022.878881 |
| [20] |
REVELES-FÉLIX S, CARREÓN-NÁPOLES R, MENDOZA-ELVIRA S, et al. Emerging strains of porcine epidemic diarrhoea virus (PEDV) in Mexico[J]. Transbound Emerg Dis, 2020, 67(2): 1035-1041. DOI:10.1111/tbed.13426 |
| [21] |
LEE S, LEE C. Genomic and antigenic characterization of porcine epidemic diarrhoea virus strains isolated from South Korea, 2017[J]. Transbound Emerg Dis, 2018, 65(4): 949-956. DOI:10.1111/tbed.12904 |
| [22] |
张坤, 何启盖. 猪流行性腹泻病毒、猪传染性胃肠炎病毒和猪A群轮状病毒多重RT-PCR检测方法的建立及临床应用[J]. 畜牧兽医学报, 2010, 41(8): 1001-1005. ZHANG K, HE Q G. Establishment and clinical application of a multiplex reverse transcription-PCR for detection of porcine epidemic diarrhea virus, porcine transmissible gastroenteritis virus and porcine group a rotavirus[J]. Acta Veterinaria et Zootechnica Sinica, 2010, 41(8): 1001-1005. (in Chinese) |
| [23] |
修金生, 周伦江, 陈如敬, 等. 猪流行性腹泻病毒SYBR Ⅰ实时荧光定量RT-PCR检测方法的建立[J]. 中国兽医科学, 2012, 42(2): 160-165. XIU J S, ZHOU L J, CHEN R J, et al. Development of a real time RT-PCR assay based on SYBR Green Ⅰ for rapid diagnosis of porcine epidemic diarrhea virus[J]. Chinese Veterinary Science, 2012, 42(2): 160-165. (in Chinese) |
| [24] |
GERBER P F, LELLI D, ZHANG J Q, et al. Diagnostic evaluation of assays for detection of antibodies against porcine epidemic diarrhea virus (PEDV) in pigs exposed to different PEDV strains[J]. Prev Vet Med, 2016, 135: 87-94. DOI:10.1016/j.prevetmed.2016.11.005 |
| [25] |
NEFEDEVA M, TITOV I, MALOGOLOVKIN A. Molecular characteristics of a novel recombinant of porcine epidemic diarrhea virus[J]. Arch Virol, 2019, 164(4): 1199-1204. DOI:10.1007/s00705-019-04166-4 |
| [26] |
WANG X W, WANG M, ZHAN J, et al. Pathogenicity and immunogenicity of a new strain of porcine epidemic diarrhea virus containing a novel deletion in the N gene[J]. Vet Microbiol, 2020, 240: 108511. DOI:10.1016/j.vetmic.2019.108511 |
| [27] |
LI N, WANG L, WANG F, et al. Rapid detection of Klebsiella pneumoniae carrying virulence gene rmpA2 by recombinase polymerase amplification combined with lateral flow strips[J]. Front Cell Infect Microbiol, 2022, 12: 877649. DOI:10.3389/fcimb.2022.877649 |
| [28] |
YANG X H, ZHAO P P, DONG Y, et al. An isothermal recombinase polymerase amplification and lateral flow strip combined method for rapid on-site detection of Vibrio vulnificus in raw seafood[J]. Food Microbiol, 2021, 98: 103664. DOI:10.1016/j.fm.2020.103664 |
| [29] |
KUBO S, NIIMI H, KITAJIMA I. Rapid detection of blood and semen mRNA markers by reverse transcription-recombinase polymerase amplification[J]. Forensic Sci Int Genet, 2022, 58: 102665. DOI:10.1016/j.fsigen.2022.102665 |
| [30] |
IVANOV A V, POPRAVKO D S, SAFENKOVA I V, et al. Rapid full-cycle technique to control adulteration of meat products: integration of accelerated sample preparation, recombinase polymerase amplification, and test-strip detection[J]. Molecules, 2021, 26(22): 6804. DOI:10.3390/molecules26226804 |
| [31] |
DING G M, FU Y G, LI B Y, et al. Development of a multiplex RT-PCR for the detection of major diarrhoeal viruses in pig herds in China[J]. Transbound Emerg Dis, 2020, 67(2): 678-685. DOI:10.1111/tbed.13385 |
| [32] |
蔡应奎, 刘新生, 张丽萍, 等. 猪嵴病毒重组酶聚合酶扩增结合侧流层析试纸条(RPA-LFD)快速诊断方法的建立与应用[J]. 中国兽医科学, 2020, 50(7): 820-824. CAI Y K, LIU X S, ZHANG L P, et al. Development and application of a recombinase polymerase amplification combined with a lateral flow dipstick (RPA-LFD) assay for rapid diagnosis of porcine kobuvirus[J]. Chinese Veterinary Science, 2020, 50(7): 820-824. (in Chinese) |
| [33] |
WANG H B, DONG J J, ZHANG T, et al. A novel rapid detection of Senecavirus A using recombinase polymerase amplification (RPA) coupled with lateral flow (LF) dipstrip[J]. Anal Biochem, 2022, 646: 114627. |
(编辑 白永平)



