2. 西北农林科技大学 动物科技学院, 杨凌 712100;
3. 吉林大学动物科学学院, 长春 130000;
4. 固原富民农业科技发展有限公司, 固原 756000;
5. 中国农业科学院北京畜牧兽医研究所, 北京 100193
2. College of Animal Science and Technology, Northwest Agricultural and Forestry University, Yangling 712100, China;
3. College of Animal Science, Jilin University, Changchun 130000, China;
4. Guyuan Fumin Agricultural Technology Development Co. Ltd., Guyuan 756000, China;
5. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China
固原黄牛是分布于宁夏回族自治区六盘山周边地区的一种地方黄牛种群,主要分布于中卫市海原县、固原市原州区、西吉县、彭阳县、隆德县、泾源县,亦称为“六盘山黄牛”。固原黄牛肉具备蛋白质含量高、谷氨酸含量丰富、胆固醇含量低、肉质细嫩多汁等特点[1]。近年来,国内外肉牛品种冻精的推广导致本地黄牛无序杂交、纯种固原黄牛数量急剧下降,进而降低了其肉品质和品牌效益。因此,亟待分析现存固原黄牛的遗传背景,挖掘固原黄牛生长发育、肉用性能的群体规律,为固原黄牛良种繁育和遗传改良提供一定的参考数据。
据《固原县志》、《固原地区志》和《固原百科》等记载,固原地区黄牛群体与周边蒙古牛和秦川牛群体相互影响,经过长期选育形成了体型外貌一致的独特类群。上世纪90年代,常洪等[2-6]通过研究固原黄牛群体遗传变异,认为固原黄牛为中国黄牛北方型和中原型之间的过渡类型,是宁夏特有地方黄牛类群。谷朝勇[7]通过相对贴近系数(relative approach coefficient,RAC)判定固原黄牛与中亚东南家牛群体相对群体集团亲缘关系,发现固原黄牛与蒙古利亚牛系之间基因交流更为频繁。刘丽等[8]基于线粒体DNA对中国部分地方黄牛群体进行了系统发育研究,发现固原地方黄牛群体经过长期的自然选择和人工选择,已经形成了独特的遗传结构。综上所述,借鉴生态、地理、历史、考古等多方面的研究资料,结合现代遗传学标记技术可知:固原黄牛是分布于宁夏回族自治区六盘山周边地区的一种固有地方黄牛类群,主要含有本地黄牛、秦川牛和蒙古牛血液。但对现存固原黄牛群体的遗传背景、体型外貌特征、体重、体尺、背膘厚和眼肌面积等表型数据仍缺乏系统全面研究。
本研究对固原黄牛全基因组进行基因芯片检测,同时收集12个品种(巴厘岛爪哇野牛、吉安牛、晋南牛、雷琼牛、利木赞牛、鲁西牛、蒙古牛、南阳牛、秦川牛、红安格斯牛、皖南牛和延边牛)的全基因组基因芯片数据,通过多维标度(multidimensional scaling,MDS)分析和构建邻接法(neighbor-joining,NJ)系统发育树,分析固原黄牛遗传背景。同时测定不同生长发育阶段固原黄牛和红安格斯牛×固原黄牛杂交牛的体重、体尺、背膘厚和眼肌面积等表型数据,参照《中国畜禽遗传资源志·牛志》公开发表的秦川牛和蒙古牛的表型数据,计算不同牛群体的体尺指数和肉用指数(beef-purpose index,BPI),深入挖掘固原黄牛相关指标的群体规律,以期为我国肉牛新类群(品种)的选育提供一定的参考和依据。
1 材料与方法 1.1 试验动物及数据收集通过对中卫市海原县、固原市原州区、西吉县、彭阳县、隆德县、泾源县等地本土黄牛进行调查,收集66头纯种固原黄牛(公牛18头、母牛48头)饲养于固原市富民农业科技发展有限公司,采集53头(公牛11头,母牛42头)纯种固原黄牛血液,使用上海纽勤生物科技有限公司GGP Bovine 100K基因芯片进行SNP分型[9]。随机选择固原市富民农业科技有限公司养殖场的红安格斯牛×固原黄牛杂交牛(公牛10头,母牛186头)为对照群体,测定不同生长发育阶段的固原黄牛和红安格斯牛×固原黄牛杂交牛的体重、体尺、背膘厚和眼肌面积等表型数据,用于描述固原黄牛的生长发育规律和肉用性能。
为更好地了解固原黄牛在牛种资源中的系统发育地位,本研究根据Chen等[10]提出的5个核心类群(欧洲普通牛、欧亚普通牛、东亚普通牛、中国瘤牛和印度瘤牛)从公共数据库下载部分普通牛、瘤牛及中国五大地方黄牛等国内外牛群基因型数据。牛群样本信息如表 1所示,其中包括WIDDE数据库中的秦川牛(N=15)、延边牛(N=26)、蒙古牛(N=31)、红安格斯牛(N=19)、利木赞牛(N=21)、巴厘岛爪哇野牛(N=20)、南阳牛(N=23)、鲁西牛(N=11)、皖南牛(N=31)、晋南牛(N=14)芯片数据[11-14]和NCBI数据库中的吉安牛(N=18)、晋南牛(N=14)、雷琼牛(N=12)芯片数据,提取收集到的牛群体基因数据集共有的SNPs位点用于固原黄牛遗传背景分析[15]。
|
|
表 1 13个牛群体试验样本相关信息 Table 1 Samples information of 13 cattle populations |
使用PLINK(v1.9)软件对包括固原黄牛在内的13个国内外群体共337头牛合并基因型数据集进行质控,剔除包含以下条件的样本和SNP位点[16]:①个体检出率 < 0.90;②SNP检出率 < 0.95;③最小等位基因频率 < 0.05;④性染色体位点;⑤哈代-温伯格平衡检验P值< 1×10-6;⑥重复SNP位点;⑦亲缘关系较近个体(PI_HAT值>0.25)。最终获得了53头固原黄牛,总计329头国内外群体牛进行后续分析。
1.3 多维标度分析多维标度分析基于降维的方式清晰展示个体之间的聚类情况,了解群体内不同个体间遗传关系[17]。本研究对包括固原黄牛在内的13个牛群体进行多维标度分析,使用PLINK(v1.90)-mds-plot4参数构建个体间成对的状态同源(identity by state,IBS)距离矩阵,并通过R(v3.6.3)的ggplot2包对第一、第二、第三主成分(PC1、PC2、PC3)结果进行可视化。
1.4 系统发育树构建为鉴定个体及群体间的聚类情况,本研究使用PLINK(v1.90)软件-distance-marix参数对基因型数据质控后的SNP位点构建个体间成对的遗传距离矩阵[16],并将该矩阵导入PHYLIP(v3.69)软件构建NJ树[18],然后通过FigTree(v1.3.1)可视化系统发育树[19],本研究参考前人的报道,将与固原黄牛等其他牛群体同一牛属但不同种的近缘种巴厘岛爪哇野牛(Bos javanicus)作为外群[11]。
1.5 体重、体尺数据测定和体尺指数、肉用指数计算体重、体尺按《肉牛生产性能测定技术规范》(NY/T 2660-2014)进行测定[20],包括体重、体高、十字部高、体斜长、胸围、腹围、管围、胸深、腰角宽、髋宽、坐骨宽及背高。参照《中国畜禽遗传资源志·牛志》公开发表的秦川牛和蒙古牛体重、体尺数据,根据文献计算不同牛群的体尺指数[21](体长指数、胸围指数、管围指数、体躯指数和肢长指数)和肉用指数[22],计算公式如下:①体长指数=体斜长/体高;②胸围指数=胸围/体高;③管围指数=管围/体高;④体躯指数=胸围/体斜长;⑤肢长指数=(体高-胸深)/体高;⑥肉用指数(kg·cm-1)=体重/体高。
1.6 背膘厚和眼肌面积测定按照肉牛背膘与眼肌的超声波活体测定操作方法[23],使用活体测膘仪(日本/HONDA公司,型号:HS-1600V,HLS-882 MT探针)扫描第12和第13肋骨之间、距脊椎3~5 cm处横向面,根据B超自身所带软件对背膘厚和眼肌面积进行测量。参照《中国畜禽遗传资源志·牛志》公开发表的秦川牛和蒙古牛背膘厚和眼肌面积数据,分析固原黄牛肉用性能。
2 结果 2.1 固原黄牛基本特征固原黄牛被毛以红色、黄褐色和黄色为主,少数为黧色(图 1);公牛头型粗重,额头微微凹陷,头小狭长;母牛清秀,眼睛小而温和有神,不突出。鼻镜小呈粉红色和青色,鼻梁较高,嘴巴小,角大多较小,角根部多为肉色、角尖多为青色。角型多样,有萝卜角、倒八字角、龙门角、笋角、疙瘩角和无角(图 2)。颈部垂皮较小不发达,鬐甲较为高而窄,颈部薄而细,肩长而斜;两前腿间距离较宽,前躯发育好于后驱。胸宽深,颜色浅者窄背,长腰,背略微凹陷,中躯较长。荐骨较高,尻部下垂的屋脊型(斜尻),腰角宽且突出,尾细,尾帚较小,毛长,过飞结。四肢较短,关节大,管骨较细,蹄小而结实,颜色多为青色。

|
A.固原黄牛毛色为红色;B.固原黄牛毛色为黄褐色;C.固原黄牛毛色为黄色;D.固原黄牛毛色为黧色 A.Hair color of the Guyuan cattle is red; B.Hair color of the Guyuan cattle is yellowish brown; C.Hair color of the Guyuan cattle is yellow; D.Hair color of the Guyuan cattle is dark and sallow 图 1 固原黄牛毛色 Fig. 1 Hair color of Guyuan cattle |

|
A.固原黄牛角型为萝卜角;B.固原黄牛角型为倒八字角;C.固原黄牛角型为龙门角;D.固原黄牛角型为笋角;E.固原黄牛角型为疙瘩角;F.固原黄牛角型为无角 A.Horn type of the Guyuan cattle is radish horn; B.Horn type of the Guyuan cattle is inverted splayed horn; C.Horn type of the Guyuan cattle is Gantry horn; D.Horn type of the Guyuan cattle is bamboo shoot horn; E.Horn type of the Guyuan cattle is lump horn; F.Horn type of the Guyuan cattle is hornless 图 2 固原黄牛角型 Fig. 2 Horn type of Guyuan cattle |
本研究对13个牛群体基因型质控后的18 929个SNPs位点开展多维标度分析,结果发现所有试验个体均按照品种类型进行聚类。不同颜色的点代表不同的群体(图 3),第一主成分(PC1)显示普通牛和瘤牛分布与进化树两端,可以解释普通牛和瘤牛之间的差异。第二主成分(PC2)结果显示来自东亚牛群体(蒙古牛、延边牛)和欧洲普通牛群体(利木赞牛、红安格斯牛)存在一定的差异,并且能够与其地理位置相对应。第三主成分(PC3)结果显示红安格斯牛和利木赞牛存在较大的遗传变异。
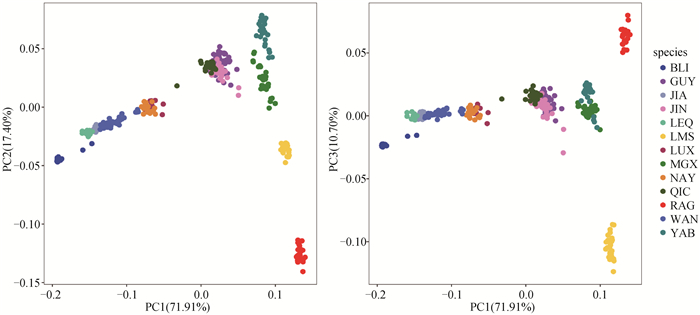
|
QIC.秦川牛;GUY.固原黄牛;JIN.晋南牛;YAB.延边牛;MGX.蒙古牛;RAG.红安格斯牛;LMS.利木赞牛;BLI.巴厘岛爪哇野牛;JIA.吉安牛;LEQ.雷琼牛;WAN.皖南牛;NAY.南阳牛;LUX.鲁西牛。下同 QIC. Qinchuan cattle; GUY. Guyuan cattle; JIN. Jinnan cattle; YAB. Yanbian cattle; MGX. Mongolian cattle; RAG. Red Angus cattle; LMS. Limousin cattle; BLI. Javanese bison; JIA. Jian cattle; LEQ. Leiqiong cattle; WAN. Wannan cattle; NAY. Nanyang cattle; LUX. Luxi cattle. The same as below 图 3 13个牛群个体进行聚类的散点图 Fig. 3 Scatter diagram for clustering of individuals of 13 cattle populations |
为检测固原黄牛个体间的遗传关系和分化程度及其在牛遗传资源中的系统发育地位。本研究以爪哇野牛群体作为外群,计算所有个体常染色体SNPs的遗传距离,构建邻接法系统发育树(图 4)。结果显示,包括固原黄牛在内的中国地方黄牛群体(南阳牛、鲁西牛、秦川牛、晋南牛、延边牛和蒙古牛),位于瘤牛和普通牛之间。固原黄牛与秦川牛和晋南牛遗传关系较近,但与多维标度分析结果相比,固原黄牛群体的个体能单独聚集在一起,且与其它品种牛明显分离,这表明固原黄牛和中国其他黄牛品种一样,同属于普通牛和瘤牛血缘杂交群体,并且固原黄牛与邻近的蒙古牛、秦川牛、晋南牛均存在一定的遗传距离,是相对独立的资源群体。
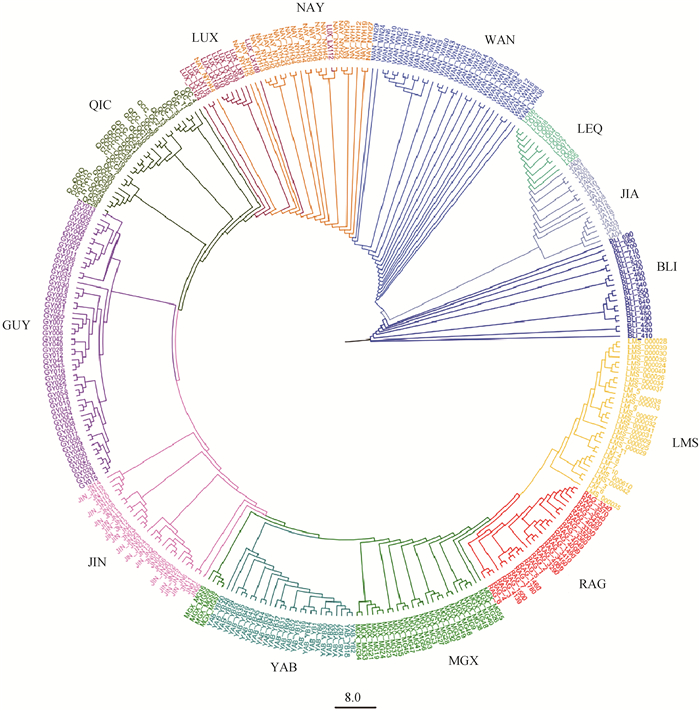
|
图 4 邻接法构建的13个牛群体系统发育关系 Fig. 4 Phylogenetic relationships of 13 cattle populations constructed by Neighbor-Joining |
由表 2可知,与固原黄牛青年母牛相比较(N=5),固原黄牛成年母牛的体重、腰角宽、髋宽、腹围、胸围、胸深、体高、十字部高、背高、管围、体斜长、坐骨宽分别提升了99.12%、26.17%、25.20%、21.71%、21.64%、19.25%、14.30%、11.03%、10.58%、9.88%、4.87%、3.94%;与固原黄牛成年母牛相比较,红安格斯牛×固原黄牛杂交牛成年母牛的体重、胸围、腹围、体高、体斜长、十字部高、腰角宽分别提升了41.01%、15.12%、13.42%、2.70%、2.48%、0.77%、0.45%,而坐骨宽下降了10.64%。参照《中国畜禽遗传资源志·牛志》公开发表的秦川牛母牛和蒙古牛母牛体重、体尺数据,与秦川牛母牛相比较,固原黄牛成年母牛的体重、体高、体斜长、胸围、管围分别下降了20.98%、2.78%、4.63%、4.38%、3.64%;与蒙古牛母牛相比较,固原黄牛成年母牛的体重、体高、胸围、管围分别提升了12.93%、8.86%、26.90%、10.55%,而体斜长下降了17.57%。
|
|
表 2 固原黄牛母牛与不同品种牛母牛体重、体尺(平均值±标准差) Table 2 Body weight and body size of Guyuan cattle and cows of different breeds(mean±SD) |
由表 3可知,较固原黄牛公牛(N=18),红安格斯牛×固原黄牛杂交牛公牛(N=10)的体重、体高、十字部高、体斜长、胸围、腹围、腰角宽、坐骨宽分别提升了57.60%、2.97%、1.51%、19.28%、22.38%、18.85%、9.49%、10.53%。同样,参照《中国畜禽遗传资源志·牛志》公开发表的秦川牛公牛和蒙古牛公牛体重、体尺数据,与秦川牛成年公牛相比较,固原黄牛成年公牛的体重、体高、体斜长、胸围、管围分别下降41.81%、11.31%、12.43%、16.88%、9.81%;与蒙古牛成年公牛相比较,固原黄牛公牛的体重、体高、胸围、管围提升3.44%、4.99%、26.16%、0.74%,而体斜长下降了14.42%。
|
|
表 3 固原黄牛公牛与不同品种牛公牛体重、体尺 Table 3 Body weight and body size of bulls of Guyuan cattle and different breeds |
基于体重、体尺数据,进一步分析固原黄牛、固原黄牛杂交牛、秦川牛和蒙古牛体尺指数和肉用指数(表 4),结果发现:与固原黄牛青年母牛相比较,固原黄牛成年母牛的体躯指数提升了9.57%;用红安格斯牛杂交改良后,固原黄牛杂交牛母牛的胸围指数和体躯指数分别提升了11.59%、12.70%,固原黄牛杂交牛公牛的体长指数、胸围指数分别提升了15.15%、19.26%;固原黄牛成年母牛的肉用指数为2.65±0.46(N=43),固原黄牛成年公牛的肉用指数为2.87±0.35,均属役用牛。
|
|
表 4 固原黄牛与不同品种牛体尺指数和肉用指数 Table 4 Body size indexes and beef-purpose index of Guyuan cattle and different breeds of cattle |
由表 5可知,固原黄牛公牛背膘厚为(5.29±1.41)mm,高于蒙古牛公牛背膘厚(4.00±1.00)mm;固原黄牛公牛眼肌面积为(62.17±8.51)cm2,介于秦川牛公牛眼肌面积(79.80±9.70)cm2和蒙古牛公牛眼肌面积之间(50.40±9.80)cm2。用红安格斯牛杂交改良后,固原黄牛杂交公牛背膘厚和眼肌面积大幅度提升,表明固原黄牛杂交公牛肉用性能改良效果显著。
|
|
表 5 固原黄牛与不同品种牛背膘厚和眼肌面积 Table 5 Backfat thickness and loin eye area of Guyuan cattle and different breeds of cattle |
本研究通过历史文献和调查研究分析固原黄牛的演变历史,发现固原黄牛是历史上通过长期自然选择形成的地方黄牛类群和当地肉牛重要种质资源,但其未被《中国畜禽遗传资源志·牛志(2011年版)》和《国家畜禽遗传资源品种名录(2021年版)》收录。依据《第三次全国畜禽遗传资源普查实施方案(2021—2023年)》重点任务,分析现存固原黄牛的遗传背景对中国黄牛遗传资源的保护、开发和利用具有重要意义。
动物遗传资源鉴定的研究方法有细胞学、形态学、生化和DNA分子标记4种[24-26]。最初动物遗传资源鉴定的DNA分子标记方法主要是应用限制性片段长度多态性(restriction fragment length polymorphism,RFLP)和短串联重复序列(short tandem repeat,STR)两种遗传标记[27]。全基因组SNP芯片具有标记密度高、均匀覆盖全基因组、测定准确率高、易实现标准化和自动化检测等诸多优势,为畜禽遗传资源的鉴定提供了可靠的技术保障。Xu等[28]通过牛HD SNPs芯片对代表中国北部、西北、西南和南部的8个黄牛品种进行研究,发现南方黄牛与北方黄牛显著分离,8个品种之间有清晰的遗传距离。本研究使用GGP Bovine 100K基因芯片检测固原黄牛的全基因组,同时收集12个品种的全基因组基因芯片数据,进行个体SNP分型,通过多维标度分析和构建邻接法系统发育树,发现固原黄牛与地理位置相对较近的秦川牛和晋南牛的遗传关系较近,但均存在遗传距离,是相对独立的资源群体。该结果为后期固原黄牛新遗传资源鉴定及肉用性能研究提供了理论支撑。
形态学标记是一种简单直观,易于观察记录的物种分类方法,主要是通过种群的体型外貌、生理性状、对环境的适应性等遗传特征进行研究分类[29-30]。本研究在现代基因组的群体遗传学方法基础上,通过形态学标记进一步分析了固原黄牛的遗传背景。由于遗传物质不同及其所处自然环境不同,中国黄牛的毛色表现出丰富的多样性。毛色遗传学研究表明,毛色是多基因的共效应,导致同一品种具有相同毛色或差异毛色[31]。中国黄牛的全基因组重测序结果显示,齿状蛋白1(jagged 1,JAG1)、BarH样同源框2(barH-like homeobox,BARX2)、KIT原癌基因受体酪氨酸激酶(KIT proto-oncogene receptor tyrosine kinase,KIT)和血小板衍生生长因子受体α(platelet-derived growth factor receptor alpha,PDGFRA)基因与毛色黄色有关,催乳素受体(prolactin receptor,PRLR)、桥粒胶蛋白3(desmocollin,3DSC3)和KIT配体(KIT ligand,KITLG)基因与毛色红色有关,促分裂原活化蛋白激酶激酶1(mitogen-activated protein kinase kinase 1,MAP2K1)基因与毛色黑色有关[32]。晋南牛和秦川牛毛色以红色为主,蒙古牛毛色为黄、黑棕及各色毛相间的杂色。本研究通过统计纯种固原黄牛群体毛色,发现其毛色以黄色、黄褐色和红色为主,少数为黧色。早期中国黄牛毛色遗传标记研究中,张文举和吕永锋[33]也发现,固原黄牛和蒙古牛群体黧色毛色个体比例占到13%~25%,而其它黄牛群体中没有或比例极低。形态学标记表明,固原黄牛含有周边蒙古牛和秦川牛血液,这与历史文献和调查研究等记载描述相一致。
现代分子育种仍离不开表型数据的测定,了解不同牛群体体尺指数、肉用指数、背膘厚和眼肌面积有助于制定准确的育种目标[21]。Meng等[34]比较了中国土牛黄牛文山牛与国外专门化肉牛品种西门塔尔牛的体尺、体重的差异,发现青年文山牛公牛体重为(160.3±32.2)kg,成年文山牛公牛体重为(312.7±50.1)kg,体重提升了95.07%;青年西门塔尔牛公牛体重为(312.2±38.3)kg,成年西门塔尔牛公牛体重为(526.2±38.6)kg,体重提升了68.55%。本研究发现,固原黄牛公牛体重为(361.33±53.53)kg,与文山牛公牛体重相近,用红安格斯牛杂交改良后,固原黄牛杂交牛公牛体重为(569.44±88.77)kg,达到国外专门化肉牛品种体重。根据张英汉[22]提出的牛肉用指数与肉用型牛和役用型牛的关系,国外专门化肉牛品种安格斯牛、海福特牛、利木赞牛和夏洛莱牛的公牛、母牛的肉用指数平均分别为7.43和5.40,国内五大良种黄牛品种晋南牛、秦川牛、鲁西牛、南阳牛和延边牛的公牛、母牛的肉用指数平均分别为3.650和3.113。本研究发现,固原黄牛公、母牛的肉用指数平均为2.87、2.65,远低于国外专门化肉牛品种和国内五大良种黄牛品种,表明固原黄牛适应了当地的地理环境和经济条件,逐渐转变为役用型牛,后续需要进一步加强选育,提高固原黄牛的肉用性能。固原黄牛公牛背膘厚高于蒙古牛公牛,固原黄牛公牛眼肌面积介于秦川牛公牛和蒙古牛公牛之间,表明固原黄牛具有较好的肉用性能育种潜力。
4 结论本研究通过历史文献、调查研究、现代群体遗传学分析方法和形态学标记均表明固原黄牛起源于秦川牛和蒙古牛。虽然固原黄牛来源于秦川牛与蒙古牛,但经过长期的杂交演变,固原黄牛与秦川牛和蒙古牛出现了一定的遗传距离,形成了独特的遗传结构特点,至此认为,固原黄牛可以作为一种相对独立的遗传资源进行进一步研究。进一步分析三者体尺指数、肉用指数、背膘厚和眼肌面积等表型数据,显示固原黄牛的体重、体高、胸围、管围介于蒙古牛和秦川牛之间,而体斜长低于蒙古牛和秦川牛,建议在肉用选育时,体尺指标以体斜长为主;依据肉用指数判断,固原黄牛属役用黄牛,但背膘厚高于蒙古牛,眼肌面积介于蒙古牛和秦川牛之间,表明固原黄牛具有较好的肉用性能育种潜力。固原黄牛后期需要加强选育,并进行良种扩繁和遗传改良,进一步发挥其种质资源特性。
| [1] |
于利子. 固原市肉牛饲料资源调查与营养成分分析[J]. 湖北畜牧兽医, 2021, 42(11): 7-10. YU L Z. Investigation on feed resources and nutrient composition analysis of beef cattle in Guyuan City[J]. Hubei Journal of Animal and Veterinary Sciences, 2021, 42(11): 7-10. (in Chinese) |
| [2] |
秦国庆, 常洪, 耿社民, 等. 固原黄牛毛色及外形特征的研究[J]. 黄牛杂志, 1997, 23(2): 17-19, 22. QIN G Q, CHANG H, GENG S M, et al. Studies on coat colour and exterior characters of Guyuan Yellow Cattle[J]. Journal of Yellow Cattle Science, 1997, 23(2): 17-19, 22. (in Chinese) |
| [3] |
秦国庆, 常洪, 孙金梅, 等. 固原黄牛系统地位的研究[J]. 西北农业学报, 1997, 6(4): 8-11. QIN G Q, CHANG H, SUN J M, et al. Phylogeny status of guyuan yellow cattle population[J]. Acta Agriculturae Boreali-Occidentalis Sinica, 1997, 6(4): 8-11. (in Chinese) |
| [4] |
秦国庆, 常洪, 孙金梅. 固原黄牛群体遗传变异的研究[J]. 黄牛杂志, 1994, 20(S2): 13-17. QIN G Q, CHANG H, SUN J M. Study on genetic variation of Guyuan cattle[J]. Journal of Yellow Cattle Science, 1994, 20(S2): 13-17. (in Chinese) |
| [5] |
秦国庆, 孙金梅, 常洪, 等. 固原黄牛头骨变异的研究[J]. 黄牛杂志, 1997, 23(4): 24-26. QIN G Q, SUN J M, CHANG H, et al. Study on skull variation of Guyuan cattle[J]. Journal of Yellow Cattle Science, 1997, 23(4): 24-26. (in Chinese) |
| [6] |
秦国庆, 常洪, 耿社民, 等. 固原黄牛血液多态性的研究[J]. 黄牛杂志, 1997, 23(3): 11-12, 19. QIN G Q, CHANG H, GENG S M, et al. The research of gu yuan yellow cattle polymorphism[J]. Journal of Yellow Cattle Science, 1997, 23(3): 11-12, 19. (in Chinese) |
| [7] |
谷朝勇. 中亚东南家牛群体相对群体集团亲缘关系的判别研究[J]. 黑龙江畜牧兽医, 2010(11): 64-65. GU C Y. Discriminant study on the genetic relationship of cattle population in Central Asia and Southeast Asia[J]. Heilongjiang Animal Science and Veterinary Medicine, 2010(11): 64-65. (in Chinese) |
| [8] |
刘丽, 赵生国, 蔡原, 等. 早胜牛及其杂交群体遗传多样性研究[J]. 农业生物技术学报, 2014, 22(3): 317-325. LIU L, ZHAO S G, CAI Y, et al. The genetic diversity of Zaosheng native cattle (Bos taurus) and associated crossbred population[J]. Journal of Agricultural Biotechnology, 2014, 22(3): 317-325. (in Chinese) |
| [9] |
范婷婷, 王文翔, 马毅, 等. 西门塔尔牛、和牛与荷斯坦牛杂种优势预测及实际杂交效果分析[J]. 畜牧兽医学报, 2022, 53(8): 2568-2577. FAN T T, WANG W X, MA Y, et al. Prediction and effect analysis of heterosis in Simmental, Wagyu and Holstein[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(8): 2568-2577. (in Chinese) |
| [10] |
CHEN N B, CAI Y D, CHEN Q M, et al. Whole-genome resequencing reveals world-wide ancestry and adaptive introgression events of domesticated cattle in East Asia[J]. Nat Commun, 2018, 9(1): 2337. DOI:10.1038/s41467-018-04737-0 |
| [11] |
DECKER J E, MCKAY S D, ROLF M M, et al. Worldwide patterns of ancestry, divergence, and admixture in domesticated cattle[J]. PLoS Genet, 2014, 10(3): e1004254. DOI:10.1371/journal.pgen.1004254 |
| [12] |
GAO Y, GAUTIER M, DING X D, et al. Species composition and environmental adaptation of indigenous Chinese cattle[J]. Sci Rep, 2017, 7(1): 16196. |
| [13] |
GAUTIER M, LALOË D, MOAZAMI-GOUDARZI K. Insights into the genetic history of French cattle from dense SNP data on 47 worldwide breeds[J]. PLoS One, 2010, 5(9): e13038. |
| [14] |
MATUKUMALLI L K, LAWLEY C T, SCHNABEL R D, et al. Development and characterization of a high density SNP genotyping assay for cattle[J]. PLoS One, 2009, 4(4): e5350. |
| [15] |
ZHANG Y R, HU Y, WANG X G, et al. Population structure, and selection signatures underlying high-altitude adaptation inferred from genome-wide copy number variations in Chinese indigenous cattle[J]. Front Genet, 2020, 10: 1404. |
| [16] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. |
| [17] |
XIA X, YAO Y, LI C, et al. Genetic diversity of Chinese cattle revealed by Y-SNP and Y-STR markers[J]. Anim Genet, 2019, 50(1): 64-69. |
| [18] |
RETIEF J D. Phylogenetic analysis using PHYLIP[M]//MISENER S, KRAWETZ S A. Bioinformatics Methods and Protocols. Totowa: Humana Press, 2000, 132: 243-258.
|
| [19] |
XU L Y, BICKHART D M, COLE J B, et al. Genomic signatures reveal new evidences for selection of important traits in domestic cattle[J]. Mol Biol Evol, 2015, 32(3): 711-725. |
| [20] |
中华人民共和国农业部. NY/T 2660-2014肉牛生产性能测定技术规范[S]. 北京: 中国农业出版社, 2015. Ministry of Agriculture of the People's Republic of China. NY/T 2660-2014 Technical specification of beef cattle performance test[S]. Beijing: China Agriculture Press, 2015. (in Chinese) |
| [21] |
杨亮, 张文举, 张晓羊, 等. 不同糖源对肉用犊牛生长性能、体尺指数及营养物质表观消化率的影响[J]. 中国畜牧兽医, 2017, 44(6): 1726-1733. YANG L, ZHANG W J, ZHANG X Y, et al. Effect of different carbohydrate sources on growth performance, body Size indexes and nutrients apparent digestibility of beef calves[J]. China Animal Husbandry & Veterinary Medicine, 2017, 44(6): 1726-1733. (in Chinese) |
| [22] |
张英汉. 牛的肉用指数及影响因素[J]. 中国牛业科学, 2006, 32(2): 4-7. ZHANG Y H. Beef performance index for cattle and its affecting factors[J]. China Cattle Science, 2006, 32(2): 4-7. (in Chinese) |
| [23] |
魏成斌, 施巧婷, 冯亚杰, 等. 肉牛背膘与眼肌的超声波活体测定操作方法[C]//中国畜牧兽医学会2010年学术年会——第二届中国兽医临床大会论文集(下册). 长春: 中国畜牧兽医学会, 2010. WEI C B, SHI Q T, FENG Y J, et al. Operation method of ultrasonic in vivo measurement of beef backfat and loin eye area[C]//Proceedings of the 2010 Academic Annual Meeting of the Chinese Society of Animal Husbandry and Veterinary Medicine and the Second Chinese Veterinary Clinical Congress. Changchun: Chinese Association of Animal Science and Veterinary Medicine, 2010. (in Chinese) |
| [24] |
WEISSMAN T A, PAN Y A. Brainbow: new resources and emerging biological applications for multicolor genetic labeling and analysis[J]. Genetics, 2015, 199(2): 293-306. |
| [25] |
BANNASCH D L, BAES C F, LEEB T. Genetic variants affecting skeletal morphology in domestic dogs[J]. Trends Genet, 2020, 36(8): 598-609. |
| [26] |
ZHANG J J, WU M H, MA Z G, et al. Species-specific identification of donkey-hide gelatin and its adulterants using marker peptides[J]. PLoS One, 2022, 17(8): e0273021. |
| [27] |
刘继强, 郝晓东, 武丽娜, 等. 全基因组SNP分型技术在畜禽遗传育种研究中的应用[J]. 畜牧兽医学报, 2022, 53(12): 4123-4137. LIU J Q, HE X D, WU L N, et al. Application of whole genome SNP genotyping technology in livestock and poultry genetics and breeding[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(12): 4123-4137. (in Chinese) |
| [28] |
XU L Y, YANG L, ZHU B, et al. Genome-wide scan reveals genetic divergence and diverse adaptive selection in Chinese local cattle[J]. BMC Genomics, 2019, 20(1): 494. |
| [29] |
GELAYE G, BAYE G, MASHO W, et al. Morphometric traits and structural indices of indigenous cattle reared in Bench Sheko zone, southwestern Ethiopia[J]. Heliyon, 2022, 8(8): e10188. |
| [30] |
LI X M, DING X T, LIU L L, et al. Copy number variation of bovine DYNC1I2 gene is associated with body conformation traits in chinese beef cattle[J]. Gene, 2022, 810: 146060. |
| [31] |
VOß K, BLAJ I, TETENS J L, et al. Roan coat color in livestock[J]. Anim Genet, 2022, 53(5): 549-556. |
| [32] |
MEI C G, JUNJVLIEKE Z, RAZA S H A, et al. Copy number variation detection in Chinese indigenous cattle by whole genome sequencing[J]. Genomics, 2020, 112(1): 831-836. |
| [33] |
张文举, 吕永锋. 中国黄牛毛色遗传标记的研究进展[J]. 甘肃畜牧兽医, 1998, 28(4): 29-31. ZHANG W J, LV Y F. Research progress on genetic markers of hair color in Chinese yellow cattle[J]. Gansu Animal Husbandry and Veterinary Medicine, 1998, 28(4): 29-31. (in Chinese) |
| [34] |
MENG X R, GAO Z W, LIANG Y S, et al. Longissimus dorsi muscle transcriptomic analysis of simmental and Chinese native cattle differing in meat quality[J]. Front Vet Sci, 2020, 7: 601064. |
(编辑 郭云雁)



